多房棘球蚴抗原蛋白TSP3抗原表位的生物信息学预测#
庞明泉,汤 锋,周 虎,万陈飞,阳丹才让,4*,樊海宁,4**
(1.青海大学附属医院肝胆胰外科,青海 西宁 810001;2.青海大学医学院高原医学研究中心,青海 西宁 810001;3.青海大学高原人畜共患病研究所,青海 西宁 810001;4.青海省包虫病研究重点实验室,青海 西宁 810001)
多房棘球蚴抗原蛋白TSP3抗原表位的生物信息学预测#
庞明泉1,3,汤 锋2,3,4,周 虎1,3,万陈飞1,3,阳丹才让1,3,4*,樊海宁1,3,4**
(1.青海大学附属医院肝胆胰外科,青海 西宁 810001;2.青海大学医学院高原医学研究中心,青海 西宁 810001;3.青海大学高原人畜共患病研究所,青海 西宁 810001;4.青海省包虫病研究重点实验室,青海 西宁 810001)
目的 预测多房棘球绦虫抗原蛋白TSP3的二级结构和优势抗原表位(T细胞和B细胞表位)。方法 Genbank获取TSP3的氨基酸序列后通过生物信息学软件SOPMA预测二级结构特征,进一步通过在线软件IEDB、SYFPEITHI、Bcepred和ABCpred预测TSP3的T细胞和B细胞表位。同时对TSP3的亲水性、柔韧性、抗原性及蛋白表面暴露区域的特征进行预测。结果 无规则卷曲和β转角分别占TSP3蛋白质的二级结构的25.68%和4.05%,说明潜在优势抗原性表位存在。通过多种生物信息学方法分析出TSP3潜在的T细胞表位为T33-42、T45-55、T53-63、T68-77、T80-90、T92-104、T110-122、T134-144;TSP3潜在的B细胞表位为T18-33、T45-55、T53-63、T64-75、T80-90、T92-104、T110-122。结论 TSP3的二级结构特征显示潜在优势抗原性表位存在,预测出8种T细胞抗原表位和7种B细胞抗原表位,为后续表位疫苗的设计奠定了理论基础。
多房棘球蚴 TSP3 生物信息学 二级结构 抗原表位
据报道,用重组蛋白疫苗免疫细粒棘球绦虫的中间宿主,免疫作用可达95%~100%。因此,免疫预防为预防包虫病疫情的有效措施[1]。表位(抗原决定簇)是决定抗原的特异性的化学基团,可分为T或(和)B细胞表位[2,3],分别与T细胞抗原受体TCR或(和)B细胞抗原受体BCR特异性结合,最终刺激机体产生免疫反应,形成对病原微生物的免疫能力。目前表位疫苗已被应用于抗细菌[4,5]、病毒等感染与抗肿瘤中[6],相比较于传统疫苗,表位疫苗更为安全、无毒、稳定,可以直接刺激机体产生特异性免疫反应,因此表位疫苗更符合未来疫苗的发展方向,在免疫预防中的作用越来越重要[7]。基于此,研制多房棘球蚴表位疫苗可为治疗和预防多房棘球蚴病的发生提供新的有效方法。
表位疫苗的研发的最大难点在于寻找具有良好免疫原性的抗原位点[8]。Pfaff等[9]证明了146-154氨基酸(AA)和200-213a氨基酸多肽含有抗口蹄疫病毒(FMDV)的表位,这些表位肽可为表位疫苗的制备提供基础。此外,Kouguchi等[10]研究显示Emy162重组抗原可对大鼠产生74.3%的免疫保护作用。Sugimoto C[11]在2009年报道称棘球蚴表面抗原TSP家族具有良好的免疫原性。Oku Y课题组[12]在2012年报道称TSP家族中TSP-3是该家族中针对棘球蚴感染免疫原性最好的蛋白,认为FBP融合表达TSP3可以增强TSP-3的免疫原性从而更好地预防棘球蚴感染[13]。这些结果表明,通过分子疫苗来预防包虫病是可行的。
本研究拟利用生物信息学软件来预测和分析TSP3的二级结构特征,进一步预测潜在的T细胞抗原表位和B细胞优势抗原表位,为后续基于TSP3蛋白的表位疫苗的设计奠定基础。
1 材料与方法
1.1 获取TSP3蛋白的氨基酸序列
从GenBank中(GenBank no.ACJ02404.1;http://www.ncbi.nih.gov/ genbank/)获得TSP3的核苷序列及相应的氨基酸序列。根据GenBank记录,TSP3 蛋白由148个氨基酸组成,由447 bp 的mRNA编码而成。氨基酸序列见图1。

图1 TSP3蛋白的氨基酸序列
Figure 1 Amino acid sequence of the TSP3 protein. The protein is composed of 148 amino acid residues
1.2 预测TSP3蛋白质的二级结构特征
用生物信息学软件SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_a-utomat.plpage=/NPSA/npsa_sopma.html)[14]分析TSP3蛋白质序列的二级结构。输入前述TSP3蛋白的氨基酸序列,对蛋白质二级结构的四个构象状态—螺旋、折叠、转角和卷曲分别进行分析。相似性阈值和窗口宽度的参数分别设置为8和17,余参数为默认值。
1.3 预测TSP3蛋白的T细胞抗原表位
在明确了TSP3的二级结构后,采用生物信息学软件IEDB(http://tools.immuneepitope.org/main/index.html)[15]和Syfpeithi(http://www.syfpeithi.de)分析人源MHC1抗原HLA-A*02:01以预测分析潜在的T细胞抗原表位。将前述TSP3蛋白的氨基酸序列输入,并且调整参数如下:“MHC allele(s)”设定为HLA-A *02:01,“length”设定为8、9、10,余参数不变。
1.4 预测TSP3蛋白的B细胞抗原表位
采用生物信息学软件Bcepred(http://www.imtech.res.in/raghava/bcepred/bcepred_submission.html)和ABCpred(http://www.imtech.res.in/raghava/abcpred/)[16]分析TSP3潜在的B细胞抗原表位。将前述TSP3蛋白质的氨基酸序列输入,然后设置Bcepred参数值:亲水性为2;灵活性为1.9;暴露的表面积为2.4;抗原性倾向为1.8,余参数不变。设置ABCpred软件的抗原表位长度为10~16,余参数不变。
2 结果
2.1 预测的TSP3蛋白质二级结构
为了评价TSP3蛋白的抗原特性,我们使用在线生物信息学软件SOPMA预测其二级结构。蛋白质中的延伸链和无规则卷曲结构部分最有可能出现潜在的优势抗原表位。预测的二级结构特征见图2。分析结果显示,TSP3二级结构的无规则卷曲、β转角、α螺旋和延伸链(β折叠)的比例分别为25.68%、4.05%、60.81%、9.46%。
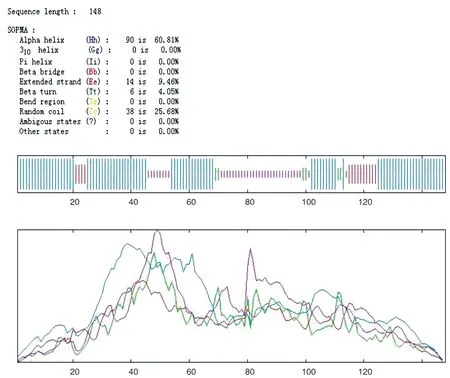
Parameters:Window width:17 Similarity threshold:8 Number of states:4
不同颜色的线代表不同的二级结构:蓝色,α螺旋;绿色,β转角;红色,延伸链;紫色,无规卷曲.在蛋白质延长链和无规则卷曲有利于形成抗原表位
Lines in different colors represent different secondary structures:Blue,αhelix;green,β turn;red,extended strand;and purple,random coil.An increased numberof extended strands and random coils in the protein corresponded with an increased likelihood of the protein forming an antigenic epitope
图2 TSP3蛋白的二级结构预测分析图
Figure 2 Secondary structure prediction results for the TSP3 protein
2.2 预测所得TSP3蛋白的T细胞抗原表位
确定抗原表位的精确位置对于研发表位疫苗非常重要。在本研究中,MHC-I 型HLA-A*02:01限制性T细胞表位使用两种在线软件—IEDB和SYFPEITHI进行T细胞表位预测。通过不同位点区域的分值高低表示所预测的T细胞表位概率大小,在分配给该区域的分数越高,则该区域为抗原表位的可能性越大。虽然这两个预测软件采用的评分系统不同,但共同处是分值较高的位点区域即为被预测的潜在抗原表位。用IEDB软件预测分较高的T细胞表位为T29-40、T37-48、T53-64、T66-77、T81-92(表1)。而SYFPEITHI软件预测的相对优势的表位位于T118-126、T114-122、T55-63、T121-129(表2)。将两软件预测结果组合,并结合前述TSP3的蛋白二级结构特征,我们最终确定的8个T细胞潜在优势抗原表位为T33-42、T45-55、T53-63、T68-77、T80-90、T92-104、T110-122、T134-144。
表1 生物信息学软件IEDB预测的TSP3的T细胞抗原表位
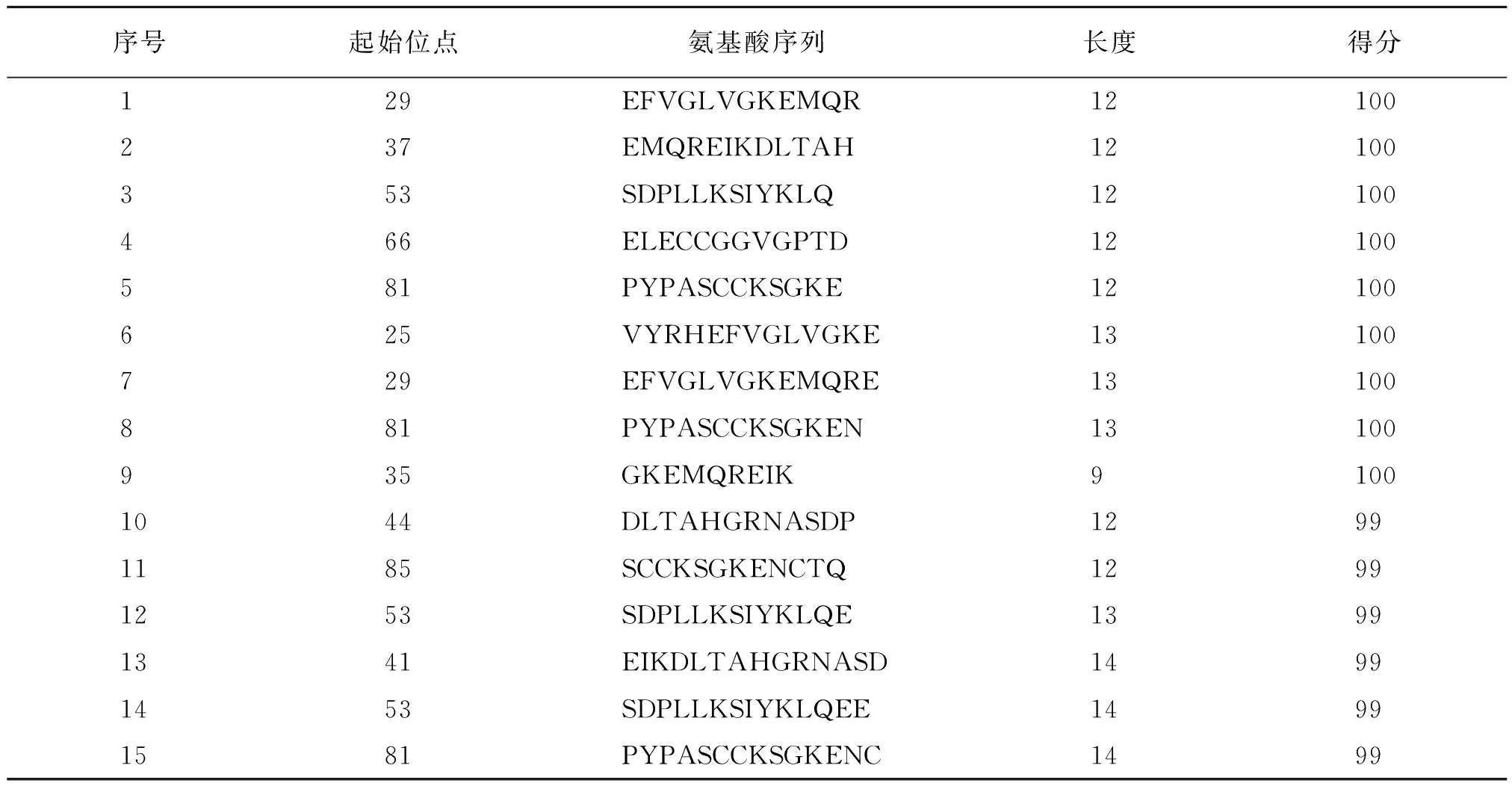
Table 1 Analysis of the T cell epitopes of TSP3 protein using IEDB online prediction software.
表2 生物信息学软件SYFPEITHI预测的TSP3的T细胞抗原表位
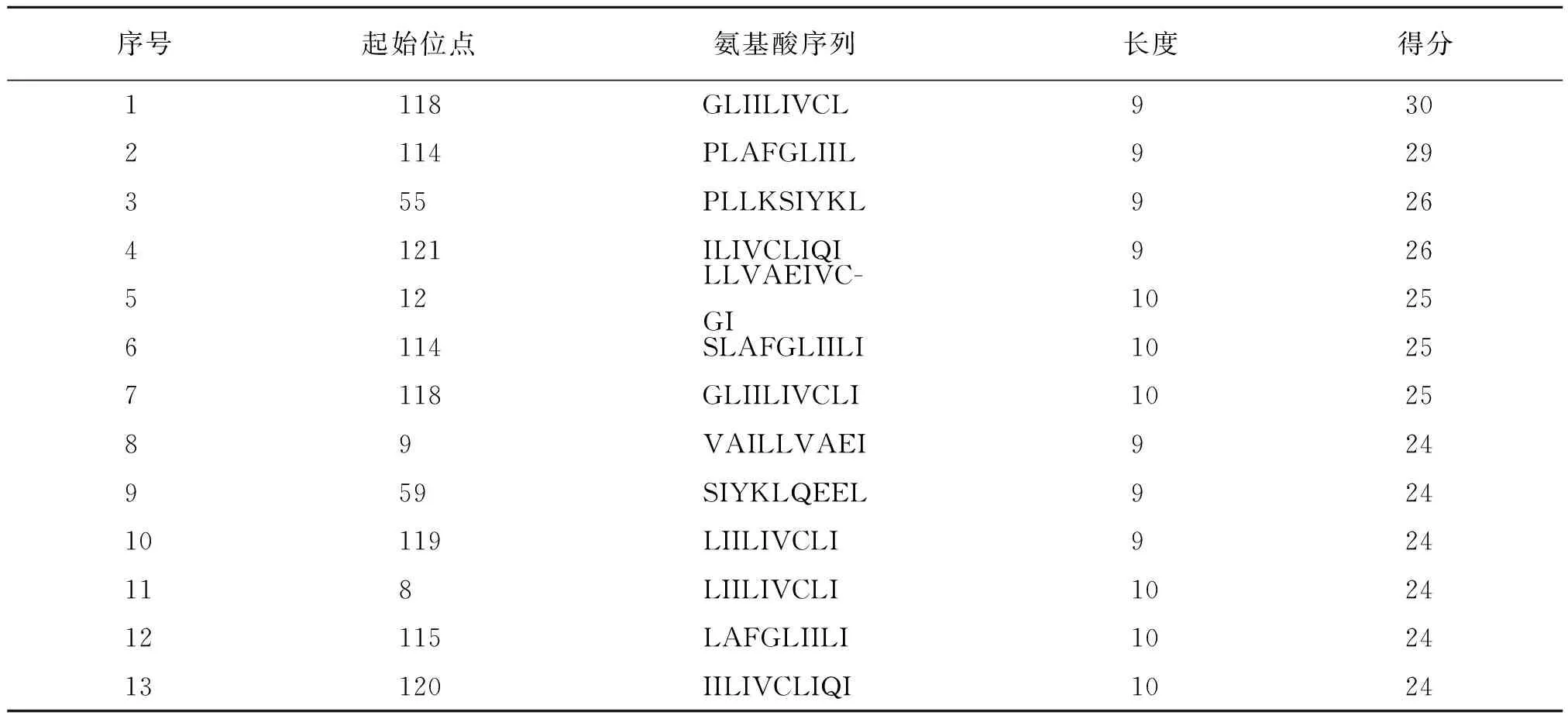
Table 2 Analysis of the T cell epitopes of TSP3 protein using Syfpeithi online prediction software.
2.3 预测所得TSP3抗原的B细胞表位
B细胞表位使用Bcepred在线软件预测,对TSP3的氨基酸序列的亲水性、弹性、抗原倾向和抗原的暴露表面面积进行分析。预测分析结果见图3,预测分析出TSP3蛋白的有高亲水性的4个区域(见图3A)是T35-41(氨基酸序列:GKEMQRE)、T48-55(氨基酸序列:HGRNASDP)、T85-97(氨基酸序列:SCCKSGKENCTQP)、T108-114(氨基酸序列:EQIKDSS);预测出的2个弹性区域(图3B)是T46-52(氨基酸序列:TAHGRNA)、T84-92(氨基酸序列:ASCCKSGKE);4个可能的抗原区域(图3C)是T16-36(氨基酸序列:EIVCGIVLLVYRHEFVGLVGK)、T55-75(氨基酸序列:PLLKSIYKLQEELECCGGVGP)、T117-130(氨基酸序列:FGLIILIVCLIQIG)、T132-138(氨基酸序列:VICACCL);3个暴露的表面区域(图3D)是T35-45(氨基酸序列:GKEMQREIKDL)、T62-68(氨基酸序列:KLQEELE)、T139-148(氨基酸序列:AKKVNEYEKV)。为了进一步准确预测出潜在的表位,我们同时通过在线软件ABCpred预测,高分的地区是T59-73、T39-54、T80-95、T74-87、T129-144(表3)。结合TSP3蛋白的二级结构特征及两种预测方法的综合结果,所预测的潜在优势B细胞表位为T18-33、T45-55、T53-63、T64-75、T80-90、T92-104、T110-122。
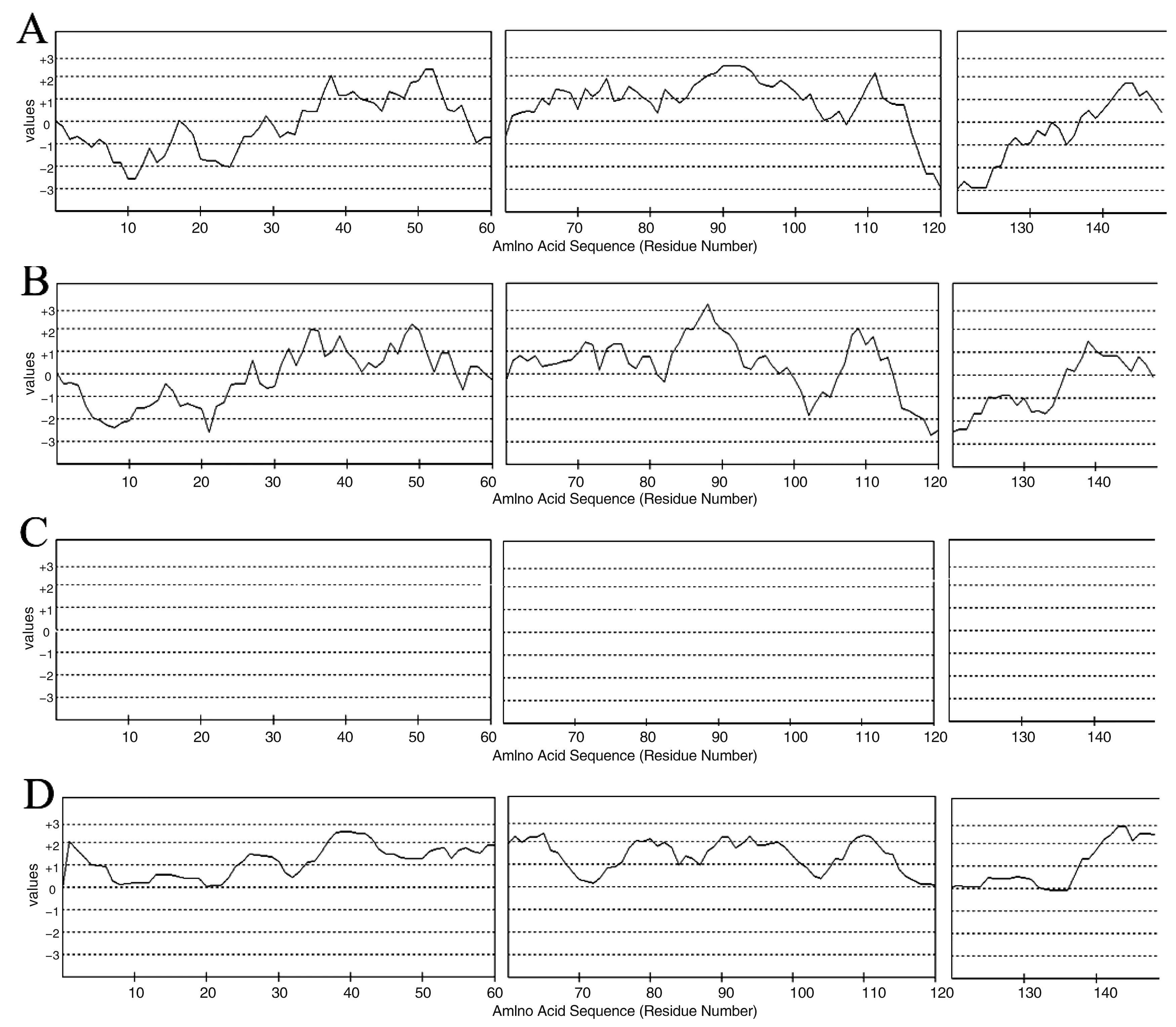
A亲水性,B弹性,C抗原倾向性、D抗原的暴露表面面积
Predictions of(A)hydrophilicity,(B)flexibility,(C)antigenic propensity,(D)exposed surface
图3 生物信息学软Bcepred预测的TSP3的B细胞抗原表位图
Figure 3 B cell epitope prediction results for the TSP3 protein
表3 生物信息学软ABCpred预测的TSP3的B细胞抗原表位
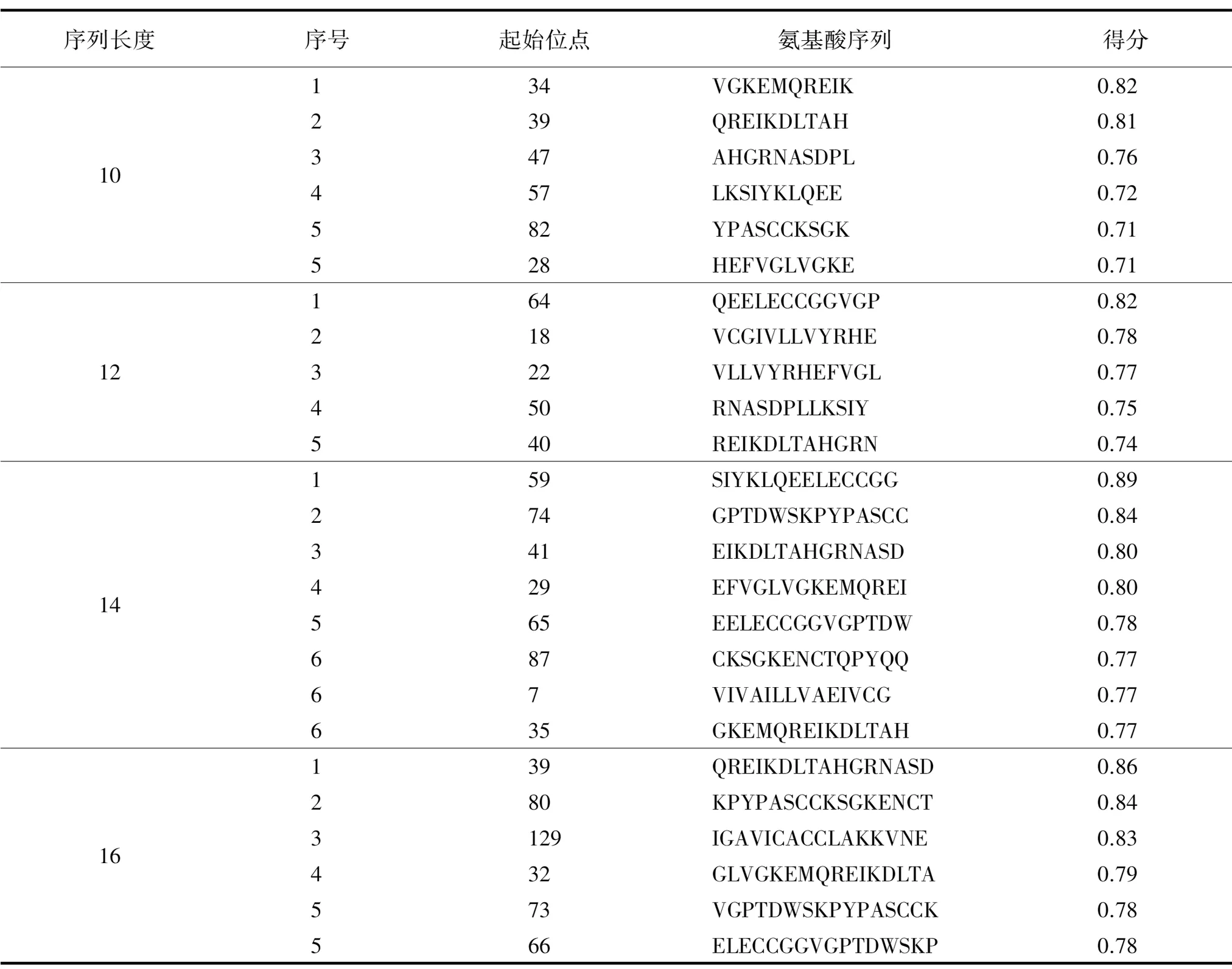
Table 3 Analysis of the B cell epitopes of TSP3 protein using ABCpred online prediction software
3 讨论
表位疫苗制备中的首要难题是获得有关表位的必要信息。
近年来,随着生物信息学的发展,表位预测的操作简便性和准确性有了较大提高,其在各项研究中的意义受到越来越多人的重视。马秀敏等[17]以Eg的AgB1基因序列为基础,首先用软件PredictProtein预测了AgB1抗原蛋白的二级结构;应用Bcepred、Abcpred、IEDB及SYFPEITHI四种软件预测出了4个B表位及4个T表位。李玲玲等人[18]根据EB病毒核蛋白-1(EBNA-1)的基因序列,使用SOPMA、GOR、HNN三种软件对EBNA-1的B细胞优势表位进行了预测,并对跨膜结构域以及各种参数(如亲水性)进行了分析。进而使用blastp对EBNA-1预测的B细胞表位的序列和人类的自身相关抗原的序列进行对比分析。
蛋白质的二级结构与其表位的分布密切相关,亲水性和抗原性是表位形成的最主要因素,但柔软性、暴露的表面区域和二级结构的构象在表位形成中也很重要。因此,我们分析了TSP3蛋白的二级结构以获得该蛋白质的抗原特性。机体存在T细胞免疫和B细胞免疫以彻底消除抗原,因此本研究中我们使用多种方法、设置多种参数对TSP3的B细胞表位和T细胞表位进行预测分析。通过对TSP3蛋白的多种性质采用不同算法进行综合分析,以提高所预测的抗原表位的准确性和特异性。
α螺旋和β折叠在维持蛋白质的二级结构稳定中起重要作用,但α螺旋和β折叠通常位于该蛋白质的内部,因此与配体结合相对困难。与此相反,β转角和无规卷曲位于蛋白质的表面,容易与配体结合,因而β转角和无规卷曲所在的氨基酸序列出现抗原表位的几率较大。
通过SOPMA服务器软件分析,α螺旋和β折叠的比例分别为60.81%、9.46%,表明TSP3抗原具有良好的稳定性。有利于形成抗原表位的无规则卷曲和β转角分别占蛋白质的25.68%和4.05%。TSP3有6个潜在的抗原区域,其中无规则卷曲区域在47-54(氨基酸序列:AHGRNASD)和72-99(氨基酸序列:GVGPTDWSKPYPASCCKSGKENCTQPYQ)比例较高,显示这两个区域具有较强的抗原性。
MHC-I型的表位在预测的T细胞表位中的预测准确率高达90%[19]。在中国人群,HLA-A *02:01是最常见的HLA-I类分子,呈55%的阳性率。因此,在本研究中,TSP3抗原的HLA-A *02:01限制性表位使用IEDB和SYFPEITHI在线软件进行分析。根据两种软件及二级结构的综合预测结果,被预测的TSP3蛋白的8个T细胞表位位于T33-42、T45-55、T53-63、T68-77、T80-90、T92-104、T110-122、T134-144。
我们采用多种方法和多种参数分析TSP3蛋白的B细胞表位,以提高预测的准确度。亲水性参数的预测反映了亲水性残基在整个抗原氨基酸序列中的位置。该蛋白质的氨基酸残基可被分为两种类型:亲水残基和疏水残基。一般情况下,疏水残基位于蛋白质的内部,而亲水残基位于蛋白的表面上。蛋白质的这个构象是有利的亲水性残基结合溶液中的极性分子,以中和蛋白质本身的电荷,从而使蛋白质保持最小能量的状态。因此,亲水性区域都与表位相关联[20]。弹性参数反映出蛋白质的弯曲和折叠的能力。弹性程度增加,则蛋白质的多肽骨架具有较好的折叠和弯曲能力,便于二级结构构象的改变[21]。抗原倾向反映了抗原潜在的免疫原区,潜在的免疫原区最有可能出现具有抗原倾向的表位。对暴露的表面区域的分析反映残基在蛋白质[22]的外层中的分布,增加了溶剂分子与蛋白接触的几率。应用两个在线软件对TSP3的表位参数综合分析可预测B细胞表位。我们共预测分析出7个B细胞潜在优势抗原表位,分别为T18-33、T45-55、T53-63、T64-75、T80-90、T92-104、T110-122。
本研究的目的是为了获得TSP3蛋白的生物信息学特性。生物信息学软件SOPMA被用来预测TSP3蛋白的二级结构,通过生物信息学软件IEDB和SYFPEITHI预测分析T细胞抗原表位,B细胞表位则通过生物信息学软件Bcepred和ABCpred综合预测分析而得。TSP3预测的二级结构表明,TSP3存在形成抗原表位的结构区域。T细胞表位和B细胞表位预测结果表明,T细胞表位位于T33-42、T45-55、T53-63、T68-77、T80-90、T92-104、T110-122、T134-144;B细胞表位位于T18-33、T45-55、T53-63、T64-75、T80-90、T92-104、T110-122。并且存在T45-55、T53-63、T80-90、T92-104、T110-122五个同时有B/T双细胞性的表位。
本研究为TSP3蛋白优势表位的鉴定和筛选提供了基础,并为免疫预防和治疗多房棘球蚴病的表位疫苗研发奠定了理论基础。
[1]Nunnari G,Pinzone MR,Gruttadauria S,et al.Hepatic echinococcosis:clinical and therapeutic aspects[J].World J Gastroenterol,2012,18(13):1448-1458.
[2]Almeida RR,Rosa DS,Ribeiro SP,et al.Broad and cross-clade CD4(+)T-cell responses elicited by a DNA vaccine encoding highly conserved and promiscuous HIV-1 M-group consensus peptides[J].PloS One,2012,7(9):e45267.
[3]Zhang Z,Chen J,Shi H,et al.Identification of a conserved linear B cell epitope in the M protein of porcine epidemic diarrhea virus[J].Virol J,2012,9(1):225-233.
[4]Li HB,Zhang JY,He YF,et al.Systemic immunization with an epitope-based vaccine elicits a Th1-biased response and provides protection against Helicobacter pylori in mice[J].Vaccine,2012,31(1):120-126.
[5]Mollica A,Stefanucci A,Costante R.Strategies for developing tuberculosis vaccines:emerging approaches[J].Curr Drug Targets,2013,14(9):938-951.
[6]Hazama S,Maeda K,Oka M.Epitope peptide vaccine with oncoantigen for cancer and its biomarker[J].Nihon Rinsho,2012,70(12):2189-2193.
[7]Ben-Yedidia T,Arnon R.Towards an epitope-based human vaccine for influenza[J].Hum Vaccin,2005,1(3):95-101.
[8]You L,Brusic V,Gallagher M,et al.Using Gaussian process with test rejection to detect T-cell epitopes in pathogen genomes[J].IEEE/ACM Trans Comput Biol Bioinform,2010,7(4):741-751.
[9]Pfaff E,Thiel HJ,Beck E,et al.Analysis of neutralizing epitopes on foot-and-mouth disease virus[J].J Virol,1988,62(6):2033-2040.
[10]Kouguchi H,Matsumoto J,Yamano K,et al.Echinococcus multilocularis:purification and characterization of glycoprotein antigens with serodiagnostic potential for canine infection[J].Exp Parasitol,2011,128(1):50-56.
[11]Dang Z,Yagi K,Oku Y,et al.Evaluation of Echinococcus multilocularis tetraspanins as vaccine candidates against primary alveolar echinococcosis[J].Vaccine,2009,27(52):7339-7345.
[12]Dang Z,Yagi K,Oku Y,et al.A pilot study on developing mucosal vaccine against alveolar echinococcosis(AE)using recombinant tetraspanin 3:Vaccine efficacy and immunology[J].PLoS Negl Trop Dis,2012,6(3):e1570.
[13]Dang Z,Feng J,Yagi K,et al.Mucosal adjuvanticity of fibronectin-binding peptide(FBP)fused with Echinococcus multilocularis tetraspanin 3:systemic and local antibody responses[J].PLoS Negl Trop Dis,2012,6(9):e1842.
[14]Geourjon C,Deléage G.SOPMA.significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J].Comput Appl Biosci,1995,11(6):681-684.
[15]Vaughan K,Peters B,Larche M,et al.Strategies to query and display allergy-derived epitope data from the immune epitope database[J].Int Arch Allergy Immunol,2013,160(4):334-345.
[16]Sollner J,Grohmann R,Rapberger R,et al.Analysis and prediction of protective continuous B-cell epitopes on pathogen proteins[J].Immunome Res,2008,7(1)4:1.
[17]马秀敏,胡晓安,阿尔孜古丽·吐尔逊,等.细粒棘球绦虫AgB1抗原表位的生物信息学预测[J].科技导报,2013,31(27):27-30.
[18]李玲玲,朱珊丽,李文姝,等.EB病毒核蛋白-1 B细胞表位的预测及其同源性分析[J].生物医学工程学杂志,2011,28(2):371-375.
[19]Testa JS,Shetty V,Hafner J,et al.MHC class I presented T cell epitopes identified by immunoproteomics analysis are targets for a cross reactive influenza specific T cell response[J].PLoS One,2012,7(11):e48484.
[20]Zhang W,Liu J,Zhao M,et al.Predicting linear B-cell epitopes by using sequence-derived structural and physicochemical features[J].Int J Data Min Bioinform,2012,6(5):557-569.
[21]Mahdavi M,Mohabatkar H,Keyhanfar M,et al.Linear and conformational B cell epitope prediction of the HER 2 ECD-subdomain III by in silico methods[J].Asian Pac J Cancer Prev,2012,13(7):3053-3059.
[22]Xue M,Shi X,Zhang J,et al.Identification of a conserved B cell epitope on reticuloendotheliosis virus envelope protein by screening a phage-displayed random peptide library[J].PLoS One,2012,7(11):e49842.
Bioinformatic prediction of epitopes in the TSP3 antigen ofEchinococcusmultilocularis
Pang Mingquan1,3,Tang Feng2,3,4,Zhou Hu1,3,Wan Chenfei1,3,Yangdan Cairang1,3,4*,Fan Haining1,3,4**
(1.Department of Hepatopancreatobiliary Surgery,Affiliated Hospital of Qinghai University,Xining,Qinghai 810001;2.Research Center for High Altitude Medical Sciences,Qinghai University School of Medicine,Xining,Qinghai 810001;3.Research Institute for High Altitude Zoonosis of Qinghai University;
4.Qinghai Provincial Key Laboratory for hydatid,Xining,Qinghai 810001)
Objective The present study aims to predict the secondary structure and the T-and B-cell epitopes for theEchinococcusmultilocularisTSP3 antigen,in order to reveal the dominant epitopes of the antigen.Methods The secondary structure of the protein was analyzed using SOPMA server.The T-cell and B-cell epitopes of TSP3 were predicted using IEDB,Syfpeithi,Bcepred and ABCpred online software.The characteristics of hydrophilicity, flexibility, antigenic propensity and exposed surface area were predicted.Results The random coils and β sheets accounted for 25.68% and 4.05% of the secondary structure of the TSP3 protein,respectively.This was indicative of the presence of potential dominant antigenic epitopes in TSP3.Following bioinformatic analysis,numerous distinct antigenic epitopes of TSP3 were identified.The high-scoring T-cell epitopes were located at positions T33-42,T45-55,T53-63,T68-77,T80-90,T92-104,T110-122 and T134-144;whilst the likely B-cell epitopes were located at positions T18-33,T45-55,T53-63,T64-75,T80-90,T92-104 and T110-122.Conclusions Eight T cell and seven B cell dominant epitopes of the TSP3 antigen were revealed by the bioinformatic methods,which may be useful for the development of a dominant epitope vaccine.
EchinococcusmultilocularisTSP3 Bioinformatics Secondary structure Epitopes
R392-3
A
10.13452/j.cnki.jqmc.2016.01.001
2015-09-03
#:青海省科技厅项目(编号:2014-ZJ-716);青海大学中青年团队项目(编号:2014-QYT-1);
*:第一通讯作者,副教授,硕士研究生导师,yangdancairang@163.com;**:第二通讯作者,教授,博士研究生导师,fanhaining@medmail.com.cn.
庞明泉(1989~),男,汉族,山东籍,在读硕士,青海大学普通外科学专业

