弗氏链霉菌tylF基因的克隆及其生物信息学分析
闫明亮
(中国人民解放军海军工程大学电气工程学院,湖北武汉 430000)
弗氏链霉菌tylF基因的克隆及其生物信息学分析
闫明亮
(中国人民解放军海军工程大学电气工程学院,湖北武汉 430000)
摘要[目的]研究弗氏链霉菌tylF基因的克隆及其生物信息学分析。[方法]利用RT-PCR技术、巢式PCR技术和RACE技术从弗氏链霉菌B-62169菌株中克隆获得tylF基因的全长cDNA序列,并对其生物信息学进行分析。[结果]经Vector NTI 11.0软件拼接获得tylF基因全长cDNA序列长度为1 245 bp,并带有19 bp长的Poly(A)尾巴,包含927 bp的开放读码框(ORF),编码一个含309个氨基酸残基的蛋白质。生物信息学分析结果表明,tylF基因编码的酶是大菌素-O-甲基转移酶,参与分子功能中甲基转移酶活性和生物学途径中甲基化过程。对tylF基因全长cDNA序列进行蛋白质结构域分析,证实该基因编码TylF蛋白,并具有223个氨基酸蛋白结构域。[结论]该研究为阐明泰乐菌素生物合成过程中甲基化反应机理,更进一步研究大环内酯类抗生素生物合成代谢途径提供生物信息学数据支持。
关键词tylF;泰乐菌素;弗氏链霉菌;基因克隆
链霉菌(Streptomyces)属于放线菌目链霉菌科原核生物,是一类具有丝状分枝细胞的革兰氏阳性细菌[1]。泰乐菌素[2](Tylosin),又名泰乐星,是一类16元大环内酯类畜禽专用抗生素,其产生菌[2]有弗氏链霉菌(Streptomyces fradiae)、龟裂链霉菌(Streptomyces romosus)和吸水链霉菌(Streptomyces hygroscopicus),其中弗氏链霉菌是泰乐菌素的主要产生菌,同时也是工业化生产泰乐菌素的工程菌株。泰乐菌素是一种畜禽专用抗生素,其主要用于饲料添加剂和动物治疗如猪气喘病、鸡慢性呼吸道病、动物消化道疾病、地方性动物支气管炎等,能明显促进禽畜生长、提高饲料利用率[3]等,具有广阔的市场前景。
tylF基因编码的大菌素-O-甲基转移酶,催化泰乐菌素C3位羟基的甲基化反应[4]。泰乐菌素C3位羟基的甲基化反应是泰乐菌素生物合成的最后一步,也是限速反应[5]。目前关于弗氏链霉菌tylF基因克隆的研究较少,弗氏链霉菌研究主要集中于工程菌株的构建和发酵生产方面,2012年范亮等[6]利用基因重组技术,构建了具有双拷贝tylF基因的泰乐菌素基因工程菌株,结果比相同条件下未转化菌种生产泰乐菌素的能力提高了32.7%。但这些研究并未获得tylF基因全长cDNA序列,只是根据NCBI上的序列设计引物进行后续试验。笔者通过对tylF基因全长序列克隆及其生物信息学分析,不仅可以从分子角度进一步阐明泰乐菌素的生物合成过程,还可以更进一步研究大环内酯抗生素生物合成过程的未知代谢机理,明晰抗生素生物合成代谢途径中的未知假定。
1材料与方法
1.1材料
1.1.1菌种和试剂。弗氏链霉菌(Streptomyces fradiaeB-62169)购于北京微生物菌种保藏中心。RNA提取试剂盒购自美国Bio-Rad公司,RACE克隆试剂盒购自美国Bio-Rad公司,pMD18-T载体、RT-PCR以及大肠杆菌E.coliDH5α等相关试剂购自大连宝生物公司。
1.1.2培养基。大肠杆菌的培养基为LB培养基,弗氏链霉菌的固体培养基为AS-2(Yeasteztract)培养基,弗氏链霉菌的液体培养基为TSB(Tryptonesoyabroth)培养基。
1.2方法
1.2.1弗氏链霉菌总RNA的提取。将弗氏链霉菌在AS-2平板上活化培养,再从AS-2平板上挑取单菌落接种至含有25mLTSB培养基的250mL三角瓶中,在培养温度37 ℃、摇床220r/min下振荡培养10~12h后,将三角瓶静止放置10min,然后在12 000r/min下离心10min收集菌体细胞。按照RNA提取试剂盒说明书提取弗氏链霉菌总RNA,电泳检测RNA质量,并将其保存,为下一步反转录备用。
1.2.2tylF基因的克隆。
1.2.2.1cDNA第一条链合成。参考Bio-Rad公司RevertAidTMfirststrandcDNASynthesisKit反转录试剂说明书进行cDNA第一条链的合成,在42 ℃下反应1h,反应结束后在70 ℃下处理5min使逆转录酶失活,-20 ℃保存备用。
1.2.2.2RT-PCR反应体系。根据GenBank中tylF基因的氨基酸保守序列设计该基因简并引物PF和PR,引物序列见表1。PCR反应体系25.0μL:2.0μL模板cDNA,2.5μL10×TaqDNAPolymerasBuffer,0.9μL10mmol/LdNTPs,1.0μL引物F1,1.0μl引物R1,0.3μLTaqDNAPolymeras,ddH2O补齐至25.0μL。PCR反应条件:94 ℃预变性5min;94 ℃变性40s,59 ℃退火60s,72 ℃延伸60s,30个循环;72 ℃延伸10min。
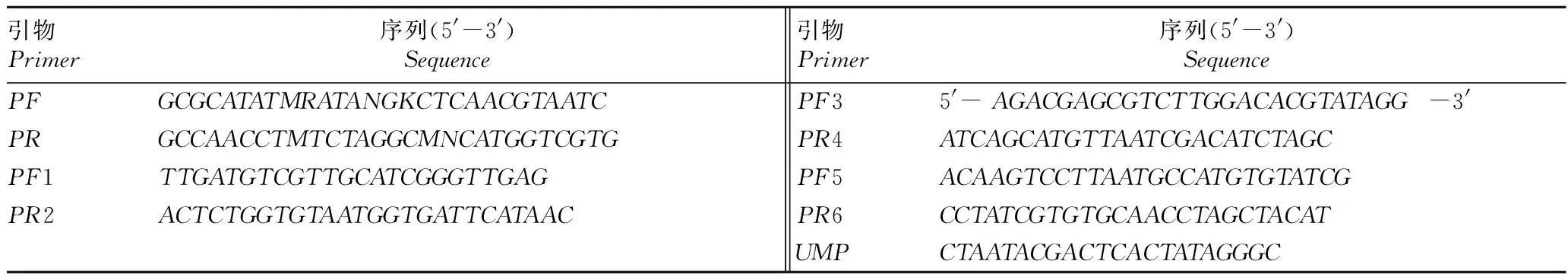
表1 弗氏链霉菌tylF基因克隆的引物
1.2.2.33′-RACE克隆。按照RACE克隆试剂盒(美国Bio-Rad公司)说明书进行RACE克隆。利用试剂盒合成3′-ReadycDNA并根据RT-PCR获得的已知序列设计2条反向嵌套PCR引物PF1、PR2(表1)。正向引物为试剂盒中所带UMP。用引物UMP和PF1配对3′-ReadycDNA进行第1轮PCR;并取第一轮PCR产物1.0μL为模板,用引物UMP和PR2配对进行第2轮PCR。
1.2.2.45′-RACE克隆。按照RACE克隆试剂盒(美国Bio-Rad公司)说明书进行RACE克隆。利用试剂盒合成5′-ReadycDNA并根据RT-PCR获得的已知序列设计2条反向嵌套PCR引物PF3、PR4(表1)。正向引物为试剂盒中所带UMP。用引物UMP和PF3配对5′-ReadycDNA进行第1轮PCR;并取第一轮PCR产物1.0μL为模板,用引物UMP和PR4配对进行第2轮PCR。
1.2.2.5全长cDNA的扩增。将3′-RACE和5′-RACE扩增获得的3′端和5′端cDNA序列在软件VectorNTI11.0上进行序列拼接,通过软件拼接获得全长cDNA序列,并利用获得的序列设计两端引物PF5、PR6(表1)扩增tylF基因全长的cDNA序列。PCR反应体系25.0μL:2.0μL模板cDNA,2.5μL10×TaqDNAPolymerasBuffer,0.9μL10mmol/LdNTPs,1.0μL引物F1,1.0μL引物R1,0.5μLTaqDNAPolymeras,ddH2O补齐至25.0μL。PCR反应条件:94 ℃预变性5min;94 ℃变性40s,60 ℃退火60s,72 ℃延伸60s,30个循环;72 ℃延伸10min。
1.2.2.6测序及序列生物信息学分析。PCR产物经1%琼脂糖凝胶电泳,在紫外灯下切下目的条带,采用AxyPrepDNA凝胶回收试剂盒回收后,将纯化回收的目的基因片段与pMD18-T载体连接。将连接好的产物转化大肠杆菌感受态细胞进行蓝白斑筛选,并进行PCR验证。将验证成功转化感受态的菌液应用M13通用引物进行测序,测序由上海生工生物工程技术服务公司完成。
1.2.3测序及测序结果生物信息学分析。将测序结果在GenBank中进行BLAST比对,利用Blast2go软件[7-8](http://www.blast2go.com/b2ghome)进行生物信息学分析,利用Pfam在线软件[9](http://pfam.xfam.org/)进行蛋白质结构域预测。
2结果与分析
2.1弗氏链霉菌tylF基因保守序列cDNA克隆与分析以弗氏链霉菌的cDNA为模板,利用设计好的引物PF和PR进行RT-PCR,结果获得片段约为800bp的目的条带。将该片段纯化后转化至大肠杆菌感受态中,筛选阳性菌落进行PCR检验和测序分析,结果表明,RT-PCR获得的目的片段长度为821bp(图1)。将该片段的核苷酸序列与NCBI中的序列进行比对,其结果与耻垢分枝杆菌和鸟型分枝杆菌中的tylF基因同源性分别达70%和76%,初步说明已经成功获得了tylF基因的部分序列。
注:M.DL2 000 Marker,1.阴性对照,2.tylF基因保守序列阳性克隆。Note:M.DL2 000 Marker, 1.Negative control, 2.Positive clone of tylF gene conserved sequence.图1 tylF基因保守序列电泳图谱Fig.1 Agarose gel electrophoretic analysis of tylF gene PCR products
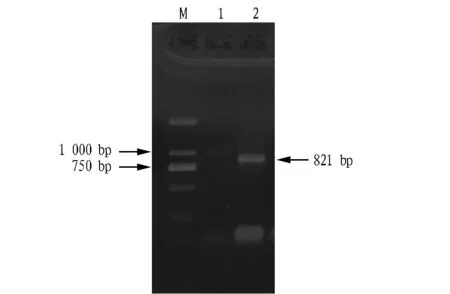
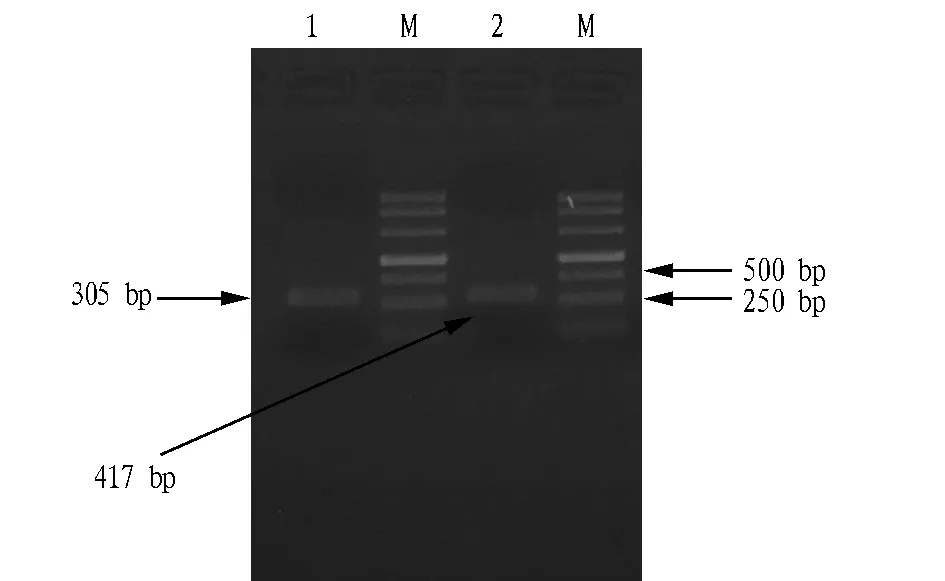
2.2弗氏链霉菌tylF基因全长cDNA序列的克隆与分析由图2可知,利用3′-RACE技术克隆获得cDNA片段长度为417bp,其中有196bp与已经克隆获得的821bp中间片段具有重叠区,同时带有19bp长的Poly(A)尾巴。利用5′-RACE技术克隆获得cDNA片段长度为305bp,其中有102bp与已经克隆获得的821bp中间片段具有重叠区。通过VectorNTI11.0软件对已经获得的3′-RACE片段序列、5′-RACE片段序列以及RT-PCR片段序列进行比对和拼接,从而获得弗氏链霉菌tylF基因全长cDNA序列。由图3可知,弗氏链霉菌tylF基因全长cDNA序列全长为1 245bp,并带有19bp长的Poly(A),tylF基因起始密码子ATG位于203nt处,终止密码子TAG位于1 130nt处。全长1 245bp序列中包含203bp的5′端非翻译区(UTR,untranslatedregion)和115bp的3′端非翻译区,以及927bp的开放读码框(ORF),编码一个含309个氨基酸残基的蛋白质,经预测编码TylF蛋白。
注:M.DL 2 000 Marker,1.5′-RACE PCR产物,2.3′-RACE PCR产物。Note:M.DL 2 000 Marker,1.5’-RACE PCR products,2.3’-RACE PCR products.图2 tylF基因RACE扩增电泳图谱Fig.2 Agarose gel electrophoretic analysis of tylF gene RACE PCR products
注:M.DL2 000 Marker,1和2.tylF基因全长cDNA序列阳性克隆,3.阴性对照。Note:M.DL 2 000 Marker,1 and 2.The cDNA sequence positive cloning of the whole tylF gene,3.Negative control.图3 tylF基因全长cDNA序列电泳图谱Fig.3 Agarose gel electrophoretic analysis of tylF gene cDNA sequence
2.3弗氏链霉菌tylF基因全长cDNA序列生物信息学分析将已经获得的弗氏链霉菌tylF基因全长序列编辑成fasta格式文件,利用生物信息学软件Blast2go对tylF基因进行功能注释分析和KEGG代谢途径分析。由图4可知,tylF基因被功能注释为大菌素-O-甲基转移酶。同时GO功能注释结果显示,其被注释到参与分子功能中甲基转移酶活性和生物学途径中甲基化过程。由图5可知,tylF基因被注释上的酶标号为EC2.1.1.101,是大环内酯类衍生物合成过程中的关键酶基因。由图6可知,获得的tylF基因全长cDNA序列包含了整个TylF蛋白保守结构域,其长度为223个氨基酸。
图4 tylF注释信息Fig.4 tylF gene annotation information
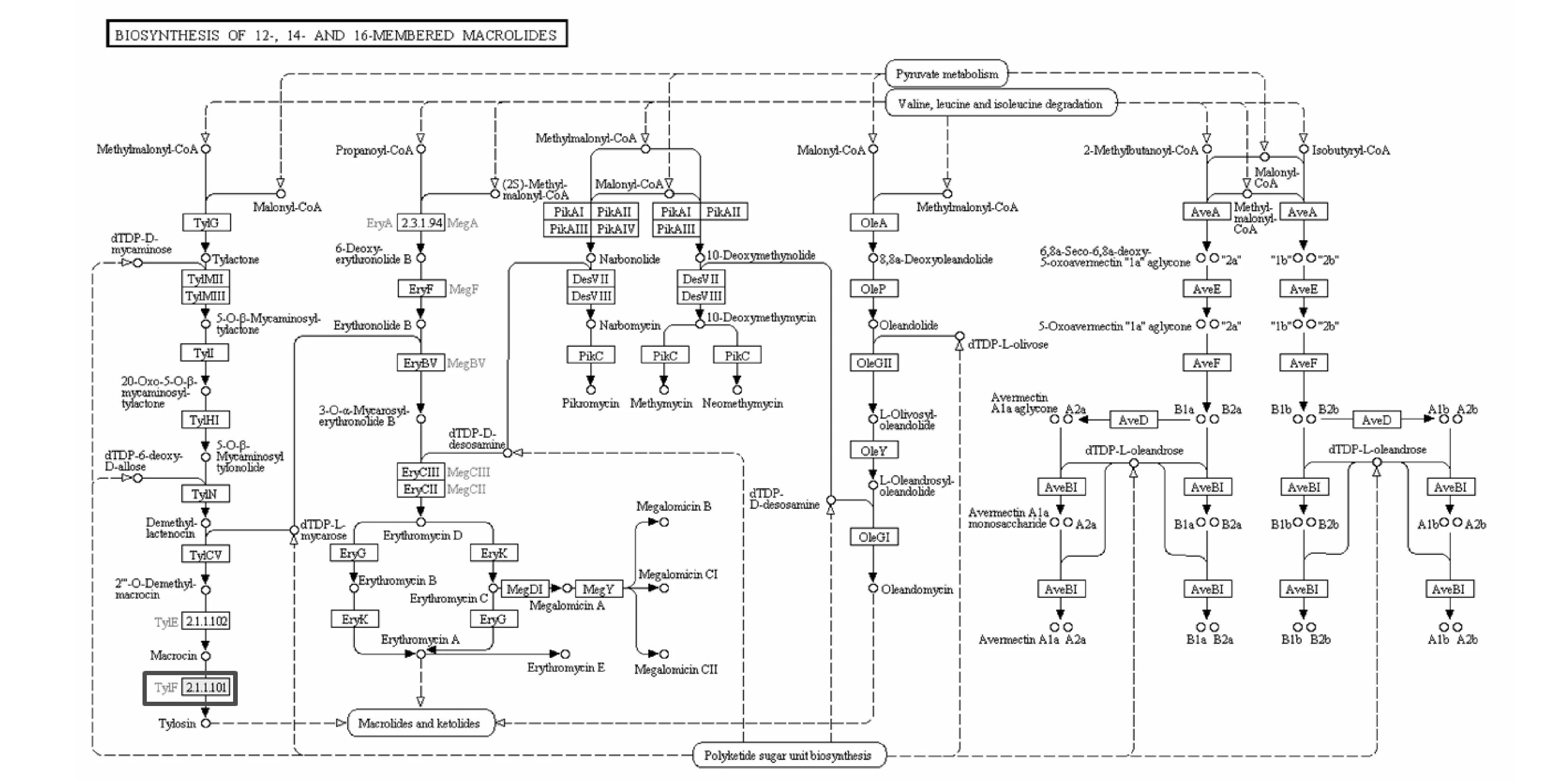
注:红色框标记为tylF基因所参与大环丙酯类衍生物合成的作用位置。Note: The red box was the interaction site of tylF gene participated in macrolide derivatives synthesis. 图5 大环内酯类衍生物合成途径Fig.5 Biosynthesis of meberde macrolides

图6 TylF蛋白保守域分析Fig.6 Analysis protein domain of TyLF gene
3结论与讨论
该研究利用RT-PCR技术从弗氏链霉菌B-62169菌株中克隆获得tylF基因的保守序列,同时利用巢式PCR技术、3′-RACE和5′-RACE技术获得tylF基因的全长cDNA序列。通过VectorNTI11.0软件拼接获得的弗氏链霉菌tylF基因全长cDNA序列全长为1 245bp,并带有19bp长的poly(A)尾巴,该基因起始密码子ATG位于203nt处,终止密码子TAG位于1 130nt处,全长序列中包含203bp的5′端非翻译区和115bp的3′端非翻译区,以及927bp的开放读码框(ORF),编码一个含309个氨基酸残基的蛋白质,经预测编码TylF蛋白。通过生物信息学分析软件对该基因进行了全面系统的分析,tylF基因编码的酶是大菌素-O-甲基转移酶,对该基因功能注释结果显示其参与分子功能中甲基转移酶活性和生物学途径中甲基化过程。tylF基因在KEGG代谢过程中被注释的酶标号为EC2.1.1.101,是大环内酯类衍生物合成过程中的关键酶基因。将获得tylF基因全长cDNA序列进行蛋白质结构域分析,证实该基因编码TylF蛋白,并具有223个氨基酸蛋白结构域。
在NCBI尚未公布有关弗氏链霉菌中tylF基因全长cDNA序列的信息,仅有耻垢分枝杆菌、鸟型分枝杆菌等分歧杆菌中tylF基因mRNA序列。该研究结合生物学知识和生物信息学分析,更进一步对tylF基因信息进行研究,重点从转录组学层面上解释了tylF基因参与大环内酯类衍生物合成途径中的关系,明晰了泰乐菌素抗生素合成代谢的积累机制,为今后定向提高大环内酯抗生素的产量提供参考。
参考文献
[1]KIESERT,BIBBMJ,BUTTNERMJ,etal.Practicalstreptomycesgenetics[M].Norwich,UK:JohnInnesFoundationNorwich,2000.
[2]HAMILLRL.Tylosin,anewantibiotic[J].Antib&Chemoth,1960,11:328-334.
[3] 闵勇.弗氏链霉菌及耐热链霉菌的遗传改造[D].武汉:华中农业大学,2006.
[4]ARISAWAA,KAWAMURAN,TSUNEKAWAH,etal.Cloningandnucleotidesequencesoftwogenesinvolvedinthe4″-O-acylationofmacrolideantibioticsfromStreptomyces thermotolerans[J].BiosciBiotechBiochem,1993,57:2020-2025.
[5] ARISAWA A,TSUNEKAWA H,OKAMURA K,et al.Nucleotide sequence analysis of the carbomycin biosynthetic genes including the 3-O-acyltransferase gene fromStreptomycesthermotolerans[J].Biosci Biotech Biochem,1995,59:582-588.
[6] 范亮,杜宝华,陈光,等.泰乐菌素基因工程菌的构建[J].沈阳药科大学学报,2012,29(5):398-401.
[7] CONESA A,GOTZ S,GARCA-GOMEZ J M,et al.Blast2go:A universal tool for annotation,visualization and analysis in functional genomics research[J].Bioinformatics,2005,21(18):3674-3676.
[8] APARICIO G,GOTZ S,CONESA A,et al.Blast2go goes grid:Developing a grid-enabled prototypefor functional genomics analysis[J].Stud Health Technol Inform,2006,120:194-204.
[9] FINN R D,BATEMAN A,CLEMENTS J,et al.Pfam:The protein families database[J].Nucleic acids research,2014,42:222-230.
CloningandBioinformaticsAnalysisoftylFGeneinStreptomyces fradiae
YANMing-liang
(CollegeofElectricalEngineering,NavalUniversityofEngineering,PLA,Wuhan,Hubei4300000)
Abstract[Objective] To research the cloning and bioinformatics analysis of tylF gene in Streptomyces fradiae. [Method] The partial cDNA of tylF was cloned from Streptomyces fradiae B-62169 through RT-PCR technique by specific primers designed on the basis of tylF gene in GenBank website. And its bioinformatics analysis was carried out. [Result] A 1245 bp full length cDNA sequence of tylF was obtained using nested PCR and RACE technique. The cloned tylF cDNA contained an open reading frame (ORF) of 927 bp and was predicted to encode a deduced protein with 309 amino acid residues. Bioinformatics analysis results showed that the gene encoding enzymes was macrocin-O-methyl transferase, which participated in the methylation process of methyltransferase activity and biological pathways in molecular function. Full-length cDNA sequence of the tylF structural gene for protein domain analysis confirmed that the gene encoding TylF protein having 223 amino acids was completed and the protein domains. [Conclusion] This research provides data support for the methylation reaction mechanism of tylosin biosynthetic process, and the further researches on biosynthesis and metabolic pathways of macrolides antibiotics.
Key wordstylF; Tylosin; Streptomyces fradiae; Gene cloning
作者简介闫明亮(1993- ),男,吉林通化人,本科生,专业:电气工程。
收稿日期2016-03-30
中图分类号Q 81
文献标识码A
文章编号0517-6611(2016)12-126-04

