基于线粒体COI基因的毛蚶群体遗传多样性
田吉腾, 侯 丫, 2, 刘志鸿, 杨爱国, 吴 彪, 周丽青, 董春光, 2(. 农业部海洋渔业可持续发展重点实验室, 中国水产科学研究院 黄海水产研究所, 山东 青岛26607; 2. 上海海洋大学 水产与生命学院, 上海 20306)
基于线粒体COI基因的毛蚶群体遗传多样性
田吉腾1, 侯 丫1, 2, 刘志鸿1, 杨爱国1, 吴 彪1, 周丽青1, 董春光1, 2
(1. 农业部海洋渔业可持续发展重点实验室, 中国水产科学研究院 黄海水产研究所, 山东 青岛266071; 2. 上海海洋大学 水产与生命学院, 上海 201306)
采用PCR 技术, 扩增了大连、乳山、烟台、舟山4个毛蚶(Scapharca subcrenata)地理群体共38个个体的线粒体COI 基因部分序列, 并分析了4个毛蚶群体的遗传多样性和系统发育关系。研究结果显示: 38个毛蚶COI部分序列经处理得到长度均为625bp的基因片段, 共分为30种单倍型; 基于COI部分序列的分析结果, 毛蚶4个地理群体总的变异位点为301个, 多样性指数Pi为0.15048, 平均核苷酸差异数为92.242, 单倍型多样性指数S为241。聚类分析显示毛蚶大连群体、乳山群体和烟台群体具有高度的遗传多样性, 3个群体交叉聚在一起, 没有明显的群体分化; 舟山群体单独聚为一支, 与其他3个群体分化明显。研究表明, 线粒体COI 基因不能单独做为毛蚶大连、乳山和烟台群体的遗传标记, 但可以作为毛蚶舟山群体的有效群体遗传标记, 为线粒体COI基因在群体遗传学的应用提供了基础资料。
毛蚶(Scapharca subcrenata); COI 基因; 群体; 遗传多样性
[Foundation : National Basic Research Special Foundation of China (2013FY110700); Special Scientific Research Funds for Central Non-profit Institutes, Yellow Sea Fisheries Research Institutes (20603022013032)]
毛蚶(Scapharca subcrenata), 俗称瓦楞子或毛蛤,属软体动物门(Mollusca), 双壳纲(Bivalvia), 翼形亚纲(Pteriomorphia), 蚶目(Arcoida), 蚶科(Arcidae),毛蚶属(Scapharca), 广泛分布于中国、朝鲜和日本沿海[1], 在中国的渤海、黄海、东海、南海均有大量分布, 是中国北方重要经济贝类之一。毛蚶普遍生活在低潮线以下, 至水深几十米的海域, 喜欢栖息于软泥质底或泥沙质底[2-3]。20世纪60~70年代, 毛蚶资源丰富, 曾为主要的捕捞对象之一。但由于捕捞强度过大, 毛蚶资源遭到了严重的破坏, 资源衰退。近年来, 毛蚶的增养殖工作以及各方面的研究已经引起人们的重视[4]。迄今人们对于毛蚶的研究, 主要集中在亲贝的促熟、苗种的培育和养成技术, 以及生理生化、生态习性、饵料需求等方面[4-7], 分子遗传多样性方面未见报道。
群体遗传多样性水平与生物的生长速度、抗病能力和物种的进化速度有重要的关系。近年来, 线粒体基因组的研究在一定程度上促进了群体遗传学的发展,并且实现了从分子水平上对自然群体的保护[8]。线粒体中的COI基因, 即线粒体细胞色素C 氧化酶亚基Ⅰ, 作为一种分子标记, 具有多种优势: (1)COI基因在物种进化中属于中度变异, 其进化速率一般大于ITS-1的进化速率, 能够从DNA分子水平上成功的区分物种。(2)COI基因的引物通用性比较强, 可操作性强。(3)COI基因密码子的第3个位点存在更多的碱基置换, 并且长度约为650bp, 能够表现出足够物种的的差异和变化, 同时也刚好适宜进行扩增和后期检测。
在海洋贝类中, 线粒体COI基因也广泛用于分析群体的遗传多样性, 如对文蛤[9], 光滑河蓝蛤[10],泥蚶[11], 缢蛏[12]等研究表明, COI基因可以用于分析物种的群体内和群体间的遗传距离, 分析个体差异,从而推断群体的遗传分化水平及基因交流的可能性。本实验通过测定毛蚶群体的线粒体的COI基因部分序列, 进行遗传分析, 构建系统进化树, 旨在分析毛蚶遗传多样性水平和不同地理群体的系统进化关系, 并推测线粒体COI基因能否作为区分毛蚶不同群体的有效分子标记, 同时为毛蚶的资源保护及群体遗传学研究提供基础资料。
1 材料与方法
1.1 实验材料
本研究所用的4个毛蚶地理群体样品分别取自辽宁大连旅顺(2013年6月1日), 山东乳山白沙滩(2013年4月2日), 山东烟台芝罘岛(2013年4月1日), 浙江舟山普陀(2013年4月18日)近海, 样品是活体加冰运输, 迅速解剖, 取出闭壳肌部分, 置于–80℃超低温冰箱保存。
1.2 基因组DNA提取和PCR扩增
每个群体随机选取9~10个样品, 采用常规的酚氯仿法提取基因组DNA[13], 最终得到的DNA模板稀释至100ng/μL。使用COI通用引物(F: GGT CAA CAA ATC ATA AAG ATA TTG G; R: TAA ACT TCA GGG TGA CCA AAA AAT CA[14])进行毛蚶4个群体COI基因片段的扩增。PCR采用Easy Taq DNA聚合酶进行扩增, PCR反应在Eppendorf PCR仪上进行。反应体系为30μL, 包括3μL PCR缓冲液(Mg2+plus), 4μL 高纯度的脱氧核糖核苷三磷酸(2.5 mmol/L), 10μmol/L的正反方向引物各2μL,1μL Taq酶(5 U/μL), 2μL DNA模板(80 ng/μL),16μL灭菌水。PCR反应程序为: 94℃预变性5min;(94℃: 20s, 52℃: 50s, 72℃: 1min; )30个循环, 最后72℃延伸10 min。所得PCR产物用1%琼脂糖凝胶电泳检测后利用美国UVP凝胶成像系统进行观察拍照。
1.3 测序和数据处理
将琼脂糖凝胶电泳检测结果中条带较单一的PCR扩增产物样品送于北京华大基因生物有限公司进行PCR产物纯化测序。所得的结果进行NCBI BLAST比对验证同源性, 保证测序结果的可靠性。利用DnaSP 5软件[15]分析毛蚶群体单倍型数(Number of Haplotype, H)、平均核苷酸差异(Average number of nucleotide difference, K)及核苷酸多样性指数(Nucleotide diversity, Pi)等遗传多样性参数, 运用MEGA 5.0软件[16]对序列的碱基组成(Nucleotide composition)、多态位点(Number of polymorphic sites, S)、变异位点(Variable sites)进行分析, 采用NJ(Neighbor-joining)法构建毛蚶4个地理群体的系统发育树, Bootstrap置信值设为1000。
2 结果与分析
2.1 序列的碱基组成
测序后获得的COI基因片段长度不一, 经序列对准去除序列两端部分碱基后, 得到38条长度均为625 bp的COI基因片段。4个群体毛蚶COI基因部分片段的碱基组成(表1), A+T含量普遍高于G+C含量, 这与Medina等的研究一致[17]。大连、乳山、烟台3个群体COI序列碱基组成相差不大, 而舟山群体尽管在A+T含量上与其他3个群体相近, 但其碱基A的含量比其他3个群体低19.9%, T的含量高16.7%。说明舟山群体在遗传系统分化上可能占有较为特殊的地位。

表1 毛蚶不同群体COI基因部分序列的碱基组成Tab. 1 Base compositions of COI gene partial sequences of four populations of S. subcrenata
2.2 遗传多样性分析
毛蚶群体COI基因片段遗传多样性指数(表2),大连、乳山和烟台群体序列之间差异不大, 多态位点分别占总数的28.8%, 35.0%和28.0%, 多样性丰富。乳山群体的平均核苷酸差异指数最高, 为106.58。舟山群体9个个体的COI基因部分序列中仅有2个多态位点, 群体特征明显, 群体遗传分化很小, 平均核苷酸差异指数最低, 仅为0.44。
由于大连群体中DL1和烟台群体中YT7单倍型是相同的, 因此本文研究的38个毛蚶个体中共存在30个单倍型, GenBank序列号为KP253046-KP253075,对应的个体依次为DL(2、3、4、6、7、8), RS(6、5、8、10), YT(5、8、9、10), ZS(2、1、5、6), YT(1、2、3、4), RS(3、4、1、2), DL(5、9), RS(9、7)。30个单倍型的核苷酸多态位点见表3。
2.3 遗传距离
根据4种毛蚶群体COI基因部分序列计算的群体内和群体间的遗传距离如表4所示。乳山群体内遗传距离最大, 为0.21, 舟山群体内遗传距离最小,为0.0011。群体间遗传分析表明, 毛蚶舟山群体和大连群体间的遗传距离最大, 为0.26, 烟台群体和大连群体之间遗传距离最小, 为0.15。
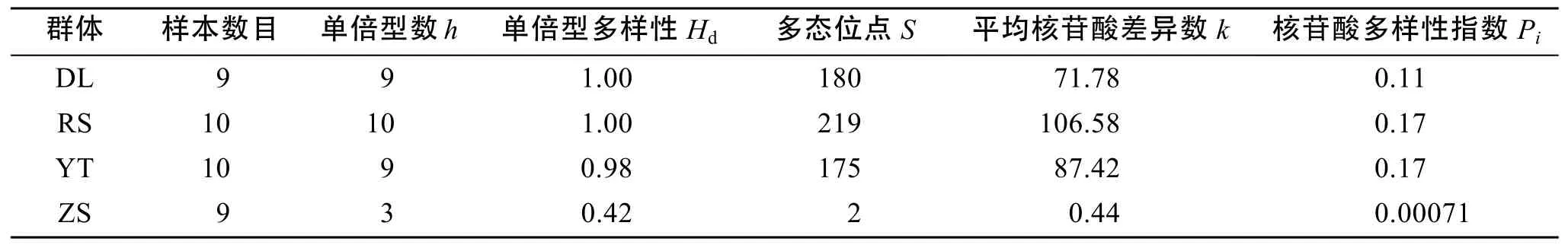
表2 毛蚶不同群体COI基因片段的遗传多样性指数Tab. 2 The genetic diversity parameters of COI gene partial sequences in different populations of S. subcrenata
2.4 分子系统进化树的构建
根据4种毛蚶群体COI基因部分序列建立的系统进化树(图1)。可以看出, 舟山群体首先单独聚在一起, 乳山群体、烟台群体和大连群体则相互交叉聚合在一起, 没有明显的先聚在一起的情况。
3 讨论
3.1 毛蚶4个群体的遗传多样性
本实验中毛蚶群体的多态位点较多, 核苷酸多样性指数较高, 可能的原因分以下两种: (1)选取样品的时候要先调查其来源, 距离较近的物种可能受到外来物种的污染或人为因素的干扰, 如群体中的个体经过捕捞作业和商户之间的转手买卖可能群体之间的交叉所致。(2)烟台和乳山地理位置较近, 在遗传分化上不明显, 两群体和大连群体均与黄海相邻, 地理群体之间可能存在一定的基因交流。
李旭光等[18]对毛蚶江苏海州湾群体、浙江象山港群体和辽宁辽东湾群体的同工酶遗传多样性进行了研究, 发现毛蚶群体具有较为丰富的遗传多样性,这与本文的研究结果相一致。毛蚶大连、烟台和乳山群体均具有较高的遗传变异水平, 这说明毛蚶对环境的适应能力很强, 也是毛蚶能够在中国沿海地区广泛分布的原因之一[19]。王晓梅等[20]对毛蚶3个群体核糖体ITS区进行了RFLP分析, 发现遗传多样性最低的为宁波样本, 而宁波群体和舟山群体距离很近, 存在一定的基因流, 从而与本实验中舟山群体遗传多样性的研究结果一致。
王晓梅等[20]发现毛蚶ITS区限制性片段长度多态性很低, ITS区564 bp的序列中多态位点仅为3.37%, 与本研究中高多态性的结果不一致, 原因是在线粒体基因组中, 不同基因的进化速率并不相同,而COI基因的进化速率一般大于ITS-1的进化速率,因此所得出的结论并不矛盾。
3.2 群体间的遗传进化关系
陈蓉[1]等采用形态性状多变量分析方法, 对中国5个野生毛蚶群体进行了分析, 发现在形态学方面, 毛蚶天津塘沽和山东青岛群体最为接近, 广西北海群体与其他群体差异最大。形态最为一致说明具有较近的进化关系, 与本研究结果中山东青岛与辽宁大连群体遗传关系相近的结果基本一致。
王晓梅等[20]构建的NJ系统发育树中显示毛蚶大连群体和塘沽群体没有明显的群体分化, 宁波群体单独聚为一支。李旭光等人[18]的聚类分析中, 江苏海州湾群体首先与宁波象山港群体相聚, 后与辽东湾群体聚类。系统发育关系均表明环渤海湾的群体遗传距离较近, 和东海海岸附近的群体遗传关系较远。
3.3 COI基因作为群体之间遗传进化关系标记的讨论
本研究分析表明, 线粒体COI基因的进化速度相对适中, 不仅对相近的物种进行识别鉴定, 也可以对具有地理隔离的群体进行分类鉴定。
4 结论
基于COI基因片段的研究结果表明, 中国北部的毛蚶群体如烟台、大连、乳山群体具有较高的遗传多样性, 群体内差异大, 群体间差异不大。而舟山群体遗传多样性较低, 群体内遗传差异很小, 与北方群体具有较大的遗传距离。
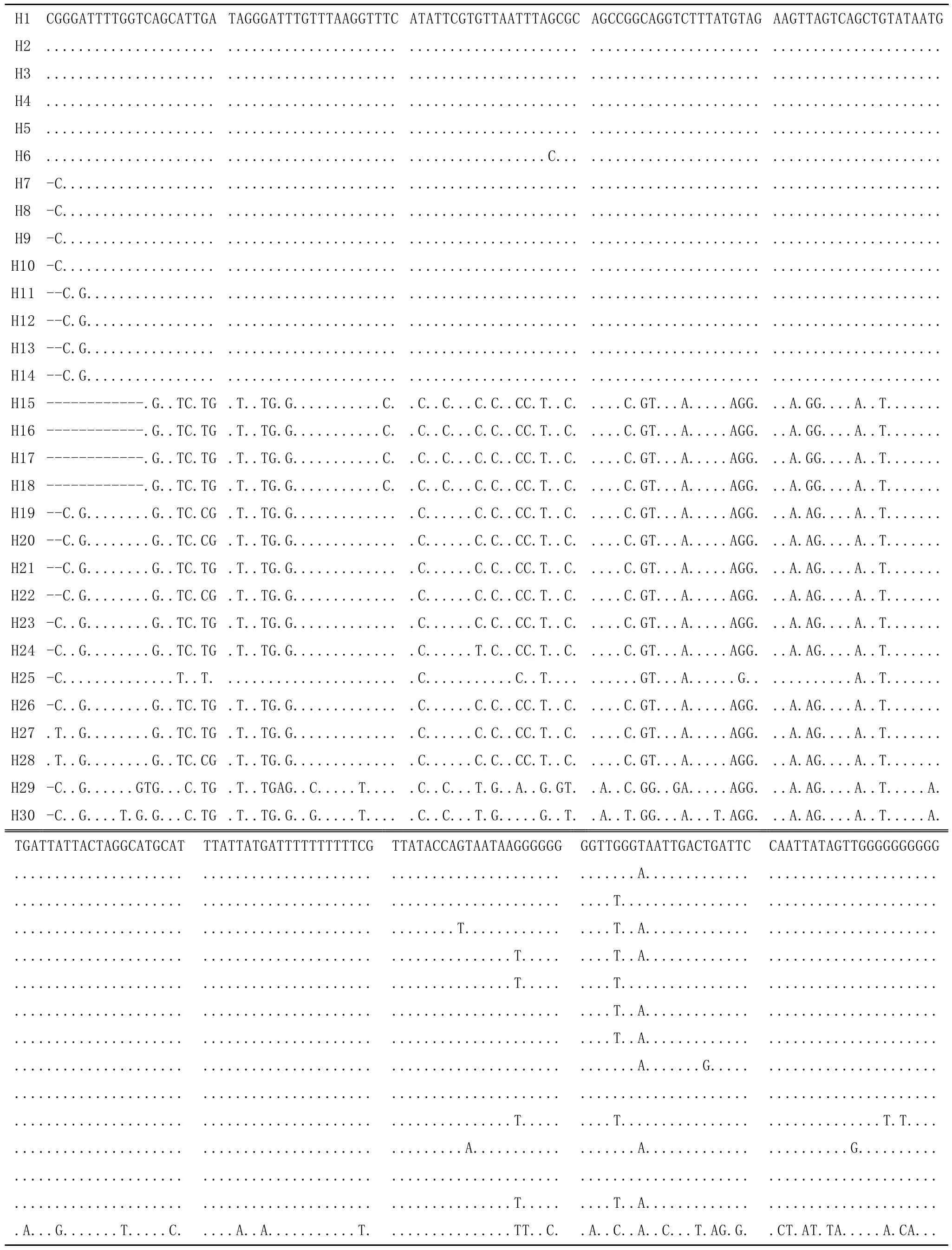
表3 毛蚶4个地理群体30个单倍型的核苷酸多态位点Tab. 3 Nucleotide polymorphism in four geographical populations of 30 haplotypes of S. subcrenata

续表

续表

续表
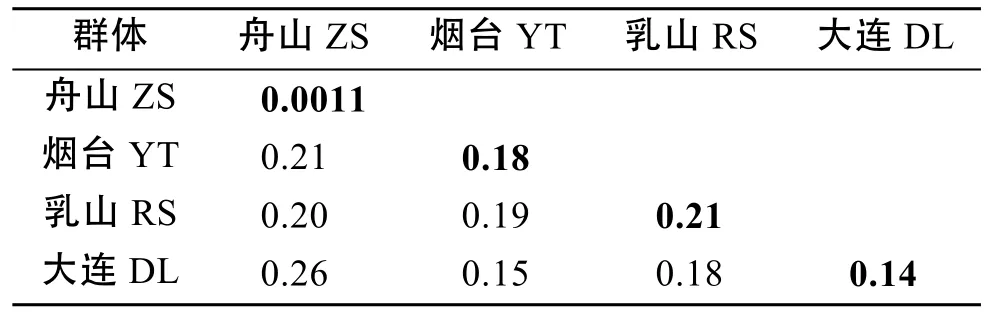
表4 毛蚶4个群体内和群体间的遗传距离Tab. 4 The genetic distance within and between different populations of S. subcrenata

图1 基于毛蚶4个群体COI部分序列建立的系统发育关系Fig. 1 Phylogenetic relationships among four groups of S. subcrenata based on COI gene partial sequences
[1] 陈蓉, 刘建勇, 唐连俊, 等. 中国5个海区不同群体毛蚶形态差异分析[J]. 海洋科学, 2009, 33(7): 64-69. Chen Rong, Liu Jianyong, Tang Lianjun, et al. Morphological variations analysis of five different populations of Scapharca subcrenata in China [J]. Marine Sciences, 2009, 33(7): 64-69.
[2] 齐钟彦. 中国经济软体动物[M]. 北京市: 中国农业出版社, 1998. Qi Zhongyan. Economic Mollusc of China [M]. Beijing:Chinese Agricultural Press, 1998.
[3] 赵汝翼. 大连海产软体动物志[M]. 北京市: 海洋出版社, 1982. Zhao Ruyi. Zoography of Dalian Marine Mollusc [M]. Beijing: Ocean Press, 1982.
[4] 陈建华, 阎斌伦, 高焕. 毛蚶生物学特性及其研究进展[J]. 河北渔业, 2006, 9(1): 24-26. Chen Jianhua, Yan Binlun, Gao Huan. Biological characteristics of ark shell Scapharca subcrenata and the research progress[J]. Hebei Fisheries, 2006, 9(1): 24-26.
[5] 赵新生, 徐国成, 万东善. 毛蚶升温促熟人工育苗技术研究[J]. 中国水产, 2008, 3: 58-61. Zhao Xinsheng, Xu Guocheng, Wan Dongshan. Study on artificial seeding technique of Scapharca subcrenata by promoting temperature for sexual maturity [J]. China Fisheries, 2008, 3: 58-61.
[6] 杨光, 杨玉香. 毛蚶幼体下沉原因分析及解决方法[J].河北渔业, 2012, 6: 32-48. Yang Guang, Yang Yuxiang. Analysis and solution of the Scapharca subcrenata larvae subsidence[J]. Hebei Fisheries, 2012, 6: 32-48.
[7] 阎斌伦, 许星鸿, 郑家声, 等. 毛蚶的性腺发育和生殖周期[J]. 海洋湖沼通报, 2005, 4: 92-98. Yan Binlun, Xu Xinghong, Zheng Jiasheng, et al. Gonadial development and the reproductive cycle of ark shell Scapharca subcrenata[J]. Transactions of Oceanology and Limnology, 2005, 4: 92-98.
[8] Mao Yangli, Gao Tianxiang, Takashi Yanagimoto, et al. Molecular phylogeography of Ruditapes philippinarum in the Northwestern Pacific Ocean based on COI gene[J]. Journal of Experimental Marine Biology and Ecology. 2011, 407: 171-181.
[9] 程汉良, 夏德全, 吴婷婷, 等. 6种帘蛤科贝类及4个地理种群文蛤线粒体COI基因片段序列分析[J]. 海洋学报(中文版), 2007, 5: 109-116. Cheng Hanliang, Xia Dequan, Wu Tingting, et al. Sequence analysis of mitochondrial COI gene fragment of six Veneridae clams (Mollusca: Bivalvia) and four populations of Meretrix meretrix[J]. Acta Oceanologica Sinica. 2007, 5: 109-116.
[10] 孙超, 刘志鸿, 杨爱国, 等. 光滑河蓝蛤3个野生群体线粒体COI基因遗传多样性研究[J]. 湖南农业科学, 2013, 7: 4-7. Sun Chao, Liu Zhihong, Yang Aiguo, et al. Genetic diversity of mitochondrial COI gene in three wild populations of Potamocorbula laevis[J]. Hunan Agricultural Sciences. 2013, 7: 4-7.
[11] 郑文娟, 朱世华, 沈锡权, 等. 基于线粒体COI基因序列探讨泥蚶的遗传分化[J]. 动物学研究, 2009, 1: 17-23. Zheng Wenjuan, Zhu Shihua, Shen Xiquan, et al. Genetic differentiation of Tegillarca granosa based on mitochondrial COI gene sequences[J]. Zoological Research, 2009, 1: 17-23.
[12] 牛东红, 李家乐, 沈和定, 等. 缢蛏六群体线粒体DNA-COI基因序列变异及群体遗传结构分析[J]. 海洋学报(中文版), 2008, 3: 109-116. Niu Donghong, Li Jiale, Shen Heding, et al. Sequence variability of mitochondrial DNA-COI gene fragment and population genetic structure of six Sinonovacula constricta populations[J]. Acta oceanologica sinica. 2008, 3: 109-116.
[13] Green M R, Sambrook J. Molecular cloning: a laboratory manual[M]. Cold Spring Harbor Laboratory Press Cold Spring Harbor, New York, 2012.
[14] Vrijenhoek R. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates[J]. Molecular marine biology and biotechnology, 1994, 3(5): 294-299.
[15] Rozas J, Sánchez-DelBarrio J C, Messeguer X, et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods [J]. Bioinformatics, 2003, 19: 2496-2497.
[16] Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using Maximum Likelihood, Evolutionary distance, and maximum parsimony methods[J]. Mol Biol Evol, 2011, 28: 2731-2739.
[17] Medina M, Walsh P J. Molecular systematics of the order Anaspidea based on mitochondrial DNA sequence (12S, 16S, and COI) [J]. Mol Phylogenet Evol,2000, 15(1): 41-58.
[18] 李旭光, 阎斌伦, 许广平, 等. 毛蚶三个地理群体生化遗传特征分析[J]. 海洋渔业, 2007, 3: 207-213. Li Xuguang, Yan Binlun, Xu Guangping, et al. Analysis on biochemical genetic variation in three geographical populations of Scapharca subcrenata[J]. Marine Fisheries, 2007, 3: 207-213.
[19] Buroker N E. Population genetics of the American oyster Crassostrean Ⅱalong the Atlantic coast and the Gulf of Mexico[J]. Mar Bio1, 1983, 75, 99-112.
[20] 王晓梅, 郭永军, 戴伟, 等. 不同群体毛蚶rDNA转录间隔区RFLP及序列分析[J]. 四川动物, 2008, 5: 774-776. Wang Xiaomei, Guo Yongjun, Dai Wei, et al. RFLP and Sequence Analysis of Ribosomal DNA Internal Transcribed Spacers of Different Geographical Populations of Scapharca subcrenata[J]. Sichuan Journal of Zoology, 2008, 5: 774-776.
(本文编辑: 梁德海)
Genetic diversity of different populations of Scapharca subcrenata based on mitochondrial COI gene
TIAN Ji-teng1, HOU Ya1, 2, LIU Zhi-hong1, YANG Ai-guo1, WU Biao1, ZHOU Li-qing1,DONG Chun-guang1, 2
(1. Key Laboratory of sustainable development of marine fisheries, Ministry of agriculture, the Yellow Sea Institute of fisheries, Chinese Academy of Fishery Sciences, Qingdao 266071, China; 2. College of Fisheries and Life Sciences, Shanghai Ocean University, Shanghai 201306, China)
May, 9, 2015
Scapharca subcrenata; COI gene; population; diversity
General PCR technology was used for the amplification of mitochondrial COI gene partial sequences of four groups of Scapharca subcrenata (Dalian, Rushan, Yantai, and Zhoushan). A total of 38 sequences from different populations were determined and analyzed. The results are as follows: (a) After excluding both sides of the inaccurate sequences, 38 COI gene partial sequences of S. subcrenata, 625 bp each, were eventually identified, among which 30 haploid types were detected. (b) Based on partial COI gene sequence analysis of the four groups of S. subcrenata, the total variation loci, haploid type diversity index S, nucleotide diversity index Pi, and average nucleotide difference index were 301, 241, 0.15048, and 92.242, respectively. (c) Cluster analysis showed that ark shells from three groups, Rushan, Dalian, and Yantai, had a high degree of genetic diversity, and they clustered across, without the obvious characteristics of the groups. However, ark shells from the Zhoushan group gathered themselves together, apart from the other three groups obviously. The genetic diversity index of Zhoushan group was much lower than that of other groups. (d) Mitochondrial COI gene could not separate the ark shells from Rushan, Dalian, and Yantai but can be a marker for Zhoushan populations of S. subcrenata, which provides more basic data for the application of mitochondrial COI gene in population genetics.
S917.4
A
1000-3096(2016)01-0001-09
10.11759/hykx20140429002
2015-05-09;
2015-09-10
国家科技部基础工作专项 (2013FY110700) ; 黄海水产研究所级基本科研业务费资助(20603022013032)
田吉腾(1986-), 男, 助理研究员, 主要从事海洋生物种质资源与遗传育种的研究, E-mail: 0708jixiang@163.com; 刘志鸿(1972-),通信作者, 女, 研究员, E-mail: liuzh@ysfri.ac.cn, 电话: 0532-85836340

