中间锦鸡儿转录因子基因CiNAC1的克隆及功能分析
岳俊燕,岳文冉,杨 杞,于秀敏,杨飞芸,王光霞,李国婧,王瑞刚,丛靖宇
(内蒙古农业大学, 呼和浩特 010018)
中间锦鸡儿转录因子基因CiNAC1的克隆及功能分析
岳俊燕,岳文冉,杨杞,于秀敏,杨飞芸,王光霞,李国婧,王瑞刚,丛靖宇*
(内蒙古农业大学, 呼和浩特 010018)
NAC转录因子家族是植物特有的、最大的转录因子家族之一,在植物应答非生物胁迫和生长发育过程中有重要的功能。该研究通过PCR技术,克隆得到了中间锦鸡儿CiNAC1基因1 066 bp的cDNA全长序列。生物信息学分析显示,CiNAC1基因的开放阅读框(ORF)为921 bp,编码306个氨基酸,推导的蛋白分子量为34.57 kD,等电点为8.35,是一种亲水性蛋白,N端具有保守的NAM结构域,具有26个磷酸化位点和7个糖基化位点。实时荧光定量PCR检测显示,中间锦鸡儿CiNAC1基因表达受干旱、高盐、脱水、高pH诱导;亚细胞定位发现CiNAC1定位到细胞核中,这与它作为转录因子的功能是一致的;转CiNAC1基因拟南芥株系侧根数目显著多于野生型,根长也明显比野生型长。研究认为,CiNAC1基因可能与中间锦鸡儿响应逆境胁迫机制有关。
中间锦鸡儿;NAC转录因子;非生物胁迫;功能分析
植物在生长发育过程中,会遭受到一些生物胁迫或非生物胁迫因素的影响,如干旱、高温、盐碱、冷、虫害等。植物在受到这些胁迫以后,细胞会受到不同程度的损伤[1]。植物为了更好地适应环境,经过长期的进化形成了一些机制来抵制各种生物和非生物胁迫。当植物受到胁迫刺激时,就会引发一系列生理生化反应。转录因子(Transcription factor,TF)在植物响应逆境胁迫的分子信号通路中,具有极为重要的调控作用。通过调控相应的应答基因的表达,从而使植物产生应对逆境胁迫的能力。
NAC转录因子是近10年新发现的最大的一类植物特有的转录因子[2]。NAC家族的命名源于矮牵牛(Petuniahybrida)的NAM(no apical meristem)和拟南芥(Arabidopsisthaliana)的ATAF1、ATAF2,以及CUC2(cup-shape cotyledon)基因[3-4]。1996年,Souer从矮牵牛中克隆得到了第1个NAC转录因子[5],随后在拟南芥、水稻、烟草、小麦和大豆等物种中相继都有发现。迄今为止,NAC转录因子已在拟南芥中至少发现117个,水稻中有151个[6],大豆[7]和烟草[8]中都至少有152个,杨树中至少含有163个[9]。
NAC转录因子包含一个N端高度保守的DNA结合结构域和C端高度变异的转录激活结构域。N端高度保守,由大约160个氨基酸残基组成,可分为A、B、C、D和E等5个亚结构域。其中,B和E亚结构域的保守性不强,而A、C、D亚结构域则高度保守,并且C、D亚结构域中包含有核定位信号[10]。N端是DNA结合域,主要负责与DNA及其他蛋白的结合;C端是高度变异和具有转录激活功能的调控区,为转录激活域[11],此区域的特点是频繁的出现简单氨基酸的重复序列,如丝氨酸、苏氨酸、脯氨酸和谷氨酸,或酸性残基[12-13]。
NAC转录因子在植物的生长发育过程中具有重要的作用,它主要参与的过程有:植物细胞次生壁的生成,如ANAC012既能增加木质部导管的细胞壁增厚,又能抑制拟南芥木纤维细胞中次生壁的生成[14];植物顶端分生组织形成,如CUC1、CUC2与植物顶端分生组织的形成有关,在拟南芥CUC1和CUC2的双突变体中,子叶、雄蕊和萼片融合,难以形成顶端分生组织[15];植物侧根发育,如AtNAC2过表达促进侧根发育,乙烯前体ACC能够诱导AtNAC2表达[16],此外,AtNAC1在TIR1下游为侧根的发育转导生长素信号[17];细胞分裂,如当拟南芥NTM1的突变体中组蛋白H4的合成受到抑制时,细胞分裂就会受到抑制,植物生长就会变缓慢[18];种胚的发育,如当降低拟南芥中AtNAC102基因的表达,种子的萌发率显著降低[19],芜菁BcNAC2基因在嫩角果和胚珠中的表达量较高,这与种子或胚的形成有关[20];果实的成熟与衰老,如番茄SiNAC3基因通过延迟果实软化和改变类胡萝卜素的合成来调控果实采摘后的成熟[21],MaNACs通过与乙烯信号元件的相互作用来正调控香蕉果实采后的成熟[22],拟南芥AtNAP基因促进植物衰老[23]。
中间锦鸡儿(Caraganaintermedia)主要分布于干旱半干旱的荒漠地区,是一种多年生豆科灌木[24],具有良好的防风固沙和保持水土功能[25],同时也是优质的饲料资源,具有较高的饲用价值[26]。本研究从本实验室建立的中间锦鸡儿干旱转录组数据库中获得一条CiNAC1基因序列,进一步研究克隆得到该基因的cDNA全长,初步研究发现CiNAC1基因受干旱、脱水、高盐、高pH的诱导。
1 材料和方法
1.1材料
本实验所用中间锦鸡儿种子采自于内蒙古四子王旗;拟南芥野生型(Col-0)由本实验保存;克隆载体pEASY-Blunt simple购自TransGen;表达载体pCanG-HA由中国科学院遗传与发育研究所谢旗研究员惠赠;表达载体pCAMBIA1302、大肠杆菌感受态、农杆菌GV3101均由本实验室提供;T4DNA 连接酶和限制性内切酶等购自Thermo公司。
1.2方法
1.2.1材料处理选取颗粒饱满的中间锦鸡儿种子种植于蛭石和营养土(2∶1)中,在23 ℃长日照的温室中培养(16 h光照/8 h黑暗),30 d后选取长势基本一致的中间锦鸡儿幼苗进行处理,检测不同非生物胁迫下CiNAC1基因的表达量。
(1) NaCl处理:将正常生长30 d的中间锦鸡儿幼苗的根部全部浸泡在200 mL浓度为300 mmol·L-1NaCl的液体MS培养基中,分别浸泡0、1、3、6、12、24和48 h后取样。
(2) 高pH处理:将正常生长30 d的中间锦鸡儿幼苗的根部全部浸泡在200 mL浓度为200 mmol·L-1NaHCO3的液体MS培养基中,分别浸泡0、1、3、6、12、24和48 h后取样。
(3) 脱水处理:将正常生长30 d的中间锦鸡儿幼苗小心从土壤中取出,洗净根部以后,置于干净的滤纸上,分别在0、1、3、6、12、24和48 h后取样。
(4) 干旱处理:正常生长29 d的中间锦鸡儿最后一次浇水24 h后,即苗龄30 d时为对照,之后停止浇水,每4 d取1次样,干旱16 d后复水,复水48 h后再取样,取样时间分别为0、4、8、12、16 和18 d。
1.2.2中间锦鸡儿总RNA的提取与反转录采取生长30 d的中间锦鸡儿幼苗,使用Trizol试剂进行总RNA提取,用超微量紫外分光光度计和琼脂糖凝胶电泳来检测提取的RNA质量,选取质量较好的RNA用M-MLV 反转录酶合成cDNA第一链。
1.2.3中间锦鸡儿CiNAC1基因cDNA全长的克隆根据本实验室干旱转录组数据库中NAC1序列设计扩增CiNAC1基因cDNA全长引物PCiNAC1-F(5′-GCGTCGACATGAGCAACATAAGCATGGTAG-3′)和PCiNAC1-R(5′-GCGAGCTCAAGAAAGCTTAAAGAGGAGTGAA-3′),以反转录中间锦鸡儿cDNA为模板,利用高保真酶Primer STAR(TaKaRa公司)进行PCR扩增反应。反应程序为98 ℃预变性3 min,98 ℃变性30 s,55 ℃退火10 s,72 ℃延伸1.5 min,72 ℃补充延伸5 min,30个循环。
1.2.4不同非生物胁迫处理下CiNAC1基因表达分析中间锦鸡儿幼苗分别用NaCl、高pH、脱水、干旱处理后,提取总RNA,反转录合成cDNA第一链,用实时定量荧光PCR(qRT-PCR)进行基因表达分析。根据CiNAC1 cDNA全长序列设计CiNAC1荧光定量表达引物qCiNAC1-F(5′-TCAAAACCAAACCAACCCAATC-3′)和qCiNAC1-R(5′-TCGGTGCTCCTCCAAATGTAG-3′),qRT-PCR采用SYBR GreenⅠ荧光染料法检测,反应体系为10 μL SYBR Primix Ex TaqTM,引物各0.4 μL(10 μmol·L-1),稀释的cDNA模板5 μL,DEPC水4.2 μL,总体系20 μL。PCR反应程序为95 ℃预变性1 min,95 ℃变性5 s,60 ℃退火30 s,72 ℃延伸10 s,95 ℃补充延伸1 min,40个循环。
中间锦鸡儿的内参引物为qCiEF1α-F(5′-CAAAAAGTCCCCTCGTTGTCTC-3′)和qCiEF1α-R(5′-AGCAATCGTTCTTCCTAATGATCTAA-3′),拟南芥内参引物为qAtEF1αF(5′-AGAAGGGTGCCAAATGATGAG-3′)和qAtEF1αR(5′-GGAGGGAGAGAGAAAGTCACAGA-3′),实验结果用2-ΔΔCt法计算。
1.2.5中间锦鸡儿CiNAC1基因的生物信息学分析利用DNAMAN 软件进行氨基酸序列多重比对;利用ExPasy在线工具ProParam和 ProtScale 分析蛋白的各种氨基酸含量,理论分子量和等电点等理化参数;NCBI CDD数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)预测蛋白的功能结构域;NetPhos(http://www.cbs.dtu.dk/services/NetPhos/)和NetGlycate(http://www.cbs.dtu.dk/services/NetGly-cate/)预测蛋白的磷酸化位点和糖基化位点;SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_opma.html)预测蛋白的二级结构;MEGA6软件进行系统进化树分析。
1.2.6构建表达载体pCanG-CiNAC1和pCAMBIA1302-CiNAC1用SacⅠ和SalⅠ限制性内切酶对CiNAC1和pCanG-HA进行双酶切,从凝胶中回收目的片段,使用T4DNA 连接酶连接,构建pCanG-CiNAC1过表达载体;用BglⅡ和SpelⅠ限制性内切酶对CiNAC1和pCAMBIA1302-GFP进行双酶切,回收目的片段,使用T4DNA 连接酶连接,构建pCAMBIA1302-CiNAC1过表达载体。将表达载体转化到农杆菌GV3101感受态,用于拟南芥转化实验。
1.2.7拟南芥遗传转化将转化有表达载体pCanG-CiNAC1的农杆菌浸染野生型拟南芥,收取种子,种植在含有25 mg·L-1卡那霉素的1/2 MS培养基上,根据孟德尔遗传定律,筛选T3代纯合体。
1.2.8CiNAC1在原生质体中的亚细胞定位选取在23 ℃、16 h光照/8 h黑暗的温室中生长3~4周龄的拟南芥幼苗叶片,用PEG介导法制备拟南芥叶肉原生质体。提取pCAMBIA1302-CiNAC1-GFP和空载体pCAMBIA1302-35S-GFP质粒各约30 μg,然后将其分别转化到拟南芥叶肉原生质体中,在六孔板中室温孵育16 h,用荧光显微镜观察GFP荧光信号。
1.2.9转CiNAC1基因拟南芥侧根分析野生型和过表达拟南芥种子(OE-5、OE-42和OE-60)经过75%乙醇灭菌10 min,用含有0.05% Tween-20的100%乙醇灭菌10 min ,待种子晾干后种在1/2 MS培养基上,4 ℃同步化处理3 d,将平板取出置于23 ℃,16 h光照/8 h黑暗条件下培养36~40 h,选取长势一致的50棵幼苗,并移到新的1/2 MS培养基上竖直培养12 d,统计侧根数,并测量根长。
2 结果与分析
2.1中间锦鸡儿 CiNAC1基因的克隆与分析
从干旱转录组得到CiNAC1基因的一段序列,通过NCBI Blast进行比对,发现具有完整的开放阅读框(ORF)。根据这段已知序列设计PCiNAC1-F和PCiNAC1-R引物,以中间锦鸡儿cDNA为模板,利用高保真酶Prime STAR进行PCR扩增,得到CiNAC1基因cDNA全长序列(图1)。CiNAC1基因cDNA序列全长1 066 bp,其中开放阅读框(ORF)921 bp,可编码306个氨基酸(图2)。
2.2CiNAC1蛋白与其他物种NAC类蛋白多重比对及系统进化树分析
应用DNAMAN 5.2软件将CiNAC1氨基酸序列与拟南芥ANAC019、苜蓿MtNAC1、大豆GmNAC35、野生大豆GaNAC21/22的氨基酸序列进行多重序列比对,结果(图3)表明CiNAC1蛋白与其他NAC类蛋白一样,N端的氨基酸序列高度保守,包含A~E 等5个亚结构域。

图1 CiNAC1基因cDNA扩增M. DL5000 1. CiNAC1Fig. 1 Amplification of CiNAC1 cDNA
利用MAGE6软件将中间锦鸡儿CiNAC1与苜蓿NAC1(Medicagotruncatula)、沙冬青(Ammopiptanthusmongolicus)、大豆NAC35(Glycinemax)、野生大豆NAC21/22(Glycinesoja)、湖北海棠(Malushupehensis)、可可树NAC21/22(Theobromacacao)、木薯NAC40(Manihotesculenta)、毛果杨NAC1(Populustrichocarpa)、欧洲山杨×银白杨NAC1(Populustremula×Populusalba)、茶树(Camelliasinensis)、拟南芥NAC1(Arabidopsisthaliana)进行系统进化树分析,结果(图4)表明中间锦鸡儿CiNAC1与苜蓿MtNAC1聚在一枝,它们的相似性达到89%,亲缘关系最近。
2.3中间锦鸡儿CiNAC1蛋白的理化性质分析及功能预测
2.3.1CiNAC1蛋白的理化性质分析利用ExPasy在线工具ProParam,分析CiNAC1蛋白编码306个氨基酸,相对分子质量34.57 kD,等电点pI为8.35,脂肪族指数为68.50,此蛋白是不稳定蛋白。疏水性预测分析发现整个多肽链一共有76个疏水性氨基酸和230个亲水氨基酸,表明CiNAC1蛋白是亲水蛋白。

起始密码子ATG用下划线表示,终止密码子TGA用星号和下划线表示图2 CiNAC1基因cDNA全长序列及编码氨基酸序列The ATG initiation codon is underlined; The terminal codon is underlined with asteriskFig. 2 The cDNA and the deduced amino acid sequence of CiNAC1

下划线部分表示NAC结构域的5个亚结构域A~E;黑色背景表示完全一样的氨基酸序列;灰色背景表示一致性的氨基酸序列; ANAC019. 拟南芥(NP_175697.1);CiNAC1. 中间锦鸡儿;MtNAC1. 苜蓿(XP_003595973.1);GmNAC35. 大豆(NP_001235901.1);GsNAC21/22. 野生大豆(KHN26800.1)图3 CiNAC1蛋白与其他NAC类蛋白保守结构域的比对The underlined sections are five conservative sub-domains A to E in NAC. Amino acid residues conserved in all sequences are boxed in black, while similar amino acids are boxed in gray. All abbreviation and GenBank Accession No.are shown as: ANAC019. Arabidopsis thaliana (NP_175697.1); CiNAC1. Caragana intermedia; MtNAC1. Medicago truncatula (XP_003595973.1); GmNAC35. Glycine max (NP_001235901.1); GsNAC21/22. Glycine soja (KHN26800.1)Fig. 3 Alignment of NAC domain between CiNAC1 and other NACs

括号内为氨基酸登录号;分支上的数字表示Bootstrap验证中基于1 000次重复该节点的可信度的百分比;标尺表示演化距离图4 CiNAC1与其他物种NACs的系统进化分析The accession No. was given in bracket; The numbers on the branches represent the reliability percent of bootstrap values based on 1 000 replications; Scaleplate represents the evolution distance of these plantsFig. 4 Phylogeneic analysis of CiNAC1 and NACs from other species
2.3.2CiNAC1蛋白的功能结构域预测与分析利用NCBI CDD(Converved Domain Database)数据库分析CiNAC1蛋白的保守结构域发现,在N端第11~135个氨基酸之间有一个由125个氨基酸组成的NAM保守结构域。因此,可以推断出中间锦鸡儿CiNAC1基因属于NAC家族。
利用NetPhos和NetGlycate在线软件预测CiNAC1蛋白的磷酸化位点和糖基化位点时发现,CiNAC1基因编码的共包含7个糖基化位点(10、32、48、121、124、226、269)和 26个磷酸化位点,其中26个磷酸化位点均为苏氨酸(Thr)磷酸化位点(50、82、112、161、199、236、267、268、334、341、379、395、429、453、455、466、485、559、584、670、686、736、737、791、877、890)。
2.3.3CiNAC1蛋白的二级结构的预测利用SOPMA在线软件预测CiNAC1蛋白的二级结构,发现CiNAC1蛋白的二级结构无规卷曲所占比例最多,其中145个氨基酸形成无规卷曲,所占比例为47.39%;70个氨基酸形成α-螺旋,所占比例为22.88%;66个氨基酸形成延伸链,所占比例为21.57%;25个氨基酸形成β-转角,所占比例为8.17%。
2.4CiNAC1基因在不同非生物逆境胁迫下的表达分析
为了进一步研究CiNAC1基因的功能,本实验采用qRT-PCR技术检测了中间锦鸡儿在经过NaCl、脱水、土壤干旱、高pH处理以后CiNAC1基因表达量的变化。实验结果显示(图5),脱水处理以后中间锦鸡儿CiNAC1基因表达量呈现先上升后下降的趋势,且在6 h时最高,达到6倍;NaCl处理后CiNAC1基因表达量逐渐升高,到12 h时达到未处理的6倍左右,并且一直到48 h仍保持高水平;
高pH处理后,CiNAC1基因表达量逐渐升高,且在48 h时达到最高水平,约为未处理的13倍;土壤干旱处理后CiNAC1基因表达量呈现先上升后下降的趋势,在16 d时达到最高水平,约为未处理的4倍。说明中间锦鸡儿CiNAC1基因可能参与对干旱、强碱和盐胁迫的响应。
2.5转基因拟南芥中 CiNAC1基因的表达分析
选取T3代纯合体转基因拟南芥幼苗的叶片,提取总RNA并反转录成cDNA,利用特异性引物PCiNAC1-F和PCiNAC1-R进行RT-PCR鉴定,结果如图6,A所示,以野生型拟南芥cDNA做的阴性对照没有扩增出目的条带,中间锦鸡儿cDNA做阳性对照有单一条带,在10个转基因株系中均扩增出目的条带,表明CiNAC1在各转基因株系中都有表达。同时利用实时荧光定量PCR检测CiNAC1在转基因株系中的表达水平(图6,B),选取表达量较高的OE-5、OE-42和OE-60纯合体株系用于下一步的表型检测实验。
2.6CiNAC1在原生质体中的亚细胞定位
选取生长3~4周龄拟南芥幼苗的叶片,制备拟南芥叶肉原生质体。将构建好的pCAMBIA1302-CiNAC1质粒和35S∶GFP空载体分别转化到拟南芥叶肉原生质体,培养16 h后观察GFP荧光信号。如图7所示,35S∶GFP空载体的荧光遍布整个细胞,而GFP-CiNAC1仅定位于细胞核中。
2.7CiNAC1促进侧根生长
将野生型拟南芥(WT)和过表达株系(OE-5、OE-42和OE-60)种子分别种在1/2 MS培养基上,春化3 d。在温室中正常生长40 h后,将其移到新的1/2 MS培养基上竖直培养12 d后,拍照(图8),统计侧根数,测量根长,并计算侧根数/根长的比值(表1),进行3次生物重复。结果表明,CiNAC1过表达株系的侧根数目明显高于野生型拟南芥,而且达到了极显著水平;CiNAC1过表达株系的根系也明显比野生型的长;CiNAC1过表达株系的侧根数/根长的比值也明显高于野生型,而且均达到了显著水平。这说明,过表达CiNAC1基因促进了拟南芥侧根的发生,同时也促进了主根的伸长。
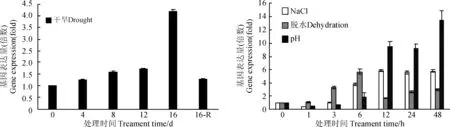
图5 qRT-PCR检测各种胁迫下 CiNAC1基因的表达Fig. 5 The expression of CiNAC1 under different stresses by qRT-PCR

A. CiNAC1转基因株系RT-PCR鉴定:M.DL2000; C-.野生型拟南芥cDNA(阴性对照);C+.中间锦鸡儿cDNA(阳性对照);其他代号为转基因株系;B.转基因株系 CiNAC1表达水平的实时定量PCR鉴定图6 CiNAC1转基因株系鉴定及表达水平鉴定A. RT-PCR identification of CiNAC1 transgenic Arabidopsis lines: C-. Arabidopsis thaliana cDNA (Negative control); C+. Caragana intermedia cDNA (Positive control); Others are transgenic lines; B. Quantitative real-time PCR analysis of CiNAC1 expression in CiNAC1 transgenic linesFig. 6 The expression level of CiNAC1 in transgenic Arabidopsis

图7 拟南芥叶肉原生质体中GFP- CiNAC1的亚细胞定位Fig. 7 The nuclear localization of GFP- CiNAC1 in Arabidopsis mesophyll protoplast

拟南芥种子在1/2 MS培养基中正常生长40 h后,转移到新的1/2 MS培养基中,竖直培养12 d(n=50)图8 过表达 CiNAC1基因促进拟南芥侧根发生The 40-hours-seedlings on 1/2 MS medium were transferred to new medium(n=50)Fig. 8 Overexpression of CiNAC1 promoted the lateral root formation in Arabidopsis

植株Plant表型特征Phenotypiccharacteristic侧根数Numbersoflateralroot/number根长Rootlength/cm侧根数/根长Lateralrootdensity(N/L)WT10.28±4.404.443±1.192.565±1.33OE-512.42±4.39*4.564±1.453.235±1.93*OE-4215.58±6.05**5.293±1.71**3.265±1.57*OE-6014.36±4.81**5.095±1.49*3.131±1.45*
注:表中所列数据为平均值±标准偏差;*表示显著性水平,*P<0.05;**P<0.01
Note: The data listed in the table as mean ±SD; Asterisks indicate a significant difference.*P<0.05;**P<0.01
3 讨 论
中间锦鸡儿经过漫长的进化过程,适应了干旱、高温、高盐碱等极端的生长环境,具有了很强的抗逆性[27]。从中间锦鸡儿中克隆参与抗逆性相关的基因,并对其进行功能研究,这对进一步从分子机制上研究中间锦鸡儿抗逆性机理提供了一定的帮助。本研究从中间锦鸡儿中克隆得到一个NAC转录因子,经过Blast比对发现,它与拟南芥NAC1基因同源性较高,因此将其命名为CiNAC1。
本研究通过观察拟南芥叶肉细胞原生质体的亚细胞定位,发现CiNAC1的表达产物定位在细胞核上,该结果与NAC转录因子的N端保守结构域中含有核定位信号的理论一致。已经有很多研究表明,NAC转录因子的定位在细胞核。如Xie等[28]发现拟南芥AtNAC1定位在细胞核;Han等[29]通过构建稳定表达融合蛋白的转基因株系,发现中间锦鸡儿GFP-CiNAC3和GFP-CiNAC4的表达产物定位于细胞核中;Zhang等[30]也通过构建稳定表达融合蛋白的转基因株系,发现胡杨GFP-PeNAC045的表达产物定位于细胞核。
本研究结果CiNAC1转基因株系侧根数目比野生型多,主根也比野生型长,侧根数/根长的比值(即侧根密度)也比野生型大。过表达CiNAC1拟南芥侧根数比野生型多的这一结果与拟南芥AtNAC1的表型一致[28],过表达CiNAC1拟南芥主根比野生型长的这一结果与过表达GmNAC004拟南芥一致[31]。在NAC转录因子家族中,有很多与生长素相关的基因。Xie等[28]发现拟南芥AtNAC1可以被生长素诱导,促进下游2个生长素相关基因DBP(DNABINDINGPROTEIN)和AIR3(AUXIN-INDUCEDINROOTCULTURES3)的表达,从而介导生长素信号,促进根尖和侧根的发育。即AtNAC1在TIR1下游传送生长素信号给AIR3从而促进侧根生长;Xie等[32]发现SINAT5促进泛素相关的NAC1降解来衰减生长素信号;Guo等[33]发现microRNA 直接切断NAC1转录因子的mRNA来下调生长素信号,从而调控拟南芥侧根的发育;Wang等[34]发现生长素和赤霉素诱导AtNAC1上游基因的表达,从而诱发侧根的形成;He等[35-36]发现拟南芥AtNAC2(ANAC092、AT5G39610)可以被生长素诱导,并且参与生长素调控的侧根形成,而且AtNAC2参与拟南芥对盐胁迫的响应,能够诱导许多衰老相关基因,参与乙烯和ABA的响应;Hao等[37]发现大豆GmNAC20基因可以促进拟南芥侧根的形成,在过表达GmNAC20拟南芥中,生长素相关的下游基因ARF2、AXR1的表达量降低,而ARF7、ARF19、LBD12、AIR1表达量降低;Quach等[31]发现无论在正常生长条件下还是在水胁迫条件下,过表达GmNAC004拟南芥与野生型相比侧根数目增加,根长较长。以上的结果表明不同物种NAC1同源性较高、功能比较保守。
[1]孙利军,李大勇,张慧娟,等. NAC转录因子在植物抗病和抗微生物胁迫反应中的作用[J]. 遗传, 2012, 34(8): 993-1 002.
SUN L J, LI D Y, ZHANG H J,etal. Functions of NAC transciption factors in biotic and abiotic stress responses in plants[J].Hereditas, 2012, 34(8): 993-1 002.
[2]陈娜,蒋晶,曹必好,等. 植物NAC转录因子功能研究新进展[J]. 分子植物育种, 2015, 13(6): 1 407-1 414.
CHEN N, JIANG J, CAO B H,etal. The latest progresses on plant NAC transcription factors function[J].MolecularPlantBreeding, 2015, 13(6): 1 407-1 414.
[3]彭辉,于兴旺,等. 植物NAC转录因子家族研究概况[J]. 植物学报, 2010, 45(2): 236-248.
PENG H, YU X W,etal. Research progress of plant transcription factors[J].ChineseBulletinofBotany. 2010, 45(2): 236-248.
[4]AIDA M, IHIDA T, FUKAKI H,etal. Genes involved in organ separation inArabidopsis: an analysis of the cup-shaped cotyledon mutant[J].PlantCell, 1997, 9(6): 841-857.
[5]SOUER E, VAN HOUWELINGEN A, KLOOS D,etal. The no apical meristem gene of petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J].Cell, 1996, 85(2): 159-170.
[6]NURUZZAMAN M, MANIMEKALAI R, SHARONI A M,etal. Genome-wide analysis of NAC transcription factor family in rice[J].Gene, 2010,465(1-2): 30-44.
[7]LE D T, NISHIYAMA R, WATANABE Y,etal. Genome-wide survey and expression analysis of the plant-specific NAC transcription factor family in soybean during development and dehydration stress[J].DNARes, 2011, 18(4): 263-276.
[8]HU R, QI G, KONG Y,etal. Comprehensive analysis of NAC domain transcription factor gene family inPopulustrichocarpa[J].BMCPlantBiology, 2010, 10: 145-178.
[9]RUSHTON P J, BOKOWIEC M T,etal. Tobacco transcription factors:novel in sights into transcriptional regulation in the Solanaceae[J].PlantPhysiol., 2008, 147: 280-295.
[10]邢国芳,王雁明,张魏斌,等. 植物NAC转录因子研究进展[J]. 山西农业科学, 2012, 40(4): 409-411.
XING G F, WANG Y M, ZHANG W B,etal. Research progress of NAC transcription factors in plant[J].JournalofShanxiAgriculturalSciences, 2012, 40(4): 409-411.
[12]李伟,韩蕾,等. 植物NAC转录因子的种类、特征和功能[J]. 应用与环境生物学报, 2011, 17(4): 596-606.
LI W, HAN L,etal. Characteristics and functions of NAC transcription factors in plants[J].ChineseJournalofAppliedandEnvironmentBiology, 2011, 17(4): 596-606.
[13]OLSEN A N, EMST H A, LEGGIO L L,etal. NAC Transcription factors: structurally distinct, functionally diverse[J].TrendsinPlantScience, 2005, 10(2): 79-87.
[14]KO J H, YANG S H, PARK A H,etal. ANAC012,a member of the plant-specific NAC transcription factor family, negatively regulates xylary fiber development inArabidopsisthaliana[J].PlantJournal, 2007, 50: 1 035-1 048.
[15]AIDA M, ISHIDA T, FUKAKI H,etal. Genes involved in organ separation inArabidopsis: an analysis of thecup-shapedcotyledonmutant[J].PlantCell, 1997, 9: 841-857.
[16]HE X J, MU R L, CAO W H,etal.AtNAC2, a transcription factor downstream of rthylene and auxin signaling pathways,is involved in salt stress response an lateral root development[J].PlantJournal, 2005, 44(6): 903-916.
[17]王瑞芳,胡银松,高文蕊,等. 植物NAC转录因子家族在抗逆响应中的功能[J]. 植物生理学报, 2014, 50(4): 1 494-1 500.
WANG R F, HU Y S, GAO W R,etal. Functions of NAC transcription factors family in stress responses in plant[J].PlantPhysiologyJournal, 2014, 50(4): 1 494-1 500.
[18]KIM S Y, KIM S G, KIM Y S,etal. Exploring memberance-associated NAC transcription factors inArabidopsis:Implications for membrance biology in genome regulation[J].NucleicAcidsResearch, 2006, 35(1): 203-213.
[19]CHRISTIANSON J A, WILSON I W, LLEWELLYN D J,etal. The low-oxygen induced NAC domain transcription factorANAC012 affects viabilityArabidopsisseeds following low-oxygen treatment[J].PlantPhysiology, 2009, 149(4): 1 724-1 738.
[20]张海娟,吴剑锋,胡帅,等. 芜菁NAC转录因子BcNAC2基因的分离及其表达[J]. 园艺学报, 2011, 38(8): 1 089-1 096.
ZHANG H J, WU J F, HU S,etal. Isolation and expression analysis ofBcNAC2, a NAC transcription factor gene in Turnip[J].ActaHorticulturaeSinica, 2011, 38(8): 1 089-1 096.
[21]KOU X H, WANG S,etal. Molecular characterization and expression analysis of NAC family transcription factors in tomato[J].PlantMolecularBiologyReporter, 2014, 32: 501-516.
[22]SHAN W, KUANG J F,etal. Molecular characterization of banana NAC transcription factors and their interactions with ethylene sigalling component EIL during fruit ripening[J].JournalofExperimentalBotany, 2012, 63(14): 5 171-5 187.
[23]KOU X H, WATKINS C B, GAN S S,etal.ArabidopsisAtNAPregulates fruit senescence[J].JournalofExperimentalBotany, 2012, 63(17): 6 139-6 147.
[24]韩晓敏. 中间锦鸡儿非生物胁迫相关转录因子的克隆与功能分析[D]. 呼和浩特:内蒙古农业大学, 2015.
[25]王峰,左忠,张浩,等. 柠条饲料加工相关问题的探讨[J]. 草业科学, 2015, 22(6): 75-80.
WANG F, ZUO Z, ZHANG H,etal. Study onCaraganafeed process and its related problem[J].PratacultuRal.Science, 2015, 22(6): 75-80.
[26]刘国谦,张俊宝,刘东庆,等. 柠条的开发利用及草粉加工饲喂技术[J]. 草业科学, 2003, 20(7): 26-30.
LIU G Q, ZHANG J B, LIU D Q,etal.Caraganaexploitation and the processing of meal feeding technique[J].PratacultuRalScience, 2003, 20(7): 26-30.
[27]杨杞,尹佳佳,王颖,等. 柠条锦鸡儿CKLEA1基因克隆及表达分析[J]. 中国生物工程杂志, 2013, 33(5): 93-99.
YANG Q,YIN J J, WANG Y,etal. Clone and expression analysis ofCkLEA4 fromCaraganakorshinskii[J].ChinaBiotechnology, 2013, 33(5): 93-99.
[28]QI X, FRUGIS G,etal.ArabidopsisNAC1 transcription auxin signal downstream of TIR1 to promote lateral root development[J].GenesandDevelopment, 2000. 14(23): 3 024-3 036.
[29]HAN X M, FENG Z Q, XING D D,etal. Two NAC transcription factors fromCaraganaintermediaaltered salt tolerance of the transgenicArabidopsis[J].BMCPlantBiology, 2015, 15, 208-219.
[30]张晓菲,路信,等. 胡杨NAC转录因子PeNAC045基因的克隆及功能分析[J]. 北京林业大学学报, 2015, 37(6): 1-10.
ZHANG X F, LU X,etal. Cloning and functional analysis ofPeNAC045 fromPopuluseuphratica[J].JournalofBeijingFoerstryUniversity, 2015, 37(6): 1-10.
[31]QUACH T N, TRAN L S, VALLIYODAN B,etal. Functional analysis of water stress responsive soybeanGmNAC003 andGmNAC004 transcription factors in lateral root development inArabidopsis[J].PLoSOne, 2014, 9(1): 1-12.
[32]QI X, GUO H S, DALLMAN G,etal. SINAT5 promotes ubiquitin-related degradation ofNAC1 to attenuate auxin signals[J].Nature, 2002, 419: 167-170.
[33]GUO H S, QI X, FEI J F,etal. MicroRNA directs mRNA cleavage of the transcriptions factorNAC1 to downregulate auxin signals forArabidopsislateral root development[J].ThePlantCell, 2005, 17: 1 376-1 386.
[34]WANG Y H, DUAN L S, LU M Z,etal. Expression ofNAC1 up-stream regulatory region and its relationship to the lateral root initiation induced by gibberellins and auxins[J].ScienceinChina, 2006, 49(5): 429-435.
[35]HE X J, MU R L, CAO W H,etal.AtNAC2, a transcription factor downstream of ethylene and auxin signaling pathways, is involved in salt stress response and lateral root development[J].PlantJournal, 2005, 44(6): 903-916.
[36]BALAZADEH S, SIDDIQUI H, ALLU A D,etal. A gene regulatory network controlled by the NAC transcription factorANAC092/AtNAC2/ORE1 during salt-promoted senescence[J].PlantJournal, 2010, 62(2): 250-264.
[37]HAO Y J, WEI W,etal. Soybean NAC transcription factors promote abiotic stress tolerance and lateral root formation in transgenic plants[J].PlantJournal, 2011, 68(2): 302-313.
(编辑:宋亚珍)
Cloning and Functional Analysis ofCiNAC1 fromCaraganaintermedia
YUE Junyan, YUE Wenran, YANG Qi, YU Xiumin, YANG Feiyun,WANG Guangxia, LI Guojing, WANG Ruigang, CONG Jingyu*
(Inner Mongolia Agricultural University, Hohhot 010018, China)
NACs are one of the largest plant-specific transcription factor families. Members in this family play important roles in growth and abiotic stress responses. In this study, the full-length cDNA sequence ofCiNAC1 fromCaraganaintermediawas cloned by PCR technique. It was 1 066 bp in length. Bioinformatics analysis showed thatCiNAC1 had an open reading frame of 921 bp, encoding 306 amino acids. The calculated molecular weight of CiNAC1 was 34.57 kD and the isoionic point was 8.35. The CiNAC1 protein was a hydrophilic protein which had a conserved NAM domain in the N-terminus. It contained 26 phosphorylation sites and 7 glycosylation sites. Real-time quantitative PCR analysis showed thatCiNAC1 was induced by drought, NaCl, dehydration, and high pH. Localization assays revealed that CiNAC1 localized in the nuclei, consistent with its role as transcription factor. Overexpression ofCiNAC1 promoted the lateral root formation and length of the primary root. These results indicated thatCiNAC1 might be related to response to environmental stress inC.intermedia.
Caraganaintermedia;NAC transcription factors;abiotic stress; functional analysis
1000-4025(2016)07-1285-09
10.7606/j.issn.1000-4025.2016.07.1285
2016-03-31;修改稿收到日期:2016-05-23
内蒙古自治区高等学校科学研究项目(NJZY055);内蒙古自治区科技创新团队(201503004);内蒙古自治区产业创新创业人才团队(草原英才工程)
岳俊燕(1990-),女,在读硕士研究生,主要从事植物分子生物学研究。E-mail:Yuejunyan13@163.com
丛靖宇,博士,副教授,硕士研究生导师,主要从事植物分子生物学研究。E-mail:congjingyuwyh@163.com
Q785;Q786
A
[11]OOKA H, SATON K, K,etal. Comprehensive analysis of NAC family genes inOryzasativaandArabidopsisthaliana[J].DNARes., 2003, 10: 239-247.

