基于液相色谱-串联质谱技术的磷酸化蛋白质组学分析
朱秀娟, 李好雨, 郭 林, 赵晓璐
(病毒学国家重点实验室,武汉大学生命科学学院,湖北武汉 430072)
蛋白质磷酸化修饰在生物体内普遍存在。真核生物中,磷酸化修饰主要发生在丝氨酸(Ser)、苏氨酸(Thr)和酪氨酸(Tyr)残基上[1]。磷酸化修饰参与调控多种细胞生命活动,如细胞增殖、发育与分化、受体介导的信号转导、细胞凋亡及肿瘤发生等。其中,磷酸化修饰对抗病毒天然免疫信号通路的调控一直是生物医学领域的一个研究热点。天然免疫反应是机体抵御外界病原微生物入侵的第一道防线。当病毒入侵时,机体通过自身的模式识别受体(Pattern Recognition Receptors,PRRs)来识别病毒的病原相关分子模式(Pathogen-associated Molecular Patterns,PAMPs),并通过一系列的信号转导事件诱导干扰素(Interferon ,IFN)表达,进而抵御病毒入侵[2]。在抗病毒天然免疫信号通路活化的过程中,磷酸化修饰起着重要的调控作用[3]。因此,磷酸化蛋白及位点的鉴定对于更好地了解抗病毒天然免疫信号通路是十分必要的。
蛋白质组学主要研究特定生理或病理条件下细胞或组织中所有蛋白的动态变化。超高静电场轨道阱傅里叶变换台式质谱(Q Exactive HF )结合了提供母离子选择的高性能四极杆和具有超高分辨率、高质量精度的超高静电场轨道阱。在数据质量无任何损失的情况下,Q Exactive HF 以更快的检测速度和更高的分辨率,在蛋白质组学领域有绝佳的表现[4]。蛋白质组学技术的飞速发展,使高通量对磷酸化蛋白进行相对定量分析成为可能。其中,稳定同位素双甲基化标记技术因操作简单而被广泛采用[5]。细胞内约30%的蛋白质能发生磷酸化修饰,但在特定生理及病理条件下,磷酸化蛋白只占总量的1%~2%[6]。夹杂在高丰度的非磷酸化肽段中,低丰度的磷酸化肽段很难被质谱检测到。因此,在样品制备过程中要对磷酸化肽段进行富集。目前比较常用的磷酸化富集方法有抗体富集、固定相金属离子螯合层析(IMAC)富集[7]以及TiO2富集(titanium dioxide)[8]。IMAC富集之后,再辅之于色谱分离技术,如强阳离子交换(SCX)、亲水相互作用色谱(HILIC)等,可以鉴定更多的磷酸化肽段[9]。
本研究运用稳定同位素双甲基化标记技术、IMAC磷酸化肽段富集技术,并结合液相色谱-串联质谱研究了病毒感染引起的宿主细胞磷酸化蛋白质组的变化。结合生物信息学分析软件的使用,对受病毒感染调控的生物学过程和信号通路进行了分析。
1 实验部分
1.1 仪器与材料
nanoHPLC-Q Exactive HF液相色谱-串联质谱仪(美国,Thermo 公司);Agilent 1200 HPLC分离系统(美国,Agilent公司)。
人胚肾细胞293T来自武汉大学典型培养物保藏中心;日本仙台病毒(Sendai Virus,SeV)来自武汉大学生命科学学院舒红兵实验室;抗p-IRF3(磷酸化IRF3)抗体购自CST公司。
1.2 实验方法
1.2.1色谱/质谱条件色谱条件:色谱柱为Easy-nano LC1000 C18柱(50 cm×75 μm×2 μm,Thermo 公司);流动相A:0.1%甲酸溶液,流动相B:0.1%甲酸的乙腈溶液;梯度淋洗程序:0~210 min,5%~28%B;210~220 min,28%~80%B;220~240 min,80%B;流速:0.3 μL/min;进样量:10 μL。质谱条件:Easy-spray 离子源;喷雾电压为2.1 kV;毛细管温度为275 ℃;S-Lens RF Level为60;Data-dependent HCD采集母离子和子离子;一级质谱(MS)扫描完成后,选取信号强度最高的20个离子(+2价到+7价)进行MS/MS分析。MS和MS/MS分析的质谱参数参见表1。

表1 质谱参数
SCX色谱分离条件:PolySULFOETHYL Aspartamide色谱柱(50×2.1 mm,5 μm);流动相A:5 mmol/L磷酸二氢钾,20%乙腈的水溶液(pH=2.69);流动相B:5 mmol/L磷酸二氢钾,500 mmol/L氯化钾,20%乙腈的水溶液(pH=2.69);梯度淋洗程序:0~20 min,0~20%A;20~35 min,20%~60%A;35~40 min,100%A;流速:200 μL/min;进样量:100 μL。
1.2.2病毒感染宿主细胞病毒感染细胞实验参照文献方法[10]。
1.2.3蛋白质提取、酶解、肽段双甲基化标记以及SCX分离用SeV分别感染宿主细胞0 h、2 h和4 h,收集细胞样品,加入500 μL裂解液(6 mol/L 尿素,2 mol/L硫脲,蛋白酶抑制剂),对其超声破碎(功率200 W,间歇3 s,工作5 s,全程时间为2 min)。4 ℃、11 400 r/min离心20 min,取上清,考马斯亮蓝法对蛋白进行定量。所用蛋白还原和烷基化、蛋白胰蛋白酶酶解、稳定同位素双甲基化标记参考文献方法[11]。等比例混合双甲基化标记样品,其中一部分样品用IMAC富集技术对磷酸化肽段进行富集(参照文献方法[9]),另一部分样品用SCX进行色谱分离成10个组分。
2 结果与讨论
2.1 病毒感染宿主细胞及蛋白组学分析流程
选择IRF3为抗病毒天然免疫信号通路激活的判定标准,第396位Ser磷酸化的IRF3能入核诱导IFN表达。如图1-A所示,Sev感染宿主细胞4 h后,蛋白免疫印迹(Western blot)检测到IRF3第 396位Ser磷酸化,表明抗病毒天然免疫信号通路被激活。
蛋白质组学实验采用SeV分别感染宿主细胞0 h、2 h(IRF3激活前)和4 h(IRF3激活后)。在每组实验中,都用Western blot确保Sev成功感染了宿主细胞,代表性Western blot检测结果如图1-B所示。后续蛋白质组学分析流程如图1-C所示,我们不仅研究了病毒感染引起的宿主细胞磷酸化组变化,还研究了全蛋白质组变化,目的是排除蛋白表达水平变化对蛋白磷酸化水平变化的影响。
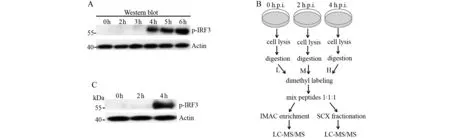
图1 (A)不同感染时间条件下Western blot检测IRF3;(B)Western blot检测IRF3以验证蛋白质组学分析细胞样品已被病毒激活;(C)基于稳定同位素双甲基化标记的定量蛋白质组学分析流程Fig.1 (A)Western blot analysis of IRF3 phosphorylation at different infection times;(B)Phosphorylation level of IRF3 detected by Western blot to validate cell activation by virus in proteomic analysis sample;(C)Workflow of quantitative proteomics based on stable isotope dimethylated labeling
2.2 磷酸化蛋白质组学数据概况分析
对于磷酸化蛋白质组学研究进行了两次生物学重复实验。重复1中,IMAC磷酸化肽段富集效率为90%(4 922个磷酸化肽段/5 496个总肽段)。重复2中,IMAC磷酸化肽段富集效率为80%(4 725个磷酸化肽段/5 904个总肽段)。IMAC磷酸化肽段富集技术的使用大大提高了磷酸化肽段的鉴定数目。被双基化标记的磷酸化肽段在MS中就可以进行定量,准确度高于MS/MS的定量。
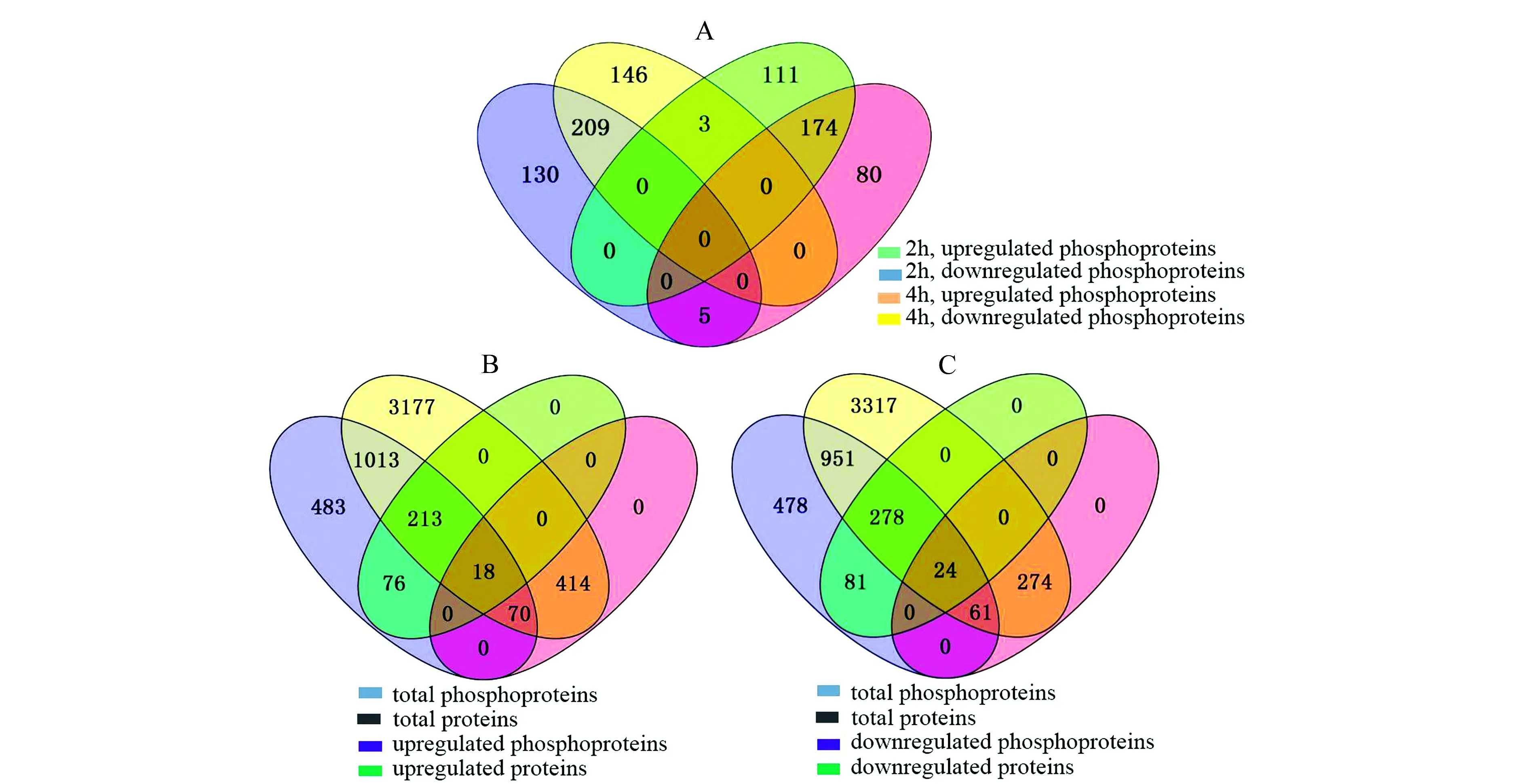
图2 (A)不同感染时间条件下检测到的显著变化磷酸化肽段数量比较分析;(B)磷酸化组和全蛋白质组两组数据中显著上调蛋白数量的比较分析;(C)磷酸化组和全蛋白质组两组数据中显著下调蛋白的数量比较分析Fig.2 (A)Comparisons of number of significantly-changed phosphopeptides at different infection times;(B)Comparisons of number of significantly-upregulated proteins in phosphoproteme and the whole proteome;(C)Comparisons of number of significantly-downregulated proteins in phosphoproteme and the whole proteome
经过对磷酸化肽段的去冗余和可信度鉴定等一系列分析,共定量到4 289个高度可信的磷酸化肽段。将这些双甲基化标记的磷酸化肽段的比值数据进行频率分布,并拟合到正态分布曲线,这里选取95%置信区间外作为显著变化的部分,即中值±1.96倍标准差(mean±1.96×SD)作为上调和下调的界限值(即cut-off值)[12]。根据计算,2 h/0 h中肽段上下调变化的cut-off值为1.60和0.55,4 h/0 h中肽段上下调变化的cut-off值为1.76和0.50。如图2-A所示,病毒感染宿主细胞2 h和4 h后,共有373个磷酸化肽段发生上调,493个磷酸化肽段发生下调。
在分析磷酸化肽段的同时,也将定量到的4 905个全蛋白(没有经过磷酸化肽段富集)的定量比值数据进行频率分布,并拟合到正态分布曲线,依然选取95%置信区间外作为显著变化的部分。根据计算,2 h/0 h 中蛋白显著变化的cut-off值分别为1.27和0.77,4 h/0 h中蛋白显著变化的cut-off值分别为1.40和0.78。病毒感染宿主细胞后,共有502个蛋白发生上调,359个蛋白发生下调。
为了进一步分析蛋白表达水平变化对磷酸化水平变化的影响,实验将磷酸化组数据和全蛋白质组数据进行了比较分析。如图2-B和2-C所示,有1 314个磷酸化蛋白在全蛋白中也鉴定到,但两组样品中共存的上调蛋白只有18个,下调的蛋白只有24个。因此,可以认为病毒感染宿主细胞后,蛋白磷酸化水平的变化基本不是由蛋白表达水平的变化引起的。
2.3 受病毒调控的天然免疫信号通路分析
SeV感染宿主细胞后,其主要的受体是RIG-I受体,但其核酸同样可以被NOD样受体和Toll样受体所识别。为了进一步探讨磷酸化修饰对抗病毒天然免疫信号通路的调控,用KEGG网站对定量的所有磷酸化蛋白进行了分类,并把参与早期抗病毒天然免疫信号通路的磷酸化蛋白都挑选出来。图3-A为RIG-I受体通路,图3-B为NOD样受体通路,图3-C为Toll样受体通路。

图3 (A)RIG-I受体信号通路;(B)NOD样受体信号通路;(C)Toll样受体信号通路Fig.3 (A)RIG-I receptor pathway;(B)NOD like receptor pathway;(C)Toll-like receptor pathway
病毒感染诱导IFN表达是宿主细胞抗病毒免疫机制的重要组成部分。在上述7个磷酸化蛋白中,对于诱导IFN产生有直接影响的是去泛素化酶DUBA。已有研究表明,去泛素化酶DUBA可以抑制RIG-I受体介导的IFN产生[13]。此外,去泛素化酶DUBA的酶活依赖自身第177位Ser的磷酸化修饰[14]。本文研究结果表明,SeV感染宿主细胞2 h后,DUBA第64位Ser磷酸化水平下调,但其表达水平无显著变化。因此,推测DUBA可能通过第64位Ser磷酸化参与调控抗病毒天然免疫信号通路。
2.4 受病毒调控的生物学过程和通路分析
我们将显著变化的磷酸化蛋白提交到PANTHER软件,进行了基于生物学过程的基因聚类(Gene Ontology,GO)分析。如图4-A所示,显著变化的磷酸化蛋白主要与代谢过程、细胞过程和发育过程等相关。使用DAVID软件进行了基于KEGG的通路富集(Pathway Enrichment)分析。如图4-B所示,当上调磷酸化蛋白与总磷酸化蛋白比较时,发现mTOR 通路发生了显著上调。此外,Toll样受体通路和NOD样受体通路等通路也发生上调。当显著下调的磷酸化蛋白和总的磷酸化蛋白比较时,发现Spliceosome通路发生了显著下调。我们进一步探讨了mTOR和spliceosome 通路在病毒感染中的作用。
通过基于KEGG网站的通路蛋白分析、STRING软件的蛋白相互作用分析和cytoscape软件分析,我们得到mTOR通路中定量蛋白的相互作用网络图。如图4-C所示,mTOR 通路中共定量到16个磷酸化蛋白,之间有87个相互作用连线。其中,有11个蛋白的磷酸化水平都发生显著变化。已有研究表明,mTOR信号通路通过调控IRF7的翻译和磷酸化修饰来参与抗病毒天然免疫信号通路[15,16]。因此,推测mTOR通路可能调控其他抗病毒蛋白的翻译。
Spliceosome通路中定量蛋白的相互作用网络如图4-D所示,定量到的34个磷酸化蛋白共有146个相互作用连线。研究表明,剪接体通路可以间接影响抗病毒天然免疫信号通路,如抗病毒蛋白的异构体会抑制此蛋白介导的抗病毒免疫信号通路[17,18]。因此,推测病毒感染引起Spliceosome 通路下调是为了抑制抗病毒蛋白异构体的产生。
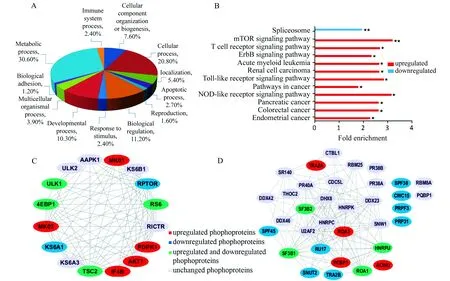
图4 (A)显著变化磷酸化蛋白基于生物学过程的GO分析;(B)信号通路富集分析;(C)mTOR通路中蛋白相互作用分析;(D)Spliceosome通路中蛋白相互作用分析Fig.4 (A)GO biological process analysis;(B)pathway enrichment analysis;(C)Protein network of mTOR pathway;(D)Protein network of spliceosome pathway
2.5 磷酸化位点基序分析
在定量到的4 289个磷酸化肽段中,有3 656个p-Ser位点,712个p-Thr位点,27个p-Tyr位点。蛋白激酶对蛋白的催化需要一定的氨基酸基序,可以通过对磷酸化基序的分析来寻找潜在的蛋白激酶,因此,对于磷酸化位点基序分析具有重要意义。将显著变化的磷酸化肽段递交到Motif-X运算程序中,对其进行磷酸化基序分析。如图5所示,有7个针对磷酸化丝氨酸(p-Ser)和2个针对磷酸化苏氨酸(p-Thr)的磷酸化基序,因为Tyr磷酸化肽段所占数目太少,很遗憾没有找到针对磷酸化酪氨酸(p-Tyr)的磷酸化基序。
为了预测提取的磷酸化基序对应的激酶,我们查阅了部分已知其激酶的磷酸化蛋白。图5-A所示基序可以被激酶MAPK、CDC2、CDK、ERK1等催化,图5-C和图5-D所示基序可以被激酶AKT催化,图5-H所示基序可以被激酶GSK-3、CDK5和ERK1所催化。这些激酶对于磷酸化蛋白进一步的功能分析具有重要意义。

图5 显著变化磷酸化肽段的磷酸化基序分析Fig.5 Phosphorylation site motif analysis of significantly regulated phosphorylation sitesA-B:p-Ser proline motif.C-F:p-Ser basic amino acid motif.G:p-Ser acidic amino acid motif;H-I:p-Thr proline motif.
3 结论
本研究运用稳定同位素双甲基化标记技术,并结合IMAC富集技术以及液相色谱-串联质谱法,研究了Sev感染引起的宿主细胞磷酸化蛋白质组的变化。借助多种生物信息学软件的分析,我们发现mTOR和spliceosome通路可能参与宿主细胞对病毒感染的免疫应答。此外,病毒感染引起一个已知具有抗病毒作用的去泛素化酶DUBA第64位Ser磷酸化下调,我们认为其第64位Ser在调控抗病毒天然免疫信号通路中的作用值得用生物学实验加以验证。液相色谱-串联质谱技术和生物信息学分析软件的结合,使我们的研究对于深入解析宿主细胞对病毒应答的分子机制提供了新线索。

