马身猪ZNF280D基因新型剪接体的鉴定及生物信息学分析
靳玉舒,李萌,张旗,高鹏飞,李步高,郭晓红
(山西农业大学 动物科技学院,山西 太谷 030801)
马身猪ZNF280D基因新型剪接体的鉴定及生物信息学分析
靳玉舒,李萌,张旗,高鹏飞,李步高,郭晓红*
(山西农业大学 动物科技学院,山西 太谷 030801)
[目的]本试验旨在探究马身猪中锌指蛋白(zinc finger protein)ZNF280D基因的剪接体类型。[方法]在猪转录组测序对ZNF280D基因剪切位置预测的基础上,采用RT-PCR和克隆测序技术对预测的剪切位点进行验证,检测该基因不同剪接体的结构并进行生物信息学分析。[结果]共检测到2个剪接体,分别是ZNF280D1和ZNF280D2,ZNF280D1为该基因的常规转录本,ZNF280D2第8外显子5’端大部分缺失。ZNF280D1含有3个C2H2型锌指结构域,属tC2H2型锌指蛋白,ZNF280D2含有4个C2H2型锌指结构域,属maC2H2型锌指蛋白。[结论]ZNF280D2与DNA的亲和力大于ZNF280D1。
猪;ZNF280D基因;剪接体
Abstract:[Objective]In this study,the splice variant of the gene of zinc finger protein 280D (ZNF280D) in Mashen pigs was investigated.[Methods]Based on the prediction of theZNF280Dgene splicing position in the pig transcriptome sequence, these splice sites were verified by RT-PCR amplification, cloning and sequencing technique.[Results]Two alternative splicing isoforms of theZNF280Dgene calledZNF280D1 andZNF280D2 were discoveried in Mashen pigs. Compared withZNF280D1, the exon 8 inZNF280D2 was missed. Three C2H2zinc finger domain structures were found in ZNF280D1 that was a tC2H2type zinc finger protein. Four C2H2zinc finger domain structures were found inZNF280D2 that was a maC2H2type zinc finger protein.[Conclusion]The greater affinity for DNA was detected inZNF280D2.
Keywords:Susscrofa, ZNF 280D gene, Alternative splicing isoforms
锌指蛋白(zinc finger protein)又名SUWH4(Suppressor of Hairy Wing Homolog 4)、 Zinc Finger Protein 634或 KIAA1584[1],是一类通过特定的保守结构域结合Zn2+折叠形成类似“手指”样结构的蛋白。根据氨基酸与Zn2+结合的空间结构不同,锌指蛋白可分成C2H2、C4和C63种主要的类型[2]。C2H2型锌指蛋白可能是哺乳动物最大的一类转录因子家族,在细胞和生物体的生长、分泌等众多生物过程中都起着重要的作用。例如,ZNF18在小鼠胚胎的心脏高表达,在成体中低表达,提示ZNF18可能与心脏发育密切相关[3]。克隆的小鼠基因组中的新基因Zfp474在睾丸中呈现高丰度表达,暗示其可能参与精子的发生[4]。另外通过体外人工构建,还发现了某些特定锌指结构如VEGF-A的转录可以促进血管生长进而加速伤口愈合[5]。
锌指蛋白家族含有大量的各类型剪接体,不同剪接体的出现增加了基因的多样性和复杂性。转录本的差异性剪接可以直接或间接的影响蛋白质的功能,也可视为蛋白的进化,这也使得研究锌指蛋白及其剪接体的种类及功能在生物体的发育成熟、免疫抗病、适应环境甚至进化等方面都变得尤为重要。
锌指蛋白280D(ZNF280D)基因是C2H2型锌指蛋白家族的成员之一,广泛存在于在自然界的多种生物中,不同物种中该基因的位置、结构和剪接体种类都有所不同。人的ZNF280D基因位于15号染色体,长288.396 kb,编码的蛋白质由979个氨基酸构成,含有5个高度保守的C2H2型锌指结构域(http://www.uniprot.org/uniprot/Q6N043)和一个DUF(domain of unknown function)4195结构域,这些锌指结构通过结合DNA发挥其转录因子的作用。DUF4195结构域是在多细胞动物蛋白N端发现的一个家族,它可以运载PHD样锌指结构域(https://www.ncbi.nlm.nih.gov/Structure/cdd/cddsrv.cgi?uid=206007)。猪ZNF280D基因位于1号染色体,含有20个外显子,该基因有67个直系同源,也同时包含C2H2型锌指结构域和DUF4195结构域(http://asia.ensembl.org)。
在自然界的多种生物中都存在ZNF280D基因的剪接体,在人上已有24个该基因的剪接变异体得到证实,且有7个可能性较大的不同启动子(https://en.wikipedia.org/wiki/SUHW4)。猪上已发现4种该基因的不同转录本,按所含碱基数由大到小排列依次为ZNF280DX2 (5609nt),ZNF280DX1 (5458nt),ZNF280DX5 (2857nt)和ZNF280DX3(2712nt)。ZNF280DX2 与ZNF280DX1相比在第2外显子起始端增添了151个碱基,但该变化发生在CDS区外,因此ZNF280DX2和ZNF280DX1编码的蛋白质中氨基酸相同,为966个。ZNF280DX1包含ZNF280DX5,ZNF280DX5包含ZNF280DX3,前一序列从后一序列的3’端向后延长,ZNF280DX5和ZNF280DX3编码的蛋白质所含氨基酸个数分别为729和791个。4个转录本都含有3个C2H2型锌指结构域和两个Zn2+结合位点。牛、鸡和鼠上也分别发现有8、4和2种该基因不同的转录本(www.ncbi.nlm.nih.gov)。但有关ZNF280D基因研究还很少,具体功能尚不清楚。
马身猪为山西地方优良品种,在抗逆性、繁殖力和肉品质等方面有较多优势。本研究采用RT-PCR扩增和克隆测序技术,鉴定马身猪ZNF280D基因剪接体类型,为进一步探讨ZNF280D基因的结构和功能奠定基础。
1 材料与方法
1.1 试验动物与样品收集
本研究试验动物来自山西省大同市种猪场,0、3、6月龄的马身猪,每阶段4头,公母各半,公猪断奶时去势。试验动物在达到目标日龄当日屠宰,立即取背最长肌,用铝箔包裹,放入离心管中,然后放入液氮速冻,-80 ℃保存备用。
1.2 主要试剂及仪器
PrimeScriptTMRT-PCR reagent Kit with gDNA Eraser(perfect Real Time)(TaKaRa, 大连);RNAiso Plus regent(TaKaRa, 大连)。
AB-9902型触摸式PCR仪(AB公司,美国);DYY-3电泳仪(六一仪器厂,北京);ND-1000核酸蛋白测定仪(Nanodrop,美国)。
1.3 RNA提取和cDNA合成
按照RNAiso Plus regent(TaKaRa, 大连)说明书提取肌肉的总RNA,利用ND-1000核酸蛋白测定仪测定总RNA 的OD 260/280值确定RNA的纯度。用非变性琼脂糖凝胶电泳检测其完整性。按照PrimeScriptTM-RT-PCR reagent Kit with gDNA Eraser(perfect Real Time)(TaKaRa,大连)反转录试剂盒操作说明进行反转录合成cDNA。第一步反应体系10 μL: RNA 500 ng,gDNA Eraser buffer 2 μL,gDNA Eraser 1 μL,加RNase-free水至10 μL。反应程序:42 ℃,2 min,4 ℃,3~5 min。第二步反应体系20 μL:第一步产物加RNAase Free H2O 4 μL,5×primer 4 μL,mixⅠ1 μL,RTmix 1 μL。反应程序:37 ℃,15 min,85 ℃,5 s,4 ℃,5 min。cDNA保存于-20 ℃。
1.4 ZNF280D引物设计与合成
本试验对ZNF280D引物的设计是基于猪转录组测序中可变剪切位点的预测所进行的。转录组测序结果显示,ZNF280DmRNA第1~9外显子中共有3处可能性较大的剪切位点,分别位于第1内含子间、第4内含子间和第7内含子5’端至第8外显子3’端。根据此预测结果和NCBI中猪ZNF280D(登录号:XM_005659574.2)基因序列,选用NCBI Primer-BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi)和 Oligo 7软件设计3对引物(表1)。
引物PL:FL位于第1外显子末端,RL位于第9外显子起始端,跨越第2~8外显子,包含3个预测的可变剪切位点。
引物PS1:FS1与PL相同,RS1位于第5外显子上,跨越2~4外显子,包含前2个预测位点。
引物PS2:FS2位于第7外显子起始端,RS2与RL相同,跨越部分第7外显子,第8外显子和部分第9外显子,包含第3个剪切位点(图1)。先用包含所有剪切位点的引物(PL)来确定ZNF280D基因是否产生多个剪接体,再分别针对不同位点设计引物(PS1和PS2)检测该基因的变异位置,增强试验的可靠性。引物由生工生物工程(上海)股份有限公司合成(表1)。

表1 猪ZNF280D克隆引物Table 1 Primers used for cloning of pig ZNF280D genes
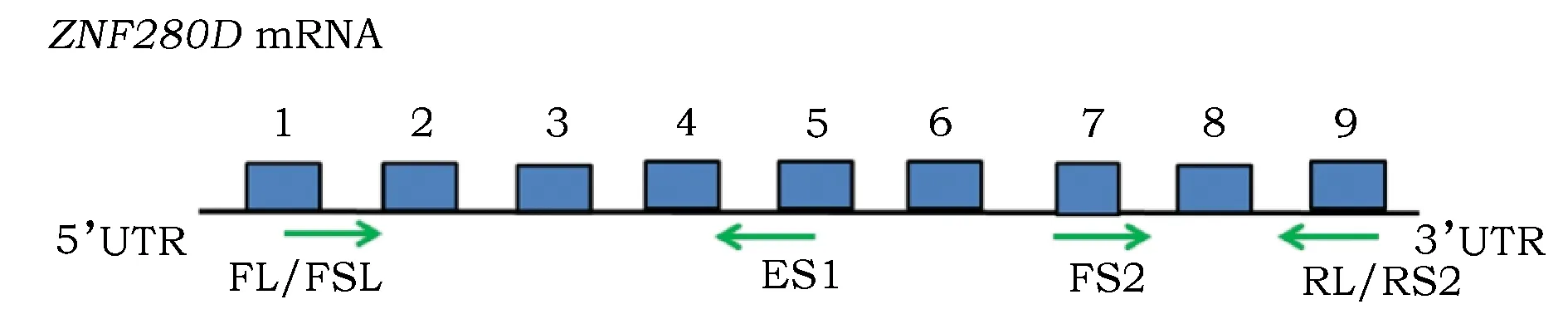
图1 猪ZNF280D基因部分结构图及引物位置Fig.1 The partial genomic structure diagram and positions of the primers of ZNF280D in pig注:(1)蓝色方框代表外显子。(2)绿色箭头代表引物。上游引物FL/FS1位于外显子1;下游引物RS1位于外显子5;上游引物FS2位于外显子7;下游引物RS2/RL位于外显子9。Note: (1) Blue box meant the exon.(2) Green arrow meant the primer. The upstream primer FL / FS1 are located in exon 1; The downstream primer RS1 is located in exon 5; The upstream primer FS2 is located in exon 7; The downstream primer RS2 / RL are located in exon 9.
1.5 PCR扩增及克隆
PCR体系10 μL:cDNA 2 μL,2×Taq PCR MasterMix 5 μL,上下游引物各1 μL,加RNAase Free H2O 至10 μL。PCR反应程序: 95 ℃ 5 min,95 ℃ 30 s,56~64 ℃ 30 s,72 ℃ 69 s 35个循环,72 ℃ 5 min。PCR产物用1.5%琼脂糖凝胶电泳检测分离。
PCR产物经OMEGA Gel Extraction Kit(OMEGA D2500-01)纯化后,连接克隆pMD-19T(TaKaRa,大连),阳性克隆菌液送华大基因测序。
1.6 生物信息学分析
采用Megalign软件对测序结果拼接并进行序列比对,用Editseq软件翻译核苷酸序列,用Megalign比对不同剪接体氨基酸序列,不同物种该基因的同源性并构建生物进化树,用NCBI(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)预测蛋白保守结构域。
2 结果与分析
2.1 猪ZNF280D可变剪接体的鉴定
以cDNA池为模板,扩增ZNF280DmRNA的前半段(外显子1~9),并进行琼脂糖凝胶电泳检测。扩增产物分3部分:引物PS1包含预测结果中的前2个可能发生变化的剪切位点,扩增产物为清晰的一条目的条带(图2a),说明在外显子1~5之间无可变剪接体产生,对于前2个剪切位点的预测结果不成立。PS2扩增产物(图 2b)与PL扩增产物(图2c)都显示为2条甚至多条带,推测ZNF280D有至少2种以上的转录本。

图2 琼脂糖凝胶电泳检测RT-PCR 扩增结果Fig.2 RT-PCR products detected by agarosegel electrophoresis
对PS2和PL引物扩增的产物进行割胶回收、纯化、连载体转入感受态细胞克隆。测序结果显示含有2条不同序列:一条是经正常剪接产生的ZNF280DmRNA,另一条则由于第7内含子3’端剪切位点后移至第8外显子内,使得第8外显子由5’端开始的222个碱基序列被剪切,仅保留第8外显子3’端最后两个碱基(5’-AG-3’)(图3,图4),分别定义常规转录本和变异转录本为ZNF280D1和ZNF280D2。ZNF280D2是不同于NCBI数据库中猪ZNF280D基因4个剪接体的新序列。
2.2 生物信息学分析
采用Megalign软件对克隆菌液测序结果拼接,并采用Editseq软件翻译这两条核苷酸序列进行比对,并对多肽链进行比对,与ZNF280D1相比,ZNF280D2缺失了第247~322个AA(图5)。并用NCBI的BLAST对两条多肽链进行蛋白结构域分析,在ZNF280D1的第347~426 AA存在着3段C2H2型锌指蛋白的保守结构域和2个Zn2+结合位点。而剪接体ZNF280D2在第229~352AA间有4段同类型的锌指蛋白结构域和3个Zn2+结合位点,但它们都属于典型C2H2型锌指蛋白(图6)。
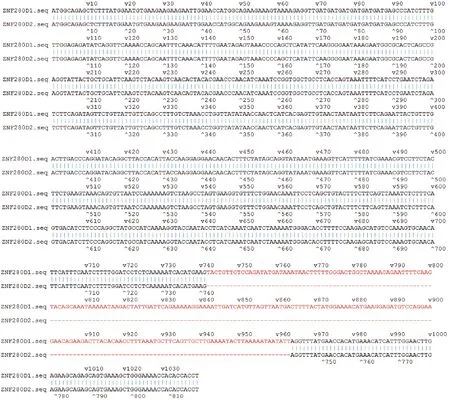
图3 猪ZNF280D1 和 ZNF280D2 的部分核苷酸序列比对Fig.3 Partial nucleotide sequence alignments of ZNF280D1 and ZNF280D2 in pig注:红色“…”代表缺失序列,蓝色“∣”代表同样的序列。Note: The red“…”meant deletion of nucleotide; The blue “∣”meant the same sequence.
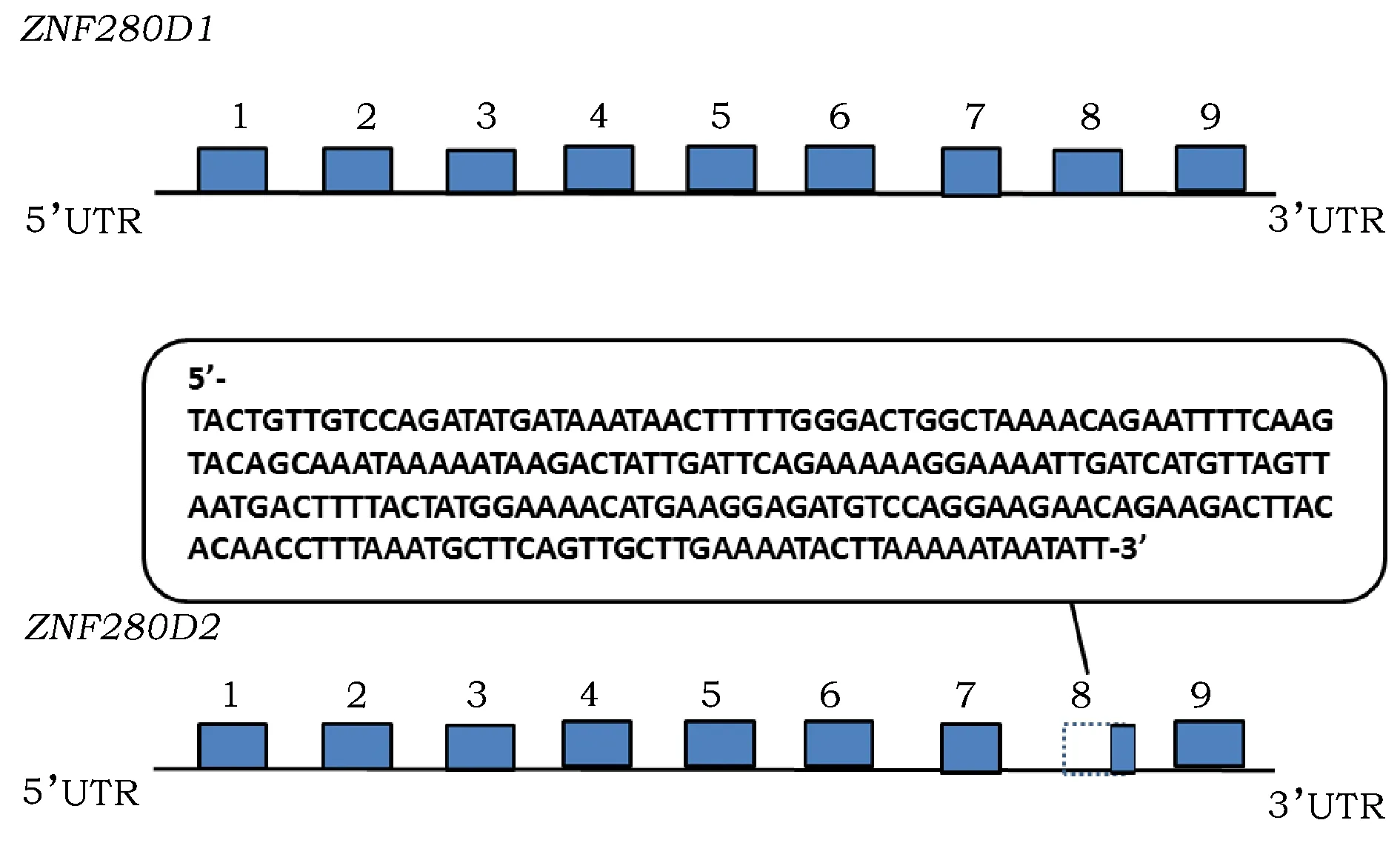
图4 猪ZNF280D1和ZNF280D2的部分基因结构图Fig.4 The partial genomic structure diagram of ZNF280D1和ZNF280D2 in Mashen pig注:虚线方框代表外显子缺失部分。Note: Dotted box meant the partial deletion of exon.
采用Megalign比对猪ZNF280D与其他物种的该蛋白AA序列的同源性并构建生物进化树,结果显示猪ZNF280D蛋白与小须鲸、牪牛和绵羊的同源性最高,其次是蒙古野马、白犀牛、雪貂和长翼蝠,而与果蝇与人的同源性较低(图6,图7)。
3 讨论
本研究发现,在马身猪中,只出现了2个ZNF280D的转录本,除了常规的ZNF280DmRNA,还发现了一个新剪接体。不同转录本出现的原因可以分两类,mRNA的选择性剪切和翻译起始位点的不同。分析该剪接体的核苷酸序列,并与常规的ZNF280DmRNA(ZNF280D1)相比,ZNF280D2变异剪切位点发生在第7内含子3’端,导致第8外显子5’端选择性缩短,该外显子的大部分序列缺失,除此之外没有内含子保留或其他的碱基增添现象,而ZNF280D基因的起始密码子位于第2外显子,该位置没有发生任何变化,因此该剪接体的启动子未发生改变,仅基因编码区内发生选择性剪切。对这2个转录本的氨基酸序列在线分析(https://blast.ncbi.nlm.nih.gov/Blast.cgi)发现,两者所编码蛋白都含有保守的C2H2型锌指蛋白结构域,但常规ZNF280DmRNA含有3个该结构域,属于tC2H2型锌指结构蛋白,新型剪接体则由于第8外显子的大部分缺失,在变异的位置碱基重新组合使得其编码的蛋白质增加了一个锌指蛋白的保守结构域,变成了maC2H2型锌指蛋白。

图5 马身猪ZNF280D1和ZNF280D2氨基酸序列对比图Fig.5 Amino acid sequence alignments of ZNF280D1 and ZNF280D2 in Mashen pig注:黑色方框代表相同氨基酸,“——”代表缺失氨基酸。Note: Black box meant the same sequence, “——”meant deletion of amino acid sequence.
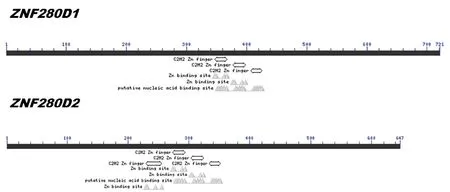
图6 ZNF280D1和ZNF280D2的氨基酸保守结构域Fig.6 Conservative amino acid structure domain of ZNF280D1 and ZNF280D2

图7 ZNF280D1和 ZNF280D2蛋白在不同物种间的系统发育树(猫头鹰猴,牦牛,小须鲸,白犀牛,果蝇,野马,人,穿山甲,长翼蝠,雪貂和绵羊)Fig.7 Phylogentic tree of ZNF280D1 and ZNF280D2 protein among different species (Aotus nancymaae, Bos mutus, Balaenoptera acutorostrata scammoni, Ceratotherium simum simum, Drosophila, Equus przewalskii, Homo sapiens, Manis javanica, Miniopterus natalensis, Mustela putorius furo, Ovis aries aries)
GAGA因子是唯一一个只含有一个C2H2型锌指结构的蛋白[6],其他的C2H2型锌指蛋白都含有3个或3个以上的锌指结构,但并不是所有的结构域都可以结合DNA,例如典型的maC2H2型转录因子TFⅢA,含有9个锌指结构,其中只有前1~3个锌指可以穿过DNA双螺旋的“大沟”与之特异性结合,而锌指4~6则游离在DNA外部形成一个开放型结构[7~10]。除此之外,很多研究显示,结合DNA的各锌指结构其结合能力也不相同,例如TFⅢA,其第7~9个锌指结构也可以结合DNA,但亲和力比锌指1~3要弱[11,12]。小鼠的转录因子tC2H2型锌指蛋白Zif268所含的3个锌指结构按亲和力由大到小排列依次为锌指1大于锌指2大于锌指3[13]。maC2H2型锌指蛋白WT1含有的4个锌指结构中,锌指1与DNA的结合能力明显低于锌指2~4,但锌指2~4对靶DNA的亲和力要高于完整的锌指1~4结构[14,15],这一现象也暗示了,不能结合DNA的锌指结构具有增强其他锌指与DNA结合力,使得整个结构更加稳定的作用。许多其他研究也表明,通常蛋白中所含锌指的数目越多, 它选择结合的能力也就越强。除此之外,锌指结构中锌的作用也不可替代,锌离子与氨基酸残基结合使肽链形成稳定的折叠结构,进而通过该结构激活蛋白与DNA结合发挥生物学功能。本研究中通过分析ZNF280D蛋白结构域发现,ZNF280D1和ZNF280D2蛋白的最后一个C2H2锌指结构域都不含有Zn2+结合位点,不能形成稳定的锌指结构与DNA结合。ZNF280D2发生变异后增加的锌指1虽含有Zn2+结合位点,但不能结合核苷酸,说明该锌指结构与DNA没有实际结合功能,但它的出现可能提升了锌指2~3与DNA的亲和力,使得该剪接体所编蛋白比传统ZNF280D蛋白更易结合DNA,形成更加稳定的结构,在该基因的编码水平达到蛋白进化的效果。
C2H2锌指蛋白在生物体生长发育过程中起着不容忽视的作用,很多研究发现它可以影响机体的免疫功能,例如参与造血干细胞、T细胞[16]和B细胞[17]等免疫细胞的分化和成熟,某些炎性因子[18]的产生,信号转导,以及肿瘤的形成[19,20]。在人的某些免疫性疾病如红斑狼疮[21]和念珠菌病等遗传病[22]中已经发现存在锌指蛋白基因的变异,在肝细胞癌、胃癌、结肠癌和非小细胞型肺癌中也都能检测到某些锌指蛋白(ZNF165)mRNA 的表达[23]。这一系列关于C2H2型锌指家族的研究暗示了ZNF280D基因在机体生长发育以及免疫过程中的潜在影响,对进一步研究ZNF280D基因在生物体中的功能具有导向作用。
4 结论
本试验在马身猪上发现了一个ZNF280D基因的新型剪接体(ZNF280D2),该剪接体第8外显子靠近5’端的大部分序列被剪切,导致其编码的蛋白质中增加了一个C2H2型锌指结构域,该结构域不能结合DNA,但可能会增强相邻锌指结构与DNA的亲和力,提高结合产物的稳定性。
[1]Nagase T, Kikuno R, Nakayama M,et al. Prediction of the coding sequences of unidentified human genes. XVIII. The complete sequences of 100 new cDNA clones from brain which code for large proteins in vitro[J].DNA Research,2000,7 (4):273-281.
[2]周畅,李麓芸.C2H2型锌指蛋白的研究进展[J].生命科学研究, 2004,8(3):215-220.
[3]郭丽丽,慈宏亮,单红爽,等.人类锌指结构新基因ZNF18的克隆和表达谱分析[J].遗传.2005,27(4):523-530.
[4]Zhou C,Li L Y,Lu G X.Molecular cloning and character analysis of the mouse zinc finger protein gene Zfp474 of exclusively expressed in testis and ovary[J].Acta Genetica Sinica,2005,32:155.
[5]Rebar E J,Huang Y,Hickey R,et al.Induction of an angiogenesis in a mouse model using engineered transcription factors [J]. Nature Medicine,2002,8(12):1427-1432.
[6]Omichinski J G,Pedone P V,Felsenfeld G,et al. The solution structure of a specific CACA factor-DNA complex reveals a modular binding mode[J]. Nature Structural and Molecular Biology,1997,4(2): 122-132.
[7]Nolte R T,Conlin RM,Harrtson S C,et al. Differing roles for zinc fingers in DNA recognition: structure of a six-finger transcription factorⅢ A complex[J]. Proceedings of the National Academy of Sciences,1998,95(6):2938-2943.
[8]Neely L,Trauger J W,Baird E E,et al.Importance of minor groove binding zinc fingers within the transcription factorⅢA-DNA complex[J]. Journal of Molecular Biology,1997,274(4):439-445.
[9]Neely L S,Lee B M,Xu J,et al.Identification of a minimal domain of 5 S ribosomal RNA sufficient for high affinity interactions with the RNA specific zinc fingers of transcription factor ⅢA[J]. Journal of Molecular Biology,1999,291 (3):549-560.
[10]Searles M A,Lu D,Klug A.The role of the central zinc fingers of transcription factor Ⅲ A in binding to 5 S RNA[J]. Journal of Molecular Biology,2000,301(1):47-60.
[11]Liao X B,Clemens K R,Tennant L,et al. Specific interaction of the first three of TFⅢA With the internal control region of the Xenopus SSRNA gene[J]. Journal of Molecular Biology,1992,223(4):857-871.
[12]Clemens K R,Zhang P,Liao X,et al.Relative contributions of the zinc fingers of transcription factorⅢA to the energetics of DNA binding[J].Journal of Molecular Biology,1994,244 (1):23-35.
[13]Choo Y. End effects in DNA recognition zinc finger arrays[J].Nucleic Acids Research,1998,26(2):554-557.
[14]Nakagama H,Heinrich G,Pelletier J,et al.Sequence and structural requirements for high-affinity DNA binding by WT1 gene product [J]. Molecular Cellular Biology,1995,15(3):1489-1498.
[15]Caricasole A,Duarte A,Larsson S H,et al.RNA binding the Wilms tumor suppressor zine finger proteins[J]. Proceedings of the National Academy of Sciences,1996,93(15):7562-7566.
[16]Carson B D,Lopes J E,Soper D M,et al.Insights into transcriptional regulation by FOXP3 [ J].Front Biosci,2006,11 (1):1607-1619.
[17]Thompson E C,Cobb B S,Sabbattini P,et al.Ikaros DNA binding proteins as integral components of B cell developmental-stage-specific regulatory circuits[J].Immunity,2007,26 (3):335-344.
[18]Fortier J M,Kornbluth J.NK lytic-associated molecule,involved in NK cytotoxic function,is an E3 ligase [J].Journal of Immunol, 2006,176 (11):6454-6463.
[19]Liyama T,Udaka K,Takeda S,et al.WT1(Wilms’ Tumor 1) peptide immunotherapy for renal cell carcinoma [J].Microbinology and Immunology, 2007,51 (5):519-530.
[20]王政,段跃强,罗德炎.DNA免疫激发针对肾母细胞瘤CTL效应初步研究[J].免疫学杂志,2007,23 (3):252-254,259.
[21]Narvi E,Nera K P,Terho P,et al.Aiolos controls gene conversion and cell death in DT40 B cells[J]. Scandinavian Journal of Immunology,2007,65 (6):503-513.
[22]O’Dwyer D T,Mcelduff P,Peterson P,et al.Pituitary autoantibodies in autoimmune poly endocrinopathy-candidiasis ectodermal dystrophy (APECED)[J].Acta bio-medica: Atenei Parmensis, 2007,78(1):248-254.
[23]Dong X Y,Yang X A,Wang Y D,et al.Zinc-finger protein ZNF165 is a novel cancer-testis antigen capable of eliciting antibody response in hepatocellular carcinoma patients[J]. British Journal of Cancer, 2004,91(8):1566-1570.
(编辑:马荣博)
IdentificationandbioinformaticsanalysisofalternativesplicingisoformsofZNF280DgeneinMashenPig
Jin Yushu, Li Meng, Zhang Qi, Gao Pengfei, Li Bugao, Guo Xiaohong*
(CollegeofAnimalScienceandVeterinaryMedcine,ShanxiAgriculturalUnivesity,Taigu030801,China)
Q78
A
1671-8151(2017)10-0726-07
2017-03-28
2017-06-06
靳玉舒(1993-),女(汉),山西长治人,硕士研究生,研究方向:动物环境调控
*通信作者:郭晓红,副教授,硕士生导师,E-mail:g_xiaohong@126.com
山西省科技创新重点团队项目(201605D131045-24);山西省农业科技攻关项目(20140311020-5)

