杂交链式反应在生物传感探针设计方面的应用
杨 婵, 王瑞嘉, 何婧琳, 肖 慧, 邓 伟, 向建南, 曹 忠
(1.长沙理工大学化学与生物工程学院,电力与交通材料保护湖南省重点实验室,微纳生物传感与食品安全检测协同创新中心,湖南长沙410114)
(2.湖南大学化学化工学院,化学生物传感与计量学国家重点实验室,湖南长沙410082)
0 引言
生物传感器是一种结合了生物敏感元件、物理化学信号转换器和电子信号处理器的新兴仪器,其工作原理是将生物敏感元件在发生特异性反应时所产生的信号经由物理化学信号转化器,转化为光、电、声等易被检测到的信号,最终通过换算等手段获知待测物的有关信息[1]。近年来,生物传感技术由于具有灵敏度高、选择性好、成本低以及能够进行连续监测等优点,成为了一类新型的传感技术。它在基因分析、遗传疾病诊断、法医鉴定和环境监测等领域具有极大的应用潜力和广阔的发展空间,因此成为科学研究者研究的重点。
提高生物传感技术的灵敏度,关键依赖于信号的放大,而信号放大的方法普遍使用的是核酸扩增技术,它是一种对特定序列DNA进行体外扩增的技术[2-3]。核酸扩增技术根据扩增是否经过温度变化主要分为两类:一类是核酸变温扩增反应,主要有聚合酶链式反应(Polymerase chain reaction,PCR)、连接酶链式反应 (Ligase chain reaction,LCR)。杂交链式反应技术(Hybridization chain reaction,HCR)、滚环等温扩增技术(Rolling circle amplification,RCA)、链替代等温扩增技术(Strand displacement amplification,SDA)等。但是,在恒温条件下,不需要酶参与的扩增技术只有HCR反应。
PCR技术是一种在DNA聚合酶的辅助下,以母链DNA作为模板,将特定引物作为延伸起点,经过高温变性、低温退火和恒温延伸三个步骤的多次循环,复制出与母链DNA互补的子链DNA[4-8]的一种核酸扩增手段。PCR技术拥有着高灵敏度、高特异性、对模板纯度要求低以及重现性好等多方面的优点,已被广泛应用于转基因、病原体定量检测、疾病分类以及药物疗效考核等领域的研究。但是该扩增技术也存在以下缺点,如:需要精密的仪器设备、易产生假阳性反应[9]、不适用于基层及现场的快速检测等。
LCR是Barany[10]在1991年从水生栖热菌中提取耐高温的DNA连接酶,并利用该连接酶成功的扩增了DNA时所发现的一种新型的核酸扩增反应方式,Barany也正式将这种方法命名为连接酶链式反应。该方法的关键点是耐高温的DNA连接酶对连接缺口具有高保真的要求,且连接酶在多次循环中始终能保持活性,提高了LCR的特异性。
相对于 PCR和 LCR反应,RCA、SDA与HCR反应在恒温条件下就可以进行核酸扩增。RCA是Landegren[11]发明的一种基于模拟自然界微生物环状DNA的滚环式扩增机制而构建的体外核酸等温扩增技术。它以环状DNA做模板,在dNTPs存在的条件下,引物与模板杂交后通过DNA聚合酶使引物沿着环状DNA模板进行复制。因为DNA聚合酶具有链置换效应,因此新合成的链可以不断延长,最终得到一段具有重复序列单元的DNA长链,实现检测信号放大的目标。RCA包括线性RCA和指数RCA两种形式,线性RCA是引物结合到环状DNA上后,在具有链置换效应的DNA聚合酶作用下被延伸,最终扩增成为具有大量重复序列的线状单链。而指数RCA相比较线性RCA而言,仅多采用了一条与环状DNA序列完全一致的第二条引物,第二条引物可与第一次线性RCA的产物结合并酶促延伸,可在很短的时间内实现指数扩增。
RCA技术具有高灵敏度、强特异性、设备要求简单、高通量等一些列优点,因而得到广泛的应用。目前主要用于痕量的分子检测领域,主要有全基因组扩增[12]、蛋白质芯片分析[13]、单核苷酸多态性[14]、细胞原位检测[15]等领域。但RCA也存在着琐式探针长度较长(一般100 nt左右)、合成费用高等缺点。
SDA技术最早由Walker等[16]提出。它是一种基于酶促反应的体外核酸等温扩增技术,其原理在于限制性内切酶剪切DNA识别位点的能力及DNA聚合酶在切口处向3'端延伸并置换出下游序列,于恒温条件谢爱实现扩增。被置换的DNA单链与引物结合,在DNA聚合酶的作用下延伸成双链。该延伸过程不断的重复,最终使得目标DNA序列呈指数增长。因SDA技术具有快速、高效、特异等优点,因而受到广泛关注并被不断改良。
HCR反应是由Pierce和Dirks在2004年提出的一种体外核酸等温信号放大技术[17],是一个没有酶参与的过程。它的原理如图1所示,HCR元件包括两个部分:引发探针、两条可杂交互补并带有粘性末端的发夹型DNA(H1和H2)。当不存在引发探针时,两条发夹探针稳定存在于溶液中。若存在引发探针时,引发探针触发HCR反应开始,发夹探针H1与H2相继打开,形成具有多个重复单元的共聚物[18-19],直到发夹探针H1与H2消耗完为止,以此来实现信号扩增的目的。HCR的优点在于不需要酶的辅助,避免了非特异性扩增对分析结果的影响,反应条件温和、易控制,等温条件下经一步反应就可以实现短链DNA的扩增,并不需要复杂的仪器设备。HCR这一信号扩增技术与各类检测技术(如:荧光法、比色法、电化学法等)都具有较高的兼容性,基于以上优点,研究人员利用HCR反应提高核酸、蛋白、小分子及重金属等生物分子检测的灵敏度。与其他核酸扩增方法相比,HCR最大的优势是无酶参与,为研究者们的研究提供了很大的便利。

图1 杂交链式反应的原理图[17]Fig.1 Schematic illustration based on hybridization chain reaction[17]
1 HCR在分子信标探针设计方面的应用
分子信标(Molecularbeacon,MB)是一种根据核酸碱基配对原则和荧光共振能量转移(Fluorescence resonance energy transfer,FRET)现象来设计的发卡型寡核苷酸探针[20]。MB由茎区部分、环区部分、荧光供体基团和猝灭受体基团四部分构成,环区部分一般有15-30个核苷酸长,并与目标序列互补;茎区部分一般有5-7个核苷酸长,并相互配对形成茎的结构。荧光供体基团和猝灭受体基团分别被标记在探针的两端。分子信标的作用原理如图2所示,MB的环状部分由和目标序列互补的核苷酸序列组成,当MB体系中未加入待检测目标序列时,MB的荧光供体基团和猝灭受体基团之间的距离很小,发生荧光共振能量转移现象,供体分子荧光被受体基团猝灭。当MB体系中加入待检测的目标序列时,由于碱基间的互补配对,使MB发夹结构发生变化,导致供体基团和受体基团之间的距离变大,FRET被破坏,荧光显现。MB法不仅操作简单、灵敏度高、特异性强,而且还可以避免核酸交叉污染,同时实现实时检测。

图2 分子信标原理Fig.2 The principle of molecular beacon
2012年,谭蔚泓等[21]利用HCR放大技术与芘MB的核酸发夹探针结合,构建一种灵敏检测目标DNA的荧光传感器。原理如图3所示,两个发夹探针H1*与H2*用芘分子进行标记。当目标DNA不存在的时候,H1*与H2*呈发夹结构,稳定存在于体系中,而芘分子以单体形式存在,荧光较低。当目标DNA存在的时候,目标DNA作为引发链,引发H1*、H2*与之杂交,发生HCR反应,而芘分子以二聚体形式存在,致使荧光强度增加,根据前后荧光强度的变化来检测目标DNA。该实验可用于复杂生物中DNA的检测。

图3 基于HCR放大技术检测DNA[21]Fig.3 Working principle behind the detection of DNA on the basis of HCR amplification[21]
2 HCR在G-quadrup lex探针设计方面的应用
Gellert小组[22]在1962年首次报道了基于碱基G的G-四分体结构(G-quartet)。富含碱基G的G-DNA序列通过对应G碱基之间形成Hoogsteen氢键,使得4条或4段富含G碱基的DNA片段肩并肩的形成G-四分体,再通过纵向鸟嘌呤杂环之间的π-π作用形成G-四链体结构(G-quadruplex),这种结构如图4所示。在G-quadruplex的中轴方向有易极性中央孔道,可以螯合适当体积的阳离子[23](如:K+、Li+、Na+),增强G-quadruplex的结构稳定性。

图4 G-四分体的多种结构[22]Fig.4 Conformations of several G-quartets[22]
2012年,Dong J等[24]利用结合 HCR与G-quadruplex构建了一种免标记检测目标DNA的荧光传感器。实验原理如图5A所示,目标DNA不存在时,没有引发链引发杂交链式反应,荧光强度较低。目标DNA存在时,目标DNA作为引发链打开两个发夹探针与其杂交,进行杂交链式反应形成长双链,释放出G-quadruplex结构,加入锌卟啉(ZnPPIX)后,荧光强度增强。体系前后荧光强度的变化可以检测目标DNA。2015年,Alexander Trifonov等[25]利用 HCR反应与 G-quadruplex结合,电化学检测目标DNA。实验原理如图5B,该传感系统把目标DNA被固定在金电极上,作为引发剂与两个发夹探针发生杂交反应形成长双链,完成杂交链式反应,释放出更多的 G-quadruplex。 加入血红素(hemin)后,导致hemin/G-quadruplex电催化H2O2还原成H2O,使电化学信号扩增,达到检测目标DNA的目的。

图5 HCR扩增(A)荧光(B)电化学检测DNA序列的原理图[24-25]Fig.5 Schematic illustration of the HCR based fluorometric(A)or electrochemical(B)sensors for DNA sequences detection[24-25]
3 HCR在DNAzyme探针设计方面的应用
1994年,Breaker和Joyce[26]利用催化洗脱和体外筛选相结合的方法,从随机单链DNA分子文库中,发现了不仅能够特异性结合且切割RNA底物,而且具备催化活性的DNA分子,称之为脱氧核酶(DNAzymes)。与蛋白质酶相比,DNAzymes更易合成、抗水解、热稳定好。根据其催化作用的不同,DNAzymes可以分为五大类:起连接作用的DNAzymes[27]、切断 DNA 的 DNAzymes[28]、切断RNA的DNAzymes[29]、催化卟啉环金属螯合反应的DNAzymes[30]、具有激酶活性的 DNAzymes[31]。其中,应用最多的是切断RNA的DNAzymes,它不仅可以催化RNA特定部位的切割,而且还可以从mRNA水平来对基因进行灭活,进行调控蛋白的表达。将HCR与DNAzyme结合,使传感平台更上一个台阶。2013年,Zhuang等[32]以DNAzyme作为识别分子,与HCR反应结合,用电化学的方法灵敏检测Pb2+。原理如图6(A)所示,将Pb DNAzyme固定在磁珠上,当Pb2+存在时,Pb2+诱导DNAzyme切割其底物,释放的底物使两条标记了二茂铁的发夹探针H1、H2聚合杂交在磁珠表面,引发磁珠表面的HCR反应,致使电化学信号放大。当Pb2+不存在时,DNAzyme稳定存在于体系中,不能释放出引物诱导HCR反应,则没有电化学信号产生。根据目标物是否存在时电化学信号的变化来检测Pb2+。2015年,Lina Freage等[33]将Pb DNAzyme作为识别分子,Mg DNAzyme作为报告基团,与HCR反应结合灵敏检测Pb2+。原理如图6(B)所示,将Pb DNAzyme固定在电极上,当Pb2+存在时,致使底物被DNAzyme切割,释放出的产物打开发夹探针,形成聚Mg DNAzyme的长链,在Mg2+的作用下切割分子信标底物,致使荧光放大。当Pb2+不存在时,底物链不能被DNAzyme切割,则不能释放HCR反应的引物,不能形成聚Mg DNAzyme的长链,无荧光产生。在有无目标物存在时荧光强度前后的变化达到检测Pb2+的目的。
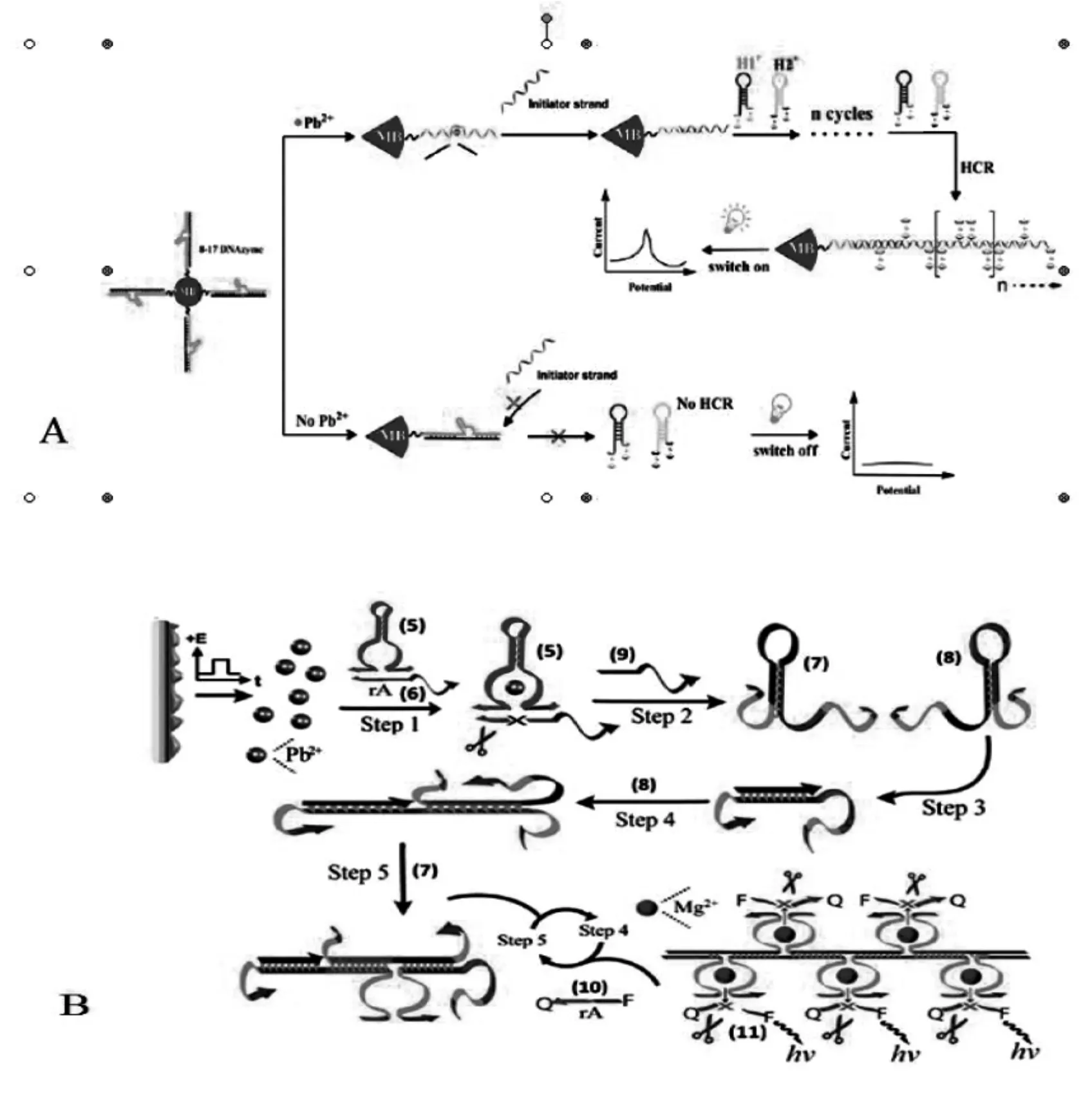
图6 基于HCR和DNAzyme的电化学(A)或荧光(B)分析方法检测Pb2+[32-33]Fig.6 Fluorescence response of electrochemical(A)or fluorescent(B)analysis detection ofPb2+based on HCR reaction and DNA zymes[32-33]
4 HCR在核酸适配体(Aptamer)探针设计方面的应用
核酸适配体(Aptamer)是一种经由体外指数级富集系统进化 (Systematic Evolution of Ligands by EX ponential Enrichment,SELEX)技术筛选所得的随机寡核苷酸片段,可以是DNA或者RNA,且该寡核苷酸片段对特定目标物质具有特异性结合的能力[34],结合的物质小至离子、分子,大至细菌、细胞。Aptamer不仅类似于其他分子识别物质对目标物特异性结合,同时还具有许多其他的独特的特点,如:(1)分子量较小,与目标物结合的空间位阻小,利于构建高表面密度的传感平台。(2)目标物质广泛,由于SELEX技术的存在,筛选出的能识别的目标物包括了金属离子、氨基酸、细菌、蛋白质、抗生素等领域。(3)核酸适配体容易合成、修饰,SELEX技术筛选的核酸适配体一般只需要2-3个月就可以被筛选出来,最快的2~3周就可以完成筛选。(4)化学性质稳定等。基于这些特点,使Aptamer在生物方面、化学方面、医学应用方面都有巨大的应用前景。
Aptamer与靶向分子结合时,通过核酸链内某些碱基之间的互补配对、氢键以及静电等作用,导致单链核酸的结构和空间构象发生变化,形成一些稳定的空间结构,比如:发卡结构、假结结构、茎-环结构等。目前,基于Aptamer的生物传感器方法有很多,其中光学Aptamer传感器、电化学Aptamer传感器、比色法Aptamer传感器备受研究人员的关注。
光学Aptamer传感器是一类检测Aptamer与靶物质作用时引起光学信号变化的传感器。根据其原理不同分为荧光、化学发光、磷光等,应用范围很广泛。根据荧光检测的原理,目前主要有两种方法检测目标物[35],一种是用Aptamer先和一个在链中间标记了荧光基团的核酸链杂交,利用碱基自身的淬灭作用,荧光淬灭,当Aptamer与目标物结合后,被标记了荧光基团的核酸链被置换,从而发出荧光。另一种方法是将与Aptamer杂交的核酸链在两端分别标记荧光基团和淬灭基团,且两端均有一定长度的碱基互补,当目标物存在时,核酸链被置换下来,互补端结合成双链,荧光淬灭。由于光学染料很容易获得,信号大部分是在均相中检测,灵敏度高,检测下限低,光化学技术的应用越来越广泛,但是它的不足之处依然存在,如荧光技术就存在背景信号高、荧光寿命短、容易发生光漂白和易受干扰等。
电化学Aptamer传感器根据测量信号产生原理的不同,可以分为电压型、电流型和阻抗型Aptamer传感器。在这些传感器当中,由于电流型Aptamer传感器检测的电信号灵敏,且容易获得,所以是研究领域中较活泼的。根据不同的设计方向,电流型适配体传感器目前主要常用的有适配体信标法 (Aptamer beacon)和夹心法。其中,Aptamerbeacon的主要原理是通过吸附、化学修饰、或生物亲和等方法在寡核普酸链或配体上修饰一些电化学活性物质或催化活性的无机或生物分子,如二茂铁、中性红、辣根过氧化酶等,通过这些物质在电极表面发生氧化还原反应,然后再通过检测修饰物本身或其催化底物的电信号来实现物质的检测。Aptamer技术与其他一些放大技术相结合使用,可以显著地提高检测的灵敏度。夹心法是一种高灵敏度的电流型Aptamer传感器,这个方法是采用抗体和Aptamer夹心,然后将碱性磷酸酯酶修饰到适配体上,再通过滚环扩增放大电信号,低检测极限地检测了血小板生长因子。当没有目标物时,亚甲基兰标记的Aptamer处于打开状态,电活性物质远离电极表面,没有电信号产生。而当目标物存在时,Aptamer与目标物结合,电活性物质接触电极表面,产生电信号。因此,基于阻抗变化的电化学适配体传感器,同样具有高灵敏度、无需标记、简单、方便等优点。
结合Aptamer传感器和HCR的优点,研究者们设计了两者结合的方法来检测目标物。2015年,Wang等[36]利用HCR与Aptamer结合构建了一种荧光传感器用于检测血小板衍生生长因子BB(PDGF-BB),原理如图7所示,该传感体系包括三个DNA探针:HP、H1和H2探针。而HP探针主要由三个部分组成,分别是PDGF-BB aptamer、延伸 DNA(E-DNA)、与 H1 探针的 5′互补配对的序列,在这之间有10个连续的腺嘌呤碱基,利于减少它与其他DNA分子之间的空间阻效应[37]。当PDGF-BB不存在时,HCR反应不能被触发,因此HP、H1、H2探针稳定存在于溶液中,能够被吸附在氧化石墨烯(GO)的表面,同时,SG也容易通过π-π键吸附在GO表面,致使发出微弱的荧光。当PDGF-BB存在时,HP探针中的aptamer序列可以识别PDFG-BB形成PDGFBB-aptamer复合体,释放出E-DNA与H1探针的末端进行杂交,打开发夹探针,释放出的H1探针的部分序列与探针H2的部分序列杂交,从而打开H2发夹探针。通过这种方式,形成一条长双链,双链嵌入剂SG可以嵌入其中,加入GO后,由于双链DNA的骨架可以有效屏蔽带负电荷的磷酸盐中的核碱基[38], 导致GO不能吸附HCR产物,所以PDFG-BB的浓度能增强该传感体系的荧光信号。
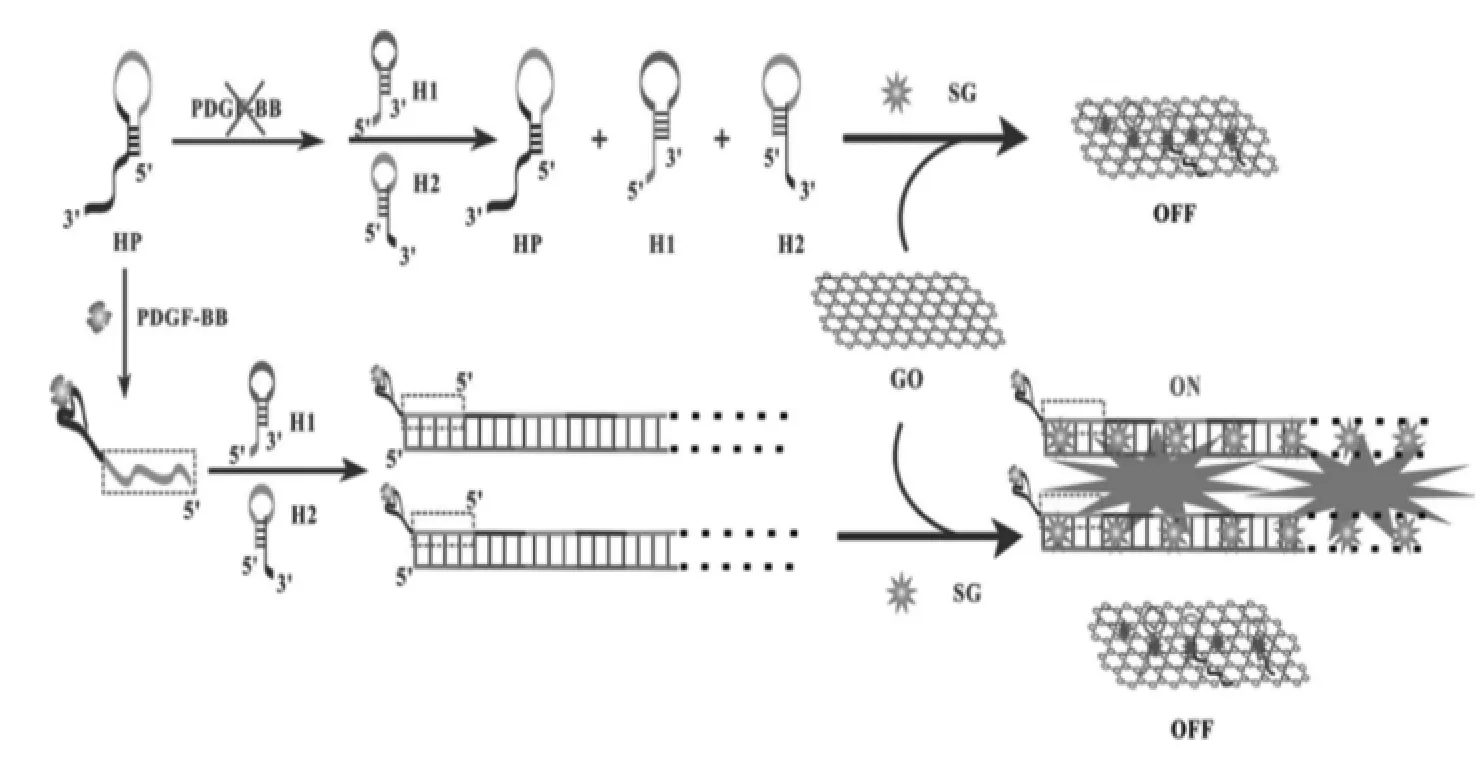
图7 基于杂交链式反应荧光法检测PDGF-BB的原理图[36]Fig.7 Schematic illustration of the fluorescence assay for PDGF-BBbased on the target-triggered hybridization chain reaction amplification[36]
5 总结
综上所述,HCR反应在生物传感探针设计方面的研究已经有了很大的进展。基于HCR的生物传感器是一种无需酶参与的、在常温下就可以进行反应的一种高灵敏、快速的生物传感器,它可以与多种检测技术结合来高灵敏检测金属离子、小分子、细胞等目标物。目前,大部分传感探针用于HCR反应的研究备受研究者们的青睐。因此,开发基于HCR反应能快速、灵敏检测目标物的生物传感探针将成为重要的研究方向。
[1]Hedenmo M,Narváez A,Domínguez E,et al.Improved mediated tyrosinase amperometric enzyme electrodes[J].Journal of Electroanalytical Chemistry,1997,425(425):1-11.
[2]Du Y,Guo S,Dong S,etal.An integrated sensing system for detection of DNA using new parallel-motif DNA triplex system and graphene–mesoporous silica–gold nanoparticle hybrids[J].Biomaterials,2011,32(33):8584-8592.
[3]Zheng X,Liu Q,Jing C,etal.Catalytic gold nanoparticles for nanoplasmonic detection of DNA hybridization[J].Angewandte Chemie International Edition,2011,50(50):12200-12204.
[4]Ho SN,HuntH D,Horton RM,et al.Site-directed mutagenesis by overlap extension using the polymerase chain reaction[J].Gene,1989,77(1):51-59.
[5]Liang P,Pardee A B.Differential display of eukaryotic messenger RNA bymeansof the polymerase chain reaction[J].Science,1992,257(5072):967-971.
[6]Bustin SA.Absolute quantification ofmRNA using realtime reverse transcription polymerase chain reaction assays[J].Journal of Molecular Endocrinology,2000,25(2):169-193.
[7]Ramakers C,Ruijter JM,Deprez R H,et al.Assumption-free analysis of quantitative real-time polymerase chain reaction (PCR)data[J].Neuroscience Letters,2003,339(1):62-66.
[8]Hua Z,Rouse JL,EckhardtA E,et al.Multiplexed realtime polymerase chain reaction on a digital microfluidic platform[J].Analytical Chemistry,2010,82(6):2310-2313.
[9]Wang X P,Yin BC,Wang P,etal.Highly sensitive detection of microRNAs based on isothermal exponential amplification-assisted generation of catalytic G-quadruplex DNAzyme[J].Biosensors and Bioelectronics,2013,42:131-135.
[10]Barany F.Genetic disease detection and DNA amplification using cloned thermostable ligase[J].Proceedings of the National Academy of Sciences,1991,88(1):189-193.
[11]Baner J,Nilsson M,Mendel-Hartvig M,et al.Signal Amplification Of Padlock Probes By Rolling Circle Replication[J].Nucleic Acids Research,1998,26(22):5073-5078.
[12]Wang G,Maher E,Brennan C,etal.DNA Amplification Method Tolerant To Sample Degradation[J].Genome Research,2004,14(11):2357-2366.
[13]Xue Q,Wang Z,Wang L,et al.Sensitive Detection of Proteins Using Assembled Cascade Fluorescent DNA Nanotags Based On Rolling Circle Amplification[J].Bioconjugate Chemistry,2012,23(4):734-739.
[14]Li J,Deng T,Chu X,et al.Rolling circle amplification combined with gold nanopartical aggregates for highly sensitive identification of single-nucleotide polymorphisms[J].Analytical Chemistry,2010,82 (7):2811-2816.
[15]Larsson C,Grundberg L,Soderberg O,et al.In situ detection and genotyping of individual mRNA molecules[J].Nature Methods,2010,7(5):395-397.
[16]Walker G T,Little M C,Nadeau JG,et al.Isothermal in vitro amplification of DNA by a restriction enzyme/DNA polymerase system[J].Proceedings of the National A-cademy of Sciences,1992,89(1):392-396.
[17]Dirks R M,Pierce N A.Triggered amplification by hybridization chain reaction[J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(43):15275-15278.
[18]Zhuang J,Fu L,Xu M,et al.DNAzyme-based magneto controlled electronic switch for picomolar detection of lead (II)coupling with DNA-based hybridization chain reaction[J].Biosensors&Bioelectronics,2013,45(2):52-57.
[19]Yang L,Liu C,Ren W,et al.A graphene surface-anchored fluorescence sensor for sensitive detection of MicroRNA coupled with enzyme-free signal amplification of hybridization chain reaction[J].Acs Applied Materials&Interfaces,2012,4(12):6450-6453.
[20]Tyagi S,Kramer FR.Molecular beacons:probes that fluoresce upon hybridization[J].Nat Biotech,1996,14(3):303-308.
[21]Huang J,Wu Y,Chen Y,et al.Pyrene-excimer probes based on the hybridization chain reaction for the detection of nucleic acids in complex biological fluids[J].Angewandte Chemie International Edition,2011,50(2):401-404.
[22]Gellert M,Lipsett M N,Davies D R.Helix formation by guanylic acid[J].Proceedings of the National Academy of Sciences,1962,48(12):2013-2018.
[23]Špacková N,Berger I,Šponer J.Nanosecond molecular dynamics simulations of parallel and antiparallel guanine quadruplex DNA molecules[J].Journal of the American Chemical Society,1999,121(23):5519-5534.
[24]Dong J,CuiX,Deng Y,et al.Amplified detection of nucleic acid by G-quadruplex based hybridization chain reaction[J].Biosensors&Bioelectronics,2012,38(1):258.
[25]Alexander Trifonov,Etery Sharon,Ran Tel-Vered,et al.Application of the Hybridization Chain Reaction on Electrodes for the Amplified and Parallel Electrochemical Analysis of DNA[J].The Journal of Physical Chemistry,2015,39(3):456-465.
[26]Breaker R R,Joyce G F.A DNA enzyme that cleaves RNA[J].Chemistry&Biology,1994,1(4):221-223.
[27]Purtha W E,Coppins R L,Smalley M K,et al.General Deoxyribozyme-Catalyzed Synthesis of Native 3‘-5‘RNA Linkages[J].Journal of the American Chemical Society,2005,127(38):13124-13125.
[28]Huizenga D E,Szostak JW.A DNA aptamer that binds adenosine and ATP[J].Biochemistry,1995,34(2):656-665.
[29]Schlosser K,Gu J,Sule L,et al.Sequence-function relation ships provide new insight into the cleavage site selectivity of the 8–17 RNA-cleaving deoxyribozyme[J].Nucleic acids research,2008,36(5):1472-1481.
[30]Li Y,Sen D.A catalytic DNA for porphyrin metallation[J].Nature Structural&Molecular Biology,1996,3(9):743-747.
[31]Travascio P,Bennet A J,Wang D Y,et al.A ribozyme and a catalytic DNAwith peroxidase activity:active sites versus cofactor-binding sites[J].Chemistry&biology,1999,6(11):779-787.
[32]Zhuang J,Fu L,Xu M,etal.DNAzyme-based magneto controlled electronic switch for picomolar detection of lead (II)coupling with DNA-based hybridization chain reaction[J].Biosensors&Bioelectronics,2013,45(2):52-57.
[33]Freage L,Trifonov A,Ran TV,etal.Addressing,Amplifying and Switching DNAzyme Functions by Electrochemically Triggered Release ofMetal Ions[J].Chemical Science,2015,6(6):3544-3549.
[34]You K M,Lee SH,Im A,et al.Aptamers as functional nucleic acids:in vitro selection and biotechnological applications[J].Biotechnology and Bioprocess Engineering,2003,8(2):64-75.
[35]LiN,Ho CM.Aptamer-based optical probes with separated molecular recognition and signal transduction modules[J].Journal of the American Chemical Society,2008,130(8):2380-2381.
[36]Wang X,Jiang A,Hou T,etal.Enzyme-free and labelfree fluorescence aptasensing strategy forhighly sensitive detection of protein based on target-triggered hybridization chain reaction amplification[J].Biosensors&Bioelectronics,2015,70(1):324-329.
[37]Song W,Zhu K,Cao Z,et al.Hybridization chain reaction-based aptameric system for the highly selective and sensitive detection of protein[J].Analyst,2012,137(6):1396-1401.
[38]He S,Song B,Li D,et al.A graphene nanoprobe for rapid,sensitive,and multicolor fluorescent DNA analysis[J].Advanced Functional Materials,2010,20(3):453-459.

