用于四种常用品系大鼠鉴定的单核苷酸多态性标记筛查和应用研究
曾玉琳,蒙裕欢,张 钰,杜红丽*
(1.华南理工大学生物科学与工程学院,广州 510006; 2.广东省实验动物监测所,广州 510663)
大鼠具有体型较大、易于研究以及在某些生理特性方面更接近人类等优点,是多种生物医学实验的首选动物模型。其中,Wistar、GK、BN及SD大鼠作为常用的实验大鼠模型广泛应用于多种药物和疾病机制等研究中。Wistar大鼠于1907年由Wistar研究所培育而来,其引进时间较早,遍布范围较广,广泛应用于世界各国实验室中[1];Goto-Kakizaki(GK)大鼠是1975年由日本东北大学的Goto和Kakizaki等[2]从Wistar大鼠中筛选高血糖个体近交繁殖数代而来,具有高血糖、高胰岛素血症、胰岛素抵抗等特性,常用于II型糖尿病的发病机制研究;Brown-Norway(BN)大鼠是Silvers和Billingham于1958年运用此前由野生大鼠培育而成的棕色突变型大鼠近交繁殖而来,其对实验过敏性脑脊髓炎及自身免疫性复合型肾小球肾炎有抗性,可作为致敏等动物模型[3-4];SD大鼠是1925年由美国的Sprague Dawley农场用Wistar大鼠培育而成,其具有较强的适应能力和抗病能力,广泛用于营养学及病理学等研究中[5]。
实验动物的遗传稳定性对实验的准确性和可靠性有着一定影响,因此在实验动物的饲养过程中,对其进行定期的遗传检测是必不可少的环节[6]。生化标记基因检测[7]、免疫标记基因检测[8]以及毛色基因检测[9]等传统的遗传质量检测方法存在着精确度不高以及检测位点少等局限性,这些检测方法已不再满足科学发展的需要[10]。随着基因组学研究的不断进步,SNP标记开始逐渐被应用于各种实验动物的鉴定及遗传检测中,相比于传统的遗传质量检测方法,利用SNP标记具有操作简单快速、能鉴别出同源品系以及能进行大规模高通量检测等显著优点[11]。然而,在国内外的研究中,应用于大鼠遗传检测的SNP标记的研究却并不多。Saar等[12]利用3 × 106个SNPs对一系列大鼠进行了基因型分析,共发掘了20 238个有效的SNPs;Guichoux等[13]通过对33 305个已知的候选SNPs进行验证,结果显示其中66%的SNPs能通用于不同大鼠品系中,并另外发掘了287个新的SNPs;蔡月花等[14]对MIJ和HFJ两个品系的大鼠进行了SNP分析研究,确定了这两个品系大鼠在9个位点的基因型。
目前,筛查到国内外广泛认可的有效遗传标记依然是实现实验动物遗传质量监测技术突破的有途径之一。因此,开发出能用于大鼠遗传检测的SNP标记具有重要意义。本文旨在于利用高通量基因组测序、生物信息技术及一代测序等方法开发出能用于快速鉴定Wistar、GK、BN和SD这四种常用品系大鼠的SNP位点,以补充大鼠遗传质量检测方法,并进一步完善常用品系大鼠等位基因信息。
1 材料和方法
1.1 实验动物
SPF级Wistar大鼠53只,体重170~240 g,1.5月龄;SPF级GK大鼠53只,体重150~220 g,1.5月龄。均购于上海斯莱克实验动物有限责任公司[SCXK (沪) 2017-0005]。SPF级BN大鼠50只,体重70~150 g,1月龄;SPF级SD大鼠50只,体重80~150 g,1月龄。均购于北京维通利华实验动物技术有限公司[SCXK (京) 2016-0011]。以上大鼠均雌雄各半。大鼠饲养在符合《中华人民共和国卫生部实验动物环境设施标准》二级标准的动物房中,恒温室内金属鼠笼,进食标准饲料,可自由饮水,大鼠的鼠尾组织取材坚持实验动物使用的3R原则,于广东省实验动物监测所动物实验设施内进行[SYXK (粤) 2016-0122],伦理审查编号为IACUC2014029。
1.2 主要试剂与仪器
组织基因组DNA提取试剂盒购自天根生化科技有限公司;引物由上海生工生物工程有限公司合成;DNA聚合酶Taq Mix购自TaKaRa宝生物工程有限公司;PCR扩增产物送于华大基因测序。PCR扩增仪和凝胶成像仪购自美国Bio-Rad公司;DNA电泳仪及电泳槽购自北京六一仪器厂;紫外-可见微量分光光度计购自美国赛默飞世尔科技公司;高速离心机购自德国Eppendorf公司。
1.3 实验方法
1.3.1 全基因组重测序
提取3只Wistar大鼠和3只GK大鼠的基因组DNA于华大基因Illumina HiSeq 2500平台进行全基因组重测序。
1.3.2 候选SNP的筛查
首先对测序的原始数据(raw reads)进行截短和过滤得到待分析数据(clean reads),然后使用Bowtie 2软件[15]将clean reads比对到BN大鼠参考基因组(www.ncbi.nlm.nih.gov/genome/?term=Rat)并生成SAM文件,利用Picard软件[16]去除PCR重复,将SAM文件用SAMtools软件[17]转换成BAM文件并进行排序。在Linux系统上,使用SAMtools软件对上述准备好的BAM文件及参考基因组建索引,使用GATK软件[18]的Haplotype Caller工具进行SNP检测并得到相应的VCF文件,最后通过阈值筛选得到最终的VCF文件。
经过整理统计,Wistar大鼠比对参考序列检测出94 800个纯合SNP,GK大鼠比对参考序列检测出106 019个纯合SNP,其中有56 216个SNP为Wistar和GK大鼠所共有,又对这些共有的SNP进行筛查,发现BN大鼠参考基因组、Wistar大鼠和GK大鼠三者基因型互不相同的位点仅有38个,从中随机挑选了15个作为候选SNP位点(如表1所示)进行后续实验。
1.3.3 基因组DNA提取与混合池构建
使用组织基因组DNA提取试剂盒提取大鼠鼠尾的基因组DNA。在每种大鼠的DNA样品中挑10个用于后续单个个体的PCR扩增(分别编号为:BN1~BN10、W1~W10、GK1~GK10、SD1~SD10),而其余40个则用于合成DNA混合池。对于合成DNA混合池的样品,先用紫外分光光度计分别对每个DNA样品的浓度测量3次,取平均值作为样品浓度值,然后用TE缓冲液稀释并定量至50 ng/μL,体积为20 μL,每10个同品系大鼠的DNA混合为一个DNA池(例如Wistar大鼠,将编号为W11~W20的DNA样品混合为W-A,将编号为W21~W30的DNA样品混合为W-B,将编号为W31~W40的DNA样品混合为W-C,将编号为W41~W50的DNA样品混合为W-D),混合好的16个池用于后续DNA混合池实验。
1.3.4 引物设计与PCR反应
提取候选SNP位点上下600 bp序列为模板,使用Primer 6.0分别设计15对引物(如表2所示)。PCR反应程序:94℃预变性5 min,94℃变性30 s,55℃退火30 s,72℃延伸1 min,循环35次后于72℃充分延伸10 min,最后于4℃保存。
1.3.5 单个个体测序及比对
对于每个品系10只大鼠的PCR扩增产物,分别取15 μL进行一代测序,然后利用Lasergene软件包中的SeqMan对测序结果进行整理与比对。根据四个品系大鼠序列的比对结果对SNP位点进行进一步的筛选:①若同品系的10只大鼠在某一SNP位点的基因型不一致,则不采用该SNP;②若GK大鼠和Wistar大鼠在某一SNP位点的基因型与表1不一致,则不采用该SNP;③对于候选SNP位点上下游序列,若出现能鉴别出某种大鼠的SNP位点,则挑选采用。
注:“AA”、“GG”、“TT”、“CC”为基因型。
Note. “AA”, “GG”, “TT”, and “CC” refer to genotypes.
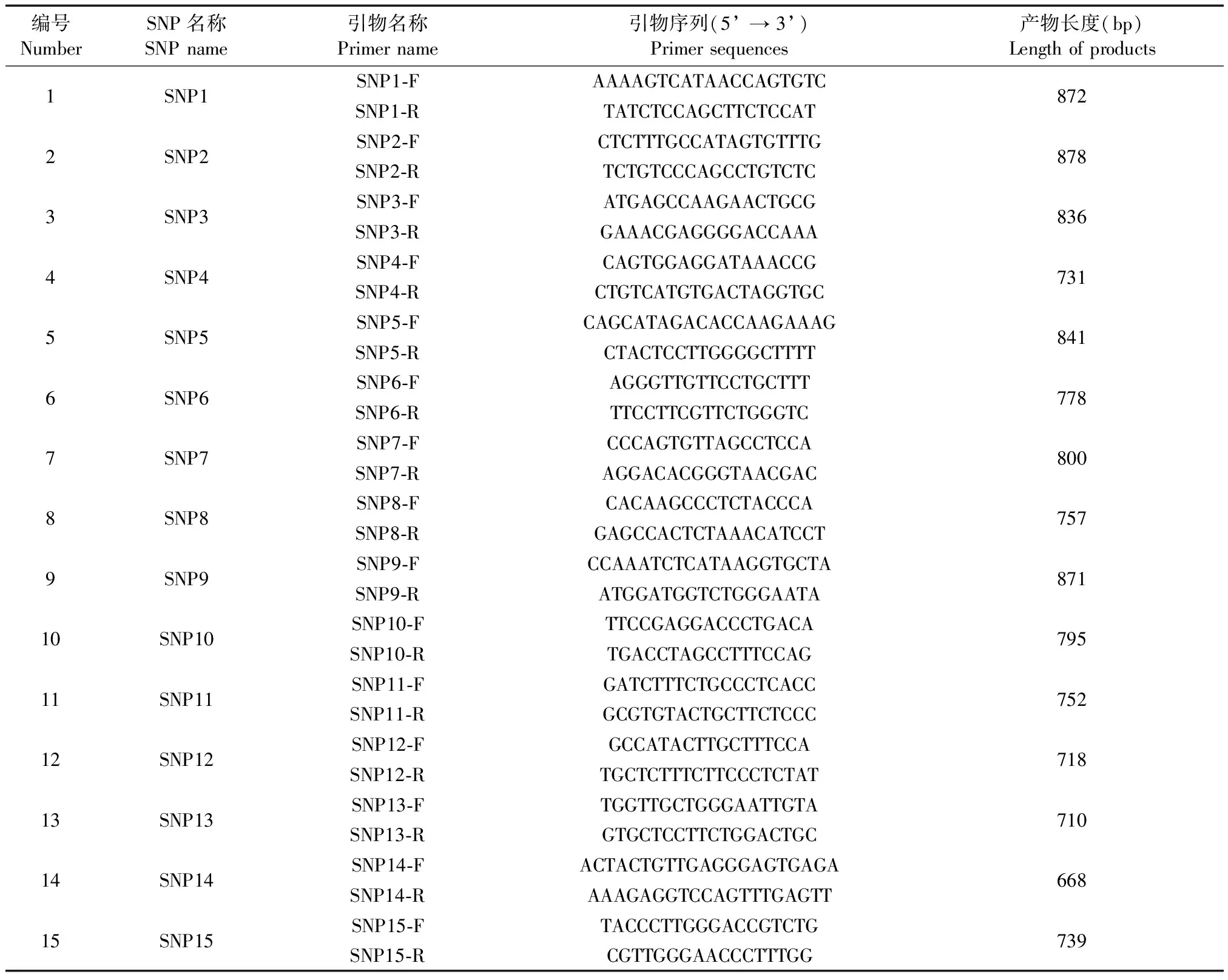
表2 候选SNP位点对应的引物信息
1.3.6 DNA混合池实验
对最终筛选的位点进行扩大样本的DNA混合池实验,以上述准备好的16个混合池为模板进行PCR扩增目的片段,反应体系与反应程序同上。对PCR产物进行测序,利用SeqMan对序列结果进行整理与比对,并查看测序情况。
2 结果
2.1 扩增结果
以W1~W10,GK1~GK10,BN1~BN10和SD1~SD10为模板,用设计的15对引物扩增目的片段。结果显示,第7对和第11对引物扩增失败,其余产物均扩增成功且可用于测序。
2.2 单个个体测序及比对结果
扩增成功的产物测序结果显示,满足上述筛选要求的位点有SNP4、SNP5、SNP6、SNP9、SNP12、SNP13、SNP14。除此之外,在这些序列中还发掘了其他可以用于快速鉴定的6个位点,分别命名为SNP16、SNP17、SNP18、SNP19、SNP20、SNP21。这13个位点在四种大鼠中的等位基因型如表3所示。
2.3 DNA混合池实验结果
对于上述13个SNP位点,DNA混合池的PCR产物测序结果显示,除SNP13和SNP16在SD大鼠中出现套峰外(如图1所示),其他位点均未出现套峰。SNP13在SD大鼠单个个体测序结果中10个个体都是杂合子,因此在混合池测序时出现套峰属于正常现象,而SNP16在SD大鼠中出现轻微G峰,说明SD大鼠群体中存在少数AG杂合子或GG纯合子的个体。
3 讨论
本研究首先利用高通量基因组测序和生物信息技术筛选了一组候选SNP位点,并继续扩大样本量以及增加大鼠种类进行测序分型,利用了一代测序对四种大鼠(Wistar、GK、BN、SD大鼠)的候选SNP位点进行进一步筛查与验证,最终得到了13个SNP位点以用于四种大鼠的快速鉴定,最后利用DNA混合池法验证了这些位点在群体中的稳定性和用于鉴定的可靠性。

表3 13个SNP位点在四种大鼠中的基因型
注:“AA”、“GG”、“TT”、“CC”为基因型;“—”为碱基缺失。
Note. “AA”, “GG”, “TT”, and “CC” refer to genotypes; “—”refers to base deletion.
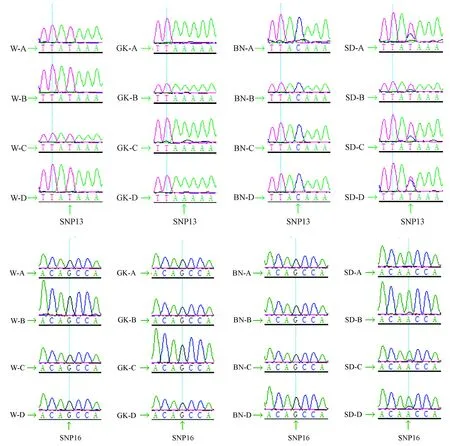
注:“W”、“GK”、“BN”、“SD”分别代表Wistar大鼠、GK大鼠、BN大鼠和SD大鼠;A~D分别代表四个DNA混合池。图1 混合池扩增产物的测序峰图Note. “W”, “GK”, “BN”, and “SD” refer to Wistar, GK, BN, and SD rats, respectively; A-D refer to the four DNA pools, respectively.Figure 1 Sequencing peak maps of DNA pooling amplification products
从这13个SNP位点在四种大鼠中的基因型可以发现,SNP13可鉴别出Wistar、GK、BN和SD四种大鼠,SNP16和SNP17可鉴别出SD大鼠,SNP18和SNP19可鉴别出GK大鼠,SNP20和SNP21可鉴别出BN大鼠,而SNP4、SNP5、SNP6、SNP9、SNP12和SNP14虽然可鉴别出GK和BN大鼠,但区分不出Wistar和SD大鼠,主要是Wistar大鼠和SD大鼠这两个品系亲缘关系较近的缘故,因此可以利用多个位点配合进行鉴别,例如,SNP4和SNP17结合也可快速鉴别出四种大鼠。此外,增加检测的SNP位点个数会大大提高鉴别的准确性和可靠性。
混合池实验显示,SNP13和SNP16在SD大鼠中出现了套峰。SNP13在SD大鼠单个个体测序结果中10个个体都是杂合子,而SNP16在SD大鼠中出现轻微G峰,说明在SD大鼠群体中存在少数AG杂合子或GG纯合子的个体,这些现象是由于SD大鼠作为封闭群在这些位点保持着一定的杂合性。鉴于以上结果,SNP13和SNP16还可以作为SD大鼠封闭群遗传评估的SNP标记,其他未出现套峰的位点说明了在四个大鼠群体中已趋于稳定,用于鉴定物种具有可靠性。
实验动物的遗传质量是衡量其品质好坏的重要因素,使用遗传特性稳定的实验动物才能保证实验结果更加具有可靠性和可重复性[19]。在生物医学研究中,使用具有明确遗传背景的实验动物是非常重要的[20],因此,筛查到能监测实验动物遗传背景的标记至关重要。SNP是基因组中最为丰富且最为常见的可遗传变异,SNP标记也将成为用于大鼠遗传质量监测的理想遗传标记[21]。目前应用于大鼠遗传检测的SNP标记的研究较少,对其展开研究并筛选能快速鉴定四种常见品系大鼠的SNP位点,以补充大鼠遗传质量检测手段,具有进一步丰富大鼠SNP数据库的重要意义。

