抑制剂RVX08 、SF2535、RVXOH与BRD4蛋白第一结构域结合模式的分子动力学研究
(山东师范大学物理与电子科学学院,250358,济南)
1 引 言
表观遗传学与人类疾病之间关系非常密切,具有遗传特性.表观遗传学是一种基因的表达调控机制,存在于多细胞生物体生长发育的始终.组蛋白乙酰化是研究表观遗传学的重要内容,它同时涉及到翻译后修饰的重要途径[1].组蛋白赖氨酸乙酰化的识别是组蛋白乙酰化能够在表观遗传调控中发挥作用的重要因素[2].Bromodomain (BRD)是一种能够通过特异性识别组蛋白乙酰化赖氨酸的保守蛋白结构域,可以参与到各种表观遗传调控,比如,激活转录因子和染色质重塑因子等[3].BRD蛋白不仅可以分开单独存在,还与其他组蛋白结构域通过协同模式调节基因转录、细胞周期和细胞凋亡[4].
迄今为止,在人类的42种蛋白质中识别了61种BRD结构域.依据BRD蛋白的序列和功能差异,将BRD结构域蛋白划分为8个家族.其中BRD家族的第二类为BET(Bromodomain and extraterminal domain)蛋白.该类BRD主要由BRD2、BRD3、BRD4和BRDT[5]构成.其中,BRD4蛋白最受关注.BET蛋白可以与乙酰赖氨酸结合,从而使染色质重塑因子、转录因子和其他蛋白质通过改变聚合酶II的活性来调节基因转录表达[3].BET蛋白在细胞恶性转化和肿瘤细胞的侵袭和转移过程起重要作用.在这种功能的作用下,BRD4可以调节细胞周期内不同组织的细胞分化[6,7].研究表明存在功能障碍的BRD4可能会使基因表达产生紊乱,所以,它在肿瘤的发生和发展过程中起着至关重要的作用[6,8].抑制BRD4的活性可以有效的治疗癌症、炎症.因此开发以BRD4为靶标的高效抑制剂已成为当前研究和治疗癌症的焦点.
1990年,通过磁共振成像技术测定出了最早的BRD结构[9,10].后来又通过晶体衍射技术测定了40种BRD结构[11],为以BRD4为靶标抑制剂的设计提供了结构基础.BRD4的结构域包括BRD4-1和BRD4-2,它们在对组蛋白和其他蛋白中赖氨酸乙酰化的鉴定中发挥重要作用.在结构方面,BRD具有高度保守性,它包括4个反向平行的α螺旋(αZ、αA、αB和αC)、连接α螺旋的疏水环结构(ZA-Loop连接αZ和αA,BC-Loop连接αB和αC).如图1(A)显示的是疏水口袋,形成在螺旋束末端赖氨酸乙酰化的识别位点.(B)、(C)、(D)为本次研究的三个小分子抑制剂的结构图.
据报道,小分子抑制剂与BRD结合时表现出很高的选择性,而且对其他基因的影响较小.通常BET小分子抑制剂包括三氮唑类、异恶唑类、嘧啶类和吡啶酮类、乙酰基吡咯类四类[12,13].这四类小分子抑制剂能在不同层次上抑制BRD活性.

图1 分子结构(A)以表面模式显示的抑制剂与BRD4-1的结合位点;(B)RVX08 ;(C)SF2535;(D)RVXOH.
近几年,运用分子动力学模拟和结合自由能预测等方法对BRD4结构域的小分子抑制剂进行了广泛的研究,如类黄酮化合物[14]、(+)-JQ1[15]、双重抑制剂EGFR-BRD4[16]等.之前的研究还表明,蛋白质的构象变化在药物设计中起着重要的作用.虽然已经揭示了抑制剂与BRD结构域的结合机制,但是仍然缺乏BRD结构域构象变化细节.故进一步研究抑制剂的结合机制和原子水平的BRD结构域的构象变化对于设计新的、有效的、针对BRD结构域的抑制剂具有重要意义.当前的研究采用分子动力学模拟(MD)[17]、结合自由能计算[18]和主成分分析(PC)探索抑制剂与蛋白的结合机制和蛋白质构象变化[19].
2 理论和方法
2.1系统准备首先,从蛋白质数据库(PDB)中获得BRD4-1与三种抑制剂RVX08 、SF2535和RVXOH的PDB文件,通过它们的晶体结构获得初始坐标.晶体结构中的所有结晶水分子都保留在初始模型中,使用Amber16中的Leap模块将所有缺失的氢原子加到晶体结构中.然后,在中性环境下,运用Propka程序来检查残基的质子化状态[20,21],运用半经验的AM1方法计算了三种抑制剂的原子电荷[22],蛋白质和水分子用FF99SB力场来描述[23],运用GAFF力场产生三个抑制剂的力场参数[24].采用截断的、TIP3P水分子的八面体盒子将每个体系都溶解,每个维度有12.0 Å的缓冲[25].最后为维持各个体系的电中性,在每个体系中加入一定量的氯离子.
2.2分子动力学模拟对于每个系统,应用Amber16中的Pmemd模块执行能量最小化和分子动力学模拟过程.为去除蛋白和配体之间的不良接触,每个系统通过两个阶段的最小化过程来产生驰豫的构象.首先,在复合物上施加强度为100 kcal/mol·Å-2的约束,以便优化水分子和中和离子.其次,移去约束,优化整个体系.在优化过程中,对每个系统分别进行了2 500步的最陡下降优化和2 500步的共轭梯度优化.接着,保持体积不变,将每一个系统在1 ns时间内从0 K升温至300 K,然后在300 K的恒温条件下执行 1 ns的动力学平衡.最后,利用系统的周期性边界条件,在恒温300 K、恒压1 atm的情况下,对系统执行120 ns无约束的分子动力学模拟.同时,每隔2 ps记录一次原子坐标.在整个MD模拟过程中,所有涉及氢原子的共价键均用Shake算法约束[26],时间步长设为2 fs.系统的温度由Langevin动力学进行控制[27],碰撞频率为2.0 ps-1.采用PME方法处理长程静电相互作用[28].同时,采用9 Å的截断距离计算非成键的范德华相互作用和静电相互作用.
2.3结合自由能的计算本工作采用MM-PBSA方法计算抑制剂与BRD4-1的结合自由能.对于每个复合系统 ,从最后60 ns的MD轨迹中,以300 ps的间隔提取200个构象用以计算结合自由能.在MM-PBSA方法中结合自由能(ΔG)可用下列方程表示:
G=ΔEele+ΔEvdw+ΔGpol+ΔGnonpol-TΔS.
(1)
其中,ΔEele和ΔEvdw分别为静电相互作用能和范德华相互作用能,通常用分子力学来计算;ΔGpol为极性溶剂化自由能,通过求解泊松-玻尔兹曼方程可以得到;ΔGnonpol非极性溶剂化自由能,可通如下经验方程计算:
ΔGnonpol=γ×SASA+β.
(2)
其中,γ和β的值分别为0.005 42 kcal/molÅ-2和0.92 kcal/mol.原子半径采用prmtop文件中缺省半径参数,溶质和溶剂的介电常数分别设为1.0和80.0.对于最后一项,熵(-TΔS)是用经典统计热力学和Normal Mode分析来计算的.
2.4主成分分析交叉相关分析(cross-correlation analysis)是研究蛋白质构象变化的重要工具,基于分子动力学模拟轨迹的相关分析可以探测出原子之间的相关运动.描述 Cα原子相对于平衡位置涨落的相关矩阵元Cij可由下列公式确定[44]:
(3)
其中,原子偏离平衡位置的位移用Δri表示,式中尖括号代表对MD轨迹上的系综平均.在这个公式中,Cij值的变化范围是-1到1.值为正表示残基i和j之间是正相关运动,反之,负相关运动则用负值表示.
本文采用Cpptraj模块对基于原子坐标构造的位置协方差矩阵C执行主成分分析块[29,30].通过协方差矩阵C可由如下方程确定:
Ci=<(qi-
(4)
式中,第i个Cα原子的笛卡尔坐标用qi表示,Cα原子的总数为N.通过移去整体的平移和旋转,并采用最小二乘拟合程序计算平均值[31].计算中利用正交坐标变换矩阵T对矩阵C对角化获得本征值和本征矢量,
Λ=TTCijT.
(5)
其中,列表示相对于运动方向的本征矢量.此外,每个本征矢量与一个本征值相关联.本征值通常用以描述系统沿着本征矢量的运动强度.
3 结果与讨论
3.1 MD模拟的稳定性为评估模拟平衡的可靠性,采用Amber中的Cpptraj模块计算了主链原子相对于最小结构的均方根偏差(RMSD), 且计算结果如图2所示.系统模拟60 ns后,四个系统均达到平衡状态.三个复合物RVX08 /BRD4-1、SF2535/BRD4-1和RVXOH/BRD4-1的RMSD的平均值分别为1.29 Å、1.06 Å和1.50 Å,而未束缚的BRD4-1蛋白的平均RMSD值为1.82 Å.该结果不仅表明MD模拟的平衡是可靠的, 而且也证明抑制剂结合抑制了BRD4-1中某些区域的运动,有利于复合物结构的稳定性.

图2 BRD4-1主链原子的均方根偏差(RMSD)随模拟时间的变化
我们也采用Cpptraj程序计算了描述原子涨落的相关矩阵,以便进一步研究抑制剂结合对蛋白质BRD4-1内部动力学的影响,结果如图3所示.从图3可以看出,抑制剂结合对BRD4-1的运动模式有明显的影响.
对于apo BRD4-1(图3(A)),在R2、R3和R4区域可以观察到红色或黄色所指示的相关运动.此外,在R1区域出现反相关运动.与apo BRD4-1相比,三个抑制剂的结合诱导了BRD4-1运动模式的明显变化.就RVX08 (图3(B))来说,RVX08 结合不仅略微削弱了R2、R3和R4区域的相关运动,而且增强了R1区域的反相关运动.对于抑制剂SF2535(图3(C)),该抑制剂在结合位点的出现导致了大部分区域的反相关运动明显增强,且R2、R3和R4区域的相关运动被削弱.如图3(D)所示,RVXOH的结合不仅削弱了R2、R3和R4区域的相关运动, 而且增强了R1区域的反相关运动.上述相关分析表明,抑制剂结构的变化对蛋白质BRD4-1的内部动力学行为产生明显影响.

图3 MD模拟平衡后Cα原子在其平衡位置附近涨落的相关矩阵(A)apo BRD4-1;(B)RVX08 /BRD4-1;(C)SF2535/BRD4-1;(D)RVXOH/BRD4-1.
3.2主成分分析为更详细地研究抑制剂结合对BRD4-1运动模式的影响,我们对MD轨迹进行了PCA分析.图4描绘了基于原子坐标构造的协方差矩阵对角化后得到的特征值与特征向量指标间的关系.结果表明,apo BRD4-1与RVX08 /, SF2535/, RVXOH/BRD4-1复合物的前6个主成分分别占MD轨道最后60 ns观测到的总运动的72.82 %、67.70 %、53.80 %和57.78%.Apo BRD4-1的特征值远高于抑制剂结合的BRD4-1,说明抑制剂结合明显抑制了BRD4-1的运动强度.与RVX08 /BRD4-1和RVXOH/BRD4-1复合物相比, SF2535/BRD4-1结合对BRD4-1的运动强度产生最强的抑制.
3.3抑制剂与BRD4-1结合能力和结合模式为能从能量的角度理解BRD4-1抑制剂的结合强度,我们使用MM-PBSA方法和基于MD轨迹最后60 ns以间隔300 ps提取的200个快照计算了抑制剂与BRD4-1的结合自由能(见表1).表1显示了当前预测的三个抑制剂结合自由能与实验值的排序相一致.因此,该结果表明我们目前对结合自由能的分析是可靠的. 表1也表明RVX08 、SF2535和RVXOH与BRD4-1结合的结合自由能分别为-7.55、-10.78和-5.14 kcal/mol, 其中SF2535与BRD4-1结合时的结合能力最强.
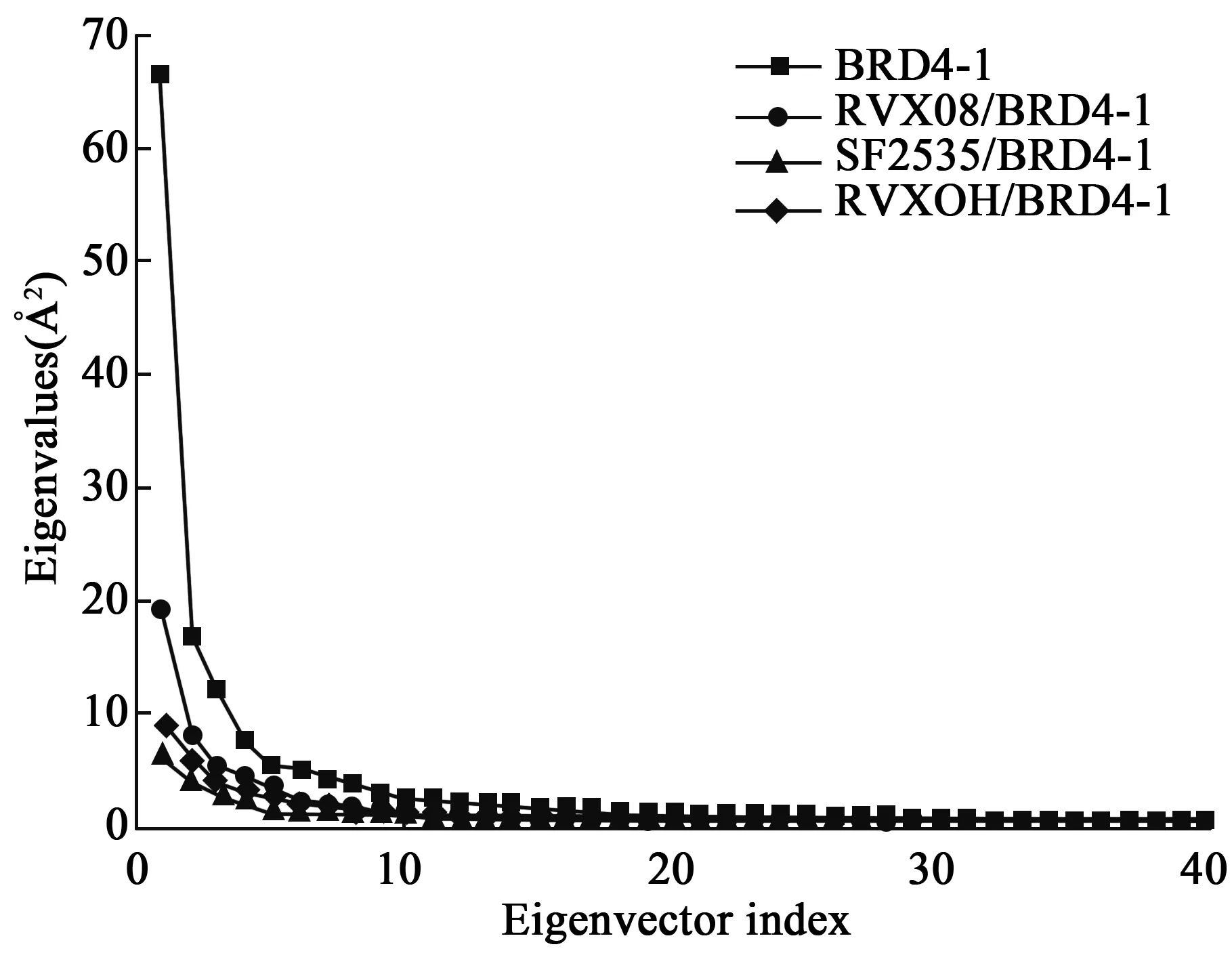
图4 特征值与从MD模拟平衡阶段构造的Cα得到的特征向量指数的比较
在表1中可以看出结合自由能分离的成分对抑制剂结合的贡献.有利于抑制剂结合的成分是分子间静电相互作用(ΔEele)、范德华相互作用能(ΔEvdw)和非极性溶剂化自由能(ΔGnopol),这些成分增强了抑制剂与BRD4-1的结合.极性溶剂化自由能(ΔGpol)和熵效应(-TΔS)削弱了抑制剂结合.虽然抑制剂与BRD4-1的静电相互作用有利于与抑制剂的结合,但这种有利的作用力完全被极性溶剂化能所抵消,从而产生了对抑制剂与BRD4-1结合的不利作用力(ΔGele+pol).

表1 用MM-PBSAa方法计算的抑制剂与BRD4-1的结合自由能
注:a除IC50外所有值的单位为kcal/mol,bΔGele+pol=ΔEele+ΔGpol,cΔGbind=ΔEele+ΔEvdw+ΔGpol+ΔGnonpol-TΔS,d结合自由能实验值是用公式ΔGexp=-TRlnIC50导出来的,IC50为参考文献中的实验值,单位为nm.
3.4分离残基对抑制剂结合自由能的贡献抑制剂-残基相互作用谱(图5)显示了将结合自由能分解为不同残基的贡献.同时利用Amber中Cpptraj程序对氢键进行了分析,结果见表2.如图5(A)所示,五个残基L94、C136、X139、N140和I146与抑制剂产生较强的相互作用,它们的能量贡献分别为-1.22、-1.0、-1.46、-2.15和-2.24 kcal/mol.它们主要是通过疏水作用和氢键作用与抑制剂结合.其中疏水作用主要来源于残基L94、C136、X139和I146,而氢键作用主要来自N140与RVX08形成的两个氢键,其占有率分别为48.07 %和46.21 %.如图5(B)所示,七个残基在SF2535与BRD4-1结合时表现出更强的相互作用,包括W81、P82、Q85、V87、L92、L94和I146.它们的相互作用能分别为-1.21、-1.82、-1.6、-1.66、-2.78、-1.21和-2.31 kcal/mol,这些能量贡献主要来自于抑制剂与蛋白之间的疏水相互作用.如图5(C)所示,RVXOH与BRD4-1结合时,其中有五个残基通过疏水作用对其贡献更大.这些残基包括W81、P82、V87、L92和I146, 相互作用能分别为-1.0、-1.0、-1.28、-2.14和-1.66 kcal/mol.

图5 三个抑制剂与BRD4-1单独残基的相互作用:(A)RVX08 /BRD4-1,(B)SF2535/BRD4-1,(C)RVXOH/BRD4-1.

复合物a 氢键键长 (Å)角度 (°)b占有率 (%)RVX08/BRD4-1N140-ND2-HD21…RVX08 -O53.09136.8548.07RVX08/BRD4-1N140-ND2-HD21…RVX08 -N13.22160.9046.21
注:a氢键是由受体…供体原子距离小于3.5Å 和受体…H-供体键角大于120°决定的,b利用占有率来评价氢键的稳定性和强度.
从上述分析可知,调控RVX08 、SF2535、RVXOH与BRD4-1结合的主要力量是抑制剂关键残基的疏水相互作用.此外,氢键相互作用在抑制剂结合中也起着比较重要作用,特别是在RVX08 /BRD4-1复合物中形成的两个氢键增强了它们的稳定性.因此,在针对BRD4-1的高效抑制剂的开发中,范德华相互作用和氢键相互作用将是重要的影响因素,应引起一定的重视.
4 结 语
本工作对BRD4-1和三个抑制剂RVX08 、SF2535和RVXOH复合体执行了MD模拟,探讨了抑制剂结合对BRD4-1构象变化的影响.结果表明,抑制剂结合增强了BRD4-1的结构稳定性.同时源自相关运动和主成分分析的结果表明抑制剂的作用对BRD4-1的运动模式有着重要影响.用MM-PBSA方法计算的结合自由能证明了范德华相互作用在抑制剂结合中起着重要作用.本文计算了抑制剂与残基的相互作用,其结果表明关键残基与抑制剂的疏水相互作用和氢键相互作用对抑制剂与蛋白BRD4-1的结合有重要贡献.此外,我们期望本文的研究结果可以为以BRD4-1为靶标的高效药物的设计提供理论辅助.

