黄麻核心种质的遴选
徐 益 张列梅 郭艳春 祁建民 张力岚 方平平 张立武,*
黄麻核心种质的遴选
徐 益1,2,3,**张列梅1,2,**郭艳春1,2,3祁建民1,2张力岚1,2,3方平平1,2张立武1,2,3,*
1福建农林大学作物遗传育种与综合利用教育部重点实验室 / 福建省作物设计育种重点实验室 / 作物科学学院, 福建福州 350002;2福建农林大学农业农村部东南黄红麻实验观测站 / 福建省麻类种质资源共享平台 / 福建省南方经济作物遗传育种与多用途开发国际科技合作基地, 福建福州 350002;3福建农林大学海峡联合研究院基因组与生物技术中心, 福建福州 350002
遴选黄麻核心种质可为黄麻种质创新及新品种选育奠定基础。本研究以300份黄麻种质资源为基础, 基于SSR分子标记和农艺性状考察, 结合地理来源构建核心种质。结果表明, 11个农艺性状变异系数变幅在13.06%~ 84.87%, 表现出丰富的遗传多样性。按农艺性状聚类分析可划分为8个类群, 按分子标记聚类可划分为10个类群。结合2个聚类分析、地理位置并按比例取样, 建立一个由108份品种(系)组成的预选核心种质。采用44对SSR引物对其进行遗传差异分析, 在遗传相似系数为0.65时, 可把108份品种(系)分成圆果种和长果种两大类。根据遗传差异分析, 剔除遗传相似系数大于或等于0.85的遗传冗余, 获得84份品种(系)的核心种质, 其中圆果种60份和长果种24份。比较84份核心种质与300份种质的农艺性状变异系数及Shannon-Wiener指数发现, 两者之间相差不大, 表明遴选的84份核心种质可以最大限度代表300份黄麻种质资源的遗传多样性加以利用和保存。
黄麻; 农艺性状; 分子标记; 核心种质
黄麻是世界上重要的次生韧皮部纤维作物, 包括圆果种黄麻(L.)和长果种黄麻(L.) 2个栽培种, 染色体数均为2= 14。主要在印度、孟加拉国、中国、尼泊尔、泰国、缅甸等国家种植, 由于黄麻纤维具有典型的高光泽、吸湿性能好、失水快、强力大、易降解等特点, 被广泛应用于纺织、造纸和吸附材料等领域[1-2]。
种质资源收集、保存, 进而鉴定评价, 对所有物种都是一项具有重要现实意义的工作[3]。目前, 全世界已有各类植物遗传资源610多万份。如此巨大的种质资源数量, 并没有加速作物育种的进展, 反而给育种家们利用、研究和评价种质资源带来许多不便, 导致许多国家的育种工作发展缓慢甚至停滞不前[4-5]。面对这一问题, 1984年澳大利亚科学家Frankel提出了“核心资源”的概念[6], 随后各国各领域的学者对核心种质的基本特征、构建核心种质的步骤和方法进一步完善, 已经在作物领域取得广泛成就, 使许多作物遗传多样性研究取得显著成就和突破性成果[7-8]。贾继增等[4]提出核心种质的遴选是“第二次绿色革命”基因挖掘的第一步。
黄麻的遗传多样性和亲缘关系研究报道较多[9-10]。陶爱芬等[9]利用SRAP结合ISSR分子标记对来自13个不同国家的96份黄麻种质资源进行起源与演化研究, 认为野生长果种黄麻及栽培长果种黄麻的起源中心均在非洲; 中国南部地区是栽培圆果种黄麻的起源中心。张加强等[10]以引进或育成的20个长果种和40个圆果种黄麻品种为供试材料, 采用亲缘系数与聚类分析的方法, 进行亲缘关系和遗传多样性分析。但在黄麻核心种质遴选方面鲜有报道, 核心种质是以最小的资源数量和遗传重复最大程度地代表整个遗传资源的多样性, 它的建立既保证了遗传多样性, 又减少了资源数量。随着黄麻种质资源的不断引进、收集和新品种的选育, 庞大的种质资源数量不但对种质资源的保存是一个挑战, 而且对种质资源的利用评价和研究也带来诸多不便。构建一批具有多样性和代表性的黄麻核心种质资源加以保存和利用, 是目前黄麻育种上亟须解决的问题。本研究旨在遴选黄麻核心种质, 促进黄麻种质资源保存和利用工作由数量保存型向质量创新型转变, 为种质创新及新品种选育奠定基础。
1 材料与方法
1.1 供试材料
来自印度、孟加拉国、中国等11个国家或地区的300份黄麻种质资源[11]均由福建农林大学麻类作物遗传育种与综合利用实验室提供。
1.2 试验设计
不同来源的300份黄麻种质资源分别于2016年5月1日和2017年4月20日在福建省三明市尤溪县福建农林大学洋中科教基地农场按2行区种植, 行长3.5 m, 行株距1.20 m × 0.10 m, 采用单因素随机区组设计, 边行种植2行保护行, 田间管理同大田。
1.3 标记来源
所用的SSR标记包括编号为CcID[11]、CcSSR[12]等引物(表1), 由生工生物工程(上海)股份有限公司合成。

表1 本研究所用的SSR引物

(续表1)
1.4 PCR体系及电泳检测
PCR体系为10 μL, 含2.0 μL DNA (50 ng μL–1)、0.5 μL引物(10 μmol μL–1)、3.75 μL 2×Master Mix (试剂购自上海进岸生物科技有限公司)、3.75 μL ddH2O。用10 μL石蜡油覆盖。PCR程序为94℃预变性3 min; 94℃变性30 s, 60℃退火30 s, 72℃延伸45 s, 共10个循环, 每个循环退火温度降低0.5℃; 94℃变性30 s, 55℃退火30 s, 72℃延伸45 s, 共35个循环; 最后72℃延伸10 min, 4℃保存。采用9%非变性聚丙烯酰胺凝胶电泳检测PCR扩增产物, 银染显带[12]。
1.5 农艺性状考察
参考《黄麻种质资源描述规范和数据标准》[13], 黄麻工艺成熟时, 从小区随机选取10株, 考察株高、分枝高、茎粗、皮厚、单株鲜茎重、单株鲜皮重、节数、分枝数等性状, 将单株鲜皮重自然晾干后称量得单株干皮重。
1.6 读带与数据统计分析
电泳结果的读带采取0/1方式, 0表示无带, 1表示有带。利用NTSYS v2.10软件对种质资源进行聚类分析。
1.7 核心种质挑选的原则与方法
11个农艺性状分别按从高到低将300份种质资源按7个等级进行划分, 根据农艺性状聚类图挑选核心种质, 从每个类群挑选若干种质, 确保每个农艺性状的每个等级都有种质被挑选, 将挑选出来的核心种质对应到分子标记聚类图, 继续挑选核心种质, 确保分子标记聚类图中每个类群都有种质被挑选。在以上挑选过程中, 尽量挑选来源地不同的种质。
2 结果与分析
2.1 黄麻种质资源农艺性状的遗传多样性分析
以株高、分枝高、茎粗、鲜皮厚、节数、分枝数、单株鲜茎重、单株鲜皮重、单株干皮重、始花天数、种子成熟天数等农艺性状对288份黄麻种质资源进行农艺性状聚类(由于有12份材料长出的单株较少, 故未考种)。以欧式距离0.036为分界线, 可将288份黄麻种质资源分为8个类群。第1个类群含34份种质资源, 其主要特征是开花期和成熟期均较晚; 第2个类群含21份种质资源, 其主要特征是开花期适中但成熟期较晚; 第3个类群含68份种质资源, 其主要特征是株高适中且分枝数较少; 第4个类群含57份种质资源, 其主要特征是株高和分枝高均较低; 第5个类群含45份种质资源, 其主要特征是开花期和成熟期均较早; 第6个类群含29份种质资源, 其主要特征是株高较高且鲜皮厚较厚; 第7个类群含31份种质资源, 其主要特征是分枝高较高但节数却适中; 第8个类群含3份种质资源, 其主要特征是单株鲜茎重较大。
2.2 SSR聚类分析
利用44对SSR引物对300份黄麻种质资源进行聚类分析, 得到300份黄麻种质资源的遗传相似系数。从整个黄麻群体结构分析中, 300份供试黄麻种质资源总共可以组成44,850种组合, 其遗传相似系数变化范围在0.105~1.000之间, 遗传相似系数总共为28,305.110, 平均值为0.631, 说明供试的300份黄麻种质资源有丰富的遗传多样性。以遗传相似系数0.674为分界线, 可将300份黄麻种质资源分为10个类群, 其中有3个类群为长果种黄麻, 其余7个类群为圆果种。第1个类群含140份圆果种黄麻, 其遗传相似系数变化范围在0.424~1.000之间, 遗传相似系数总共为7355.212, 平均值为0.756; 第2个类群含17份长果种黄麻, 遗传相似系数变化范围在0.697~ 0.970之间, 遗传相似系数总共为112.727, 平均值为0.829; 第3个类群含12份长果种黄麻, 遗传相似系数变化范围在0.737~1.000之间, 遗传相似系数总共为60.105, 平均值为0.835; 第4个类群含2份圆果种黄麻, 其遗传相似系数为0.788; 第5个类群含19份圆果种黄麻, 遗传相似系数变化范围在0.485~1.000之间, 遗传相似系数总共为125.242, 平均值为0.732;第6个类群含32份圆果种黄麻, 遗传相似系数变化范围在0.364~0.970之间, 遗传相似系数总共为365.818, 平均值为0.738; 第7个类群含23份长果种黄麻, 遗传相似系数变化范围在0.515~0.970之间, 遗传相似系数总共为201.606, 平均值为0.797; 第8个类群含10份圆果种黄麻, 遗传相似系数变化范围在0.606~1.000之间, 遗传相似系数总共为33.909, 平均值为0.754; 第9个类群含30份圆果种黄麻, 遗传相似系数变化范围在0.394~0.970之间, 遗传相似系数总共为324.608, 平均值为0.746; 第10个类群含15份圆果种黄麻遗传相似系数变化范围在0.576~1.000之间, 遗传相似系数总共为81.910, 平均值为0.780。
2.3 核心种质的聚类分析
根据农艺性状聚类图和分子标记聚类图的聚类结果, 分别在2个聚类图的每个类群挑选不同品系和不同来源的若干个种质资源构成预选黄麻核心种质, 共选出108份黄麻种质资源(图1)。剔除遗传相似系数大于等于0.85的遗传冗余, 得到84份黄麻核心种质资源(图2和附表1), 遗传相似系数变化范围在0.211~0.970之间, 遗传相似系数总和为2181.78, 平均值为0.626, 84份黄麻核心种质在分子标记聚类图中可划分为8个类群, 其中第1个类群含35份圆果种黄麻, 第2个类群含9份长果种黄麻, 第3个类群含12份圆果种黄麻, 第4个类群含12份长果种黄麻, 第5个类群含2份长果种黄麻, 第6个类群含3份圆果种黄麻, 第7个类群含4份圆果种黄麻, 第8个类群含7份圆果种黄麻。

图1 108份预选黄麻核心种质聚类分析
2.4 核心种质与所有种质资源的多样性比较
从表2可以看出, 288份黄麻种质资源群体各性状的变异系数为13.06%~84.87%, 最大的是分枝数, 最小的是始花期, 表现出丰富的遗传多样性。而84份黄麻核心种质资源各性状的变异系数为13.27%~ 90.42%, 最大的是节数, 最小的是始花期。与288份种质资源相比, 各性状的变异系数变化均不大。因此, 遴选出的84份黄麻核心种质可以最大限度代表288份黄麻种质资源的遗传多样性。
与300份黄麻分子标记聚类图相比, 最终得到的84份黄麻核心种质中(图2), 有34份来自第1个类群, 8份来自第2个类群, 2份来自第3个类群, 1份来自第4个类群, 4份来自第5个类群, 10份来自第6个类群, 12份来自第7个类群, 3份来自第8个类群, 7份来自第9个类群, 3份来自第10个类群。300份黄麻种质资源和84份黄麻核心种质的Shannon-Wiener指数分别为-2.574,192,203、-2.481,606,678, 两者相差较小, 再次验证84份黄麻核心种质可以代表300份黄麻种质资源。
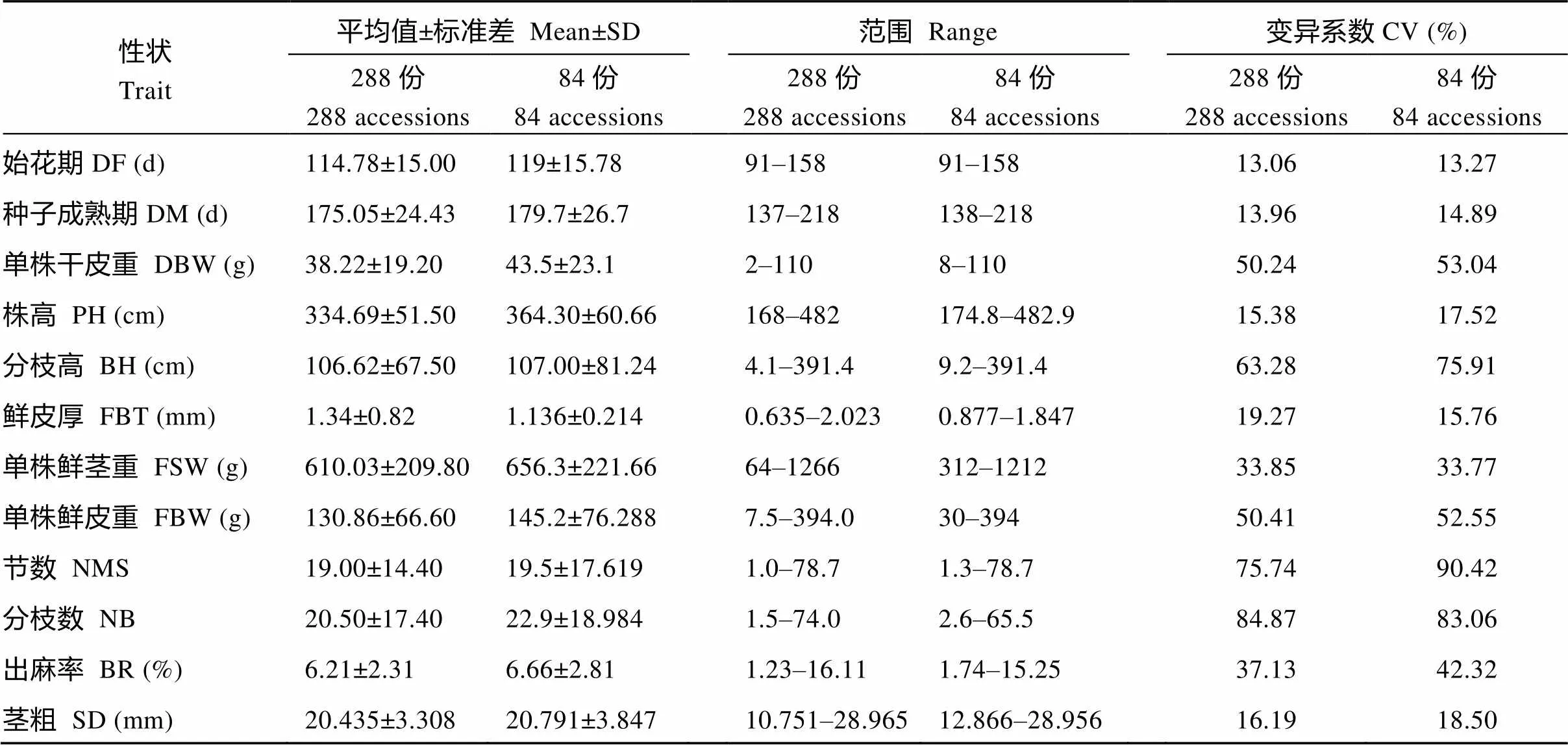
表2 黄麻主要农艺性状统计分析
DF: days to flowering; DM: days to seeds mature; PH: plant height; SD: stem diameter; FBT: fresh bark thickness; NMS: nodes of main stem; FSW: fresh stem weight per plant; FBW: fresh bark weight per plant; DBW: dry bark weight per plant; BR: bark rate; BH: branching height; NB: number of branches.
3 讨论
利用分子标记对种质资源进行分类鉴定在诸多领域已经完善成熟[14-15], 合适的分子标记直接关系到能否准确评价物种遗传多样性和亲缘关系等[3]。Basu等[16]利用10对引物和49份黄麻种质资源来分析黄麻遗传多样性, 能将黄麻圆果种和长果种完全区分开来, 本研究在增加引物数量和黄麻种质资源数量的情况下, 得到的结论一致, 说明圆果种黄麻与长果种黄麻之间遗传差异较大、亲缘关系较远, 这应该与圆果种黄麻和长果种黄麻有着不同的起源地有关。圆果种黄麻种间遗传距离小于长果种, 说明圆果种的遗传多样性小于长果种, Huq等[17]用27对引物对16个黄麻品种进行遗传多样性分析也得到了相同结论, 这应该是因为圆果种黄麻属于自花授粉作物, 而长果种黄麻属于常异花授粉作物。
核心种质构建中取样方式和比例至关重要。李自超等[18]认为, 取样比例应根据具体物种遗传结构及数量规模状况而定, 总资源份数多的物种其核心种质所取比例可小一些, 总资源份数少的物种其核心种质所取比例可相对大一些。国内外不同物种构建的核心种质, 占总资源的比例一般为5%~ 40%[19-21]。而构建核心资源的关键因素是材料的分组, 常见的分组方法有按种质资源地理分布、系谱来源、农艺性状和分子标记等[22-23]。任丽平等[3]按种质资源地理来源结合分子标记分类, 在500余份甘蓝型油菜种质中遴选出87份核心种质资源。刘艳阳等[19]利用表型数据和分子标记在5020份芝麻种质资源中遴选出501份核心种质, 便于作物核心种质的管理、研究利用, 为后续育种提供了优异基因资源。本研究利用11个农艺性状聚类及分子标记聚类结果, 在300份黄麻种质资源中遴选出84份核心种质资源, 核心种质的构建将有助于加速黄麻种质创新的相关研究进程。比较84份核心种质与300份大群体的多样性, 发现84份黄麻核心种质遗传多样性丰富; 且两者变异系数和Shannon-Wiener指数比较接近, 表明84份核心种质资在遗传多样性方面可以最大限度代表300份黄麻种质资源。但是, 聚类分析发现黄麻种质资源并不总是与其地理来源一致[24]。因此广泛引种, 深入研究, 对核心种质实时进行动态调整, 不断补充新搜集的黄麻种质资源, 不断优化核心种质结构, 使其遗传变异最大化将更有利于其开发利用。
核心种质建立后, 要建立完善的繁种、供种体制, 可以保证核心种质的有效利用。加强对所构建核心种质的后续评价研究, 挖掘资源特异基因; 同时加强资源应用研究, 建立各类专项核心种质, 满足生产、育种对不同资源类型的需要[19]。

图2 84份黄麻核心种质聚类分析
4 结论
本研究利用44对核心引物对300份黄麻种质资源进行聚类分析, 可将300份黄麻种质资源分为10个类群; 农艺性状聚类分析可分为8个类群。根据2个聚类图, 挑选出108份预选黄麻核心种质, 然后对其进行遗传差异性分析, 剔除遗传相似系数大于等于0.85遗传冗余, 得到84份核心种质, 可最大限度代表300份黄麻种质资源, 应加以保存和利用。
[1] 熊和平. 麻类作物育种学. 北京: 中国农业科学技术出版社, 2008. pp 156–207. Xiong H P. Breeding Sciences of Bast and Leaf Fiber Crops. Beijing: China Agricultural Science and Technology Press, 2008. pp 156–207 (in Chinese).
[2] Tao A F, Huang L, Wu G F, Reza K A, Qi J M, Fang P P, Lin L H, Zhang L W, Lin P Q. High-density genetic map construction and QTLs identification for plant height in white jute (L.) using specific locus amplified fragment (SLAF) sequencing., 2017, 18: 355.
[3] 任丽平, 倪西源, 黄吉祥, 雷伟侠, 曹明富, 赵坚义. 甘蓝型油菜一个代表性核心种质的遴选. 中国农业科学, 2008, 41: 3521–3531. Ren L P, Li X Y, Huang J X, Lei W X, Cao M F, Zhao J Y. Core collection of a representative germplasm population in., 2008, 41: 3521–3531 (in Chinese with English abstract).
[4] 贾继增, 张启发. 为第二次“绿色革命”发掘基因资源——国家重点基础研究发展规划项目“农作物核心种质构建、重要新基因发掘与有效利用研究”总体设计及研究进展. 中国基础科学, 2001, (7): 4–8. Jia J Z, Zhang Q F. Exploring gene resources for “the Second Green Revolution”— the overall design and research progress of “crop core germplasm construction, new important genes discovery and effective utilization researches” in the national key basic research and development planning project., 2001, (7): 4–8 (in Chinese with English abstract).
[5] 陈学军, 雷刚, 周坤华, 方荣. 蔬菜核心种质研究进展. 江西农业大学学报, 2015, (1): 60–66. Chen X J, Lei G, Zhou K H, Fang R. Review of the studies on core collection for vegetables.2015, (1): 60–66 (in Chinese with English abstract).
[6] Frankel O H. Genetic Perspectives of Germplasm Conservation. Cambridge: Cambridge University Press, 1984. pp 161–170.
[7] Coimbra R R, Miranda G V, Cruz C D, Silvad J H, Vilelar A. Development of a Brazilian maize core collection., 2009, 32: 538–545.
[8] 邱丽娟, 李英慧, 关荣霞, 刘章雄, 王丽侠, 常汝镇. 大豆核心种质和微核心种质的构建、验证与研究进展. 作物学报, 2009, 35: 571–579. Qiu L J, Li Y H, Guan R X, Liu Z X, Wang L X, Chang R Z. Establishment, representative testing and research progress of soybean core collection and mini core collection., 2009, 35: 571–579 (in Chinese with English abstract).
[9] 陶爱芬, 祁建民, 粟建光, 方平平, 林荔辉, 徐建堂, 吴建梅, 林培清. 黄麻种质资源遗传多样性的SRAP 分析. 植物科学学报, 2012, 30(2): 178–187. Tao A F, Qi J M, Li J G, Fang P P, Lin L H, Xu J T, Wu J M, Lin P Q. Analysis of genetic diversity of jute (L.) germplasm revealed by SRAP., 2012, 30(2): 178–187 (in Chinese with English abstract).
[10] 张加强, 陈常理, 骆霞虹, 金关荣. 中国育成黄麻主要品种间的亲缘关系分析. 中国农业科学, 2015, 48: 4008–4020. Zhang J Q, Chen C L, Luo X H, Jin G R. Analysis of the coefficient of parentage among major jute cultivars in China., 2015, 48: 4008–4020 (in Chinese with English abstract).
[11] 张力岚. 黄麻InDel标记开发与纤维品质相关性状的关联分析. 福建农林大学硕士学位论文, 福建福州, 2018.Zhang L L. Development of InDel Markers and Association Analysis of Fiber Quality Related Traits in Jute (spp.). MS Thesis of Fujian Agriculture and Forestry University, Fuzhou, Fujian, China, 2018 (in Chinese with English abstract).
[12] 张立武, 袁民航, 何雄威, 刘星, 方平平, 林荔辉, 陶爱芬, 徐建堂, 祁建民. GenBank数据库中黄麻EST-SSR标记的开发及其通用性评价. 作物学报, 2014, 40: 1213–1219. Zhang L W, Yuan M H, He X W, Liu X, Fang P P, Lin L H, Tao A F, Xu J T, Qi J M. Development and universality evaluation of EST-SSR markers from GenBank in jute., 2014, 40:1213–1219 (in Chinese with English abstract).
[13] 粟建光, 龚有才. 黄麻种质资源描述和数据标准. 北京: 中国农业出版社, 2015. pp 7–27. Su J G, Gong Y C. Descriptors and Data Standard for Jute (L. &L.). Beijing: China Agriculture Press, 2005. pp 7–27 (in Chinese).
[14] 马冰, 代帅帅, 程亚增, 蒋彩虹, 任民, 程立锐, 杨爱国. 烤烟种质资SSR 核心引物的筛选及验证. 中国烟草科学, 2016, 37(5): 1–5. Ma B, Dai S S, Cheng Y Z, Jiang C H, Ren M, Cheng L R, Yang A G. Screen and identification of SSR core primers for flue-cured tobacco germplasm., 2016, 37(5): 1–5 (in Chinese with English abstract).
[15] 杨美, 付杰, 向巧彦, 刘艳玲. 利用AFLP分子标记技术构建花莲核心种质资源. 中国农业科学, 2011, 44: 3193–3205. Yang M, Fu J, Xiang Q Y, Liu Y L. The core-collection construction of flower lotus based on AFLP molecular markers., 2011, 44: 3193–3205 (in Chinese with English abstract).
[16] Basu A, Ghosh M, Meyer R, Powell W, Basak S L, Sen S K. Analysis of genetic diversity in cultivated jute determined by means of SSR markers and AFLP profiling., 2004, 44: 678–685.
[17] Huq S, Islam M S, Sajib A, Ashraf N, Haque S, Khan H. Genetic diversity and relationships in jute (spp.) revealed by SSR markers., 2009, 38: 153–161.
[18] 李自超, 张洪亮, 曾亚文, 杨忠义, 申时全, 孙伟清, 王象坤. 云南地方稻种质资源核心种质取样方案研究. 中国农业科学, 2000, 33(5): 1–7. Li Z C, Zhang H L, Zeng Y W, Yang Z Y, Sheng S Q, Sun W Q, Wang X K. Study on sampling schemes of core collection of local varieties of rice in Yunnan, China., 2000, 33(5): 1–7 (in Chinese with English abstract).
[19] 刘艳阳, 梅鸿献, 杜振伟, 武轲, 郑永战, 崔向华, 郑磊. 基于表型和SSR分子标记构建芝麻核心种质. 中国农业科学, 2017, 50: 2433–2441. Liu Y Y, Mei H X, Du Z W, Wu K, Zheng Y Z, Cui X H, Zheng L. Construction of core collection of sesame based on phenotype and molecular markers., 2017, 50: 2433–2441 (in Chinese with English abstract).
[20] Hu J B, Wang P Q, Su Y, Wang R J, Li Q, Sun K L. Microsatellite diversity, population structure, and core collection formation in melon germplasm., 2015, 33: 439–447.
[21] Xu Q J, Zeng X Q, Lin B, Li Z Q, Yuan H J, Wang Y L, Sang Z, Nyima T. A microsatellite diversity analysis and the development of core-set germplasm in a large hulless barley (L.) collection., 2017, 18: 102.
[22] 栾明宝, 陈建华, 许英, 王晓飞, 孙志明. 苎麻核心种质构建方法. 作物学报, 2010, 36: 2099–2106. Luan M B, Chen J H, Xu Y, Wang X F, Sun Z M. Method of establishing ramie core collection., 2010, 36: 2099–2106 (in Chinese with English abstract).
[23] 郎彬彬, 黄春辉, 朱博, 谢敏, 张文标, 钟敏, 曲雪艳, 陶俊杰, 徐小彪. 基于果实相关性状的江西野生毛花猕猴桃初级核心种质的构建方法研究. 果树学报, 2016, 33: 794–803.Lang B B, Huang C H, Zhu B, Xie M, Zhang W B, Zhong M, Qu X Y, Tao J J, Xu X B. Study on the method of constructing a primary core collection of Jiangxi wildbased on fruit traits., 2016, 33: 794–803 (in Chinese with English abstract).
[24] Zhang L, Cai R, Yuan M, Tao A, Xu J, Lin L, Fang P, Qi J. Genetic diversity and DNA fingerprinting in jute (spp.) based on SSR markers., 2015, 3: 416–422.
附表1 黄麻84份核心种质类型及来源

Supplementary table 1 Type and origin of 84 core germplasms in jute
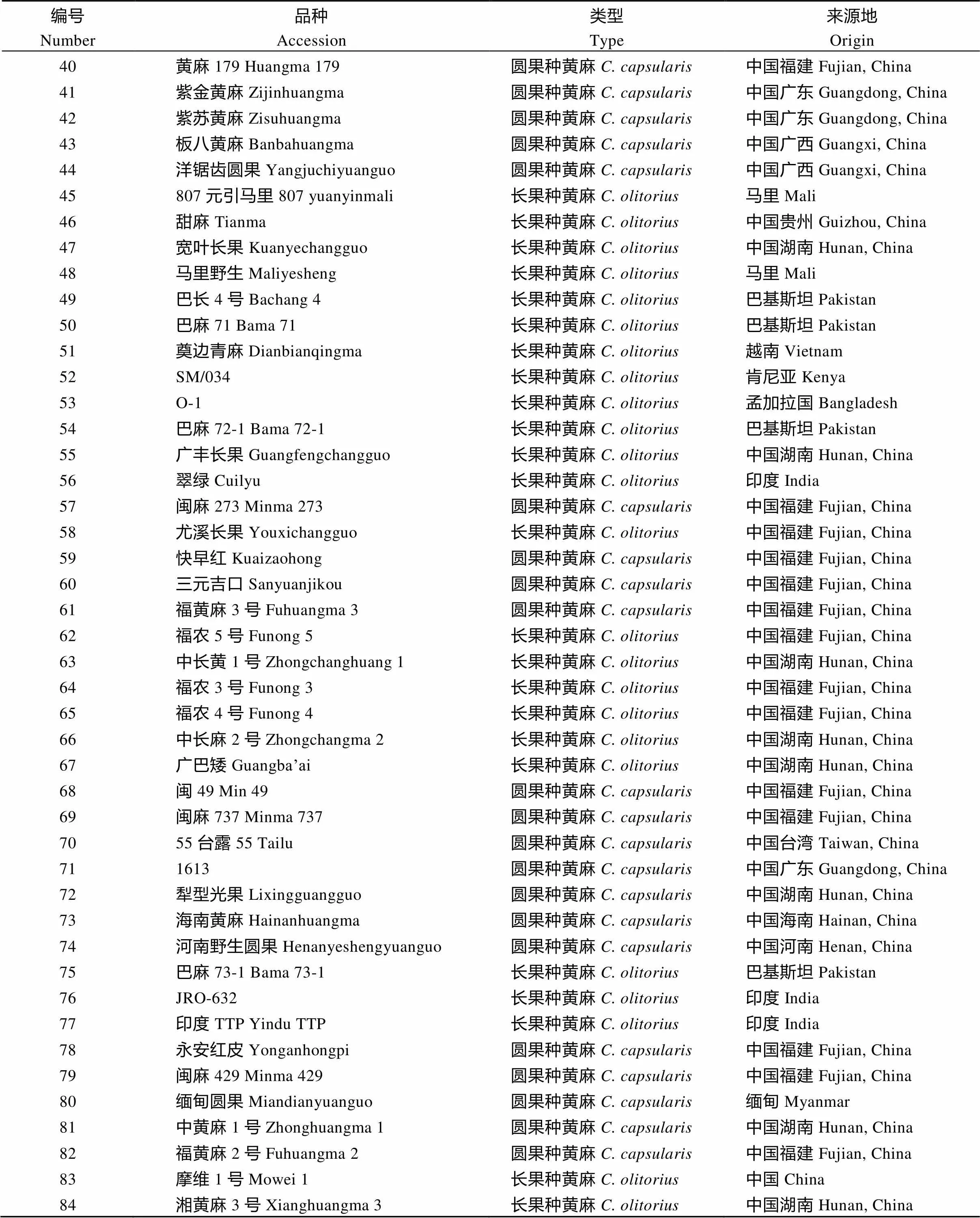
(续附表1)
Core collection screening of a germplasm population in jute (spp.)
XU Yi1,2,3,**, ZHANG Lie-Mei1,2,**, GUO Yan-Chun1,2,3, QI Jian-Min1,2, ZHANG Li-Lan1,2,3, FANG Ping-Ping1,2, and ZHANG Li-Wu1,2,3,*
1Key Laboratory for Genetics, Breeding and Multiple Utilization of Crops of Ministry of Education / Fujian Key Laboratory for Crop Breeding by Design / College of Crop Science, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;2Experiment Station of Jute and Kenaf in Southeast China of Ministry of Agriculture and Rural Affairs / Fujian Public Platform for Germplasm Resources of Bast Fiber Crops / Fujian International Science and Technology Cooperation Base for Genetics, Breeding and Multiple Utilization Development of Southern Economic Crops, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;3Center for Genomics and Biotechnology of Haixia Institute of Science and Technology, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China
Innovation of jute germplasm and breeding new varieties are based on core collection. In this study, 300 jute accessions were systematically identified. The core collections were constructed by SSR molecular markers, agronomic traits and geographical sources. The variation coefficient of 11 agronomic traits ranged from 13.06% to 84.87%, indicating an abundant genetic diversity. These jute germplasm was divided into eight groups based on agronomic traits while ten groups based on the clustering analysis of molecular markers. Combining the two cluster analyses, and geographic location of these jute accessions, a preselected core collection including 108 accessions was established. Furthermore, 44 pairs of SSR primers were used to analyze the genetic differences. The 108 varieties were divided into white jute and dark jute at the genetic similarity coefficient of 0.65. According to the analysis of genetic differences, genetic redundancy with genetic similarity greater than or equal to 0.85 was excluded and 84 core collections, including 60 white jute accessions and 24 dark jute ones, were obtained. By comparison in the coefficient of variation and Shannon-Wiener index of agronomic traits between 84 core collections and 300 germplasm, it was found that there was no significant difference between them, indicating that the 84 jute core collections could represent the genetic diversity of 300 jute germplasm resources to the maximum extent.
jute (spp); agronomic traits; molecular marker; core collections
本研究由国家自然科学基金项目(31771369)和国家现代农业产业技术体系建设专项(CARS-19-E06)资助。
This study was supported by the National Natural Science Foundation of China (31771369) and the China Agriculture Research System (CARS-19-E06).
张立武, E-mail: lwzhang@fafu.edu.cn, zhang_liwu@hotmail.com
**同等贡献(Contributed equally to this work)
徐益, E-mail: 1275924118@qq.com; 张列梅, E-mail: zhangliemei@126.com
2019-01-11;
2019-05-12;
2019-05-20.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20190520.0924.004.html
10.3724/SP.J.1006.2019.94008

