浮游软体动物长角螺属(Clio)种类的分类学和谱系地理学
高阳,李海涛*,姜重臣,陈志云,李宏俊
( 1. 国家海洋局南海环境监测中心,广东 广州 510300;2. 中国科学院南海海洋研究所 热带海洋生物资源与生态重点实验室,广东 广州 510301;3. 国家海洋环境监测中心,辽宁 大连 116023)
1 引言
海洋浮游软体动物是一类终生营浮游生活的类群,一般属于腹足纲(Gastropoda)。据估计,终生浮游腹足类仅有约140 种[1]。浮游腹足类大多栖息于上层200 m 水体,并有明显的昼夜垂直迁移现象[2–3]。作为初级消费者、被捕食者及钙化生物,浮游腹足类在海洋生态系统的生物地球化学过程中扮演着十分关键的作用[4–5]。此外,由于对海洋酸化的极度敏感性,浮游腹足类成为良好的指示物种[6–7]。
自半个多世纪前张福绥[8]开展中国海的浮游软体动物分类以来,迄今鲜见其他分类报道,其研究结果影响深远。由于研究的匮乏,我国浮游腹足类的分类严重滞后,一些分类错误延续至今,无法得到订正,如:归在螔螺科(Limacinidae)下的强卷螺属(Agadina)种类实际上是成体营底栖生活的轮螺科(Architectonicidae) 的面盘幼虫[9–10];厚唇螺属(Diacria) 由D.trispinosa 复合群和D. quadridentata 复合群组成,其中D. trispinosa 复合群中的种类根据壳面花纹的分布来区分[11],我国记录的D. trispinosa 缺乏壳面花纹分布这一关键分类特征的描述,实际上无法确定其为何种[8]。此外,一些物种的形态描述和图示与实物有一定出入;基于形态变化的考虑而提出的一些亚种或变种, 尚无后续研究的证实。
大多数浮游腹足类的贝壳缺乏雕刻或生长纹等特征,其形态鉴定主要依据一些简单的分类性状,单纯依靠形态特征很难进行准确的种类鉴定。基于mt-COI 基因的DNA 条形码技术[12]目前已被广泛应用于浮游腹足类的种类鉴定和多样性分析[13–15]。浮游软体动物分布广泛,很多种类为接近全球分布或环球分布[13,16],是开展生物地理学或谱系地理学研究的良好材料[16–17]。本研究以长角螺属(Clio)物种为材料,通过对mtCOI 基因和核18S rRNA 基因序列的分析,结合数据库中已有的数据,对其进行了分类学和谱系地理学研究,以期了解该属种类的地理遗传分化和分布状况,同时为其他浮游软体动物的多样性研究提供参考。
2 材料与方法
2.1 样品采集
实验样品于2016 年5 月至2019 年1 月采自西北太平洋(含南海、吕宋海峡及其周边海域和新几内亚岛以北海域) 及北印度洋海域。使用大型浮游生物网(网目0.505 mm)采用垂直拖网和水平拖网相结合的方式采集浮游动物样品。为增加样品的采获几率,西北太平洋海域的采样均在夜间进行。浮游动物样品经无水酒精固定后冰冻保存带回实验室,体视镜下分选出长角螺属动物样品,对其进行初步的形态鉴定,并编号拍照。除吕宋海峡的样品为部分测序外,其余采样点的样品全部用于测序,共计55 个标本用于DNA 序列的分析,测序标本的采样信息见表1。
2.2 DNA 提取、PCR 扩增和测序
剪取长角螺游泳鳍,采用碱裂解法[18]提取DNA。使用引物LCO-1490(5′-GGT CAA CAA ATC ATA AAG ATA TTG G-3′)和HCO-2198 (5′-TAA ACT TCA GGG TGA CCA AAA AAT CA-3′)[19]扩增mt-COI 基因片段;使用引物18A1 (5′- CTG GTT GAT CCT GCC AGT CAT ATG C-3′)和1800 (5′-GAT CCT TCC GCA GGT TCA CCT ACG-3′)[20]扩增18S rRNA 基因。
PCR 反应体系总体积为50 μL,包括:2×buffer 25 μL,dNTP 400 μmol/L,引物各0.3 μmol/L,KOD FX 高保真PCR 酶(TOYOBO 公司)1.0 U,模板DNA1 μL,加去离子水补足至50 μL。PCR 反应条件为:94℃预变性2 min;98℃变性15 s,52℃退火30 s,68℃延伸60 s (mtCOI)或120 s (18S rRNA),共30 个循环;最后68℃延伸10 min。PCR 产物经1%琼脂糖凝胶电泳检测后,送广州艾基生物技术有限公司进行回收纯化和测序,测序引物与扩增引物相同。为保证18S rRNA基因片段测通,中间引物1155R (5′-CCG TCA ATTCCT TTA AGT TTC AG-3′)也被用于测序[20]。

表 1 测序标本的采样信息Table 1 Collection information for specimens analyzed in this study
2.3 系统学分析
拼接、校对后的测序结果,连同GenBank 中下载的序列,在BioEdit 软件中进行多重序列比对。使用MEGA 7.0 计算碱基的组成,基于Kimura 2-Parameter(K2P)参数模型计算遗传距离,并采用邻接法(Neighbor-joining,NJ)构建系统进化树,可靠性经过1 000 次自展(Bootstrap, BS)检验。采用MrBayes 3.2[21]构建贝叶斯树(Bayesian Inference, BI),分子进化模型使用jModelTest 2.1[22]根据Bayesian Information Criteria(BIC)标准计算得出。mtCOI 和18S rRNA 基因的最佳进化模型分别为HKY+I+G 和K80+I。在马尔可夫链蒙特卡罗(Markov Chain Monte Carlo,MCMC)参数下运行300 万代,每100 代抽样一次,前2 500 代作为老化样本舍去。BI 树各分支置信度以后验概率(Posterior Probabilities, PP)表示。使用FigTree 1.4.3 显示BI 树的拓扑结构图。
从GenBank 中下载的长角螺属COI 基因序列包括C. pyramidata(MH101647~55、FJ876874~75、MF048921~22、KC774065~68、KP292791、KC788279、GQ861823)、C. pyramidata lanceolate (FJ876872~73、FJ876877~79)、C. pyramidata antarctica (FJ876876、MF048919)、C. cuspidata (FJ876869~71、KP292789、KC774064)、C. convexa (KC774062~63、KC774069)、C.recurva (FJ876880~86、KP292790、MF048923)、Clio sp.(FJ876887)。
2.4 分子可操作分类单元估计
采用GMYC(Generalized Mixed Yule Coalescent)模型[23]、ABGD(Automatic Barcoding Gap Discovery)方法[24]和3%K2P 遗传距离阈值进行分子可操作分类单元(Molecular Operational Taxonomic Units, MOTUs)估计。
GMYC 模型:利用BEAST v1.10.1[25]构建超度量系统发育树;采用uncorrelated log-normal clock 模型,1×108次MCMC 分析,1 000 次burn in;利用BEAST 软件包中的TreeAnnotator 分析超度量系统发育树,并生成最大谱系置信树;随后用FigTree 1.4.3 将最大谱系置信树转化为APE 识别的格式(Newick);最后利用R 环境中的“SPLITS”软件包进行单阈值GMYC 物种界定[23],利用gmyc 函数获得零模型与GMYC 模型之间的似然比值、种−种群的转折点,以及转折点对应的MOTU 的数量。
ABGD 方法:样本在线提交至ABGD 网站(http://wwwabi.snv.Jussieu.fr./pubilc/abgd/abgdweb.html),采用K2P 模型计算遗传距离,其他参数均使用网络版本的默认值,基于遗传距离对样本进行划分。
3%K2P 遗传距离阈值:将K2P 遗传距离小于或等于3%(0.03)的序列划分为同一MOTU,遗传距离大于3%的序列划分为不同的MOTU。
3 结果
3.1 形态鉴定结果
西北太平洋和印度洋海域采集的长角螺属标本,经形态鉴定,属于矛头长角螺(C. pyramidata)(图1a–c)、尖棘长角螺(C. cuspidata) (图1d–f)和膨凸长角螺(C. convexa) (图1g)3 个形态种,以及个别通过形态难以定种的幼体(图1h)。在采样过程中,较大个体的标本极易破损,棘刺发达的尖棘长角螺很难得到完整的标本。吕宋海峡采集到的尖棘长角螺(图1d–e)明显较新几内亚岛以北海域采集到的个体(图1f)宽大,后者壳体明显纤细。
3.2 序列分析
测序共获得55 条mtCOI 序列(GenBank 登录号MK749610~64,表1) 和9 条18S rRNA 基因序列(GenBank 登录号MK749665~73,表1),其中55 条mt-COI 序列定义了51 种单倍型。mtCOI 序列长658 bp,A、T、G、C 含量分别为22.4%、44.3%、19.0% 和14.3%,A+T 的含量明显高于G+C 的含量。mtCOI 序列共有变异位点154 个,简约信息位点113 个。
对18S rRNA 基因序列两端做截齐处理后,序列长1 728~1 740 bp,存在两段分别为8 bp(TTTCGGGG)和5 bp(TGAAG)的插入/缺失片段,以及1 个碱基(T)的插入/缺失位点。A、T、G、C 含量分别为24.0%、25.2%、27.7%和23.2%,A+T 和G+C 的含量大体相当。3 个形态种均分别测定了3 个个体的18S rRNA 基因序列,其中尖棘长角螺和膨凸长角螺在种内没有变异位点,矛头长角螺在种内有3 个变异位点。
3.3 系统发育和谱系地理结构
mtCOI 和核18S rRNA 基因的BI 树和NJ 树有大体一致的拓扑结构,本文只显示BI 树(图2,图3)。mtCOI 基因系统分析显示,矛头长角螺和尖棘长角螺存在明显的谱系地理结构,分别形成4 个明显分化的支系;膨凸长角螺(含印度洋、红海和西太平洋样品)和曲形长角螺(C. recurva,含南大西洋、北大西洋和南大洋样品)形成单系群,无地理遗传分化。矛头长角螺的4 个支系(A–D)均获得较高的支持率(PP≥95%,BS≥90%),本文测序的标本分属2 个不同的支系(A 和C)。支系A 基本上呈全球分布,支系B 目前仅见于大西洋(含加勒比海)和印度洋,支系C 目前仅见于西太平洋和东印度洋,支系D(C. pyramidata antarctica)目前仅见于南大洋的高纬度地区(图4)。
尖棘长角螺的4 个支系(E–H)也获得较高的支持率(PP≥99%,BS>85%),本文测序的标本分属支系E 和F。不同支系的地域性十分明显,支系E 分布于吕宋海峡,支系F 分布于西太平洋和印度洋,支系G分布于大西洋,支系H 分布于南大洋(图5)。
18S rRNA 基因系统树中(图3),矛头长角螺的支系A、B 和C 形成一个独立的分支,与支系D 区分开来。膨凸长角螺和尖棘长角螺的个体形成单系群,尖棘长角螺的支系E 和F 在18S rRNA 基因系统树中无法得到显现。
3.4 遗传距离分析
K2P 遗传距离分析表明(因C. pyramidata(GQ861823)、C. recurva(MF048923)和Clio sp.(FJ876887)可能存在种类误定或序列太少,未参与分析),形态种的种内遗传距离在0~0.089 之间,种间遗传距离在0.070~0.185 之间,两者发生较大范围重叠(表2)。矛头长角螺支系内的遗传距离为0~0.026,支系间的遗传距离为0.031~0.089;尖棘长角螺支系内的遗传距离为0~0.014,支系间的遗传距离为0.048~0.079。
3.5 MOTUs 估计
ABGD 比较稳定的初始划分结果与GMYC 和3%K2P 遗传距离阈值的MOTUs 估计结果完全相同(图2, GMYC 和ABGD 原始划分结果未给出),矛头长角螺和尖棘长角螺的4 个支系均被划分为独立的MOTU。
4 讨论
4.1 长角螺属的分类学

图 1 长角螺属种类的贝壳形态(比例尺=5 mm)Fig. 1 Shell morphology of Clio species (scale bars=5 mm)
由于长角螺外壳薄脆,在采样过程中极易造成破损,相当部分的标本为非完整的个体,如尖棘长角螺发达的棘刺很难完整保留(图1),给形态鉴定带来很大的难度。外形上的相似性,也难以区分不同大小的个体是种间差异还是同种不同发育阶段的个体,如矛头长角螺支系C 中2 个不同大小的个体在形态上有较大的差异(图2),支系A 和C 的成体在形态上则难以区分。张福绥[8]描述了中国近海长角螺属3 个种(含变种),其中包括一新变种小尾长角螺(C. pyramidata var. microcaudata,原文Euclio pyramidata var. microcaudata),该变种仅采集到1 个个体。该变种能否被视为一个有效的分类单元,尚需进一步的研究证实。我们检视了采集到的大量长角螺标本,未发现类似个体。新几内亚岛以北海域采集到的尖棘长角螺与吕宋海峡分布的尖棘长角螺在形态上有一定的差异,且两者的mtCOI 基因序列也有明显分化。然而由于前者仅采集到一个不太完整的标本,尚无法判断这种形态差异是种间的差异还是同种个体间的差异。
相比膨凸长角螺和曲形长角螺两个种的种内遗传距离,矛头长角螺和尖棘长角螺两个形态种各支系间的遗传距离已经达到种间的分化水平。矛头长角螺的支系D(C. pyramidata antarctica)与其他3 个支系在18S rRNA 基因序列上也存在明显的差异,并在系统树上与其他支系区别开来。因此,C. pyramidata antarctica 这一亚种应提升为种,该亚种(或变种)的形态[2,26]也明显有别于典型的矛头长角螺,前者明显修长。18S rRNA 基因系统树并不支持其他支系的划分,这可能是由于18S rRNA 基因较为保守的缘故。矛头长角螺和尖棘长角螺是否为隐存种还需要进一步研究的证实(如ITS 序列)。
4.2 长角螺属的谱系地理学

图 2 基于mtCOI 基因构建的长角螺属贝叶斯系统发育树Fig. 2 Bayesian inference phylogenetic tree of genus Clio based on mtCOI gene sequences
海洋生物,无论是终生浮游生物还是其他海洋生物的浮游幼体,可以借助洋流进行扩散,开放的海洋环境也没有阻碍基因交流的屏障,因而通常认为海洋生物会在很大的地理尺度内表现出较低的遗传分化[27]。目前,越来越多的证据改变了我们这种传统的观点,即使在开放海域,海洋学和生态学过程也会对海洋生物的基因流产生阻碍作用,从而导致在不同尺度上产生遗传分化[28–30]。
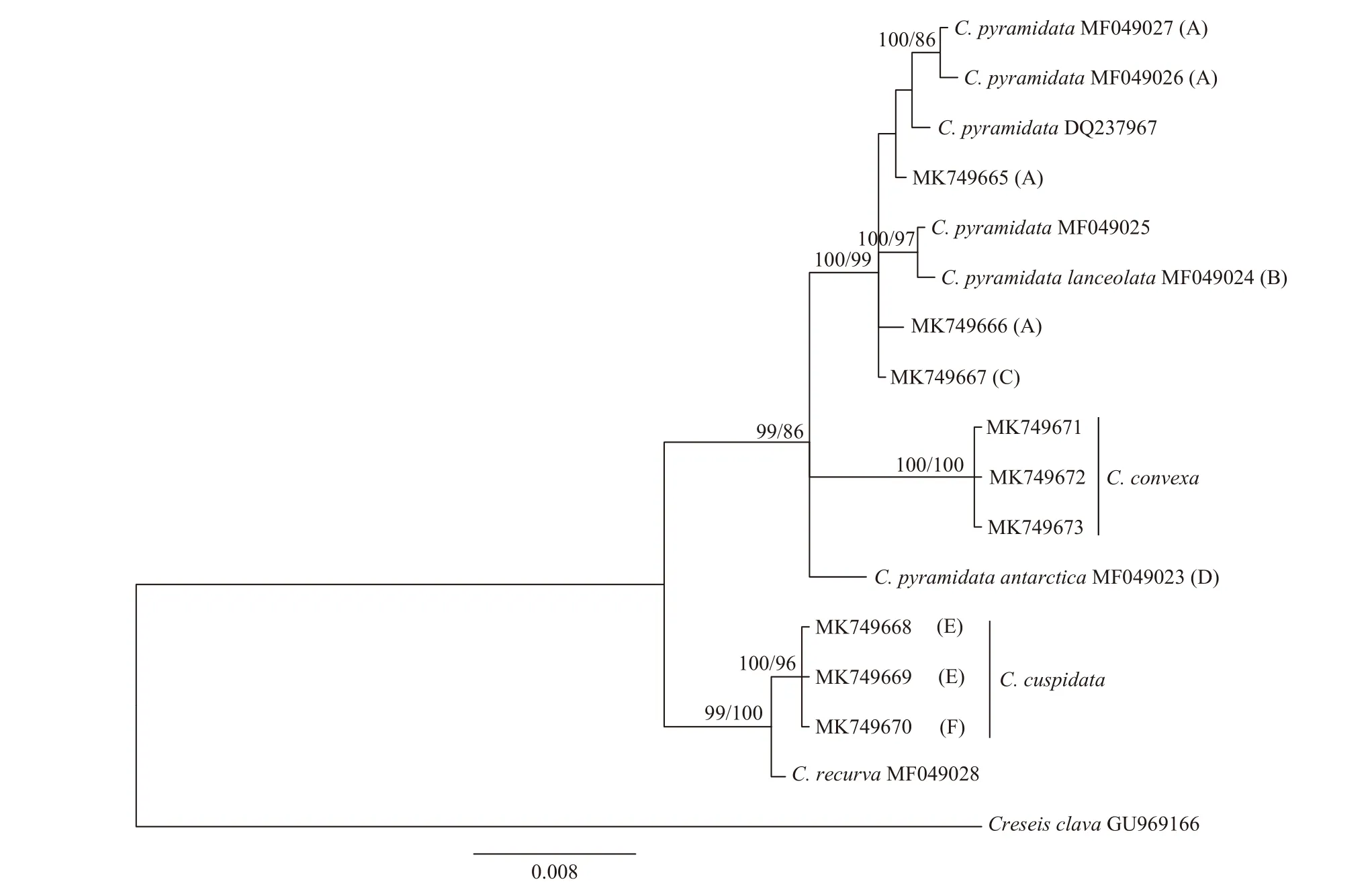
图 3 基于18S rRNA 基因构建的长角螺属贝叶斯系统发育树Fig. 3 Bayesian inference phylogenetic tree of genus Clio based on nuclear18S rRNA gene sequences

图 4 矛头长角螺4 个mtCOI 支系的地理分布Fig. 4 Geographical distributions of four mtCOI lineages for C. pyramidata
结合本文和数据库中已有的数据分析表明,矛头长角螺和尖棘长角螺存在较为明显的地理群体遗传分化,中国海及其邻近海域可能只有矛头长角螺支系A 的存在,分布于吕宋海峡的尖棘长角螺支系E为一新的谱系分支。从全球尺度来讲,尖棘长角螺的采样范围和采样数量较为有限,其谱系地理结构还需更广泛的采样进行研究。南海–吕宋海峡及其周边海域与新几内亚岛以北海域具有完全不同的长角螺种类和支系组成,除分布着矛头长角螺和尖棘长角螺完全不同的支系外,新几内亚岛以北海域分布的膨凸长角螺在南海–吕宋海峡及其周边海域(10 余航次调查)未有发现。北赤道流到达菲律宾群岛东岸后发生分叉,形成南、北流向的棉兰老流和黑潮[31],相反的流向可能阻碍了两海域间的物种扩布及群体间的基因交流。可以推测,不同遗传背景的浮游软体动物群体可能对海洋酸化等环境变化产生不一样的响应[32]。
4.3 海洋生物的DNA 条形码鉴定需要建立本地数据库
我们分析了印度洋和西北太平洋海域数百个浮游软体动物的mtCOI 基因序列和核18S、28S 基因序列,并通过与其他海域序列的比较,发现浮游软体动物中普遍存在mtCOI 基因的地理遗传分化现象(结果另文发表)。如按照DNA 条形码遗传距离的10 倍法则[33]或3%阈值[12]标准,这些具有地理遗传分化的形态种会被认为存在隐存种。
DNA 条形码技术是一种基于种内及种间DNA序列变异程度的距离法(Distance-Based Method),它要求种间遗传距离明显大于种内遗传距离,形成“条形码间隙(Barcoding Gap)”[34–35]。然而,DNA 条形码技术也受到一些质疑和批判。如单独的线粒体DNA 序列不足以识别物种,因为这种遗传分化并非是划定物种界限的必要条件[36–37];“条形码间隙”是人为的不充分取样造成的[38]。显然,取样范围的扩大(全球取样)、测序样本数量的增加会使种内遗传距离增大,测序物种数量的增加会使种间遗传距离缩小。取样数量和地理尺度的大小有时会显著影响DNA 条形码的分析结果,甚至可能得出错误的结论。单纯依据mtCOI 基因序列来揭示隐存种,证据可能不够充分,还需要核基因证据的支持。
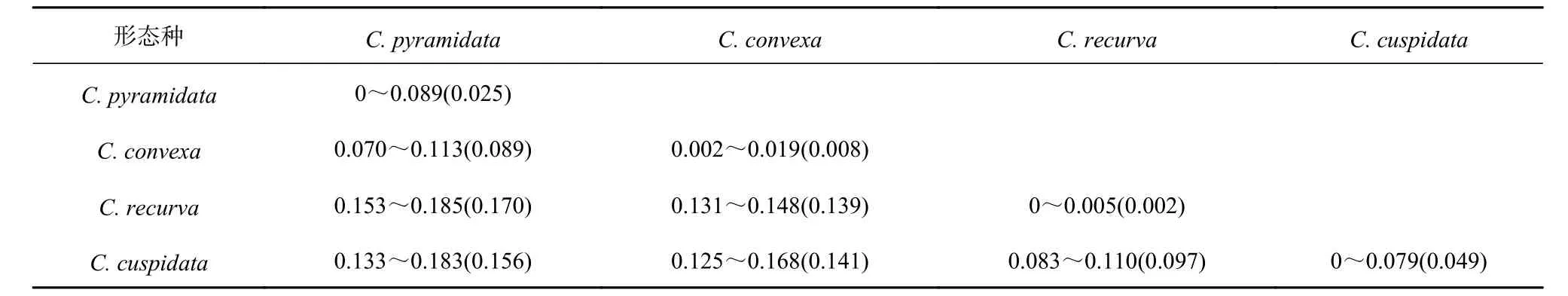
表 2 基于mtCOI 基因的长角螺属4 个形态种的K2P 遗传距离Table 2 The K2P distances of four Clio morphospecies based on mtCOI sequences
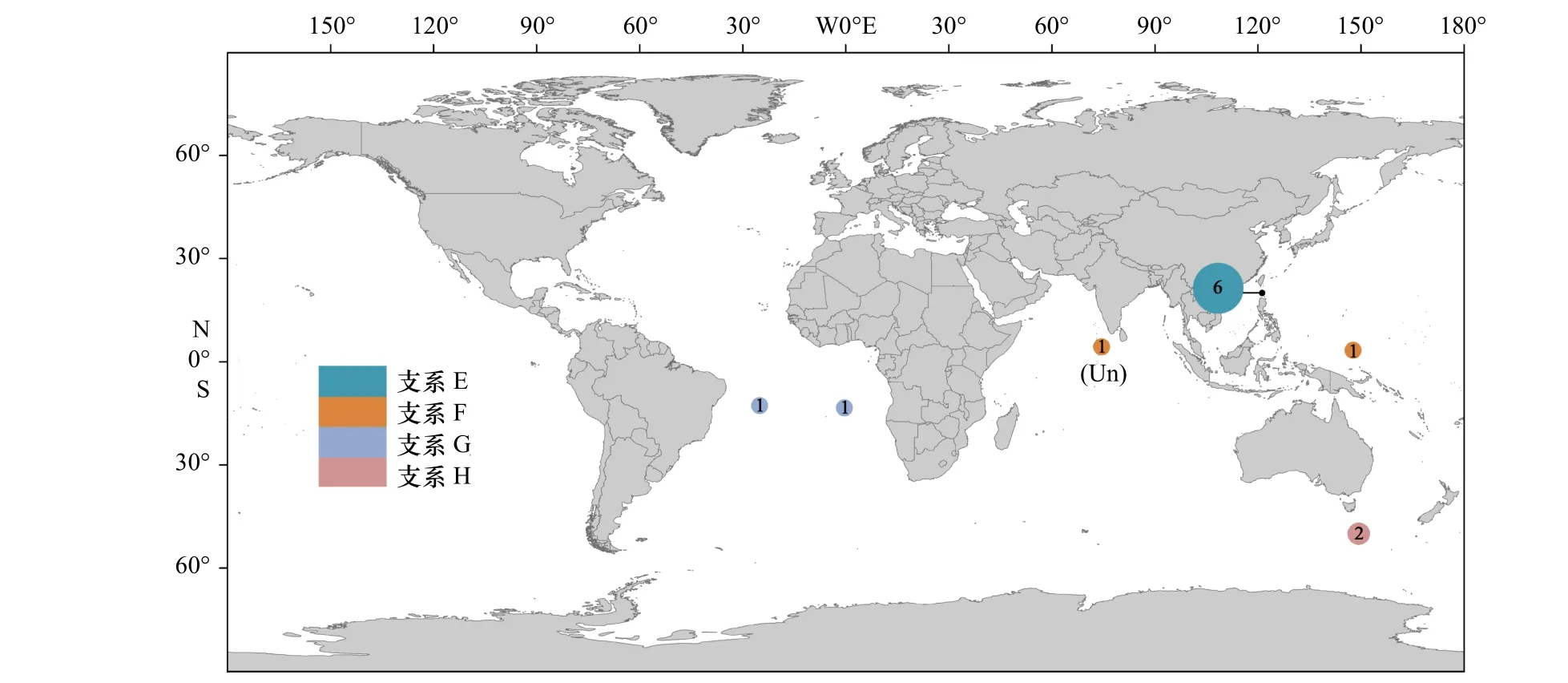
图 5 尖棘长角螺4 个mtCOI 支系的地理分布Fig. 5 Geographical distributions of four mtCOI lineages for C. cuspidata
形态种内的遗传分化广泛存在于海洋动物中,除浮游软体动物[16]外,还有如水母[39–40]、甲壳类[41–43]、毛颚类[44]和鱼类[45]等。这种遗传分化不仅存在于大的地理尺度,也存在于小的区域尺度。
现有公共数据库(如GenBank、BOLD 等)中的参考序列缺乏相对应的物种形态照片,并且数据库中还存在误定的种类。来自不同海域的序列,即使与数据库中已有的序列属于同一形态种,由于不同地理群体的遗传分化,仍然无法通过DNA 条形码序列的比对来定种。如本文测序的吕宋海峡尖棘长角螺序列,无法在数据库中与已有的其他海域的尖棘长角螺序列形成高精度匹配。因此,建立我国海域本地物种的DNA 条形码数据库是极为必要的,且测序个体应有足够的数量并覆盖较大的地理尺度。
致谢:中国科学院南海海洋研究所周林滨博士采集了印度洋样品,李开枝研究员在实验过程中给予了帮助和支持,谨致谢忱。

