国际奶牛遗传评估体系概况
郑伟杰 ,李厚诚,苏丁然,闫青霞,陈绍祜,张胜利,孙东晓*
(1.中国农业大学动物科学技术学院,北京 100193;2.中国奶业协会,北京 100193)
奶牛育种主要是在现有的优良品种中系统地开展选育工作,实现群体的持续遗传改良。20 世纪80 年代,奶牛育种便已普遍应用以青年公牛后裔测定为核心的常规遗传评估技术,后裔测定虽有较高的准确性,但存在世代间隔长、育种进程缓慢等缺点。2001 年,Meuwissen 等[1]首次提出基因组选择技术,并于2009年被正式应用。通过基因组选择技术可实现青年公牛早期准确选择,从而大幅度缩短世代间隔,加快群体遗传进展。目前基因组选择技术已被广泛应用于世界各国的奶牛遗传评估体系中,世界范围内奶牛育种工作已进入基因组选择时代。
我国于2012 年正式在奶牛育种中应用基因组选择技术,起步较晚,目前存在参考群规模不大、验证公牛数量太少等问题,这也是我国奶牛基因组选择发展的主要限制因素。本文概述了北美、欧洲和澳洲等12 个奶业发达国家的奶牛常规遗传评估和基因组遗传评估体系,包含参考群规模、评估所用芯片、每年评估次数、遗传评估模型、负责部门等信息,旨在为给我国奶牛遗传评估工作提供有利借鉴。
1 常规遗传评估和基因组遗传评估的核心技术要点
常规遗传评估的主要核心技术是以线型混合模型为基础的最佳线性无偏预测法(Best Linear Unbiased Prediction,BLUP)[2],主要评估产奶、体型、繁殖力、长寿性、乳房健康、产犊难易和泌乳指数共7 大类性状,根据不同的评估性状构建不同的模型,利用BLUP 法综合亲缘信息来计算个体估计育种值(Estimated Breeding Value,EBV)及其准确性。2001 年,Interbull 官方公布的《The Interbull Guidelines》指出,针对生产性状而言,动物模型优于公畜模型、多性状模型优于单性状模型、多泌乳期模型优于可重复性模型、测定日模型优于单泌乳期模型;针对阈性状而言,公畜模型和公畜-外祖父模型估计结果更为准确。Weller 等[3]、Boettcher等[4]和Hozé 等[5]的研究也得到了一致的结果;但对于其他非生产的线性性状,Interbull 并无更好的推荐。后裔测定技术的应用大力推动了奶牛育种的发展,时至今日,其仍是评估公牛种用价值最可靠的方法。
基因组选择技术是利用覆盖全基因组的SNP 遗传标记来估计个体育种值的方法。根据统计模型的不同,目前基因组育种值(GEBV)的计算方法可分为两类:①构建一定规模的参考群,利用参考群个体的表型信息和芯片信息估计每一个SNP 标记对选择性状的效应值,再检测候选个体的标记基因型,将参考群体估计的标记效应累加间接获得个体的GEBV;②用标记构建个体间关系矩阵(G 矩阵),替代用系谱信息构建的分子血缘关系矩阵(NRM 或A 矩阵)放入混合模型方程组(MME)中直接获得个体的GEBV。虽然估计基因组选择技术可靠性低于后裔测定育种值,但它可将后裔测定的5~6 年缩短为1.5 年左右,显著加快遗传进展,大幅加快奶牛育种进程。
基因组选择于2001 年被首次提出,但受限于当时的测序技术还相对落后,成本太高,导致在实际中应用甚少。2006 年,Schaeffer[6]基于加拿大荷斯坦奶牛群体的研究结果表明,实施基因组选择可以节省约92% 的育种成本。随着高通量测序技术的发展,2007年,首款商业化奶牛50K 全基因组SNP 芯片(Illumina BovineSNP50 assay)问世[7]。2009年,美国农业部和加拿大奶业网分别于1 月和8 月正式官方颁布包含GEBV 和传统育种值(系谱指数)的合并育种值,称为GPTA(Genomic Predicted Transmitting Ability),并将之应用于青年公牛选择,具有GPTA 的青年公牛可不经后裔测定直接作为种公牛使用。之后,世界各国陆续在奶牛育种中应用基因组选择技术。2012 年,中国农业大学奶牛育种团队及全国畜牧总站等单位合作成功建立了中国荷斯坦牛基因组选择技术平台,被农业农村部指定为我国荷斯坦青年公牛遗传评估的唯一方法并在全国各大公牛站推广应用。
2 北美部分国家奶牛遗传评估概况
2.1 美国奶牛遗传评估概况
2.1.1 常规遗传评估 早在20 世纪60 年代初,美国便开展奶牛常规遗传评估工作,随着技术的不断研发和工作的持续推进,目前已建立了较为完善的奶牛育种体系。美国的常规遗传评估由美国奶牛育种委员会(Council on Dairy Cattle Breeding,CDCB)组织执行,主要对荷斯坦牛、爱尔夏牛、瑞士褐牛、更赛牛和娟姗牛共5个品种进行遗传评估,每年评估3 次,分别在每年的4 月、8 月和12 月公布评估结果。
美国奶牛常规遗传主要评估除泌乳指数性状外的6大类性状,其中针对产奶性状,使用多性状重复力动物模型BLUP 法(MT-RP-AM-BLUP)进行评估;针对体型和繁殖力性状,使用MT-AM-BLUP 法;针对乳房健康性状,使用单性状重复力动物模型BLUP 法(ST-RPAM-BLUP);针对长寿性状,使用ST-BLUP-BLUP;产犊难易性状使用单性状公畜-外祖父模型BLUP 法(ST-S MGS-AM-BLUP)。各性状育种值估计可靠性使用综合了亲本、自身记录及后代信息的迭代方程进行计算。美国用综合性能指数(Total Performance Index,TPI)来衡量奶牛个体的种用价值,用终生净值(Lifetime Net Merit Dollars,NM)来衡量其经济价值。终生净值计算公式:
NM($)=16%×3.41×PTAP+19%×2.89×PTAF+(<1%)×0.0001×PTAM+22%×35×PL-10%×182×(SCS-3)+7%×32×UDC+4%×15×FLC-6%×23×SC+11%×27×DPR+5%×(25%×SCE+15%×DCE+15%×SSB+45%×DSB)其中,PTAP为乳蛋白量预期传递力,PTAF为乳脂量预期传递力,PTAM为产奶量预期传递力,PL为生产寿命预期传递力,SCS为体细胞评分预期传递力,UDC为乳房综合指数,FLC为肢蹄综合指数,SC为体型综合指数,DPR为女儿妊娠率预期传递力,SCE为种公牛产犊易产性(难产率)预期传递力,DCE为女儿产犊易产性(难产率)预期传递力,SSB为种公牛产犊死胎预期传递力,DSB为女儿产犊死胎预期传递力。本文所有公式均引自国际公牛组织(Interbull,https://interbull.org/)。
近期美国荷斯坦协会更新了TPI 公式,并将于2020 年4 月起用于官方评估。主要更新内容:①将乳脂量(PTAF)权重由17%上调至19%;②将乳蛋白量(PTAP)权重由21%下调至19%;③增加健康性状指数(HT),权重为2%;④生产寿命(PL)权重由4%上调至5%,增加的1%由乳用特征(DF)的权重转移而来;⑤繁殖力指数(FI)中增加首次产犊日龄(EFC),总权重保持不变仍为13%。更新后生产、健康与繁殖、体型这三类主要性状权重由此前的46%:28%:26%调整为46%:29%:25%。更新前的TPI 计算公式:其中,PTAP为乳蛋白量预期传递力,PTAF为乳脂量预期传递力,FE为饲喂效率,PTAT为体型预期传递力,DF为乳用特征预期传递力,UDC为乳房综合指数,FLC为肢蹄综合指数,PL为生产寿命预期传递力,HT为健康性状指数,LIV为奶牛生存力预期传递力,SCS为体细胞评分预期传递力,FI为繁殖力指数,DCE为女儿产犊易产性(难产率)预期传递力,DSB为女儿产犊死胎预期传递力。
2.1.2 基因组遗传评估 美国的奶牛基因组遗传评估工作同样由CDCB 组织执行,与常规遗传评估同时进行。其采用Illumina Bovine SNP50 芯片进行基因型检测,缺失基因型使用findhap.f90 软件填充,标记基因型效应采用传统的BLUP 法和改进的Bayesian 法进行估计,GEBV 计算采用选择指数法综合了系谱信息与基因组信息。在美国,奶牛各性状基因组育种值估计的平均可靠性达50% 以上[8],远高于传统系谱指数估计的可靠性。
美国于2009 年1 月首次官方公布奶牛基因组遗传评估结果,其基因组遗传评估体系相对成熟,参考群体的建立也更加完善。研究表明,预测青年公牛GEBV及其准确性时,使用由多个品种奶牛组建的核心群要优于单一群体来源组成的核心群[9-11],且核心群规模越大其估计准确性越高[12-13]。故针对不同的品种和性状,美国建立了相对应的参考群体,具体群体信息如表1 所示。
由于美国和加拿大的奶牛遗传联系性较高,故两国联合构建了荷斯坦验证公牛资源群体,以提高基因组选择的准确性。从2008 年开展奶牛全基因组选择育种开始,到2010 年两国已联合建立了由5 484 头荷斯坦验证公牛构成的资源群体,2019 年参考群规模已达到56 970 头荷斯坦公牛和391 037 头荷斯坦母牛。两国参加基因组检测的荷斯坦牛头数也逐年攀升,具体增长趋势如图1所示。

表1 美国基因组遗传评估参考群体
2.2 加拿大奶牛遗传评估概况
2.2.1 常规遗传评估 加拿大的荷斯坦育种工作由加拿大奶业网(CDN)与美国相关机构合作同平台开展,主要评估7 大类性状,评估时间相同。其中针对产奶、乳房健康性状,使用多性状极大似然法随机回归测定日模型BLUP 法(MT-ML-RR-TD-AM-BLUP)进行评估,体型、繁殖力性状使用MT-AM-BLUP,长寿性状使用ST-AM-BLUP,产犊难易性状使用阈模型BLUP法(TM-BLUP)。估计准确性计算遵循Meyer 的近似估计方法[14],评估结果常用生产性能指数(Lifetime Profit Index,LPI)来表示,计算公式:
LPI=51%×EBV产量+34%×EBV持续力+15%×EBV健康和繁殖
公式各模块中性状所占比例如表2 所示。
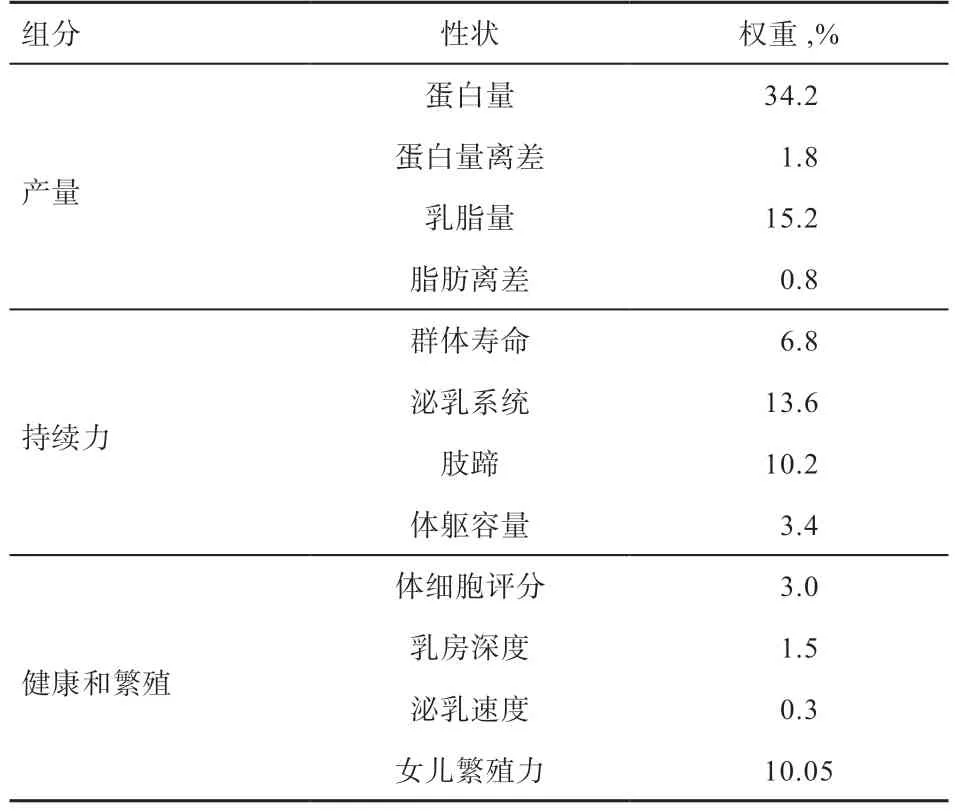
表2 2019 年加拿大LPI 各性状权重[15]
2.2.2 基因组遗传评估 加拿大于2009 年8 月首次官方公布荷斯坦奶牛的基因组育种值评估结果。其奶牛基因组遗传评估工作也由CDN 负责展开,同样使用Illumina SNP50K 芯片进行基因型检测,使用Flmpute软件进行基因型填充。对于青年公牛和青年母牛,加拿大公布的基因组育种值综合考虑系谱信息与直接基因育种值(Direct genomic value,DGV),其权重比为35:65,平均可靠性达60%;对于无后代女儿信息的成年母牛,以45:55 的权重比综合考虑EBV 和DGV,平均可靠性达70%;对于具有后代女儿信息的成年母牛和验证公牛,EBV 和DGV 的权重比为50:50,平均可靠性达90%[16]。
目前加拿大官方同时公布奶牛母牛的传统后裔测定育种值排名及基因组育种值排名;而对公牛仍使用传统后裔测定育种值进行排序,仅对各公牛进行基因组育种值的标注(数据来源于Interbull 和CDN)。
3 欧洲部分国家奶牛遗传评估概况
欧洲的奶业发达国家众多,奶牛养殖育种历史久远,但其基因组育种同样面临着挑战。自2008 年8 月荷兰首次公布奶牛基因组选择成绩后,欧洲各国纷纷将基因组选择应用于本国的奶牛遗传评估中,并于2009年秋季形成了以荷兰、德国、法国以及北欧三国(丹麦、瑞典、芬兰)为主的欧洲联合基因组选择体系。该体系融合了北美与欧洲地区地荷斯坦牛遗传血统,构建起几乎全覆盖荷斯坦牛遗传变异的基因组选择资源群体,成功克服了部分国家参考群体规模有限(尤其是优秀验证公牛的后裔测定成绩积累)的难题。据初步结果显示,欧洲联合基因组选择体系将育种值估计的可靠性提高约10%,极大地提升了欧洲在奶牛育种方面的竞争优势[17]。
3.1 荷兰奶牛遗传评估概况
3.1.1 常规遗传评估 荷兰奶牛常规遗传评估由荷兰奶牛育种公司(CRV)组织执行,主要评估7 大类性状,每年评估3 次,使用的育种值估计模型为ST-ML-RRTD-BLUP-AM-BLUP,统计多性状模型下的有效女儿贡献值(Multiple trait-Effective daughter contribution,MT-EDC)用于计算估计育种值的准确性[18]。其选择指数NVI 计算公式:
NVI=0.35×(-0.3×EBVlactose+2.1×EBVfat+4.1×EBVpro)+0.09×EBVlongevity+5.2×(EBVfertility-100)+5.2 ×(EBVSCS-100)+5.2×(EBVMS- 100)+6×(EBVF&L- 100)+2×(EBVcalving-100)
其中,lactose为乳糖,fat为乳脂量,pro为乳蛋白量,longevity为长寿性,fertility为繁殖力,SCS为体细胞评分,MS为乳房评分,F&L为肢蹄评分,calving为产犊难易。
3.1.2 基因组遗传评估 荷兰奶牛育种技术的研发处于国际领先水平,于2008 年8 月首次官方公布奶牛基因组遗传评估结果,整个奶牛基因组选择遗传评估工作交由CRV 组织负责。其采用自主设计的Illumina SNP60K芯片进行基因型检测,其中用于基因组选择的有效标记数达4.75 万个;基因组遗传评估频率为每周评估1 次,评估产奶、体型、繁殖力、长寿性、乳房健康、产犊难易共7 类性状。
2009 年荷兰奶牛基因组选择的参考群群体便有将近1 600 头验证公牛,并基于Bayesian 多重QTL 模型[19],使用从国家MACE(Multiple Across Country Evaluation)评估中获得的EBV,综合考虑系谱信息和基因组信息计算得到GEBV。研究结果表明,相对于父母均值(PA),乳蛋白率基因组育种值可靠性提升33%,其他性状的可靠性均有显著提升(数据来源于Interbull 和CRV,https://www.crv4all.be/)。
3.2 德国遗传评估概况
3.2.1 常规遗传评估 德国的奶牛常规遗传评估体系由VIT 德国奶牛数据中心负责,主要评估品种为荷斯坦牛、娟珊牛和丹麦红牛,主要评估7 大类性状,每年评估3 次,分别在每年的2 月、4 月和8 月公布评估结果;其遗传评估模型多采用MT-AM;使用MT-EDC 计算准确性。德国奶牛遗传评估结果常用综合育种值(German Total Merit Index,RZG)来衡量优劣,计算公式:
RZG=0.45×(100+0.24 × EBVfat+0.48×EBVpro)+0.2×EBVlongevity+0.07×EBVSCS+0.1×EBVfertility+0.075×EBVudder+0.075×EBVF&L+0.03×EBVcalving
其中,fat为乳脂率,pro为乳蛋白率,longevity为长寿性,SCS为体细胞评分,fertility为繁殖力,udder为乳房评分,F&L为肢蹄评分,calving为产犊难易。
3.2.2 基因组遗传评估 德国于2009 年8 月首次官方公布荷斯坦奶牛的基因组育种值评估结果。受到参考群规模的限制,其奶牛基因组遗传评估体系同样由德国、卢森堡与澳大利亚3 个国家组成,并统一交予VIT 负责;评估频率为每周1 次。评估所用的参考群体来自于欧洲基因组学国家(德国、法国、荷兰、北欧国家、西班牙和波兰)以及美国和加拿大的荷斯坦验证公牛,也包括了表型合格的德国已分型母牛。
德国采用包含了多基因效应和具有相同方差的随机SNP 效应的线性GBLUP 模型进行遗传评估;采用Illumina 50K 芯片,缺失的基因型通过Flmpute 软件填充。基于传统评估与MACE 评估相结合,计算有后代女儿的公牛的逆回归-MACE 育种值作为基因组遗传评估的表型数据;对于无MACE 估计育种值的公牛,则采用传统逆回归育种值作为基因组遗传评估的表型数据(数据来源于Interbull 和VIT,https://www.vit.de/en)。
3.3 英国奶牛遗传评估概况
3.3.1 常规遗传评估 英国的奶牛常规遗传评估由EGENES 公司负责,主要对荷斯坦牛、丹麦红牛、娟珊牛和瑞士褐牛共4 个品种进行遗传评估,主要评估7大类性状。公司使用MT-AM-BLUP 模型(根据不同性状会对模型进行一定的调整),准确性计算方法同MACE 评估的准确性计算方法[20];每年评估3 次,分别在每年的2 月、4 月和8 月公布评估结果。其遗传评估结果用选择指数(Profitable Lifetime Index,PLI)表示,计算公式:
PLI=PTAmilk- 0.027+PTAfat×0.08+1.71×PTApro+25.4×PTAlongevity+PTASCS- 0.19+2.16 × PTANR56+PTACI- 0.35+1.18×PTAudder+1.13 ×PTAF&L
其中,fat为乳脂率,pro为乳蛋白率,longevity为长寿性,SCS为体细胞评分,NR56为56 天不返情率,CI为产犊间隔,udder为乳房评分,F&L为肢蹄评分。
3.3.2 基因组遗传评估 英国并未参与欧洲联合基因组选择体系,其公牛基因组遗传评估工作由EGENES 公司负责,于2009 年夏季首次官方公布荷斯坦奶牛的基因组育种值评估结果,每年进行3 次评估。EGENES 公司选用Illumina 50K 芯片,用Flmpute 软件填充缺失基因型;采用20% 多基因效应下的线性模型,将基于传统评估与MACE 评估计算得到的各性状的逆回归育种值作为表型数据,用于基因组育种值的估计。
英国奶牛基因组选择参考群选取来自于英国、美国和加拿大的丹麦红牛公牛群,针对不同性状制定相应的群体大小,其中产奶性状和长寿性状群体大小为6 452头,繁殖力性状为5 013 头,乳房健康性状为4 869 头,产犊难易性状为5 491 头(数据来源于Interbull)。
3.4 法国
3.4.1 常规遗传评估 法国的奶牛常规遗传评估工作由GenEval 公司负责,主要对荷斯坦牛、诺曼底牛、西门塔尔牛等3 个品种进行遗传评估,主要评估7 大类性状。GenEval 公司使用ST-AM-BLUP 模型进行遗传评估,每年评估3 次,评估结果用综合效益指数(Index de Synthèse UPRA,ISU)来表示,计算公式:
其中,PY为乳蛋白率,FY为乳脂量,FP为乳脂率,PP为乳蛋白率,longevity为长寿性,SCS为体细胞评分,fertility为繁殖力,heifer fertility为青年牛繁殖力,udder为乳房评分,Milking speed为泌乳速度,CI为产犊间隔,conf为体型评分。
3.4.2 基因组遗传评估 法国于2009 年6 月首次官方公布荷斯坦奶牛的基因组育种值评估结果,法国奶牛基因组遗传评估工作由GenEval 公司负责,每年评估6 次(其中3 次为官方评估,分别在4 月、8 月和12 月)。其评估所用芯片为Illumina 50K SNP 芯片,通过Flmpute软件填充缺失基因型;采用单性状混合线性模型(包含了1 个多达3 000 个QTL 位点以及1 个残余多基因效应相关的同一状态回归)估计GEBV。估计GEBV 所需的表型数据来源于传统遗传评估获得的DYD 数据,如果缺少相应的DYD 数据(如外国公牛),则使用MACE 评估下的逆回归育种值数据。
评估使用的参考群体中,包含了来自于欧洲基因组学联盟的3 700 头荷斯坦公牛与6 015 头瑞士褐牛公牛以及来自于法国的2 650 头西门塔尔公牛和 2 300 头诺曼底公牛(数据来源于Interbull)。
3.5 北欧部分国家遗传评估概况
3.5.1 常规遗传评估 北欧奶牛常规遗传评估体系包含丹麦、芬兰与瑞典3 个国家,由北欧牛遗传评估中心负责评估。公司主要对荷斯坦奶牛进行遗传评估,主要评估7 大类性状,使用动物模型BLUP 法,通过综合了后代数量、遗传相关和经济权重的选择指数法计算准确性,每年进行4 次评估。所有评估性状均应用于北欧奶牛总经济指数计算中。
3.5.2 基因组遗传评估 北欧三国(丹麦、瑞典、芬兰)受到参考群体的限制,因此形成联合育种体系,于2009 年7 月官方公布奶牛基因组育种值评估结果,由北欧牛遗传评估中心负责评估工作。中心选择无多基因效应的线性模型与Illumina 50K SNP 芯片及更低密度填充的芯片,缺失的基因型通过Flmpute 软件填充,评估中的表型数据为基于传统育种和MACE 评估下的逆回归育种值,每年进行12 次评估。
评估中心在评估工作中,根据评估性状的不同,选择对应的参考群体,具体群体信息如表3 所示。
4 澳洲部分国家奶牛遗传评估概况
4.1 新西兰常规遗传评估 新西兰是澳洲主要的奶牛养殖国,其奶牛常规遗传评估主要评估的品种为荷斯坦牛、更赛牛、瑞士褐牛和丹麦红牛,评估除长寿性状外的6大类性状,其中生产性状每年评估14 次,泌乳指数性状每年评估5 次,体型性状每年评估4 次,其余3 个性状每年评估4 次,分别于2 月、3 月、5 月和11 月。主要采用MT-AM-BLUP 来估计育种值,其评估结果用育种价值(Breeding Worth,BW)用表示,BW 计算公式:
BW=12%×EBVfat+39%×EBVpro-15%×EBVmilk-6.5%×EBVSCS-14%×EBVweight+7.5%×EBVfertility+6%×EBVcalving
其中,fat为乳脂率,pro为乳蛋白率,milk为产奶量,SCS为体细胞评分,weight为活重,fertility为繁殖力,calving为产犊难易。
4.2 新西兰基因组遗传评估 新西兰于2011 年4 月首次官方公布奶牛基因组遗传评估结果,基因组遗传评估工作由新西兰家畜遗传改良公司(LIC)负责,每年评估2 次(2 月和5 月)。其参考群包含2 626 头荷斯坦公牛、1 639 头娟珊公牛和642 凯威公牛(数据来源于Interbull;DairyNZ,https://www.dairynz.co.nz/ 和LIC,https://www.licnz.com/),采用Illumina SNP54K芯片进行标记基因型检测,并使用VanRaden[21]提出的混合线性模型估计GEBV。LIC 在 GEBV 计算中还考虑了系谱指数信息,针对无表型数据的青年公牛生产性状、体重、繁殖性能、体细胞数和长寿性状,其 GEBV的估计可靠性在50%~67%,远高于采用系谱指数得到的估计育种值可靠性(34%)[22]。
5 总结
目前,全世界范围内奶牛育种工作已进入基因组选择时代,基因组选择技术的实施大幅度缩短了世代间隔并加快了遗传进展。目前,美国与加拿大合作开展北美联合评估,建立了“ 公牛为主+母牛”模式的大规模参考群,以及较为完善的育种体系,是基因组选择运用最为成熟、遗传进展最快的国家之一,其TPI 呈现的信息是多个国家奶牛育种规划的重要参考。由于基因组选择准确性较高,故美国对公牛使用基因育种值进行排序,将具有GPTA 的青年公牛不经后裔测定便直接作为种公牛使用,而加拿大和大部分欧洲国家则仍持谨慎态度,对公牛仍使用EBV 进行排序,仅补充GEBV 的标注。而欧洲七国的 EuroGenomics 合作项目使得各国的参考群规模得到极大提高,其GEBV 可靠性提升了约10%,基因组选择效果显著,极大地提升了欧洲在奶牛育种方面的竞争优势。同时,Interbull 在保证各个国家和公司的育种数据相对独立和保密的情况下整合数据,让所有参与者均从中受益。以新西兰为代表的澳洲国家,目前建立了含2 626 头荷斯坦公牛的参考群,其官方公布的公牛基因组育种值达60% 左右。这些奶业发达国家的育种工作经验和国际合作育种案例为我国的奶牛育种工作提供了良好的借鉴和思路。

表3 北欧基因组遗传评估参考群体
2012 年,我国开始在全国范围内启动荷斯坦公牛基因组遗传评估工作,在验证公牛数量太少的情况下另辟蹊径,建立了“ 母牛为主+公牛 ”模式的参考群,张哲等[23]、Koivula 等[24]均证实了在参考群中加入母牛有利于提高GEBV 估计准确性。截至2019 年12 月,我国奶牛基因组选择参考群规模已达9 227 头,主要性状的基因组育种值估计准确性达到了65%。但与欧美奶业发达国家相比,我国自育种牛较少以及由于参测牛场存在不连续性、取样操作不规范等造成的DHI 资料可靠性不高,导致基因组选择核心群公牛数量太少,截至2018 年12 月仅有273 头,而且每年参加基因组遗传评定的青年公牛数量也不足300 头,因此,我国基因组选择工作仍然需要进一步完善(数据来源于中国奶牛数据中心https://www.holstein.org.cn/)。建议:①持续扩大参考群体规模,以进一步提高我国荷斯坦奶牛GEBV 准确性;②推进GS 在青年公牛、核心群母牛和商业牧场的应用力度;③积极寻求参与国际合作育种项目的机会。
基因组选择在奶牛育种应用中还面临着很多机遇与挑战:①如何优化计算模型,缩短计算时长,提高估计准确性;②如何整合企业资源,扩大参考群规模及参考群中验证公牛数量,提高估计准确性;③如何充分利用基因组数据,整合品种资源、控制近交程度,获得更高的遗传进展等。

