基于U-net的直肠癌智能分割
谭俊杰 钟妤 黄泽斌
摘要:近年来,随着医学影像与大数据的发展,通过人工智能来处理图像,从而给医生带来辅助性的诊断参考成为广大学者的研究热点。文章利用U-net网络对直肠癌CT影像进行智能分割,加入了图像增强、批归一化等技巧缓解过拟合现象,并通过多次实验确定最佳初始学习速率和卷积核数目,在验证集上的Dice系数达到0.9329。实验表明,U-Net对小数据集的医学图像分割有很好效果,对正负样本极度偏斜的数据集,使用Dice系数可以准确地衡量分割的相似程度。
关键词:直肠癌;图像分割:U-net:Dice系数
中图分类号:TP301.6 文献标识码:A 文章编号:1006-8228(2020)08-18-03
0 引言
近年来,我国直肠癌发病率呈显著上升趋势,特别是在大城市,已跃居恶性肿瘤发病率排行榜前三位。研究显示,有65%的患者的发病时间是在40岁之后,其中男女比例为23:1,并且发病率最高的人群年龄分布在40岁-50岁。
医学图像分割是临床诊断和治疗的关键技术,能够提供准确可靠的诊断依据。随着深度学习中图像翻译算法的普遍应用,医学图像分割技术也获得了巨大的进展。尤其在U-Net网络结构被提出后,该网络一直在医学图像分割领域表现优秀,众多研究者利用此模型进行医学图像分割[1],并在此基础上提出改进。本文研究来自于第七届“泰迪杯”数据挖掘挑战赛B题,对直肠CT影像中的肿瘤分割问题提出一种基于深度学习U-Net网络结构的分割方法。
1 数据预处理
1.1 数据清洗与划分
本数据集共有107个病例,3029张CT影像及对应的掩模图,数据样本为512×512像素。而且每个病例有1745张CT影像序列。其中,有肿瘤区域的CT影像仅有860张且肿瘤区域在CT影像中占比较小,属于正负样本不平衡的数据集。因此,在划分数据集时,为使训练集、验证集和测试集的正负样本分布尽可能地相近,将病例数作为划分对象,训练集、验证集、测试集的个数分别设置为65个(即0.6)、16个和26个。
通过掩模图,可观察出肿瘤位置均在图像的一定区域内,因此,本文运用OpenCV库计算出肿瘤区域的最大最小像素值。经统计,长为126-382,宽为202-458的像素范围能包含所有肿瘤特征。因此,最终将数据集裁剪成256x256大小的影像,即过滤了无关特征,同时节省资源,减少计算量。
1.2 图像增强
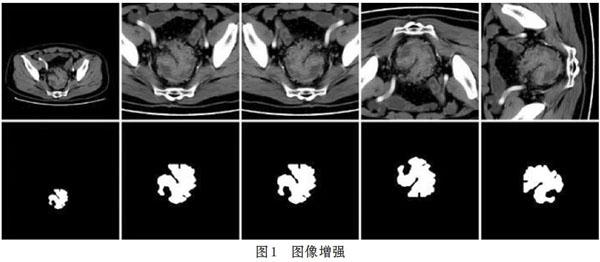
由于划分后的训练集只有1820张CT影像,对深度学习而言远远不足。因此,需要通过图像增强[2]技术扩充训练集样本量,本文使用水平翻转、垂直翻转、90度旋转以及翻转和旋转的组合将训练集样本量扩充至原来的6倍。图像增强对模型性能的训练有很大的提升,既可以减少过拟合现象的发生,又能提升模型的泛化能力。效果如图l所示。
2 U-Net模型训练
2.1 窗宽和窗位
由于CT影像的格式为DICOM,首先利用RadiAntDICOM Viewer软件得出每个病例CT序列的最佳窗宽和窗位,其次利用SimpleITK库读取CT影像的HU值,将HU值储存为数组后设置其最佳窗宽和窗位,使用灰度直方图均衡化技术提高具体器官的对比度。最后在模型训练前,对样本数据做均值方差归一化,大幅度减少计算量。
2.2 U-Net结构
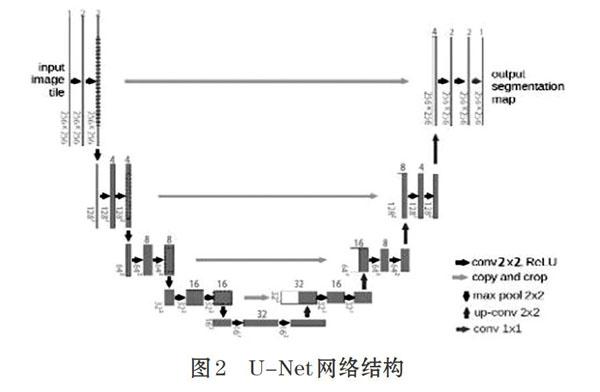
U-Net网络结构是全卷积神经网络[3](FCN)基础的改进,主要包括卷积、池化和反卷积三大步骤。其中,卷积操作中每一个卷积块包括两次卷积,然后是最大值池化操作,通过10次卷積操作后先结合对应大小的特征图进行反卷积操作使图像大小翻倍而通道数缩减一倍,再进行两次卷积操作,激活函数为ReLU。
本文针对医学图像数据集对U-Net网络结构进行超参数调整,由于肿瘤占图像范围区域小,提取的特征数太多易导致过拟合。经过多次实验,本文对256×256大小的输入图像初始提取两个特征,每经过一次最大值池化,提取的特征数翻倍。每个卷积核的大小设置为3x3,其中步长为l,Padding设置为same,这样即可保证卷积后的图像与原来图像大小一致。反卷积操作后再经过一层卷积层,卷积核大小为1x1,利用Sigmoid函数进行二分类。U-net网络结构如图2所示。
本文在卷积和反卷积操作后加入批归一化( Batch_Normalization)的操作,使上一层传递到下一层的参数分布保持一致,这既能加快训练速度,防止低层次神经网络的梯度在反向传播中消失,又能减少参数初始化的影响。
2.3 衡量指标
考虑到这个数据集中阳性和阴性样本的极端不平衡,因此本文模型的衡量指标选择使用Dice系数Ⅲ,模型的损失函数是1-Dice系数。两个轮廓区域所包含的点集分别用A、B来表示,通过计算公式可知,它可以衡量A、B的相似程度。
2.4 超参数
本文使用Adam算法优化损失函数,它结合了AdaGrad和RMSProp两种优化算法的特点,能对每次梯度的学习速率进行调整,同时后期不会使学习速率衰减至很小而导致模型无法学习。
经过多次实验,本文设置的初始学习速率为1x10-5,虽然小的学习速率需要更多的训练次数,但模型结果会比较好;而较大的学习速率使学习曲线大幅震荡,甚至还可能导致梯度朝着错误的方向更新。
每次迭代传入8张CT图和对应的掩模图进模型中训练,若批量( Batch)设置过小,模型需要消耗更多时间训练,且会使批归一化的效果不明显;若批量设置过大,虽然加快训练速度,但更容易出现过拟合。
3 实验结果
3.1 模型结果
本文使用Tensorflow为后端的Keras框架结构搭建U-Net网络[5],并在服务器上使用GPU对加速模型训练。模型参数共有31507个,训练一次所花费的时间为44秒,最终在第45次训练结束后,训练集上的Dice系数达到0.9803,验证集上的Dice系数达到0.9329,训练曲线如图3所示。
3.2 结果分析
通过观察训练曲线,模型没有明显过拟合,且梯度更新较大,在前10次训练中,训练集和验证集上的Dice相似,甚至出现欠拟合,原因在于使用图像增强技巧加大了模型的训练难度。训练10次后,学习速率出现缓和,但后来又比之前更加陡峭地上升,并且在第40次训练结束后过拟合越来越明显。
学习率越来越陡峭,这说明过拟合程度已经逐渐增大。并且当Dice值大于1后,模型会学习更多其他区域的特征而导致严重过拟合。因此当前训练集上的Dice系数大于1时,应停止训练。
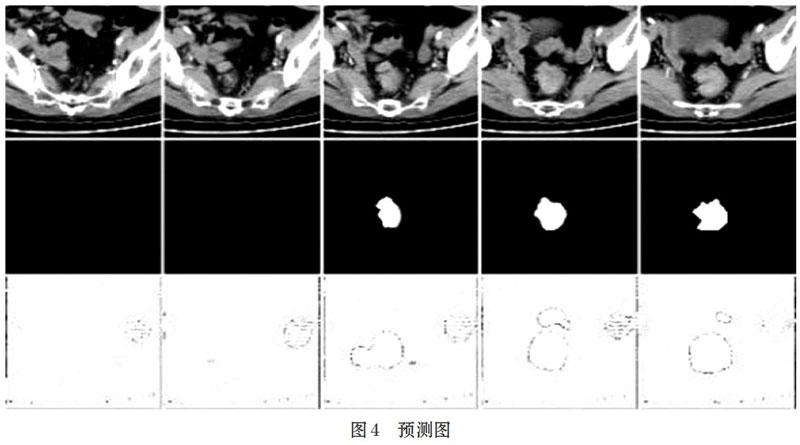
在图4中,第一行为测试集中随机一个病例的5张CT影像,第二行是对应的掩模图,第三行是模型的预测结果。通过观察得出U-Net模型能准确分类出有无肿瘤的CT影像,但预测的肿瘤边缘不够细致,而且存在一定的过拟合。同时,预测图中非肿瘤区域是灰白色,说明CT图像与轮廓滤波器进行卷积运算时,还不能把肿瘤区域和非肿瘤区域的轮廓区分得很明显,模型还需要进一步改进。
4 总结
本文基于深度学习中U-Net网络对直肠癌肿瘤区域进行图像智能分割。经过超参数调整后,验证集上的Dice系数达到0.9329,可以准确区分有无肿瘤的CT影像,一定程度上能够给予医生辅助性的诊断参考意见,减轻医生的工作负担。但预测的肿瘤区域轮廓不够细致,且模型容易出现过拟合,主要原因在于模型的训练样本还不够充足和丰富,相信在样本量增大后,模型一定有显著提升。
参考文献(References):
[1]吳锐帆,代海洋,杨坦,江颖,蔡志杰.直肠癌淋巴结转移的智能诊断研究[J].数学建模及其应用,2019.8(4):30-37
[2]李雯.基于深度卷积神经网络的CT图像肝脏肿瘤分割方法研究[D].中国科学院深圳先进技术研究院,2016.
[3]周鲁科,朱信忠.基于U-net网络的肺部肿瘤图像分割算法研究[J].信息与电脑(理论版),2018.5:41-44
[4]袁甜,程红阳,陈云虹,张海荣,王文军.基于U-Net网络的肺部CT图像分割算法[J].自动化与仪器仪表,2017.6:59-61
[5]张凯文.基于U-Net的肝脏CT图像分割研究[D].华南理工大学,2019.
作者简介:谭俊杰(1997-),男,广东佛山人,本科,主要研究方向:深度学习、数据挖掘
通讯作者:黄泽斌(2000-),男,广东普宁人,本科,主要研究方向:数据科学,数据挖掘

