吡虫啉降解菌群结构解析及菌株资源挖掘
排孜丽亚·帕尔哈提, 车 娟, 阿孜古力·库尔班, 张 伟
(新疆师范大学生命科学学院 新疆特殊环境物种保护与调控生物学实验室, 新疆 乌鲁木齐 830054)
吡虫啉(imidacloprid,IMI)又称咪蚜胺、灭虫精、蚜虱净,化学名称为1-(6-氯-3-吡啶甲基)-N-硝基亚咪唑烷-2-基胺,被广泛用于防治蚜虫、叶蝉、蓟马等刺吸式口器害虫[1-3]。由于具有杀虫谱广、低毒(对哺乳类动物)、高效等特点[4],有望取代高毒类有机磷、有机氯类农药。目前,已在世界120多个国家使用,并已成为应用最广的杀虫剂之一[5]。研究表明,IMI具有土壤滞留性,使用3年后,仍可在未直接施药的作物中检出残留物[3],虽然对哺乳动物毒性不大,但是其在作物和土壤中的残留对有益昆虫蜜蜂造成的“种群崩溃综合症”、对陆生无脊椎动物的致死作用造成的土壤动物生态系统的破坏等危害越来越被关注[6-8]。新疆地处亚洲腹地,是典型的大陆干旱半干旱环境,气候干旱少雨,土壤盐渍化严重,但非常适合棉花的栽培种植[9]。自20世纪80年代起,棉花在新疆大面积种植[10]。阿克苏等棉花主栽区连作现象极为突出,为了杀灭棉蚜等害虫,吡虫啉等农药被广泛、大量、常年施用,因此当地土壤农药残留及土壤生态环境问题引起了广泛关注[11]。由于生物修复具有低成本、无二次污染等优势被认为是解决土壤污染的很好方式,至今,国内外学者已经从自然界分离得到多种能降解吡虫啉的细菌及真菌等微生物[12-14],同时对其降解途径等也做了深入研究[15-16],但因为新疆特殊的土壤及气候条件,类似的研究成果并不一定适合用来解决当地的土壤农药污染问题。本研究拟从新疆当地土壤环境中分离吡虫啉降解菌株,为解决当地土壤农药污染提供菌株资源的同时,结合菌株功能系统地开展新疆特殊土壤环境特有微生物资源的研究。
1 材料与方法
1.1 材料
1.1.1 土样采集 2015年6月5日采集自阿克苏棉区连作10年以上的土壤(79°45′81″~79°46′55″E ,39°31′47″~39°45′50″N 之间)。按五点取样法使用内径6 cm的土钻垂直钻取1~20 cm深度土壤。将5个相同连作年限土壤合并为1个土样,按上述方法分别取连作10、15、20和30年的4个土样,-4 ℃保存,带回实验室-20 ℃保存。
1.1.2 培养基 ①基础无机盐培养基(g):硝酸铵 1.0,硫酸镁 0.2,硫酸二氢钾 0.5,磷酸氢二钾 1.5,氯化钠 0.5,硫酸铵 0.5,蒸馏水1 000 mL, 1×105Pa灭菌30 min;②PDA培养基[17];③高氏一号培养基[18];④牛肉膏蛋白胨培养基[19];⑤LB培养基[20]; ⑥Trace element solution培养基(g): 乙二胺四乙酸二钠 5.2, 氯化亚铁 1.5, 氯化锌 0.07, 四水氯化锰 0.1, 硼酸 0.062, 氯化钴 0.19, 二水合氯化铜 0.17, 氯化镍 0.24,蒸馏水1 000 mL,1×105Pa灭菌30 min;⑦Vitamin solution培养基(mg): 维生素H 2.00, 叶酸 2.00,盐酸吡哆醇10.00,硫胺素5.00, 维生素B25.00, 烟酸 5.00, D-泛酸钙5.00, 维生素B120.10, 对氨基苯甲酸 5.00,硫辛酸 5.00,蒸馏水1 000 mL,1×105Pa灭菌30 min。以上培养基用氢氧化钠调pH为8.0。固体培养基添加1.8%琼脂粉。吡虫啉直接加到培养基使其浓度为100 mg/L。
1.1.3 主要试剂 Ex-Taq酶、引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)、1492R(5′-GGTTACCTTGTTACGACTT-3′)和T-载体克隆试剂盒均购自上海生工。QIAGEN®DNeasy R ULtraClean®Microbial Kit提取试剂盒和胶回收试剂盒等均购自TIANGEN。吡虫啉原药购自广西南宁广酞农业化工有限公司(纯度≥98%)。
1.1.4 仪器与设备 BIOTECH-5BG-7000超净工作台(上海百典仪器设备有限公司);SIGMA 2-16P离心机(上海百典仪器设备有限公司);MLS-37500电子天平(北京欣凯隆生物科技有限公司);S1000PCR仪(Bio-Rad生物公司);ChemiDocTM XRS+凝胶成像系统(Alpha innotech);DYCZ-24DN电泳仪(北京六一仪器厂);2800H高压灭菌锅(武汉沙特利生物器材有限公司)。
1.2 方法
1.2.1 吡虫啉降解菌群的富集 称取采集到的4个土壤样品各2.5 g混匀后加入到含有100 mL基础无机盐+吡虫啉培养基(吡虫啉浓度100 mg/L)的250 mL三角瓶中,37 ℃、180 r/min摇床黑暗培养7 d后,做为液体种子按3%接种量传代于新的相同培养基中继续培养。连续传代6次以上,部分菌液保存,部分菌液用于提取菌群总DNA。按同样的方法富集高效氯氰菊酯(GX)和克百威(KBW)降解菌,与吡啉降解菌做对比。
1.2.2 吡虫啉降解菌群结构的测定 培养的菌群按照QIAGEN® DNeasyR ULtraClean® Microbial Kit步骤要求提取菌群总DNA,电泳检测合格后,委托天津诺禾致源生物科技有限公司对样品进行后继建库、数据处理和分析。具体方法参见文献[21]。
1.2.3 吡虫啉降解菌的分离、纯化 使用基础无机盐培养基、PDA培养基、高氏一号培养基、牛肉膏蛋白胨培养基、LB培养基(均添加吡虫啉100 mg/L)、通过高通量测序结果设计的培养基、参考KOMODO培养基数据库(the Known Media Database)(http://komodo.modelseed.org)设计的选择培养基(配方见1.2)从菌群中分离各种微生物。将富集的吡虫啉降解菌培养液进行适当倍数稀释,涂布于上述培养基,37 ℃、湿度55%恒温培养7~15 d后,挑取生长较快、菌落形态明显不同的单菌落,连续划线纯化培养,获得形态一致的单菌落,用于分类鉴定。
1.2.4 吡虫啉降解菌的分类鉴定 使用TIANGEN基因组DNA提取试剂盒提取各菌株的DNA作为模板,采用16S rRNA通用引物27F/1492R扩增目的片段。PCR扩增体系为:模板1.5 μL,上下游引物各1 μL,2×TaqPCR Green Mix 25 μL,无菌水补至50 μL。PCR扩增条件:95 ℃ 5 min;94 ℃ 50 s;54 ℃ 30 s;72 ℃ 30 s,共30个循环;72 ℃ 7 min。PCR扩增产物送至上海生工进行测序,其中16S rRNA序列相似度较低的菌株再通过TA克隆方法获得克隆子后再送至武汉昆泰瑞测序。 将菌株16S rRNA基因序列测序结果在NCBI网站上进行相似度比较,并提交到GenBank数据库获得菌株序列注册号(MN463036-MN463048、MN044953-MN044983、MN540804-MN540831)。利用Mega-X软件(版本10.0.5)对获得的菌株16S rRNA序列进行多序列拼接,构建系统发育树[22]。
2 结果与分析
2.1 吡虫啉降解菌群高通量测序
吡虫啉降解菌群测序结果显示,总序列数145 335条,总碱基数56 532 027 bp,有效序列133 290条,占总序列的91.71%,无效序列12 045条。序列平均长度424 bp(其他为高效氯氰菊酯和克百威菌群对比数据)(见表1)。
2.2 吡虫啉降解菌群群落结构分析
该菌群在门分类水平上,占比最高的依次为变形菌门(Proteobacteria,67.05%)、放线菌门(Actinobacteria,10.67%)、厚壁菌门(Firmicutes,9.99%)、绿弯菌门(Chloroflexi,4.1%)、酸杆菌门(Acidobacteria,2.84%);在纲分类水平上为γ变形菌纲(Gammaproteobacteria,54.79%)、杆菌纲(Bacilli,9.03%)、δ变形菌纲(Deltaproteobacteria,6.09%)、放线菌纲(Actinobacteria,5.96%)、α变形菌纲(Alphaproteobacteria,3.75%)、β-变形菌纲(Betaproteobacteria,2.28%)(图1);在科分类水平,为假单胞菌科(Pseudomonadaceae,22.55%)、肠杆菌科(Enterobacteriaceae,14.4%)、莫拉氏菌科(Moraxellaceae,12.54%)、气单胞菌科(Aeromonadaceae,4.24%)、李斯特氏菌科(Listeriaceae,2.19%)、链球菌科(Streptococcaceae,1.39%);在属分类水平,最多的依次为假羊胞菌属(Pseudomonas,16.33%)、莫拉氏菌属(Moraxella,11.14%)、埃希氏杆菌属(Escherichia,4.57%)、环丝菌属(Brochothrix,2.19%)等,未能分类占58.13%(GX和KBW分别为高效氯氰菊酯和克百威菌群数据)(图2)。
该菌群由1 377个OTU组成,与其他降解菌群相比较,特有OTU255个,核心OTU580个(图3)。
2.3 常规培养基分离吡虫啉降解菌株的分类鉴定
通过基础无机盐等常规培养基培养分离吡虫啉降解菌共分离获得48株菌。其中BP5等7株菌经16S rRNA测序并对比分别隶属于Nocardioides(BP5)、Sphingopyxis(BG5)、Shinella(BP2)、Cellulosimicrobium(BJ17)、Agromyces(BP8)、Bacillus(BJ7)、Bosea(BJW8)等属。菌株BP5与NocardioidesnitrophenolicusNSP 41同源性为98.08%,其他菌株相似度介于99.25%~100%之间(表2)。

图3 吡虫啉样品与GX和KBW样品韦恩图Fig.3 Venn diagram of imidacloprid samples with GX and KBW samples

表2 7株菌株的测序结果及同源性分析
2.4 利用Vitamin solution培养基和Trace element solution培养基分离菌株的分类鉴定
利用吡虫啉降解菌群结构数据设计的Vitamin solution培养基和Trace element solution培养基培养分离吡虫啉降细菌,共分离出25株菌株。经16S rRNA测序并对比分别隶属于Paenibacillus(BMV3)、Ammoniphilus(BMV5)、Planococcus(BMV7.1)、Brevibacillus(BMV14.1)、Paenibacillus(BLE1)、Bacillus(BLE3.1)、Sphingopyxis(BLE4)、Rhodococcus(BLE5)、Bacillus(BLE10.1)等属。其中BMV5与AmmoniphilusresinaeCC-RT-E同源性为98.5%,BMV7.1与PlanomicrobiumokeanokoitesIFO 12536同源性为98.29%,BLE3.1与BacillushalosaccharovoransE33同源性为98.02%,其他菌株同源性介于98.22%~99.59%之间(表3)。为确保测序的准确性,对相似度较低的10株菌株再次进行了16S rRNA克隆测序,序列比对结果分别隶属于Novosphingobium(BMVC4.1)、Ammoniphilus(BMVC5)、Planomicrobium(BMVC7.1)、Nocardioides(BMVC9)、Brevibacillus(BMVC10)、Nitratireductor(BLEC7)等属。克隆测序并对比后,其中BMVC4.1与NovosphingobiumacidiphilumDSM 19966同源性为97.69%,BMVC5与AmmoniphilusresinaeCC-RT-E同源性为98.22%,BLEC7与NitratireductorindicusC115同源性为97.73%,其他菌株同源性介于98.3%~99.33%之间(表4)。

表3 9株菌株的测序结果及同源性分析
2.5 吡虫啉降解菌群分离菌株的系统分类
通过常规培养基培养纯化吡虫啉降解菌,对从中获得的BP5、BG5、BP2等7株菌株的16S rRNA序列GenBank相似性最高的菌株序列进行比对并构建系统进化树。发现BJW8、BP2和BG5与Boseathiooxidansstrain BI-42、Sphingopyxisindicastrain DS15和Rhizobiumsubbaraonisstrain JC85等菌株聚为一簇。BJ7与Bacillussubtilissubsp. inaquosorum strain BGSC 3A28聚为一簇。BP5、BP8和BJ17与Nocardioidesnitrophenolicusstrain NSP 41、Cellulosimicrobiumcellulansstrain DSM 43879等菌株聚为一簇,其中BP5与BP8完全相同,可能是重复筛出的菌株(图4)。

表4 10株菌株的克隆测序结果及同源性分析
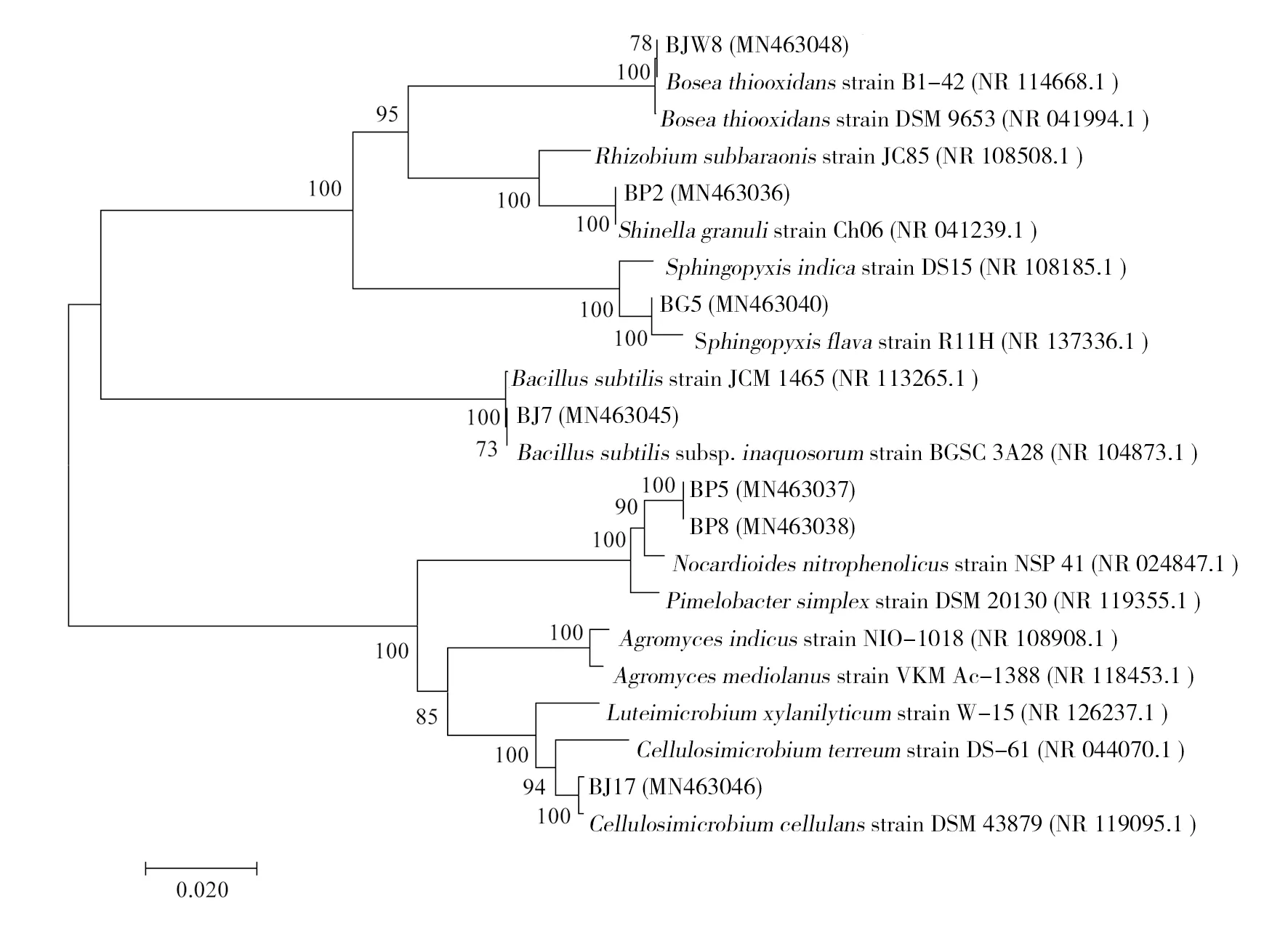
图4 基于16S rRNA基因序列构建的常规培养基分离株的系统发育树Fig.4 Phylogenetic tree construction of 16S rRNA gene based isolates in conventional medium
通过设计的培养基培养纯化吡虫啉降解菌群并对从中获得的BMV3、BMV5、BMV7.1等9株菌株的16S rRNA序列与GenBank相似性最高的菌株序列进行比对,构建系统进化树。结果表明,BLE1、BMV3分别与Paenibacilluslautusstrain JCM9073、Paenibacilluslautusstrain NBRC15380形成一簇,属于Paenibacillus属,这两株菌虽然从不同培养基分离得到,但可能是重复菌株。BLE3.1与Bacillushalosaccharovoransstrain E33、Bacillusmalikiistrain NCCP-662形成Bacillus属中的进化成员。其他各菌株也分别代表了Planomicrobium、Ammoniphilus、Sphingopyxis、Rhodococcus、Nocardioides等菌属。
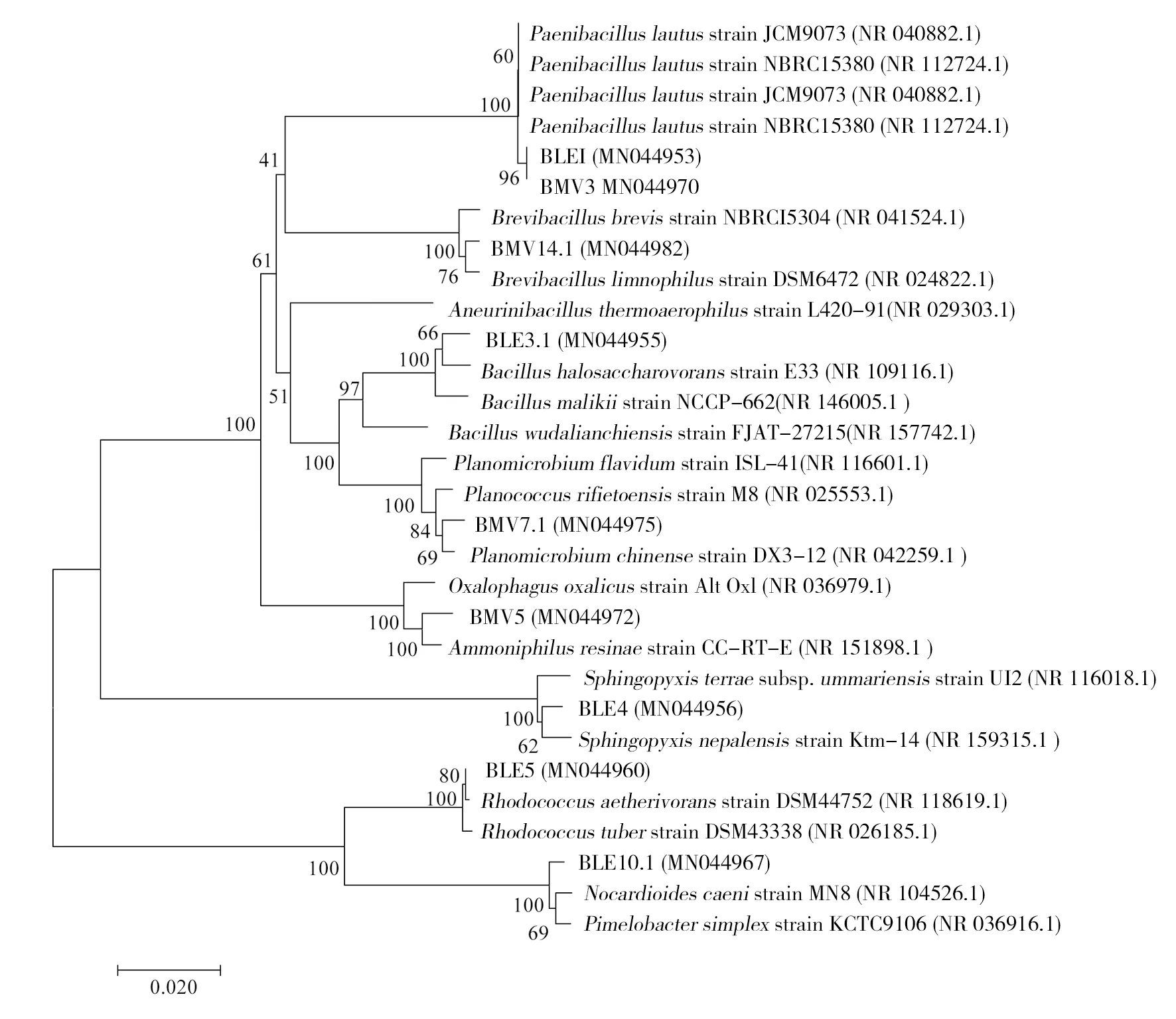
图5 基于16S rRNA基因序列构建的特殊培养基分离株的系统发育树Fig.5 Phylogenetic tree of a special medium isolated based on 16S rRNA gene sequence
3 讨 论
研究表明,作物的种植及与其相配套的栽培管理措施能极大地引起土壤微生物群落结构组成的变化[23-24]。新疆地区的气候和土壤性质造就了该地区拥有独特的土壤微生物群落组成,而棉花长达10年以上的连作和雷同的施用农药等栽培管理措施又促使该棉区土壤演替出了新的微生物群落结构组成[25-26]。本研究试图从新疆特殊土壤环境中筛选出耐受或者能降解吡虫啉农药的菌株资源,为后期盐碱土壤环境下修复吡虫啉农药污染和降解途径研究提供材料。结果发现,源自该长期连作棉田的土壤微生物群落在吡虫啉的胁迫下,富集到的菌群群落结构组成与原土壤微生物群落结构相比发生了巨大改变。首先,在门分类水平上占绝对优势的门类减少了,且所占比例发生很大变化。如Proteobacteria门所占比例由25%增加到67%以上,Firmicutes门所占比例无明显变化, Actinobacteria门所占比例减少了50%,而原来占比20%左右的Gemmatimonadetes门几乎未检测到。在属分类水平上能被分类的比例由原来的不到8%增加到41.9%,占比超过1%的Pseudomonas、Lactococcus、Moraxella等8个属都不是原土壤群落结构中的优势属。其次,与克百威和高效氯氰菊酯胁迫下形成的菌群相比,尽管菌群来源相同,但富集后的新菌群结构从门到属等任何水平上优势菌属及其丰度都截然不同。由此可见,不同的农药胁迫的确能富集到不同类型的菌群,且富集到的菌群结构组成趋于简单、优势菌属占比明显,能为进一步有目的的分离纯化微生物提供参考。
未培养微生物资源的开发利用一直是微生物领域的研究热点[27-30]。新疆特殊的地理环境孕育了特殊的微生物资源,但至今仍然缺少结合功能的系统性的开发[31]。第一,本研究结合近几年发展起来的高通量测序技术和相近菌属对培养条件和营养需求相似的特点[32-35],首先获取吡虫啉降解菌群的优势菌属特性,然后参考KOMODO网站设计了两种培养基试图从该菌群中分离到其特有的菌株。结果针对特有菌属设计的培养基与富集培养和常规培养基分离纯化获得的菌株有共同的也有不同的。如:在门水平上富集和常规培养基分离到的厚壁菌门(Firmicutes)占比最高,依次是变形菌门(Proteobacteria)、放线菌门(Actinobacteria),未分离出绿弯菌门(Chloroflexi)和酸杆菌门(Acidobacteria);在纲分类水平上,Bacilli占比最高,依次为Alphaproteobacteria、Gammaproteobacteria、Actinobacteria;在科分类水平上,Paenibacillaceae占比最高,依次为Bacillaceae、Sphingomonadaceae、Nocardiaceae。而经设计培养基分离到的BMV1菌株与汤鸣强等[3]已报道的降解吡虫啉的菌株BB-1(GU247754)均属Ochrobactrum目,BLE5、BMV9、BMV12与Kandil等[12]报道的能够降解吡虫啉的菌株MK6(KR052814)均属Mycobacteriaceae,BMV8与Sharma等[13]报道的降解吡虫啉菌株Enterobactersp. ATA1(JX233483)隶属于Gammaproteobacteria纲。通过基础无机盐培养基获得的菌株与高通量测序结果菌群结构组成设计的培养基获得的菌株的对比,BG5与BLE4Sphingopyxis属,BJ7与BLE3.1、BLE10.1同属Bacillus,其他菌株隶属不同菌属。通过基础无机盐培养基获得的菌株与克隆测序的菌株对比,BP5与BMVC9同属Nocardioides,其他菌株隶属不同菌属。第二,经设计培养基实际分离出来的菌株与高通量测序结果不完全一致,原因可能是部分菌株由于培养基或培养条件不能满足其生长需要而未能培养出来,而恰好符合另一些菌株的需求,但总体上分离获得潜在新种的概率有所提高。第三,根据高通量测序数据,吡虫啉降解菌群中依然有很多尚未分离到的菌株,因此,将第一和第二与读长能覆盖整个16S核糖体序列的三代测序结合起来,设计出更多选择培养基用来挖掘菌群中尽可能多的菌株,有待尝试。
首先,根据吡虫啉降解菌群结构组成,从门到属分类的菌群中包括尚未报道的降解吡虫啉的菌属,而实际分离到的部分菌株初步分类结果,确与已经报道的降解吡虫啉菌株有差异,原因可能是土壤样品采集地环境导致的,因为使用的培养基以及培养条件与已经报道的差异不大。来自不同环境下的不同菌属在降解吡虫啉的代谢通路上可能有所不同。其次,在分离纯化过程中发现,即使只使用不加吡虫啉的基础无机盐培养基也能分离到一些菌株。如果说在固体培养基中这些菌株是利用琼脂中少量的碳源生长,那么在菌群液体培养基中他们是如何协作的,他们存在的功能是否和吡虫啉的降解有关等问题都值得深入探究。另外,已有的降解吡虫啉的报道大多是关于单一菌株的,且具有较高的降解率,虽然是在吡虫啉作为唯一碳源胁迫下连续传代富集到的菌群,但其降解吡虫啉的能力并不强,可能该菌群中关键降解菌株的培养条件未得到满足,或菌群中存在与关键降解菌株竞争代谢中间产物的菌株。因此还需优化菌群培养条件、调整添加碳源种类,提高吡虫啉降解率,筛选关键降解菌株。

