纤维素降解菌XW08的筛选、鉴定及其降解条件优化
姜立春,朱 兰,顾相悦,费 禹,曾文佳,徐中文,李晓琴
(绵阳师范学院生命科学与技术学院,四川绵阳 621006)
0 引言
秸秆作为一种在农业生产中产生的重要的可再生生物质资源,在我国每年产量约在8.0亿t以上,占世界秸秆总产量的20%~30%[1].纤维素是其中含量最高的组分,所以家畜难以高效分解利用,故用来做家畜饲料的仅占我国秸秆总量15%~20%[2].纤维素能够利用植物的光合作用不断进行积累,是世界上分布最广、数量最大的可再生资源[3].然而,广泛的纤维素资源将被废弃或直接焚烧,造成极为严重的资源浪费,同时对环境产生巨大污染[4].为此,当前纤维素资源的开发和使用成了目前能源和环境的焦点问题.
纤维素是葡萄糖由β-1,4-糖苷键链接形成的生物大分子多聚糖,是构成植物细胞壁的重要组成部分之一,是自然界中含量最丰富的的碳水化合物[5].但是纤维素中含有大量的氢键,同时纤维素含结晶区和非结晶区,且结晶区较非结晶区更难降解.所以纤维素不易被降解,利用率低.通过微生物产生的酶作用,可以降低纤维素的聚合度生成葡萄糖[6],提高纤维素的利用率.
目前纤维素降解方式为化学水解与生物酶解法[7],化学水解法存在成本较高、操作较复杂,同时会导致二次污染等严重问题.而生物酶解法却拥有反应条件温和、设备要求简单、操作流程方便等优势,具有降解效率高、成本相对较低与安全环保等特点,是降解纤维素的较为理想的方法.生物酶解纤维素的第一步一般为纤维素酶,纤维素酶不是特称某一种酶,而是一个酶系的合称.其中包括内切和外切-β-葡聚糖酶、β-葡萄糖苷酶[8].纤维素酶广泛存在于动物、植物和微生物中.
自然界中存在大量能够产生纤维素酶的的微生物.至今,研究者已发现能分解纤维素的菌包括真菌、细菌和放线菌共53属,其中木霉菌和曲霉菌两种真菌产酶量最大、酶系最全[9],为筛选纤维素降解菌提供了重要的参考依据.尽管利用生物酶解法有众多优势,但是酶活力低一直是制约纤维素酶实际应用的一个重要原因[10].本试验从三台某木材厂存放木材下面的土壤和腐朽的木材作为研究对象,筛选出一株可高效降解纤维素的细菌.
1 材料与方法
1.1 材料
1.1.1 样本 2019年7月采集三台县某木材厂存放多年木材下面的土壤和腐朽的木材.
1.1.2 试剂和设备 羧甲基纤维素钠(CMC-Na)、刚果红、DNS试剂,PCR试剂盒,新华滤纸;紫外可见光分光度计、高速冷冻离心机、恒温培养箱、台式恒温培养摇床、超净工作台、梯度PCR仪、电泳系统、凝聚成像仪、超低温冰箱等.
1.1.3 培养基 牛肉膏蛋白胨培养基(g/L):牛肉膏3.0,蛋白胨10.0,NaCl 10.0,琼脂20.0,pH 7.2-7.4.
羧甲基纤维素钠培养基(g/L)[6]:CMC-Na 8.0,KH2PO41.5,NaH2PO4·7H2O 2.5,MgSO4·7H2O 0.3,FeSO4·7H2O 0.005,ZnCl20.001 5,MnSO40.001 5,(NH4)2SO42.0,pH 7.2.
产酶发酵培养基(g/L)[6]:NaCl 3.0 g、K2HPO41.5 g、MgSO4·7H2O 0.5 g、CaCl20.2 g,FeSO4·7H2O 5.0 mg,MnSO41.5 mg,ZnCl21.5 mg,CoCl22.0 mg,pH 调至7.2.以上培养基121 ℃高温灭菌20 min.
1.2 实验方法
1.2.1 菌株筛选 将采集土壤和腐朽木材混合浸泡在无菌0.9% NaCl溶液中,放置摇床振荡8 h.量取样本重悬液,依次梯度稀释到浓度为10-1、10-2、10-3、10-4、10-5、10-6,分别量取稀释液浓度10-4~10-6稀释度0.6 mL,涂布在羧甲基纤维素钠固体平板上,于28℃恒温培养60~96 h.将筛选获得的纤维素降解菌做好标记,在点接至CMC-Na固体平板上,28 ℃倒置培养60 h后,取1.0 mg/mL的刚果红染液适量倒入培养皿中,染色30 min,弃去染液,随后加入1.0 mol/L的NaCl溶液,浸泡20 min,根据透明圈与菌体直径比筛选高效降解纤维素的菌株[7].选择比值大菌落进行平板划线纯化,连续挑选获得纯培养的单菌落,观察菌落形态.将该菌株接种斜面上培养48 h,于4 ℃冰箱保存备用.
1.2.2 酶活性分析 (1) CMCase[9]菌液在4 ℃、6 000 r/min的条件下离心15 min,取0.50 mL上清液(粗酶液),加入0.50 mL 0.4 %羧甲基纤维素钠(CMC-Na)溶液,50℃恒温水浴锅中温育1.5 h,然后加入4 mL DNS试剂,放入沸水浴10 min终止反应,放置冷却后在520 nm下测吸光值,粗酶液沸水浴处理10 min后作为阴性对照,每个样品做3个平行试验.
(2)滤纸糖化酶(FPase)[11]将培养好的菌液在4 ℃、5 000 r/min的条件下离心20 min,取0.5 mL上清液(粗酶液).将新华滤纸剪成规格为10 mm×60 mm的条形,放入大试管中,取粗酶液0.5 mL,加入醋酸缓冲液1.0 mL,50 ℃恒温水浴1.0 h后取出,加入2.0 mL DNS试剂将反应终止,摇匀后于100 ℃水浴显色10 min,室温冷却后,在520 nm下测吸光值进行还原糖测定,粗酶液100 ℃水浴处理10 min作阴性对照,每个样品做3个平行试验.
1.2.3 酶活测定 酶活力单位定义:在1.0 min内每毫升酶液催化底物生成1.0 μg葡萄糖所需要的酶量,即1.0 U/mL.酶活力计算公式:Y=(1000×A×V)/(0.5×T),式中A:根据标准曲线获得净葡萄糖的浓度(g/L);V:总体积;0.5:测定时;量取粗酶液的毫升数;T:酶解反应时间.
1.3 菌株分子生物学鉴定
1.3.1 形态观察 菌株在高氏一号培固体养基上28 ℃培养36 h后进行菌落形态观察,放于4 ℃冰箱中保存.挑取单一菌落进行革兰氏染色,显微镜下观察其菌体形态结构.
1.3.2 基因组提取与16S rRNA基因扩增 基因组DNA提取:参考文献[12]方法提取细菌总DNA,略有修改.0.9%琼脂糖电泳检测.PCR反应体系:37.5 uL ddH2O、5.0 uL 10×PCR buffer、4.0 uL dNTPs (各2.5 mmol/L)、1.0 uL F27:(5′-AGAGTTTGATCCTGGCTCAG-3′) (20 umol/L)、1.0 uL R1492:(5′-GGTTACCTTGTTACGACTT-3′) (20 umol/L)、2.0 uL模板、0.5 uL Taq DNA聚合酶(5.0 ug/uL),反应总体积为50 uL.扩增循环体系:94 ℃预变性3.5 min,94 ℃变性35 s,57 ℃退火35 s,72 ℃延伸90 s.经33个循环后,72 ℃延伸9 min.PCR扩增产物经1.0%电泳检测后,其产物回收纯化与pMD-19T载体连接,转入感受态细胞JM109,筛选阳性重组子,邮寄成都擎科梓熙生物技术公司测序,其核酸序列用于构建系统发育树.
1.3.3 基于16S rRNA序列构建系统发育树 通过测序获得的16S rRNA序列用NCBI中BLAST搜索与GenBank数据库进行相似性分析,从而获得相应菌株16S rRNA序列,在Clustal X(1.83)程序包中进行序列比对分析.基于软件MEGA6.06中的邻接法(Neighbor-Joining)法构建系统进化树[13],确定菌株分类地位.
1.4 单因素优化
培养基组分的优化,本试验对筛选出的菌种的培养条件做了以下单因素的优化试验:培养时长、氮源及浓度、碳源及浓度、酸碱度、培养温度、接种量.培养时间的控制是在培养的发酵液每隔12 h取样测其酶活.碳源为玉米粉、微晶纤维素、蔗糖、CMC、麦芽糖、乳糖、淀粉、葡萄糖为碳源,其浓度为10 g/L;氮源为牛肉膏、NH4Cl、NaNO3、NH4NO3、KNO3、(NH4)2SO4、胰蛋白胨、尿素、酵母膏为氮源,其浓度为5 g/L.
1.5 酶学性质
1.5.1温度 采用最适条件对菌株进行培养,获得粗酶液,考察不同温度(20、30、40、50、60 ℃)对CMCase与FPase活性的影响.测定在不同温度下该菌种产生酶的稳定性,确定其温度适应范围.
1.5.2 pH 采用最适条件对菌株进行培养,获得粗酶液,考察不同pH值(4.0、5.0、6.0、7.0、8.0)对CMCase与FPase活性的影响.于最适温度下酶解30 min,测定两种酶活性.随后测定在不同pH下该菌株产生酶的稳定性,确定其pH适应范围.
1.6 综合最佳条件进行培养测定
在确定的最佳碳源和氮源及其最佳浓度、温度、pH及装液量下,将菌恒温160 r/min下摇床培养相应时间后测定Lip酶酶活,与上各单因素测定酶活比较在综合条件下其酶活是否有大的改进,从而可以了解菌产酶条件的优化情况.
2 结果与分析
2.1 菌株筛选
通过初筛培养分离得到20株菌可产生纤维素酶,对其进行纯培养菌株.在纤维素培养基上经过刚果红染色后,观察试验中菌落大小与透明圈大的大小如表1所示.以培养80 h后透明圈直径与菌落直径之比进行了评估,共有9株纤维素降解菌株水解圈大于3,其中菌株XW08透明圈直径与菌落直径最大为6.28,其次XW05为4.46和XW15为4.09,选择XW08作为目标菌株进行液体培养,测其CMCase酶活性和FPase活性.对分离出的20株菌做两种酶活力测定试验,结果如表2所示.获得菌株XW08的CMCase和FPase酶活力最大,分别为6.12和6.56,结合初筛结果,将XW08作为待测菌种进一步后续试验.

2.2 基因组提取与PCR扩增
采用常规SDS法提取DNA,然后进行琼脂糖凝胶电泳检测显示基因组提取条带比较清晰、整齐和均匀,浓度较高,可用于16S rRNA基因序列扩增.采用通用引物扩增,将16S rRNA基因邮寄到成都擎科梓熙生物技术有限公司进行测序,将测序片段采用DNA Baser软件拼接在一起,得到16S rRNA基因序列.
2.3 菌株XW08鉴定
2.3.1 形态观察 菌株XW08在含CMCase的营养琼脂固体平板上生长,有土腥味,白色,菌落小而致密,边缘不规则,表面粗糙,干而不透明,正反面颜色不一致,不易挑起,不产生色素.通过形态观察,初步判定为放线菌.经革兰氏染色后,发现细胞呈杆状,革兰氏染色为紫色,呈阳性.
2.3.2 16S rDNA系统发育树构建 将测序得到的菌株XW08的16S rDNA序列与数据库中已注册的16S rDNA序列用BLAST搜索进行序列相似性比较分析,其结果如图1所示.供试菌株的16S rDNA序列与链霉菌属的菌株同源相似度大部分在97 %以上,目前认为,当2个细菌的16S rDNA的相似性大于95%时,可将其归为同一属,而同源性较高的序列均来自Streptomyces,初步将此菌株归为链霉菌属.同时XW08在系统发育树上与StreptomycesexfoliatusHBUM174569 (FJ486375)和Streptomycessp. MJM1121 (KP137363)聚集在同一个分支中,且16S rDNA序列的同源相似性为99%以上.结合其形态特征及生理生化特性与链霉菌属较为一致,因此,将菌株XW08属于链霉菌属,命名为Streptomycessp. XW08.

图1 基于16S rDNA序列构建菌株XW08和相关菌的系统发育树Fig.1 Phylogenetic tree of strain xw08 and related bacteria based on 16S rDNA sequence
2.4 发酵条件对产酶活性的影响
2.4.1 培养时间 图2中显示从12 h开始CMCase和FPase的产量都开始缓慢上升直至60 h,60 h过后两种酶的酶活开始大幅度上升,至72 h时酶的活力达到最大,而72 h过后酶活力开始急速下降.且由图中可知CMCase相比FPase活力更高,在72 h时CMCase活性达到了5.68 U/mL,FPase活性达到4.36 U/mL.因此,后续培养时间为72 h.

图2 培养时间产酶活性的影响Fig.2 Effect of culture time on enzyme activity
2.4.2 最适碳源与浓度 碳源的种类与性能对纤维素酶活力会产生较大影响,况且菌株种类对于不同的碳源及其最适浓度也会产生不同的代谢偏好.本试验考察不同碳源对酶活的影响,由图3可知,以淀粉为唯一碳源产酶活力最高,CMCase和FPase活力都为最高分别达到了5.65和4.32 U/mL,葡萄糖次之.因此淀粉是XW08菌种的最适碳源.分别设置5、10、20、30、40、50 g/L 6个浓度考察淀粉对酶活性的影响,由图4可知当淀粉含量在5~10 g/L时,两种酶活力缓慢上升,随着淀粉浓度升高酶活力迅速上升.在20 g/L时两种酶的酶活力均达到最高分别为8.12、6.54 U/mL.当过了20 g/L时酶活力都开始下降.由此得出最适淀粉含量为20 g/L.
2.4.3 最适氮源与浓度 氮源是组成蛋白质酶和核酸的重要元素,对于纤维素酶的形成有重大影响.由图5可知,当胰蛋白胨和酵母膏作为氮源时,CMCase和FPase活性都较高,以酵母膏为唯一氮源时CMCase和FPase活性分别为7.22、5.96 U/mL.所以酵母膏是菌株XW08最适氮源.分别设置1、2、4、6、8、10 g/L 6个浓度,来考察酵母膏对酶活性的影响,由图6得到在酵母膏含量为6 g/L时CMCase和FPase的活力最高,分别为8.23、6.65 U/mL.当含量小于6 g/L时随着酵母膏浓度增加酶活力增加,而浓度高于6 g/L两种酶活力开始逐渐降低.由此得出酵母膏含量达到6 g/L时,菌株产酶的量最多.

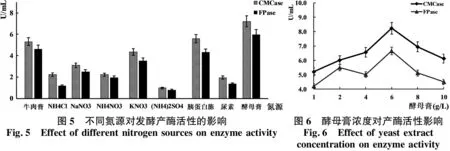
2.4.4 初始pH值 培养环境对于产纤维素酶的菌株生长有一定的影响,过酸或过碱都会有不同的影响.由图7中数据可得随着酸碱度的增加CMCase和FPase的酶活都在上升,菌株的产酶量不断增加,pH6.0时CMCase和FPase酶活达到最高,分别为14.12、10.86 U/mL,而pH高于6.0时酶活性急速下降.为此,该菌株的较适生长酸碱度为6.0.
2.4.5 温度 不同温度对微生物生存及代谢的影响极其重要,本试验中分别测定20 ℃~42 ℃温度区间对XW08菌株产酶活力的影响,见图8.温度范围在20 ℃~35 ℃范围时酶的产量相对缓慢上升,在25 ℃时两种酶的产量相差最小,在35℃时CMCase酶和FPase的产量最高,分别为27.52、21.28 U/mL,25 ℃~35 ℃的酶活力测出均在15 U/mL以上,温度适应范围较广.为此,35 ℃为该菌种的较适生长温度.
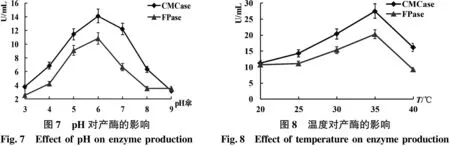
2.4.6 接种量 接种量过多,菌株生长繁殖的速度较快,资源有限,菌群生长发育则会受到限制.接种量少,菌株适应期增长,需要更多时间来繁殖达到对数生长期,影响产酶活性.由图9可得当接种量在2%~5%时两种酶的产量都比较高,均在25 U/mL以上.当接种量达到5%时,两种酶的产量达到最大,CMCase和FPase分别为38.35、32.28 U/mL.在接种量超过5%之后,接种量的多少对酶的产量造成的影响不大,则较适的接种量为5%.

图9 接种量对产酶的影响Fig.9 Effect of inoculation amount on enzyme production
2.5 酶学性质
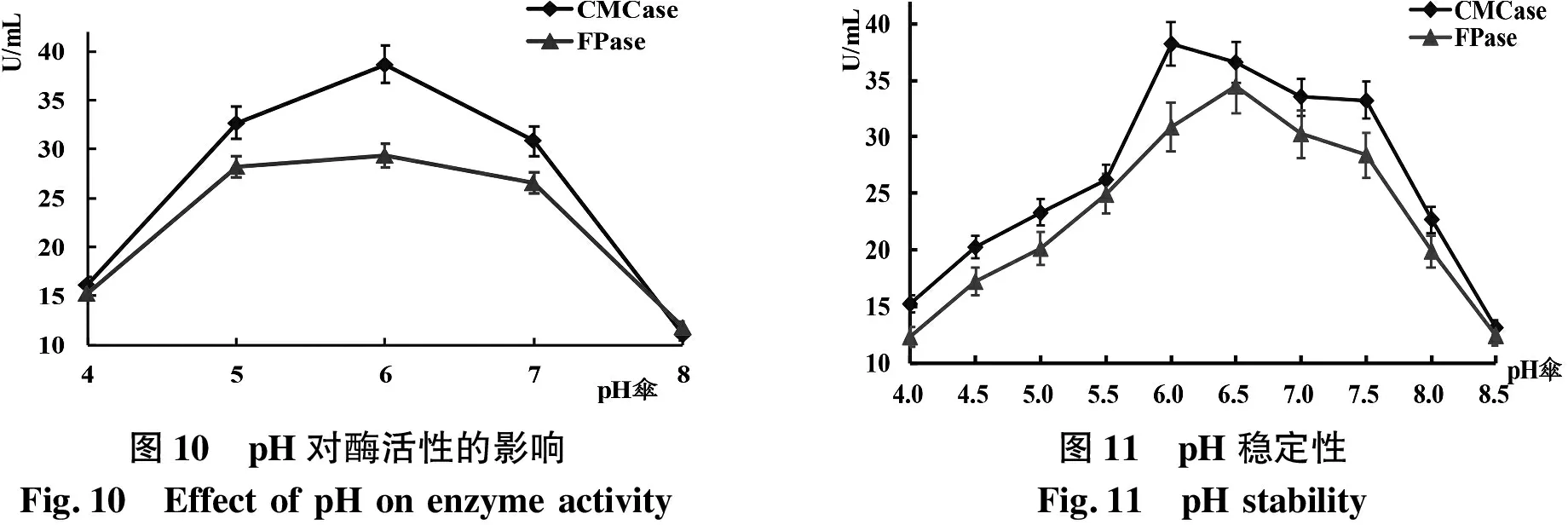
2.5.1 最适pH与稳定性 菌种生长时会受到外界条件的影响,酶是一种具有高度特异性和高效催化化学反应能力的物质,使化学反应在极为温和的条件下也能发生反应.虽然有这些优点,但其不稳定,易被破环.由图10可知,酶在pH5.5~7.5的活性都比较高,在pH为6.0时,两种酶活性最高,CMCase和FPase分别为38.68和29.32 U/mL.当pH低于5.0或高于7.0时,酶的活性下降速度较快,酶活性明显受到抑制.所以酶在中性环境下活性最佳,最适pH为6.0.酶在不同pH条件下处理1.5 h,考察酶的稳定性,由图11可知随之pH增高逐渐升高,而在pH5.5~7.5酶活力都比较高,CMCase和FPase
活性最高,分别为38.24、30.85 U/mL,最适应的pH5.5~7.5.
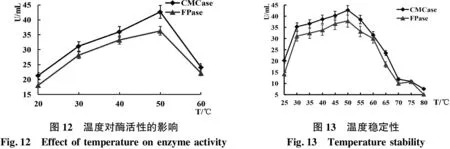
2.5.2 最适温度与稳定性 酶是一种蛋白成分,不同温度条件下对酶的活性有较大影响,温度过高会对酶产生不可逆的破坏,即蛋白质的变性.由图12可知随之温度的升高,酶的活性迅速增高,当温度为50 ℃时,CMCase和FPase的活性达到最高,分别为42.68和36.32 U/mL.高于50 ℃时酶活性迅速受到抑制,因此,温度对该酶活性有较大影响.酶在不同温度条件下处理1.5 h,考察酶的稳定性,见图13.结果表明,XW08菌株的温度适应范围较宽,30 ℃~60 ℃的温度对菌株XW08酶活性影响较小,两种酶的活性都比较高,高于60℃两种酶的活性开始大幅度受到抑制,而高于80 ℃ 1.5 h后,CMCase和FPase酶活性降低到最高酶活的17.76%以下.因此,菌株最适应的温度在30 ℃~60 ℃.
2.6 XW08综合优化条件培养
根据前面的优化结果,即20 g/L淀粉,6 g/L酵母膏,pH6.0、35 ℃和装液量为70 mL的条件下,对接种XW08后的三角瓶置于160 r/min摇床上进行摇床培养72 h,取部分5 000 r/min离心15 min得到取上清液为粗酶液.测定CMCase和FPase酶活性分别为43.56、36.72 U/mL.
3 讨论
从木材厂的土壤和朽木中分离筛选得到的Streptomycessp. XW08(纤维素分解链霉菌XW08),通过对菌株的16S rRNA系统发育分析,发现菌株XW08在遗传上与链霉菌属相似度极高,因此,将该菌命名为Streptomycessp. XW08.XW08菌株为分离的20个菌株中初始CMCase与FPase酶活最高,分别为6.12与6.56 U/mL.在经过本次试验的各项培养条件优化后,该菌株单位酶活明显增加,最优条件下培养72 h后,CMCase和FPase酶活分别达到43.56、36.72 U/mL.菌株XW08的CMCase活性明显高于王伟等[14]报道的链霉属菌株YDL3和HLF4,也高于王洪媛等[15]和李子昂等[16]报道的菌株,在CMCase活性上具有独特优势.虽然XW08菌株在条件优化后产酶活性在已报道的菌株中处于较高水平,但是酶活性大小不仅与菌株及其来源、培养基组成成分、培养时间与测定酶活采用的方法等有关,还与纤维素酶的反应条件(底物浓度、时间和温度等)有关[17],因此,对其工艺仍需进一步深入研究.这些将为采用生物法降解纤维素提供试验依据,进而提高纤维素资源利用率.

