非洲猪瘟病毒复制相关基因E165R启动子预测及其甲基化分析
张彦兵 张石磊 刘良波 谢全亮 孙延鸣*
(1石河子大学动物科技学院,新疆石河子 832003;2石河子大学生命科学学院,新疆石河子 832003)
非洲猪瘟(African Swine Fever,ASF)自从 2018年秋季传入我国,迅速在我国各地区猪场点状散发,给我国的养猪业造成了严重的损失。非洲猪瘟病毒感染猪体,会造成猪体皮肤发红、发绀,出现皮炎,强毒感染可导致猪多个脏器出血,例如,脾脏、肾脏等出血,对猪体造成严重的病理损伤。非洲猪瘟病毒(African Swine Fever Virus,ASFV)感染的靶细胞主要是猪原代肺泡巨噬细胞、树突状细胞以及骨髓细胞等[1]。
ASFV作为一类DNA病毒,基因组大小为170~190 kb,两端为可变区域,中间为保守的核心区域;由于两端高度可变使得ASFV具有较多的基因型[2]。ASFV有多层外膜包裹着病毒核酸,因此该病毒具有较强的耐受性,对酸碱不敏感;在潮湿的条件下,ASFV在猪肉中存活数年之久[3]。ASFV的灭活疫苗不能提供免疫保护,基因缺失疫苗仍然具有一定的毒力,甚至可导致猪只死亡[4]。到目前为止,仍然没有安全有效的ASF疫苗用于猪群的免疫保护。目前,为了防控ASFV的传播,主要采取生物安全防控措施来遏制病毒。
一般DNA甲基化发生在基因序列的cg位点,cg位点又称CpG位点,其甲基化主要由DNA甲基化转移酶(DNMTs)调控,哺乳动物DNA甲基化转移酶主要包括DNMT3B、DNMT3A和DNMT1,基因启动子区域高甲基化抑制基因表达,启动子区域低甲基化则利于基因转录表达[5]。ASFV复制过程中完全借助宿主细胞进行自我复制,宿主细胞自身DNA甲基化修饰是否参与限制病毒基因复制的过程,从而抑制ASFV的复制,还不清楚。
本文首次预测分析ASFV的复制相关基因E165R的启动子及其甲基化,通过生物信息学分析E165R基因的启动子活性和转录因子,进一步利用生物信息学手段预测其启动子甲基化区域。从DNA甲基化角度,初步分析病毒基因E165R启动子特性及其DNA甲基化位点,为研究DNA甲基化调控ASFV的感染奠定初步基础。
1 材料和方法
研究中所用的主要软件:DNA star、MEGA 7、NCBI Blast、BDGP、Promoter 2.0 Prediction、AliBaba 2.1、Meth Primer、QUMA等。
1.1 启动子预测及进化树构建
据报道,ASFV的E165R基因与病毒的复制相关。为了分析其启动子序列,首先从National Center for Biotechnology Information(NCBI) 获得中国 ASFV毒株 (African swine fever virus isolate Pig/HLJ/2018) 全序列(基因登录号:MK333180.1)[6],从序列中选取E165R的编码区,进一步截取其上游500 bp的序列,作为其启动子。在NCBI数据库中比对E165R的启动子并下载其他ASFV毒株的序列,利用MEGA7构建进化树。
1.2 启动子活性及转录起始位点预测
利用在线预测软件BDGP(Berkeley Drosophila Genome Project)(http://www.fruitfly.org/seq_tools/promoter.html)预测启动子活性,打开软件,输入所要预测的DNA序列,点击submit按钮,软件开始运行分析。
利用 Promoter 2.0 Prediction Server[7]软件 (http://www.cbs.dtu.dk/services/Promoter/)预测启动子活性区域,打开软件输入被预测的DNA序列。
1.3 启动子区域转录因子预测
打开在线软件AliBaba 2.1(http://gene-regulation.com/pub/programs/alibaba2/index.html),在对话框中将要预测的DNA序列粘贴进去,选择中等条件的参数,点击START按钮运行程序。
1.4 甲基化区域预测及甲基化检测引物设计
利用在线软件Meth Primer(Meth Primer-Design Primers for Methylation PCRs)[8]预测可能的甲基化区域,进一步设计甲基化检测引物,打开浏览器,输入IP地址:http://www.urogene.org/cgi-bin/methprimer/met hprimer.cgi,网址点开后会显示软件的操作界面,将被预测的DNA序列粘贴到对话框中,点击submit按钮,软件运行显示结果,会给出几组引物序列选项。
1.5 DNA甲基化序列分析
DNA甲基化数据分析quantification tool for methylation analysis[9]软件,打开浏览器并输入 IP:http://quma.cdb.riken.jp/按下Enter键打开软件,genomic对话框输入基因组DNA序列,第二和第三对话框输入,测序获得的重亚硫酸盐处理转化后的序列,具体见表1。
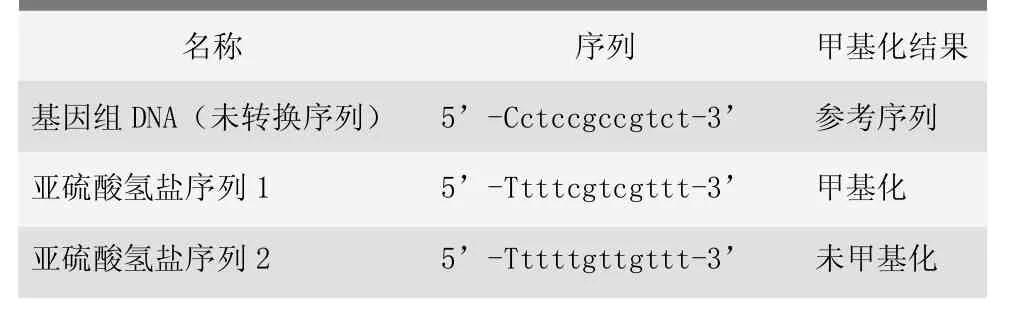
表1 重亚硫酸盐处理转化后的序列比对
2 结果
2.1 ASFV dUTPase(E165R)基因序列分析及其启动子区域预测
从NCBI数据库中,搜索ASFV的基因信息,选择下载 Pig/HLJ/2018(MK333180),发现此毒株的E165R基因ATG是从166 507位点开始的,本研究截取E165R上游500 bp左右作为其启动子(165 985~166 506)。
2.2 ASFV dUTPase(E165R)基因启动子同源进化分析
在NCBI下载35株ASFV的E165R基因启动子序列,ASFV毒株分离时间跨度为1978—2019年,利用MEGA 7软件中NJ-1000方法构建同源进化树;通过进化树可以看到,不同来源的ASFV分为2大群,中国新分离的毒株与俄罗斯远东地区分离的毒株同源关系很近,ASFV E165R基因启动子处于165 985~166 506之间,选择的E165R基因启动子非常保守,在2大群中间都较保守(图1)。在此基础上,进一步分析E165R基因的启动子活性及甲基化位点CpG才有实际意义,也能较为普遍地反映ASFV毒株的E165R基因启动子甲基化区域。

图1 E165R基因启动子同源进化树分析
2.3 ASFV dUTPase(E165R)基因启动子活性预测
软件运行完毕之后会输出预测结果,结果中score的值越高表示预测结果越可信,图2中score值在0.98~1.0之间,表明预测结果可信度较高,所预测的启动子活性可信。此外,启动子序列中每一个预测结果里字体变大的碱基,表示转录起始位点。

图2 BDGP软件预测结果显示
利用Promoter 2.0预测软件,预测启动子转录起始位点,打开软件输入被预测的DNA序列,单击submit选项运行软件。运行结束后会更新页面,显示出预测的结果。预测结果显示,在第200位附近有一个潜在的转录起始位点,score值为0.619(图3);因此,预测在第200位点上下游区域为可能的启动子活性中心。为了更加直观地显示所预测的E165R基因启动子活性,在序列上将2种软件所预测的活性区域进行标注,同时标出基因的ORF区域(图4)。

图3 Promoter2.0预测结果显示界面

图4 ASFV E165R基因启动预测活性区域及其ORF区域
2.4 ASFV dUTPase(E165R)基因启动子转录因子预测
在预测E165R基因启动子的基础上,进一步利用在线软件AliBaba 2.1预测其转录因子,共预测到7个转录因子,分别是 c-Jun、c-Fos、GCN4、2个 Sp1、Oct-1和C/EBPalp(图5)。其中较为常见的有c-Jun、Sp1和C/EBPalp。转录因子的成功预测与启动子活性的成功预测是相一致的。在转录因子Sp1、Oct-1和C/EBPalp结合序列上均含有cg甲基化位点,cg位点已标出(图 5)。

图5 E165R(dUTPase)基因启动子转录因子预测及CG位点标注
2.5 ASFV dUTPase(E165R)基因启动子甲基化检测方法
利用meth primer软件设计并预测可能的甲基化区域,打开软件输入序列,勾选亚硫酸氢盐测序法(Bisulfite sequencing PCR,BSP)选项并点击提交,可以获得5对BSP的引物,软件根据引物CG含量、退火温度等综合后进行了排名,实际情况下首选F1和R1为目标引物,如图6。BSP引物上不能含有被测的CpG位点,这样可以保证甲基化和非甲基化的片段高效扩增,保证扩增片段的准确性。BSP扩增的片段要通过TA克隆连接克隆载体,进一步挑选克隆产物进行测序分析,每个处理组克隆数目不少于15个。
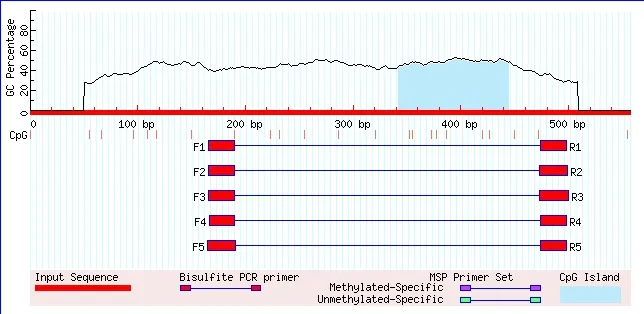
图6 BSP引物设计结果示意图
除了设计BSP引物,还可利用软件设计甲基化特异性的引物 PCR(Methylation-specific PCR,MSP),结果见图7。在同一区域有2条引物,但每条引物的3’UTR处至少包含一个CpG位点,M引物为XXXCG,则与之对应的U引物为XXXTG。MSP法可以根据甲基化或非甲基化引物扩增的条带,迅速确定基因片段是否存在甲基化。若MF扩增出条带,UF无条带则说明在这一区域存在甲基化;反过来,UF能扩增出条带,而MF不能扩增出条带,则表明此区域不存在甲基化。
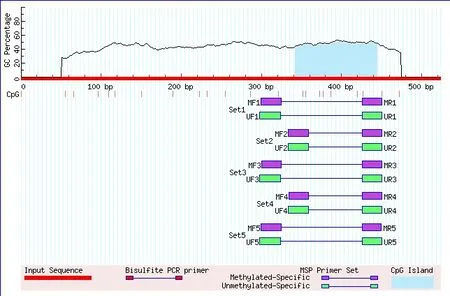
图7 MSP引物设计结果示意图
3 讨论
ASFV编码150~200种蛋白,许多病毒蛋白与病毒的复制密切相关[10-12]。如,E165R是ASFV复制所需的关键蛋白[13]。在猪的基因甲基化研究中,发现大量的重复序列处于高甲基化水平[14],而这些重复序列恰恰源于病毒基因。说明宿主一般通过高甲基化的形式来限制某些有害基因的表达。
宿主DNA甲基化修饰可能参与限制病毒基因的复制,例如乙型肝炎病毒基因中存在高甲基化与其病毒蛋白复制有关[15,16]。本文选择ASFV复制相关基因E165R为研究对象,分析其启动子甲基化等的特性。虽然ASFV基因型较多,但是ASFV的E165R基因启动子序列较保守;通过在线软件分析预测到ASFV基因的3个潜在启动子活性中心,说明预测的E165R启动子在理论上具有活性,将来还需进一步克隆其启动子并验证活性。此外,在启动子上预测到了常见的一些转录因子,例如c-Jun、c-Fos、GCN4、Sp1、C/EBPalp[17,18]等。值得注意的是,在转录因子Sp1和Oct-1的结合区域上含有cg甲基化位点,如果这些结合位点发生甲基化则会抑制转录因子与启动子的结合,最终抑制E165R基因启动子激活从而限制蛋白的合成。
DNA甲基化的检测方法有BSP(重亚硫酸氢盐测序法)和MSP(甲基化特异性检测法)等。BSP是甲基化检测的金标准,优点是检测结果准确、可信度高;缺点是成本较高[5]。MSP的检测方法主要优点是快捷迅速、成本低;但是,MSP对于半甲基化的基因不易区分甲基化水平的高低。本文针对E165R启动子序列成功设计了甲基化检测引物,包括BSP和MSP。
4 小结
研究中对E165R基因启动子进行了生物信息学分析,预测到了E165R基因的启动子活性区域、启动子转录因子及甲基化区域。此外,本文对ASFV的复制相关基因E165R启动子设计了甲基化检测引物,为将来在甲基化方向深入研究ASFV的感染复制机理打下了基础。

