基于免疫相关lncRNAs的胃癌早期诊断和预后风险模型的构建和验证
王洪英 李岩 韩鑫浩 魏孝礼 贾慧珣 袁文娟 张秋菊
胃癌是消化系统最常见的恶性肿瘤之一,全球每年约有77万人死于胃癌,我国每年因其死亡人数超过37万,肿瘤致死率高[1]。研究表明,中晚期胃癌预后普遍较差,尽管运用放化疗、靶向治疗等手段可在一定程度上延长患者的生存时间,但5年生存率仍然不足50%,而早期患者术后5年生存率超过90%[2]。因此胃癌早期筛查和预后预测对改善患者的生存状况具有重要意义。
长链非编码RNA(Long non-coding RNAs,lncRNAs)是指长度大于200个核苷酸的非编码RNA,广泛参与表观遗传、细胞周期调控等众多生命活动,在胃癌的发生发展和转移等过程中发挥重要作用[3-4]。同时lncRNAs被证实与肿瘤免疫密切相关[5]。但针对胃癌诊断和预后与免疫lncRNAs的关系研究较少。
因此本研究拟通过提取胃癌免疫相关lncRNAs,构建早期诊断和预后模型,为胃癌早期诊断和预后预测提供理论依据。
1 资料与方法
1.1 数据来源及样本筛选
下载The Cancer Genome Atlas(TCGA,https://xena.ucsc.edu/)数据库中的STAD数据集作为训练集,用于胃癌早期诊断和预后模型的构建;下载gene expression omnibus(GEO,https://www.ncbi.nlm.nih.gov/)数据库中的GSE54129[6]和GSE62254[7]数据集作为验证集,分别用于诊断及预后模型的外部验证;免疫基因列表下载自immport数据库(https:www.immport.org/)。
对TCGA-STAD和GSE62254中患者年龄、性别、肿瘤分期及总生存期信息缺失或总生存时间小于30天的样本进行剔除;利用GSE54129进行外部验证时未使用临床信息,因此纳入全部样本。
1.2 基因注释及初步基因筛选
从GENCODE(https://www.gencodegenes.org/)数据库下载人类基因注释文件(gencode.v38.annotatio版本),对3个数据集进行基因注释后取交集,获得lncRNAs 1 961个,免疫基因1 739个。过滤低丰度基因(所有样本counts之和小于1)后,最终纳入1 957个lncRNAs和1 716个免疫基因。
1.3 差异表达及免疫相关lncRNAs筛选
利用R语言DESeq2[8]包对训练集进行差异基因分析,选取调整后P<0.05且|logFC| >1.5倍的lncRNAs作为差异表达lncRNAs(Differentially expressed lncRNAs,DElncRNAs)。将其与免疫基因行Pearson相关性分析,过滤出相关系数|r| >0.5且P<0.0001的免疫DElncRNAs以供后续分析。
1.4 胃癌诊断模型的构建和评价
将免疫DElncRNAs在不同肿瘤分期(Ⅰ、Ⅱ、Ⅲ期)患者中进行t检验,筛选Ⅰ、Ⅱ、Ⅲ期两两比较均有统计学差异(P<0.05),(Ⅰ期vs.Ⅱ期)和(Ⅰ期vs.Ⅲ期)有差异,或者(Ⅱ期vs.Ⅲ期)和(Ⅰ期vs.Ⅲ期)有差异的基因作为胃癌早期诊断候选标志物,构建早期诊断logistic模型chass=∑(基因Exp×βi)。对模型进行Hosmer-Lemeshow拟合优度检验,并绘制ROC曲线评价模型的诊断价值。
1.5 胃癌预后基因标签的构建和评价
选取至少在两个分期(Ⅰ、Ⅱ、Ⅲ期)中差异表达的免疫DElncRNAs进行单因素Cox回归分析,获得影响患者总生存期的免疫lncRNAs(P<0.05),通过LASSO回归构建预后基因标签riskscore=∑(基因Exp×βi)。根据标签风险得分中位数将患者分为高、低风险组进行Kaplan-Meier生存分析。一致性指数(C-index)和ROC曲线分析用于评估基因标签的预测性能。
1.6 胃癌预后风险模型的构建和评价
将基因标签与患者临床指标(年龄、性别、肿瘤分期)进行单因素及多因素Cox回归分析,筛选出影响胃癌患者总生存率的独立预后因素,构建预后风险模型nomogramscore=∑(临床指标×βi),并绘制列线图。而后利用C-index、ROC曲线和校准曲线(Calibration curve,CV)分析来评价模型的预测能力。
1.7 统计学分析
所有统计采用R4.1.1版本完成,临床计量资料以中位数和四分位数表示,组间比较采用Wilcoxon秩和检验(数据不服从正态分布);计数资料以频数(%)表示,组间比较采用χ2检验或Fisher精确检验,P<0.05为差异具有统计学意义。
2 结果
2.1 临床信息纳入情况
经样本筛选过程,训练集STAD中共纳入320例肿瘤组织样本和32例癌旁组织样本,验证集GSE54129中共纳入111例肿瘤组织样本和21例癌旁组织样本,验证集GSE62254中共纳入300例肿瘤组织样本。由于仅在预后模型的构建与验证中使用临床信息,因此表1中仅列出预后模型训练集和验证集的临床信息。其中年龄(P=0.0014)和肿瘤分期(P<0.0001)的组间分布差异具有统计学意义。

表1 预后模型训练集及验证集胃癌患者临床信息
2.2 免疫相关DElncRNAs
经差异基因筛选,获得344个DElncRNAs,与免疫基因行Pearson相关分析后获得免疫lncRNAs 194个,其中上调基因125个,下调基因69个(图1A)。

图1 差异表达lncRNAs和免疫lncRNAs筛选Figure 1 Screening for differentially expressed lncRNAs and immune-lncRNAsNote:A.Volcano plot of differentially expressed lncRNAs in gastric and adjacent tissues;B.Venn diagram of differentially expressed immune-lncRNAs in different stages of gastric cancer.
2.3 胃癌早期诊断模型的构建和评价
通过t检验获得17个(Ⅰ期vs.Ⅱ期)和(Ⅰ期vs.Ⅲ期)有差异的lncRNAs,以及2个(Ⅱ期vs.Ⅲ期)和(Ⅰ期vs.Ⅲ期)有差异的lncRNAs,共计19个免疫lncRNAs(图1B)。利用logistic回归分析,最终筛选出9个免疫lncRNAs(P<0.05,表2)构建胃癌早期诊断模型:class=0.6409×(FAM30AExp)-1.5488×(LINC02716Exp)-1.4529×(LINC00671Exp)-1.5132×(LINC00582Exp)-1.1187×(LINC00330Exp)+0.9539×(BARX1-DTExp)-0.9510×(HAND2-AS1Exp)+0.8511×(LINC01968Exp)+1.9620×(LINC02465Exp)。模型构建及验证的拟合优度检验P值分别为0.9982 和1.0000(表2),ROC曲线下面积分别为0.991和0.958(图2),表明该模型具备出色的早期诊断能力。

表2 Logistic回归分析筛选胃癌早期诊断lncRNAs
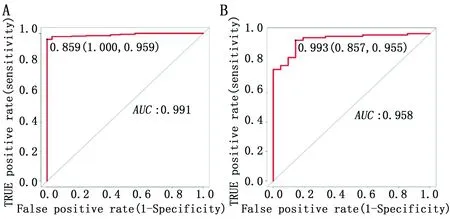
图2 胃癌早期诊断模型ROC曲线图Figure 2 ROC curves of the early diagnostic model of gastric cancerNote:A-B.ROC plot of the early diagnosis model in TCGA STAD and GSE54129 data sets.
2.4 胃癌预后基因标签构建及评价
利用t检验选取至少在两个分期中差异表达的免疫lncRNAs共计65个(图1B)。通过单因素Cox回归分析获得6个影响患者总生存时间的免疫lncRNAs(表3),并构建基于LASSO回归(图3)的胃癌预后基因标签:risk score=0.0721×(LINC01711Exp)+0.0861(LINC02544Exp)+0.1156×(TDRG1Exp)+0.1605(ERICH3-AS1Exp)+0.0207×(LINC01416Exp)+0.1868×(RPH3AL-AS1Exp)。

表3 TCGA STAD单因素Cox回归分析结果
根据该标签计算每个患者的风险得分,以其中位数将患者分为高、低风险组,Kaplan-Meier生存分析显示,训练集结果与验证集一致,即高风险组患者总生存率明显低于低风险组患者(图4A和4C)。该生存状态预测基因标签构建及验证的C-index分别为0.61和0.59(表4),1、3、5年总生存率ROC曲线下面积分别为0.623、0.623、0.677(图4B)和0.581、0.613、0.622(图4D),可见此标签具备中等水平的预后预测能力。
2.5 胃癌预后风险模型的构建及评价
将基因标签联合患者临床指标(年龄、性别、肿瘤分期)进行单因素及多因素Cox回归分析,发现基因标签、年龄和肿瘤分期是影响胃癌患者总生存率的独立预后因素(表4)。因此本研究最终采用这三个指标构建胃癌预后风险模型:Nomogram score=1.1959×risk score+0.0315×age+0.4154×(stage Ⅲ)+1.2811×(stage Ⅳ),并绘制列线图(图5)。

图3 LASSO回归分析Figure 3 The LASSO regression analysesNote:A.Plot of the six immune-lncRNAs′ regression coefficients in the LASSO regression analysis;B.LASSO regression analysis for the plot with the smallest λ values of screening variables(No.of lncRNAs=6).
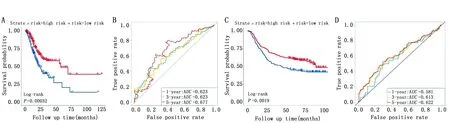
图4 基因标签Kaplan-Meier生存曲线和ROC曲线图Figure 4 The Kaplan-Meier survival curves and ROC curve of the gene signatureNote:A-B.The Kaplan-Meier survival curve and ROC curves of the gene signature in TCGA STAD;C-D.The Kaplan-Meier survival curves and ROC curve of the gene signature in GSE62254.
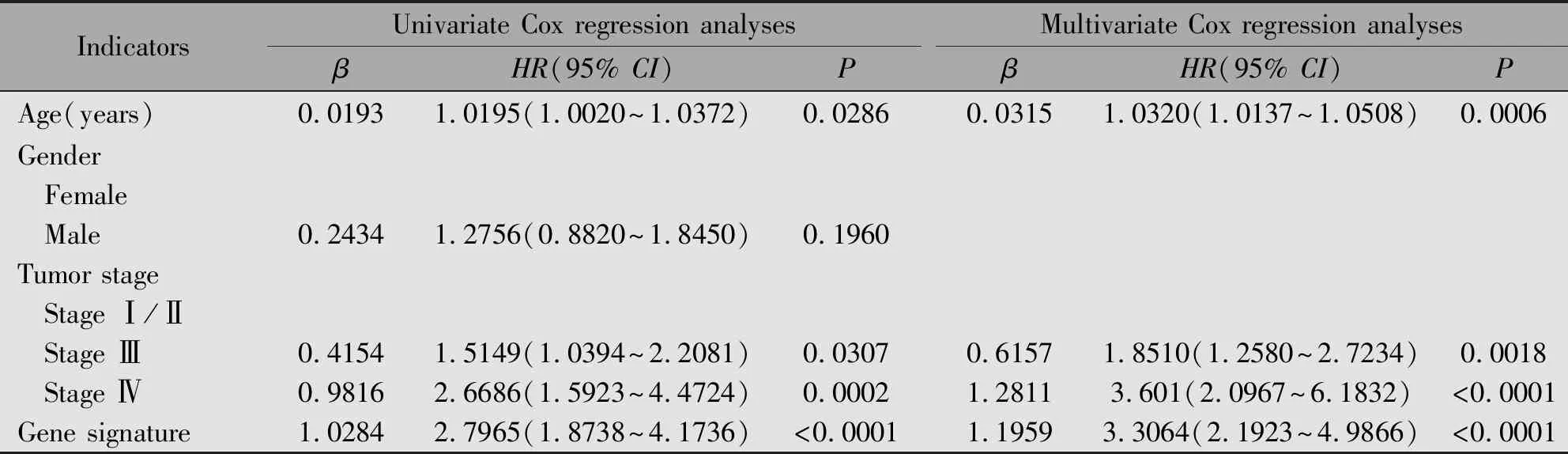
表4 TCGA STAD基因标签联合临床指标的单因素及多因素Cox回归分析结果
此用于生存状态预测的预后风险模型构建及验证的C-index分别为0.68和0.72,1、3、5年总生存率的ROC曲线下面积分别为0.707、0.705、0.755(图6A)和0.779、0.745、0.732(图6B),校准曲线分析显示模型预测一致性好(曲线斜率接近1,图7)。表明此预后模型具备较高的预测价值。

图5 预后风险模型列线图Figure 5 Nomogram of the prognostic risk model

图6 预后风险模型ROC曲线图Figure 6 ROC plot of the prognostic risk modelNote:A-B.ROC plots of the prognostic risk model in TCGA STAD and GSE62254 data sets.
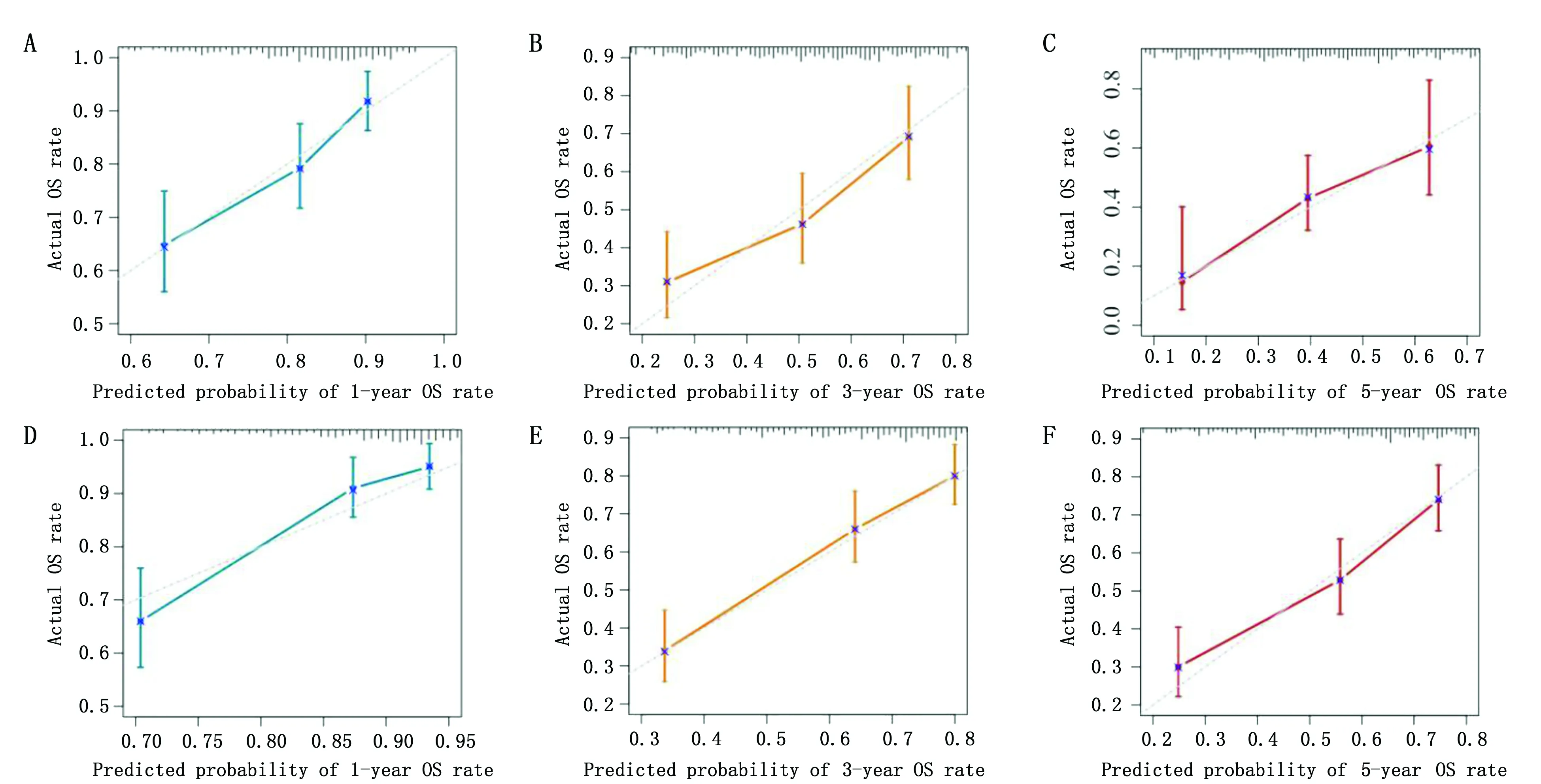
图7 预后风险模型校准曲线图Figure 7 Calibration curve of the prognostic risk modelNote:A-C.Calibration curve of 1-,3-,and 5-year overall survival rate of the prognostic risk model in TCGA STAD;D-F.Calibration curve of 1-,3-,and 5-year overall survival rate of the prognostic risk model in GSE62254.
3 讨论
胃癌是消化系统最常见的恶性肿瘤之一,致死率位列恶性肿瘤第4位[1],晚期胃癌5年生存率不足50%[2]。手术是目前唯一根治胃癌的方法,但由于早期症状不典型,且缺乏有效的早期诊断标志物,多数患者一经确诊即为中晚期,错过最佳手术时间。因此,对患者而言,挖掘有效且灵敏的早期诊断标志物意义重大。目前,临床常用诊断标志物如癌胚抗原(CEA)、甲胎蛋白(AFP)等对早期胃癌检出率不足20%[9]。研究发现lncRNAs可以稳定存在于患者的体液中可被检测到,其在肿瘤中的异常表达具有癌症特异性,对早期胃癌检出率超过60%[10],可作为新型胃癌早期诊断标志物。与此同时,lncRNAs的表达水平与胃癌分化程度、浸润深度和TNM分期密切相关[11],LINC01133等已被证明与胃癌预后有关[12],基于lncRNAs构建的预后模型也具备较高的预测能力[13]。此外研究发现,lncRNAs与肿瘤免疫也存在一定关联,lncRNAs异常表达的胃癌患者免疫检查点表达水平显著降低,这部分患者对免疫治疗不敏感,且预后较差[14]。但目前针对免疫lncRNAs与胃癌早期诊断和预后的关系研究较少。
本研究成功构建了基于9个免疫lncRNAs(LINC02465、BARX1-DT、LINC01968、FAM30A、LINC02716、LINC00582、LINC00671、LINC00330、HAND2-AS1)的胃癌早期诊断模型。实验发现,LINC02465在幽门螺旋杆菌感染的胃癌患者和健康人群的生物样本(血清、血浆、尿液或唾液)中差异表达,通过对患者血清/血浆等体液中LINC02465的检测可以增加肿瘤检测的敏感性和特异性,是胃癌早期诊断生物标志物[15];而HAND2-AS1是一种功能性癌症相关lncRNAs,广泛参与肿瘤细胞增殖、分化、凋亡等多个生物学进程,是结直肠癌的早期诊断标志物[16],虽然该基因未在胃癌早期诊断中得到证实,但在其他肿瘤中已被证实其诊断意义,在胃癌中的价值值得深入挖掘。其余7个lncRNAs少见肿瘤诊断方面的报道,有待进一步研究和证实。
本研究构建了基于6个免疫lncRNAs(RPH3AL-AS1、ERICH3-AS1、TDRG1、LINC02544、LINC01711、LINC01416)的预后基因标签,具备中等程度的预测能力。研究显示,ERICH3-AS1可能通过介导细胞周期和凋亡参与肿瘤的发生和发展,在胃癌组织中,该基因表达量上调与肿瘤复发和预后较差密切关联[17];TDRG1被证实通过miR-873-5p/HDGF轴促进胃癌细胞的侵袭,其表达水平与淋巴结转移正相关,是胃癌的预后生物标志物[18];LINC02544过表达可以促进癌细胞的增殖、侵袭和转移,定量PCR分析也证实该基因在胃癌组织中表达具有肿瘤特异性[19],以上3个免疫lncRNAs均被证实与胃癌预后相关。LINC01711、LINC01416和RPH3AL-AS1由本研究首次发现与胃癌预后的关系,有待进一步证实。
本次研究证实基因标签、年龄、肿瘤临床分期是胃癌预后的独立危险因素。基于此三个因素构建的Cox预后风险模型经验证具有较高的预后预测价值。
综上所述,本研究基于TCGA、GEO数据库分析了免疫lncRNAs与胃癌早期诊断及预后的关系,并成功构建并验证了胃癌早期诊断和预后风险模型。然而本研究仍存在诸多不足之处:由于不同数据库的基因测序问题,本研究并未纳入全部的lncRNAs进行分析,且研究结果缺乏有效的实验验证。此外,用于建模的部分基因尚无相关文献来支持本研究的结论,因此仍需进一步研究提供证据支持。

