A型塞尼卡病毒3′非编码区潜在吻环基序突变对微型基因组蛋白表达的影响
刘拂晓,王宁,赵地, 2,李静,孟海蓝,李紫薇, ,于永乐,董雅琴,尼博
(1.青岛农业大学动物医学院,山东青岛 266109; 2.内蒙古农业大学兽医学院,内蒙古呼和浩特 010018; 3.青岛市黄岛区市场监督管理局,山东青岛 266500; 4.中国动物卫生与流行病学中心,山东青岛 266032)
A型塞尼卡病毒(Senecavirus A, SVA),旧称塞尼卡谷病毒(Seneca Valley virus, SVV),属于小RNA病毒科(Picornaviridae)、塞尼卡病毒属(Senecavirus)成员。SVA目前是塞尼卡病毒属的唯一成员[1]。该病毒最初作为人胚胎视网膜细胞的污染物被发现,2007年首次在加拿大的猪场发现该病毒感染的猪群[2]。该病毒可导致猪的口吻部、蹄冠部出现水疱样病变,其临床症状与其他水疱型疫病(如口蹄疫、猪水疱病)所引起的症状难以区分。除此之外,SVA可导致流行性新生仔猪暂时性死亡。目前,SVA感染已在全球多个国家有所报道,如美国[3]、巴西[4]、泰国[5]等。我国于2015年在广东省发现该病,目前全国已有多个省份暴发了该病,给养猪业造成较大的经济损失[6]。
SVA颗粒呈二十面体对称,直径大约27 nm。病毒衣壳由三个主要结构蛋白VP1、VP2和VP3镶嵌拼接而成,另一个结构蛋白VP4则位于病毒粒子内部。病毒基因组为单股正链RNA,5′端不含有帽子结构,3′端含有多聚A序列。整个基因组本质上是一条mRNA,由5′非编码区(5′ untranslated region, 5′ UTR)、多聚蛋白的开放阅读框(open reading frame, ORF)及3′非编码区(3′ untranslated region, 3′ UTR)构成。5′ UTR含有内部核糖体进入位点(internal ribosome entry site, IRES),使得病毒蛋白的翻译不依赖于帽子结构。本课题组前期已证明,IRES内部含有对病毒复制必需的假结(pseudoknot)结构[7-8],而且假结与起始密码子AUG之间的距离较为保守[9]。多聚蛋白的ORF呈典型的“L-VP4-VP2-VP3-VP1-2A-2B-2C-3A-3B-3C-3D”分布[10]。SVA的3′ UTR较短,除了多聚A尾巴,不足70个核苷酸(nucleotide, nt),该区域目前的研究较少。Hales等[11]预测该区域含有一个潜在的吻环(kissing loop)结构。吻环是一类特殊的RNA结构,本质上属于一种RNA假结结构,由两个临近颈环结构的末端RNA环通过“吻合”作用形成。Hales等[11]的报道预测该结构可能与病毒的复制等分子机制相关,但未进行试验证实。
小RNA病毒的分子机制可借助于病毒的微型基因组(minigenome)进行初步研究。通常可将病毒的多聚蛋白ORF替换成荧光素酶或绿色荧光蛋白ORF,进而将其cDNA克隆至合适的质粒,从而构建病毒的微型基因组质粒。通过分析荧光素酶活性,从而可以初步判断5′或(和)3′ UTR的分子修饰对病毒多聚蛋白表达的影响。本研究通过构建吻环形成基序(motif)突变的SVA(CH-LX-01-2016株[12])微型基因组,研究了所有吻环结构的突变基序对NanoLuc®荧光素酶(NanoLuc®luciferase, NLuc)表达的影响。
1 材料与方法
1.1 细胞及质粒
表达T7 RNA聚合酶的BSR-T7/5细胞,中国动物卫生与流行病学中心惠赠;SVA CH-LX-01-2016株(GenBank,KX751945.1)野生型(wild-type, wt)微型基因组(M-0),青岛农业大学预防兽医学实验室构建并保存[8];萤火虫荧光素酶(firefly luciferase, FLuc)表达质粒(plasmid of FLuc, pFLuc),山东大学张友明教授惠赠。
1.2 主要试剂
DpnI、PrimeSTAR Max Premix,宝日医生物技术有限公司(TAKARA)公司;TOP10感受态细胞,上海昂羽生物技术有限公司;质粒小量提取试剂盒,天根生化科技(北京)有限公司;Opti-MEMTM减血清培养基、DMEM培养基、Lipofectine 2000转染试剂,赛默飞世尔科技公司;VivaCell胎牛血清,上海逍鹏生物科技有限公司;G418,北京索莱宝科技有限公司;Renilla-LumiTMLuciferase Assay Kit、Firefly Luciferase Reporter Gene Assay Kit,上海碧云天生物技术有限公司。
1.3 3′ UTR二级结构及吻环预测
通过在线服务器UNAFold Web Server (http://www.unafold.org/)预测SVA CH-LX-01-2016株3′ UTR RNA二级结构。其3′ UTR的吻环结构参考SVV-001株的预测结果[11]。
1.4 突变型微型基因组的构建
设计7对突变引物(表1),通过定点突变(site-directed mutagenesis, SDM)方法对野生型微型基因组M-0进行改造,共构建7个突变型微型基因组,使其吻环形成基序获得所有可能的突变组合,即3个单核苷酸突变、2个双核苷酸突变和1个三核苷酸突变。以M-0为模板,分别使用7对SDM引物进行高保真PCR反应。高保真酶为PrimeSTAR Max Premix,反应条件设定为:98 ℃ (10 s)、58 ℃ (5 s)、72 ℃ (38 s),共30次热循环。PCR反应产物进行DpnI酶切反应以降解M-0模板。酶切产物转化TOP10感受态细胞,涂布含有氨苄青霉素的LB琼脂平板,37 ℃过夜培养。次日,挑取单菌落至氨苄青霉素LB液体培养基培养,然后送派森诺公司进行突变位点的测序。测序正确的质粒,采用质粒小量提取试剂盒进行提取,并测浓度。所有提取的质粒及M-0进行琼脂糖凝胶电泳分析。

表1 用于构建突变型微型基因组的SDM引物
1.5 微型基因组及pFLuc共转染
将BSR-T7/5细胞接种24孔细胞培养板,置于37 ℃二氧化碳培养箱中培养。待细胞密度至70%时,将构建的7个微型基因组分别与pFLuc共转染BSR-T7/5细胞。首先取一无菌EP管配制转染复合液,包含170 μL Opti-MEMTM减血清培养基、1 500 ng微型基因组、1 500 ng pFLuc及6 μL Lipofectine 2000转染试剂。然后将复合液室温静置15 min后,加至细胞孔中,每个样品分别转染3个孔,每孔60 μL,置于37 ℃二氧化碳培养箱中培养6 h。最后,移弃转染液上清,每孔补加500 μL含4%胎牛血清的DMEM,置于37 ℃二氧化碳培养箱中培养48 h。
1.6 双荧光素酶报告分析
微型基因组及pFLuc共转染的24孔板在培养箱内培养24 h后将其取出,冻融2次,低速离心收获上清,转至白色不透明96孔板,利用全波长酶标仪进行双荧光素酶报告分析。使用Renilla-LumiTMLuciferase Assay Kit和Firefly Luciferase Reporter Gene Assay Kit分别测定NLuc和FLuc荧光值。NLuc荧光值对应微型基因组,FLuc荧光值为内参值。所有荧光值都减掉背景值,以消除背景荧光干扰。采用GraphPad Prism软件(8.0版)将NLuc荧光值、FLuc荧光值及二者的比值分别绘制柱状图,并进行双尾Student’st检验分析。
2 结果与分析
2.1 3′ UTR二级结构及吻环预测
SVA CH-LX-01-2016株基因组结构及3′ UTR序列如图1所示。
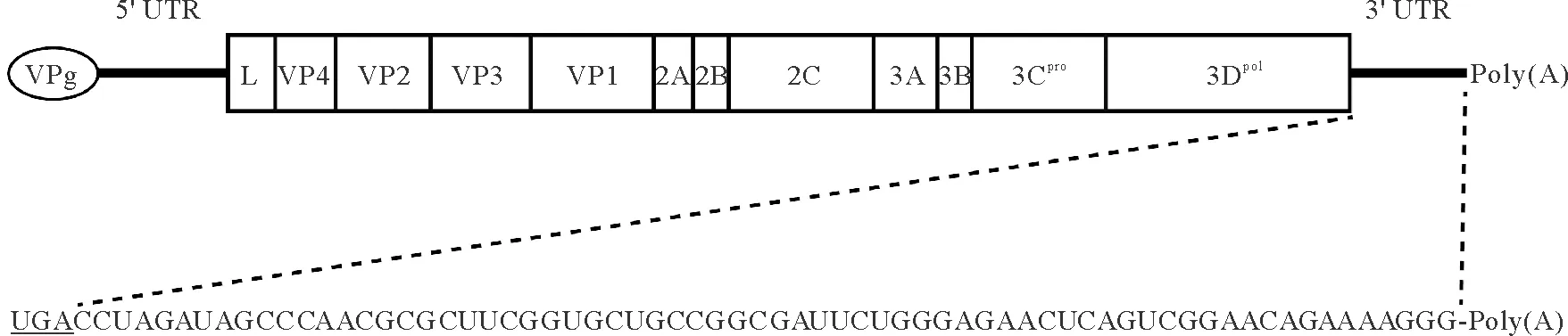
图1 SVA基因组结构及3′ UTR序列
通过在线服务器UNAFold Web Server (http://www.unafold.org/)预测该毒株3′ UTR的RNA二级结构如图2A,其包括两个相邻的颈环结构,两个末端环通过潜在的三碱基对(U-A、U-A、C-G)“吻合”途径形成吻环结构(图2B)。
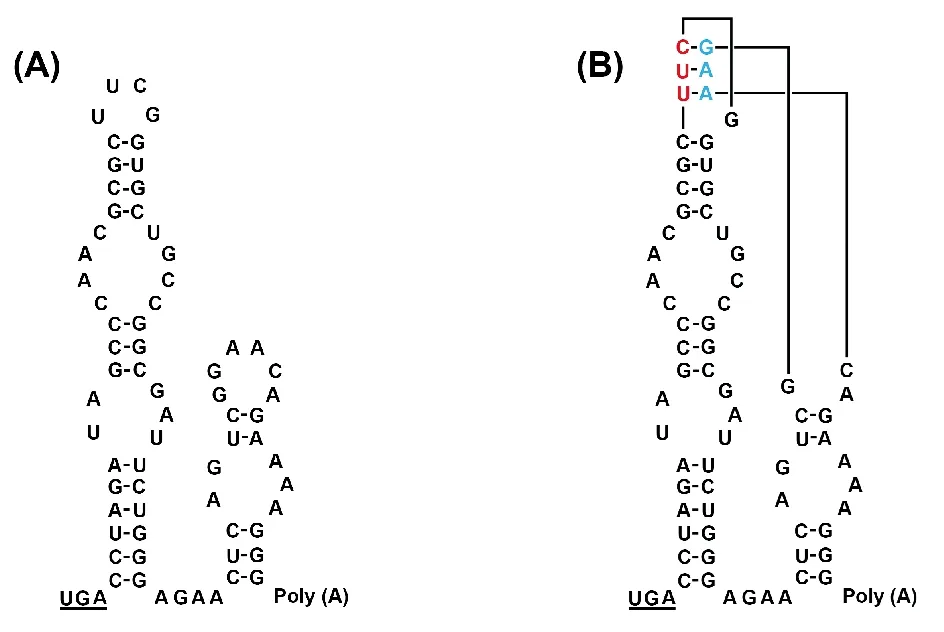
图2 SVA 3′ UTR二级结构(A)及潜在的吻环结构(B)
2.2 突变型微型基因组的构建
野生型微型基因组质粒结构如图3所示。该微型基因组骨架为pUC57质粒,上游受T7启动子(pT7)调控,下游受T7终止子(T7t)调控。BSR-T7/5细胞可表达T7 RNA聚合酶,微型基因组在胞内可以转录mRNA,进而通过IRES启动NLuc的表达。
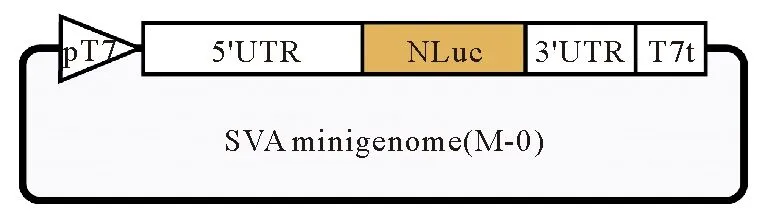
图3 野生型SVA微型基因组质粒结构
通过SDM,对吻环形成基序进行定点突变,总共获得7个突变型微型基因组(M-1至M-7),其突变位点如图4绿色字母所示。
图4 SVA微型基因组突变位点(绿色字母所示)
通过Sanger测序对M-1至M-7进行分析,如图5所示,突变位点对应的峰图以相应字母标记。
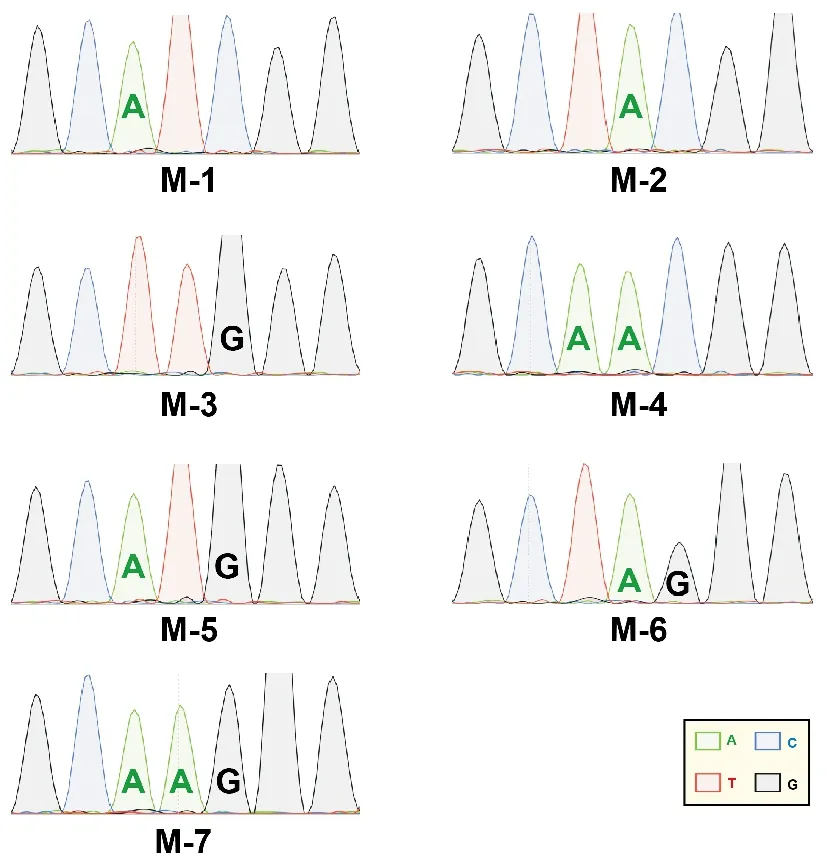
图5 SVA微型基因组Sanger测序(字母所示突变位点)
经测序鉴定正确的质粒进行琼脂糖凝胶电泳,如图6所示。

图6 SVA微型基因组质粒琼脂糖凝胶电泳
2.3 双荧光素酶报告分析
7个突变型微型基因组分别与pFLuc共转染BSR-T7/5细胞后进行双荧光素酶报告分析。NLuc和FLuc表达情况分别见图7A和7B,二者比值见图7C。图7表明,7个突变型微型基因组皆可表达NLuc。与对照组(M-0)相比,虽然M-4和M-7组表现出统计学极显著差异(p值小于0.01)(图7C),但实际NLuc发光值并未显著降低至较低水平(图7A),说明吻环基序的所有单一或组合突变,并未破坏蛋白表达。
图7 双荧光素酶报告分析
3 讨论
SVA属于小RNA病毒属成员,其基因组是一条单股、正链、线性的RNA,长约7 300 nt,两端为UTR[13]。5′ UTR长约670 nt,3′ UTR则不足70 nt。小RNA病毒的3′ UTR虽然序列较短,但某些关键基序可能影响病毒的复制。例如,肠道病毒是一类常见的小RNA病毒,其3′ UTR含有复杂的RNA二级结构[14],如6碱基配对的典型吻环结构[15-17],这种结构对病毒的复制而言是必需的。如果这种6碱基配对结构出现错配,继而导致吻环结构破坏,那么可能导致温度敏感型或致死性子代病毒的出现。因此,3′ UTR吻环结构可能决定着某些小RNA病毒的复制特性。
另有研究表明,小RNA病毒基因组复制过程中,线性基因组会出现环化现象,宿主蛋白在环化过程中充当“蛋白桥”的作用[18],“蛋白桥”两端的“桥头”蛋白分别连接基因组的5′和3′末端。因此,虽然3′ UTR远离5′ UTR,但前者的二级或高级结构发生改变,则可能导致“蛋白桥”的连接作用失活,从而导致病毒基因组复制或(和)多聚蛋白表达失败。另外,如果3′ UTR含有类似吻环结构的顺势作用元件,那么其突变后可能直接影响与反式作用因子的结合,从而潜在干扰病毒的增殖。虽然SVA 3′ UTR RNA结构影响病毒复制或蛋白表达的研究鲜有报道,但Hales等[11]预测该区域含有一个吻环结构,其可能与病毒的复制等分子机制相关,但未进行试验证实。因此,这促使我们通过微型基因组初步对该结构进行了研究。
NLuc是一种新发现的荧光素酶[19],其ORF仅有516 nt。由于NLuc ORF替换了6 546 nt的多聚蛋白ORF,SVA微型基因组方便了病毒分子机制的研究。本研究通过SDM构建了7个不同突变型微型基因组,这些微型基因组包括了所有单一和复合点突变。通过双荧光素酶报告分析,本研究试图探究预测的吻环结构如果被破坏,是否影响上游5′ UTR启动蛋白表达。结果表明,尽管双尾Student’st检验证明M0分别与M4和M7存在极显著差异(图7C),p值分别为0.008和0.003,但实质上所有突变型质粒并未将NLuc的表达量降低到一个较低水平(图7A)。换言之,吻环结构的破坏,并未破坏5′ UTR启动蛋白表达的能力。根据本研究的结论,推测存在两种可能:(1)SVA的3′ UTR不存在吻环结构;(2)即使存在吻环结构,该结构对病毒的复制是非必须的。究竟存在哪一种可能性,有待进一步研究证实。

