DNA甲基化检测研究进展
唐文悦, 孙欣楠, 张丽瑶*,
(1.武汉大学生命科学学院,杂交水稻国家重点实验室,湖北武汉 430072; 2.天津大学生命科学学院,天津 300072)
1 引言
DNA甲基化,一种重要的表观遗传修饰,指在DNA甲基转移酶的催化下,甲基供体(S-腺苷甲硫氨酸)的甲基基团被转移到DNA胞嘧啶或腺嘌呤上的过程。DNA甲基化已被发现在基因表达、胚胎发育、细胞增殖、细胞分化和染色体稳定性维持等关键生物学过程中起重要作用[1]。在原核生物中,DNA甲基化可发生在胞嘧啶或腺嘌呤上,DNA甲基化不仅参与染色质复制和修复等过程,还保护宿主自身的DNA不被自身限制性修饰系统中限制性内切酶切割,而同时使入侵的噬菌体DNA被切割降解[2]。在真核生物中,DNA甲基化仅仅发生在胞嘧啶的五位碳上,形成5-甲基胞嘧啶(5-Methylcytosine,5mC),植物中,胞嘧啶的甲基化不仅发生在CG对称位点,也发生在CHG和CHH位点(其中H为A、T或C),一般CG序列的甲基化概率最高,CHG序列次之,CHH序列最低,且不同植物中甲基化程度各不相同。拟南芥基因组中,约24%CG、6.7% CHG 和1.7%CHH位点被甲基化,而水稻对应的甲基化占比分别是54.7%、37.3%和12%,明显高于拟南芥中的值[3]。在哺乳动物中,DNA甲基化几乎只发生在对称的CG位点,约80%的体细胞基因组DNA CG位点均发生高度甲基化[4]。DNA的甲基化主要由两类DNA甲基转移酶完成,即维持型甲基转移酶(DNMT1)和从头甲基转移酶DNMT3a和3b。现已发现DNMT1和DNMT3对于哺乳动物的胚胎发育是必须的[5]。在小鼠中,DNMT1单纯合突变体或DNMT3a和3b双纯合突变体胚胎均不能存活,且DNMT3a和DNMT3b之间存在功能冗余[5]。甲基转移酶对胚胎发育的重要性不局限于哺乳动物中。如拟南芥中,MET1(DNMT1同源基因)突变体中也存在胚胎致死现象[6]。已发现人类多种疾病的发生如癌症[7.8]、心血管疾病[9]、帕金森病[10]等与DNA甲基化密切相关。如通过将整体降低小鼠基因组甲基化水平可导致T-细胞淋巴癌的发生[8]。在多种癌组织中存在异常的甲基化现象,通常表现为整体基因组的低甲基化和特异启动子区域的高甲基化,DNA甲基化图谱分析正成为临床癌症诊断的方法之一[11]。
在真核生物中,不仅存在DNA甲基化,还存在DNA去甲基化。哺乳动物中,DNA去甲基化由10/11易位(Ten-Eleven-Translocation,TET)家族蛋白完成。TET蛋白可催化5mC生成5-羟甲基胞嘧啶(5hmC),5hmC再被TET蛋白催化生成5-醛基胞嘧啶(5-Formylcytosine,5fC)和5-羧基胞嘧啶(5-Carboxylcytosine,5caC),5fC和5caC被胸腺嘧啶DNA糖苷酶(Thymine DNA glycosylase,TDG)识别切除,形成无嘧啶位点,随后该位点被碱基切除修复途径中酶修复,生成胞嘧啶,完成DNA去甲基化过程[12,13](图1)。5hmC是一种稳定的DNA修饰[14],存在于动物细胞和组织中。癌细胞基因组中存在整体的5hmC缺失,低量的5hmC已经成为癌症治疗预后差的一个标志[15]。综上所述,建立高灵敏度和可靠的甲基化检测方法对研究疾病发生机理,建立有效的诊断和治疗策略具有重要意义。目前,DNA甲基化分析与研究技术不仅可以在特定基因水平上分析DNA甲基化,还可以分析基因组特定区域以及全基因组中的DNA甲基化变化。按照研究技术原理不同,DNA甲基化分析与研究方法可分为基于碱基分离的分析技术、基于甲基化敏感限制性内切酶处理的技术、基于亚硫酸氢盐处理的技术、基于亲和富集处理的技术等。本文根据检测技术原理不同介绍了DNA甲基化分析和检测方法的研究进展,为未来深入研究DNA甲基化提供参考。
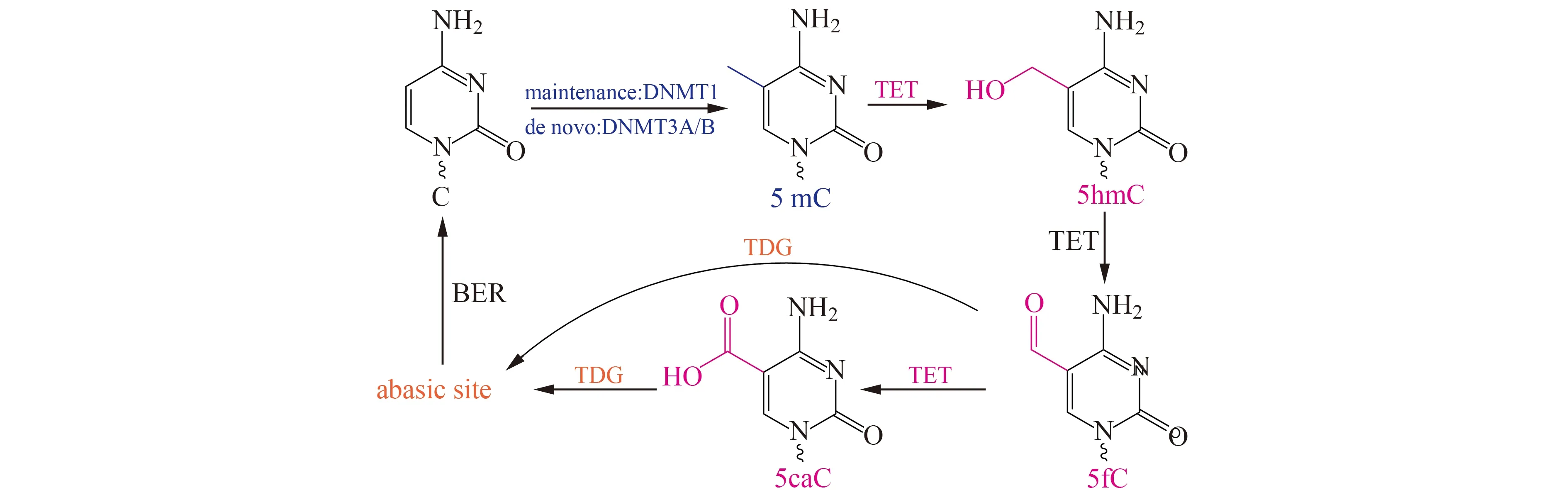
图1 DNA甲基化和去甲基化动态过程[13]Fig.1 Dynamic regulation of DNA methylation and demethylation[13]
2 DNA甲基化分析和检测技术
2.1 基于碱基分离的分析技术
高效液相色谱法(HPLC)可将DNA水解成单个脱氧核糖核苷,分离并检测脱氧胞苷和5-甲基脱氧胞苷从而计算出基因组中5mC含量[16]。1980年,Kuo等首次利用高效液相色谱紫外检测法分析基因组中5mC的含量[17]。随着质谱(MS)技术的发展,应用高效液相色谱-串联质谱法(HPLC-MS/MS)[16]和超高效液相色谱-串联质谱法(UHPLC-MS/MS)[18]使得5mC检测的选择性和灵敏度明显增强。由于具有高的准确度和灵敏度,HPLC-MS是检测全基因组5mC含量的金标准方法。
2000年,Fraga等提出一种新的甲基化胞嘧啶分离检测技术,即高效毛细管电泳法(High performance capillary electrophoresis,HPCE)[19]。与HPLC相比,HPCE具有进样量更低、成本低、分析速度快等特点,已被用于检测人全血中5mC的含量,12 min内完成分离与检测[20]。但HPLC与HPCE方法均不能了解5mC的具体位置信息,即无法得知基因组序列信息。
2.2 基于甲基化敏感限制性内切酶处理的分析技术
甲基化敏感限制性内切酶(Methylation Sensitive Restriction Enzymes,MSRE)是一类对其识别位点含有甲基化碱基敏感的限制性内切酶,利用消化处理可以特异性识别甲基化序列,这类酶一般只能识别一个甲基化碱基位点。甲基化敏感性内切酶消化后扩增方法操作简便、应用范围广,但只能定性或半定量的检测目的片段的某一位点甲基化状态,不能定量,若酶消化不完全则会影响结果。甲基化敏感性内切酶消化联合Southern杂交技术还可以获得染色质重复序列的甲基化状态[21]。Southern杂交是对基因组DNA特定序列进行定位的分析方法,通过标记探针与膜上DNA进行杂交可检测目标DNA片段中是否存在与探针同源的序列。Southern杂交不仅可用于DNA图谱分析,转基因的拷贝数检测等方面,还可用于DNA甲基化分析。采用HpaⅡ或MspⅠ酶切基因组结合Southern杂交可分析拟南芥着丝粒180bp重复序列的甲基化[21]。在众多的甲基化敏感性内切酶中,值得一提的是McrBC,一种GTP依赖的DNA内切酶,该酶作用原理和上述HpaⅡ和MspⅠ相反,对高甲基化DNA有强切割活性,而对非甲基化DNA活性低,利用McrBC消化基因组后PCR可知基因组某一片段的甲基化水平[22]。
2.3 基于亚硫酸氢盐处理的测序技术
2.3.1 经典亚硫酸氢盐测序亚硫酸氢盐测序(Bisulfite sequencing,BS-seq)是目前研究DNA甲基化最重要的方法。其原理是利用亚硫酸氢盐对非甲基胞嘧啶的高效脱氨作用,生成尿嘧啶(转化原理和过程见图2),在随后的测序中被读成胸腺嘧啶T,而5mC和5hmC均对亚硫酸氢盐不敏感,通过PCR产物直接测序或挑克隆测序可判断DNA的甲基化[23,24]。该方法精确度高,可知目的片段每一个CG、CHG、CHH位点的甲基化状态,被认为是DNA甲基化检测的“金标准”。但缺点如下:(1)不能区分5mC和5hmC;(2)出现转化不完全的情况;(3)耗时长;(4)通过克隆拷贝数计算甲基化程度有一定误差。

图2 亚硫酸氢盐转化胞嘧啶生成尿嘧啶的过程[24]Fig.2 The mechanism of nucleotide conversion from cytosine to uracil with bisulfite treatment [24]
2.3.2 甲基化特异性PCR甲基化特异性PCR(Methlation specific PCR,MSP)法是一种分析CpG岛DNA甲基化模式的方法[25]。首先亚硫酸氢盐对DNA转化处理,然后PCR扩增,为了区分甲基化或非甲基化DNA,需两对引物,一对引物对甲基化DNA有特异性,另一对引物对甲基化DNA没有特异性。如果DNA在引物覆盖位点内不含甲基化,则发生非甲基化引物对扩增,反之亦然。MSP可快速评估CpG岛的甲基化,不需要克隆或甲基化敏感的内切酶,可对少量DNA及石蜡包埋样本进行分析[25],该方法中引物设计是关键[26]。
Eads等[27]将MSP与荧光定量PCR结合,开发出甲基化定量PCR技术(命名为MethyLight)。MethyLight特异性好、敏感性强,特别适合在非甲基化DNA高背景下检测低频甲基化DNA区域[27]。将MethyLight与数字PCR(Digital PCR)相结合可明显提高DNA检测灵敏度[28]。MethyLight检测结肠癌基因组上相关位点的甲基化可预测临床上癌治疗效果,高甲基化病人预后差于低高甲基化病人[29]。
2.3.3 亚硫酸氢盐焦磷酸测序焦磷酸测序(Pyrosequencing)是一种利用酶偶联反应和生物发光实时监测伴随核苷酸掺入的焦磷酸盐释放过程的DNA测序技术[30]。亚硫酸氢盐焦磷酸测序是指首先亚硫酸氢盐处理基因组,再用焦磷酸测序技术测定目标位点中C/T的比率,以此判断目标位点的甲基化程度。该技术成本效益高,快速、且能准确定量,但最大的限制是读取片段短,可分析的CG位点少,通过连续测序可提高分析范围[31]。目前,亚硫酸氢盐焦磷酸测序已被用于多种疾病标志基因的启动子甲基化分析,如PCDH10[32]和KEAP[33]等。
2.3.4 全基因组亚硫酸氢盐测序全基因组亚硫酸氢盐测序(Whole-genome bisulfite sequencing,WGBS)是将亚硫酸氢盐处理过的DNA构建文库并与处理前的对照文库比对测序,通过比对碱基差异判断甲基化水平,可生成最全面、最高分辨率的DNA甲基化图谱,适用于所有具有参考基因组的物种[34]。WGBS通常需要30×以上覆盖范围测序,因此对于那些具有较大甲基组的样本而言(如人类和玉米),价格昂贵。但由于所获信息最全面,可靠性强,因此WGBS是目前国际上研究基因组甲基化项目的金标准[35]。当前WGBS技术已成熟,详细的实验步骤和最新的技术流程已被很好的总结比较[36],并成功用于大型植物基因组(玉米和大豆)的甲基化测序[37]。
2.3.5 简化代表性亚硫酸氢盐测序由于WGBS费用高,2005年,Meissner等[38]提出了简化代表性亚硫酸氢盐测序(Reduced representation bisulfite sequencing,RRBS)。该方法是首先利用特异性限制性核酸内切酶(如MspI)消化基因组DNA,电泳并回收所需要大小的片段,随后亚硫酸氢盐测序。相较WGBS,RRBS仅对基因组1%的区域进行测序,成本低,测序深度更大,数据利用率更高,但不能获得完整的全基因组甲基化信息[39]。由于玉米基因组上没有大量CG岛,MspI酶切不合适,改用MseI酶切,RRBS被成功用于检测玉米基因组的甲基化,尽管只有四分之一的基因组被覆盖,但84%启动子区域可被检测[40]。通过比对测序,RRBS可用于无参考基因组的不同物种之间的甲基化比较分析,为解析DNA甲基化分子机制提供工具[41],但对无参考基因组的物种来说,RRBS具有很大局限性[42]。
2.3.6 TET酶或化学试剂辅助亚硫酸氢盐测序经典亚硫酸氢盐测序技术无法区分5mC和5hmC,针对于此,Booth等建立了氧化亚硫酸氢盐测序(Oxidative bisulfite sequencing,oxBS-seq)[43,44]。该方法首先利用钌酸钾(KRuO4)氧化基因组,将5hmC氧化成5fC,随后亚硫酸氢盐处理将未甲基化胞嘧啶和5fC均转化成尿嘧啶,可特异性检测5mC,通过对比经典亚硫酸氢盐测序结果,还可获得全基因组中5hmC的情况。自2012年以来,oxBS-seq技术已被广泛应用于多种生物样品的5mC和5hmC检测,如癌组织和人胎盘等[45 - 47]。
TET辅助亚硫酸氢盐测序(TET-assisted bisulfite sequencing,TAB-seq)方法是一种仅针对5hmC直接检测的测序方法[48,49]。首先糖基化对5hmC进行保护,然后利用TET酶将5mC和5fC完全氧化为5caC,亚硫酸氢盐处理后5caC转化为尿嘧啶,PCR扩增和测序后5mC和5fC为胸腺嘧啶T,而5hmC为胞嘧啶信号C。通过该方法检测人和小鼠胚胎干细胞基因组中5hmC,结果发现5hmC几乎仅存在CG上,其丰度明显低于5mC,5hmC和5mC均富集于末端调控元件上,而少有富集在启动子和基因上[48]。
2.4 基于亲和富集处理的测序技术
2.4.1 DNA甲基化免疫沉淀芯片检测或测序在哺乳动物中,5mC占所有碱基的约1%,丰度较低,富集含有5mC的片段有助于后续的DNA建库测序,通过5mC抗体或与含5mC的DNA特异性结合的蛋白质可富集甲基化DNA。2005年,Weber等[50]提出了甲基化DNA免疫沉淀富集技术,该技术利用5mC单克隆抗体富集高度甲基化单链DNA片段。方法的基本步骤是将基因组DNA超声断裂成300~600 bp,随后95度变性,再用5mC单克隆抗体温育及蛋白A/G标记的磁珠或琼脂糖珠富集,提纯后芯片分析或测序。由于高通量测序所获信息远远高于DNA芯片,因此DNA甲基化免疫沉淀测序(Methylated DNA immunoprecipitation sequencing,MeDIP-seq)技术已成为人类疾病与健康中表观遗传学研究最重要的技术[51,52]。MeDIP-seq无需深度测序,成本低,富集后测序,灵敏度高(即DNA用量少,可低至50 ng[53]),近年来已成功用于外周血中游离DNA的甲基化分析,为寻找新的癌症标记物提供了良好工具[54,55]。但该方法的结果依赖于抗体质量,高特异性的单克隆抗体是必须的。
2.4.2 甲基化DNA特异性结合蛋白富集测序甲基化DNA特异性结合蛋白富集测序技术(Methyl-CpG binding domain protein-enriched genome sequencing,MBD-seq)原理与MeDIP-seq相似,都是基于特异蛋白对甲基化DNA的高亲和性,将其富集。甲基化CpG结合(Methyl-CpG-binding domain,MBD)蛋白家族中的某些具有相似MBD结构域蛋白能结合甲基化的双链DNA。四种MBD蛋白(MBD1,MBD2,MeCP2,MBD4)可高特异性结合甲基化双链DNA,被晶体结构和核磁共振技术证实[56,57]。由于具有强亲和性等优点,MBD2常被用于MBD-seq[58,59]。甲基CpG结合域蛋白3-like-1(MBD3L1),一种MBD2结合蛋白,在实验中常作为辅助蛋白以增加MBD2对甲基化DNA的结合力,促进甲基化DNA结合复合物的形成,提高富集效果[60,61]。同MeDIP-seq相比,MBD-seq实验中无需将DNA变性,且倾向于富集高密度的甲基化CG岛[62]。方法经济有效,可用于大批量样品的甲基化检测[63]。同MeDIP-seq技术,MBD-seq具有灵敏度高,可用于检测外周血中游离DNA的甲基化[64]。
和基于亚硫酸氢盐处理的测序技术相比,基于亲和富集处理的测序技术成本低,特异性高,灵敏度高等优点,但其缺点是不能定量,分辨率低(通常是150 bp左右,无法达到单碱基分辨率),难以获得低甲基化区域信息[57]。
2.5 不依赖于亚硫酸氢盐的DNA甲基化测序
经典的亚硫酸氢盐处理基因组DNA会导致基因组的大量降解,限制了少量样品的分析[65]。针对于此,2019年,Liu等[66]人建立了TET辅助吡啶硼烷测序法(TET-assisted pyridine borane sequencing,TAPS),避免了基因组的大量降解。首先利用TET酶将5mC和5hmC氧化成5caC/5fC,随后5caC/5fC被吡啶硼烷还原形成二氢尿嘧啶(dihydrouracil,DHU,图3),PCR扩增测序后被识别为胸腺嘧啶T,未甲基化胞嘧啶保留为胞嘧啶,从而可检测基因组中5mC和5hmC(图4),同亚硫酸氢盐测序相同,TAPS不能区分5mC和5hmC。若将5hmC利用糖基化进行保护,随后TET氧化和吡啶硼烷还原,则可特异性检测5mC,5hmC保护酶为β-糖基转移酶(β-glucosyltransferase),方法命名为TET辅助吡啶硼烷测序法β(TET-assisted pyridine borane sequencing withβ-glucosyltransferase,TAPSβ)[66]。若将TET酶用氧化剂钌酸钾(KRuO4)代替,特异性氧化5hmC成5fC,再吡啶硼烷还原将5fC转化成二氢尿嘧啶,则可特异性检测5hmC,方法命名为化学辅助吡啶硼烷测序(chemical-assisted pyridine borane sequencing,CAPS)[66]。TAPSβ和CAPS方法已被证实可分别用于全基因组的5mC特异检测和5hmC特异检测[67]。TAPS方法灵敏度高,10 ng DNA(1~3 mL血浆)[68]即可分析外周血游离DNA的甲基化。

图3 TET氧化5mC和5hmC及吡啶硼烷还原5caC生成二氢尿嘧啶的过程[66]Fig.3 The mechanism of nucleotide conversion from 5mC and 5hmC to dihydrouracil with TET and pyridine borane treatment [66]
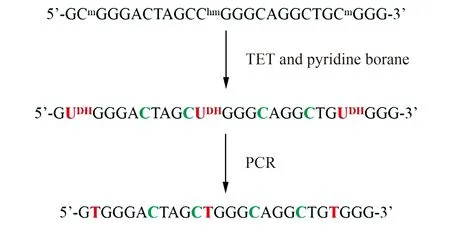
图4 TET辅助吡啶硼烷测序原理Fig.4 The sequencing principle for DNA methylation with TET and pyridine borane
同样针对亚硫酸氢盐处理的大量DNA降解问题,2021年,Sun等[69,70]建立了不依赖于亚硫酸氢盐的酶促甲基化测序技术(Enzymatic methyl-seq,EM-seq)。该方法利用TET酶和β-糖基转移酶氧化和保护5mC和5hmC,生成5caC或糖基化的羟甲基胞嘧啶,随后利用胞嘧啶脱氨酶(APOBEC)催化脱氨,未甲基化的胞嘧啶脱氨转化为尿嘧啶U,PCR测序读为胸腺嘧啶T,而5caC或糖基化的羟甲基胞嘧啶不被脱氨,PCR测序读为胞嘧啶C,从而可检测基因组中的5mC和5hmC(图5)。若利用β-糖基转移酶保护5hmC,随后胞嘧啶脱氨酶脱氨,可特异性检测5hmC,即为APOBEC酶耦合的表观测序(APOBEC-coupled epigenetic sequencing,ACE-Seq)[71]。同TAPS技术,EM-seq中DNA基本不降解,所需DNA量少(100 pg DNA即可),基因组覆盖度好,数据分析速度快等优点[70],表1总结了以上各种甲基化测序技术中胞嘧啶C、5mC和5hmC的测序结果。

图5 酶促甲基化测序技术原理Fig.5 The sequencing principle for DNA methylation by enzymatic deamination method

表1 几种测序方法中5mC和5hmC的测序比较Table 1 Comparison of different methods for 5mC and 5hmC sequencing
2.6 第三代测序技术
因价格便宜,实验流程成熟可靠,短读大规模平行测序(Short read massive parallel sequencing),又称为第二代测序(Next-generation sequencing,NGS)已成为检测DNA甲基化水平的标准工具,但短读技术有其固有的局限性,如从头组装、单倍体定相和结构差异检测困难等[72]。第三代测序技术主要包括单分子实时测序技术(Single molecule real-time,SMRT)和单分子纳米孔测序技术(Single-molecule nanopore DNA sequencing)。SMRT技术由太平洋生物科学公司(Pacific Biosciences,PacBio)研发而成[73,74],测序过程无需PCR扩增,可克服二代测序中的结构差异检测困难等缺点,具有可读取长片段DNA或RNA序列,可直接检测DNA的表观遗传修饰等优点[72,75]。该技术是基于零模波导孔(Zero Mode Waveguide,ZMW)的单分子检测技术发展而来的一种边合成边测序技术[73,74]。SMRT技术不仅可区分常规碱基,还可以检测修饰后碱基,其检测原理是根据不同的碱基有不同颜色的荧光以及有不同的荧光持续时间,如甲基化的腺嘌呤上的时间远长于未甲基化的腺嘌呤[76]。目前SMRT技术已经成功用于DNA甲基化检测[76,77]。
纳米孔测序技术也是基于单分子检测的测序技术,起源于20世纪80年代末期科学家提出了一种新型想法,即能否通过电泳将DNA通过纳米小孔进行测序,随后大量科学家进行了相关实验,获得了一系列技术上的突破[78]。该技术的基本原理是DNA单链分子通过纳米孔蛋白时,由于碱基不同导致产生的电流干扰不同,通过检测电流的变化而区分碱基[78,79]。2014年,由牛津纳米孔技术公司推出第一台商品化的纳米孔测序仪(MinION),已被用于甲基化检测以及临床上呼吸道的细菌感染等[80 - 82]。与其他长读测序仪一样,纳米孔测序的主要缺点是错误率较高(5%到20%之间),具体取决于纳米孔、文库制备及数据处理方法,错误包括替换、插入和删除[83]。相对第二代测序技术,第三代测序技术的高错误率和高费用限制了其应用。
3 总结与展望
DNA甲基化作为表观遗传最重要的一种修饰,在动物与植物中广泛存在,在多种关键生命过程中起重要作用。DNA甲基化检测技术的发展推动了生命科学领域的研究。高效液相色谱法可检测全基因组中5mC含量,准确度高,但不能了解甲基化的位置信息;限制性酶切结合PCR或Southern杂交技术一次只能检测单个位点或某些特异序列的甲基化;亲和富集技术结合二代测序条件较温和,灵敏度高,但不能提供单碱基分别率的DNA甲基化图谱。基于亚硫酸氢盐处理的测序技术存在DNA降解问题,导致比对率低。不依赖于亚硫酸氢盐处理的测序技术需要利用重组酶,酶的效果是关键因素。第三代核酸测序技术,实现了不需PCR的长度测序,但准确度不够,费用高。由于具有高准确度和低费用,亚硫酸氢盐结合二代测序仍是目前甲基化检测最主要的技术;改进现有技术或将不同方法结合开发新的技术,获得灵敏度高、特异性强、易操作、高通量的检测方法仍是未来研究的重点。

