2016-2017年中国部分地区TTSuV2流行情况调查与遗传进化分析
王德海,陈文文,朱金海,李春秋,李鹏,孙东波
(黑龙江八一农垦大学动物科技学院,大庆 163319)
猪细环病毒是一种无囊膜单股负链DNA病毒,早期曾被称为猪输血传播病毒(Transfusion transmitted virus,TTV)、猪源细环病毒(Porcine torque teno virus,PTTV)[1-2]。TTSuV在猪群中具有很高的感染率,并能通过多种传播途径进行传播,还会对生物制品及公共环境等造成污染。虽然TTSuV单独感染可能并不引发临床症状,但已许多研究显示其可与其他多种病原混合感染,促使疾病的发生、加剧疾病的临床表现。TTSuV为无囊膜、呈二十面体对称、单股负链、环状DNA病毒,其基因组全长约2.7~3.0 kb,主要包括非编码区(Untranslated region,UTR)和编码区(Translated region,TR)两个部分。TTSuV各基因型及亚型间的基因组结构比较保守。根据ICTV第10次在线报告,指环病毒科共包含12个病毒属(Alphatorquevirus-Zetatorquevirus),68种病毒;其中TTSuV分布于Iotatorquevirus和Kappatorquevirus病毒属,前者包含两个种,分别命名为TTSuV 1a和TTSuV 1b,而后者也包含两个种分别命名为TTSuV k2a和TTSuV k2b[3-4]。目前,国内外许多学者仍习惯将TTSuV分为TTSuV1(Iotatorquevirus)和TTSuV2(Kappatorquevirus)两种基因型[5-6],研究也沿用此分类名称。
TTSuV是一种变异速率很快的病毒,不同基因型毒株间同源性低于50%,但目前关于TTSuV基因组及其编码蛋白的结构与功能的研究却很少。TTSuV UTR的核苷酸片段长约800 bp,占整个基因组的28%~30%。其包含一段长度不等的高GC含量短序列(G+C含量超过90%),研究发现该区域在空间上会形成一个茎环结构,对PCR扩增及基因测序造成很大影响[7-8]。此外,UTR还包含启动子区、TATA box(ATATAA)及Poly(A)尾等转录调控元件。Cornelissen-Keijsers等[9]研究发现启动子区序列在TTSuVk2a和TTSuVk2b型毒株之间高度保守。TTSuV的UTR相比TR更为保守,且不受自然选择压力的影响,但其在不同基因型毒株中却能表现出很高的遗传多样性,因此国内外许多学者常将UTR作为鉴定及区分不同类型TTSuV毒株的一种分子标记[10-11]。研究发现TTSuV的TR存在多个相互重叠的开放阅读框(Open reading frame,ORF),病毒在转录过程中可形成三个或更多mRNA,编码至少6种蛋白,其中ORF1和ORF2由未剪辑的mRNA1编码,ORF1/1和ORF2/2由剪辑内含子1的mRNA2编码,而ORF1/1/2和ORF2/2/3则由剪辑内含子1和2的mRNA3编码[12-13]。
大量的流行病学研究表明TTSuV流行范围十分广泛,目前北美洲、南美洲、欧洲、亚洲、澳洲、非洲中至少18个国家对该病进行了报道。TTSuV在世界范围内持续存在且不断变异,给养猪行业的健康发展带来巨大威胁。因此,持续监测TTSuV的流行与遗传变异情况对其病原特性的研究及该病的防控具有重要意义。通过普通PCR方法,对2015~2016年华东地区采集的仔猪腹泻样品进行TTSuV2检测,并对该病毒在华东地区流行情况及基因型进行分析,为TTSuV2的流行情况和遗传进化情况提供基础数据,也为该病毒在我国的预防提供了可参考数据。
1 材料与方法
1.1 材料
1.1.1 样品采集
采集自2016年9月-2017年12月,中国东北、华东、华中、华北、华南、西南、西北7个地区规模化猪场发病猪只的肠组织或粪便样品共325份;将样品根据不同猪场与发病日期进行整理标记,保存于-80℃冰箱。
1.1.2 主要试剂

表1 主要生化试剂Table 1 The detail information of reagents
1.1.3 仪器设备

表2 仪器设备Table 2 The detail information of machines
1.1.4引物设计
通过巢式PCR方法扩增UTR基因,引物序列参照王礞礞等[17]研究;另外合成一对引物用于扩增TTSuV2基因组的高变区,目的基因的引物序列参照Martí等[14]研究,详细信息见表3。

表3 TTSuV2 UTR和ORF1基因扩增引物Table 3 Primers for the amplification of UTR and ORF1 genes of TTSuV2
1.2 方法
1.2.1 样品的处理与基因组的提取
称取1 g肠组织/粪便样品,加入液氮研磨至粉末状(粪便样品省略此步骤);加入3倍体积PBS,涡旋振荡5 min,4℃4 200×g离心15 min取上清液,按照病毒DNA提取试剂盒说明书提取样品中病毒DNA。提取的DNA保存在-20℃。
1.2.2 目的基因PCR扩增与测序
使用表3中设计的引物对UTR基因进行特异性巢式PCR扩增,PCR反应体系见表4。外套PCR反应条件为94℃预变性5 min,94℃变性30 sec,52℃退火30 sec,72℃延伸40 sec,共35个循环,最后72℃再延伸10 min。内套PCR反应条件为94℃预变性5 min,94℃变性30 sec,55℃退火30 sec,72℃延伸30 sec,共35个循环,最后72℃再延伸10 min。取内套扩增产物3 μL经1.5%的琼脂糖凝胶电泳,同时以DL2 000 DNA Marker作为参照,在紫外凝胶成像仪上观察并记录结果。
TTSuV2基因组高变区PCR扩增反应体系见表4。反应条件为94℃预变性5 min,94℃热变性30 sec,60℃退火45 sec,72℃延伸3 min,共40个循环,最后72℃再延伸7 min。取扩增产物3 μL经1%的琼脂糖凝胶电泳,同时以DL 2 000 DNA Marker作为参照,在紫外凝胶成像仪上观察并记录结果。将阳性产物送由哈尔滨博仕生物有限公司进行测序。
1.2.3 系统进化树分析
进化树分析方法与前研究相似[19],使用MEGAⅩ(网址:https://www.megasoftware.net/)软件对试鉴定毒株基因序列与参考序列进行多序列比对,将对齐后的序列应用MEGAⅩ软件中邻接法(Neighborjoining method,NJ法)结合最大复合似然法模型(Maximum Composite Likelihood)构建系统进化树,bootstrap(自展值):1 000。

表4 PCR反应体系Table 4 PCR reaction system
1.2.4 目的基因序列核苷酸与氨基酸同源性分析
使用DNASTARTM5.06软件包中MegAlign程序对测序得到的目的基因序列进行核苷酸同源性分析,分别选取Kappatorquevirus病毒属国内外代表性毒株作为参考毒株,其中TTSuV k2a基因型:2p(Brazil)、Porcine TTV 2 from China(China)、PTTV2c-VA(USA)、472142(Germany)、TTSuV2-KU2(Japan);TTSuV k2b基因型:38E23(New Zealand)。按照系统进化树划分体系分别与以上参考毒株进行比对,分析其同源性关系。
1.2.5 核苷酸与氨基酸序列比对
选取美洲型代表性毒株ATCC VR-2332、中国早期经典毒株CH-1a、高致病性代表性毒株JXA1以及NADC30毒株为参考毒株,使用BioEdit(v7.05)软件(网址:https://www.bioedit.com/)对56株TTSuV2型鉴定毒株以及参考毒株的Nsp2蛋白序列进行比对。
1.2.6 重组分析
使用RDP4软件中RDP、GENECONV、BootScan、MaxChi、Chimaera、SiScan和3eq方法检测潜在的重组事件及确定最可能含有重组基因片段的毒株,只有5种及以上的方法都认定为重组且其P值<0.05,才判定该事件中的毒株为重组毒株。其中BootScan检测方法的临界值百分率设为70%,窗口大小为200 bp,步长为20 bp其他参数为默认数值。使用SimPlot(v3.5.1)软件进一步检验重组事件,窗口大小设置为200 bp,步长设置为20 bp,其他参数为默认数值。
1.2.7 选择压力分析
使用在线分析服务器Datamonkey(http://www.datamonkey.org/)对鉴定的TTSuV2型毒株的ORF5基因进行选择压力分析,用SLAC、FEL、MEME、REL和FUBAR方法进行正选择压力分析,前3种方法的p值<0.1具有统计学意义且差异显著;REL方法以贝叶斯因子为统计学检验标准(Bayes Factors>50),FUBAR方法以后验概率为统计学检验标准(posteriori Probability>0.9);用SLAC、FEL、REL和FUBAR方法进行负选择压力分析,统计学检验标准和正选择压力分析相同。比较不同方法所得结果,只有2种及以上的方法检测到密码子位点受到非中性选择,且相应统计学检测方法均显示差异显著时,则认定该位点受到相对应的正选择压力或负选择压力。
2 结果与分析
2.1 目的基因PCR扩增
采用巢式PCR方法对样品的UTR基因进行特异性扩增,琼脂糖凝胶电泳显示PCR扩增得到大约250 bp目的片段(图1、图2),针对TTSuV2高变区目的基因进行特异性PCR扩增,琼脂糖凝胶电泳显示扩增得到目的片段大小约719 bp,均与预期大小相符(图3)。
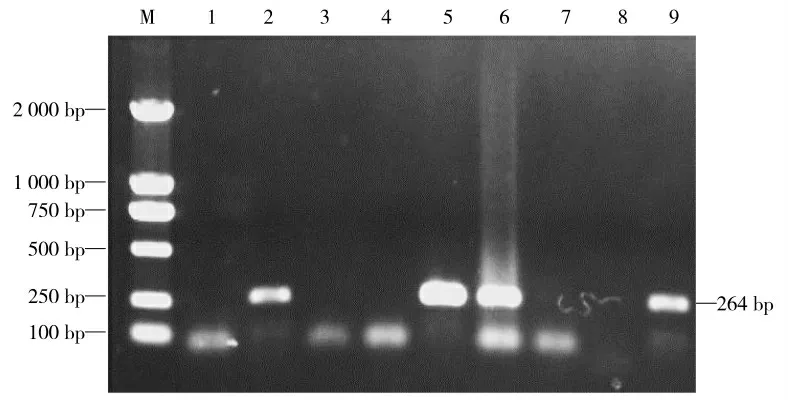
图1 TTSuV2 UTR基因第1轮PCR鉴定Fig.1 The identification for TTSuV2 UTR gene by first round PCR
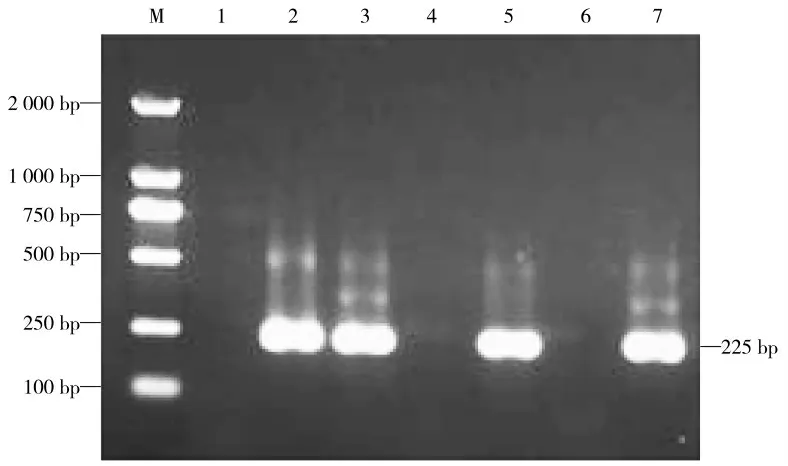
图2 TTSuV2 UTR基因第2轮PCR鉴定Fig.2 The identification for TTSuV2 UTR gene by second round PCR
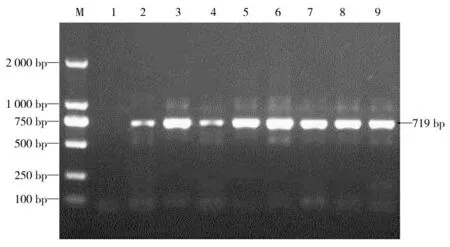
图3 TTSuV2基因组高变区PCR鉴定Fig.3 The identification for TTSuV2 genomic hypervariable region by PCR
2.2 TTSuV2样品阳性率统计
PCR检测结果显示,2016年收集中国6个地区腹泻样品共计174份,阳性样品66份,阳性率为37.93%;其中华南地区感染率最高,为56%;东北地区感染率最低,为30.26%。2017年收集中国5个地区腹泻样品共计151份,阳性样品26份,阳性率17.22%;其中华东地区感染率最高,为27.27%;华中地区感染率最低,为9.1%;相比2016年,东北、华北、华东、华中地区感染率均降低,且2017年总感染率也低于2016年。2016-2017年共计325份腹泻样品中阳性样品总数为92份,平均阳性率为28.31%(表5)。对92份阳性样品的目的基因进行克隆,结果共成功克隆出目的基因31条,并将测得目的基因序列处理后提交GenBank。
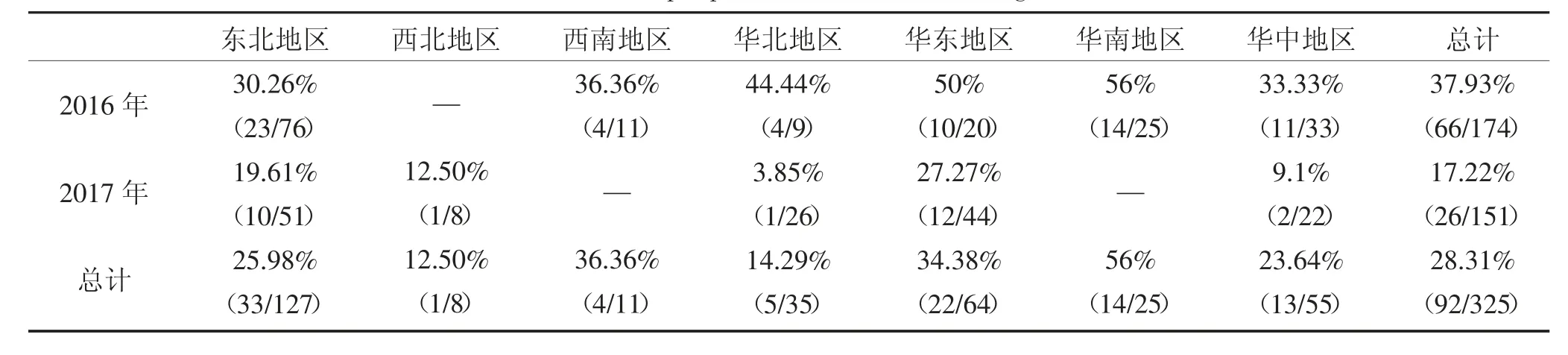
表5 2016-2017年TTSuV2样品阳性率统计Table 5 TTSuV2 sample positive rate statistics during 2016-2017
2.3 系统进化树分析
根据99株TTSuV毒株构建系统进化树结果显示,试验所鉴定31株TTSuV毒株全部属于Kappatorquevirus属,TTSuV k2a基因型。根据毒株间的亲缘关系将所有鉴定毒株划分为2个群(Group1-2)(图4)。共有来源于东北、华北、华东、华南、西南5个地区的21株TTSuV鉴定毒株分布于Group1。来源于东北、华北、华东3个地区的10株TTSuV鉴定毒株分布于Group2。Group1中前9株鉴定毒株与鉴定毒株与该属原型毒株2p及国内代表性毒株Porcine TTV 2 from China亲缘关系较近,其余12株鉴定毒株则与国内地方野毒株亲缘关系较近。Group2中前7株鉴定毒株与日本野毒株TTSuV2-KU2、TTSuV2-KU5亲缘关系最近,而鉴定毒株TTSuV2/508、TTSuV2/512与吉林野毒株TTV2Jl27亲缘关系最近,鉴定毒株TTSuV2/407则与广西野毒株TTV2Gx4亲缘关系最近。试验所有TTSuV鉴定毒株整体与2009-2011年国内野毒株亲缘关系较近,而与TTSuV k2b基因型毒株亲缘关系较远。
2.4 核苷酸与氨基酸同源性分析
全长目的基因核苷酸同源性比对结显示:31株TTSuV鉴定毒株自身相比,核苷酸同源性为87.5%~99.7%;与参考毒株相比,其与各国TTSuV k2a基因型代表性毒株的核苷酸同源性比较接近,均在86.7%~99.0%之间;与TTSuV k2b基因型代表性毒株38E23的核苷酸与氨基酸同源性很低,仅为59.9%~64.3%(表6)。
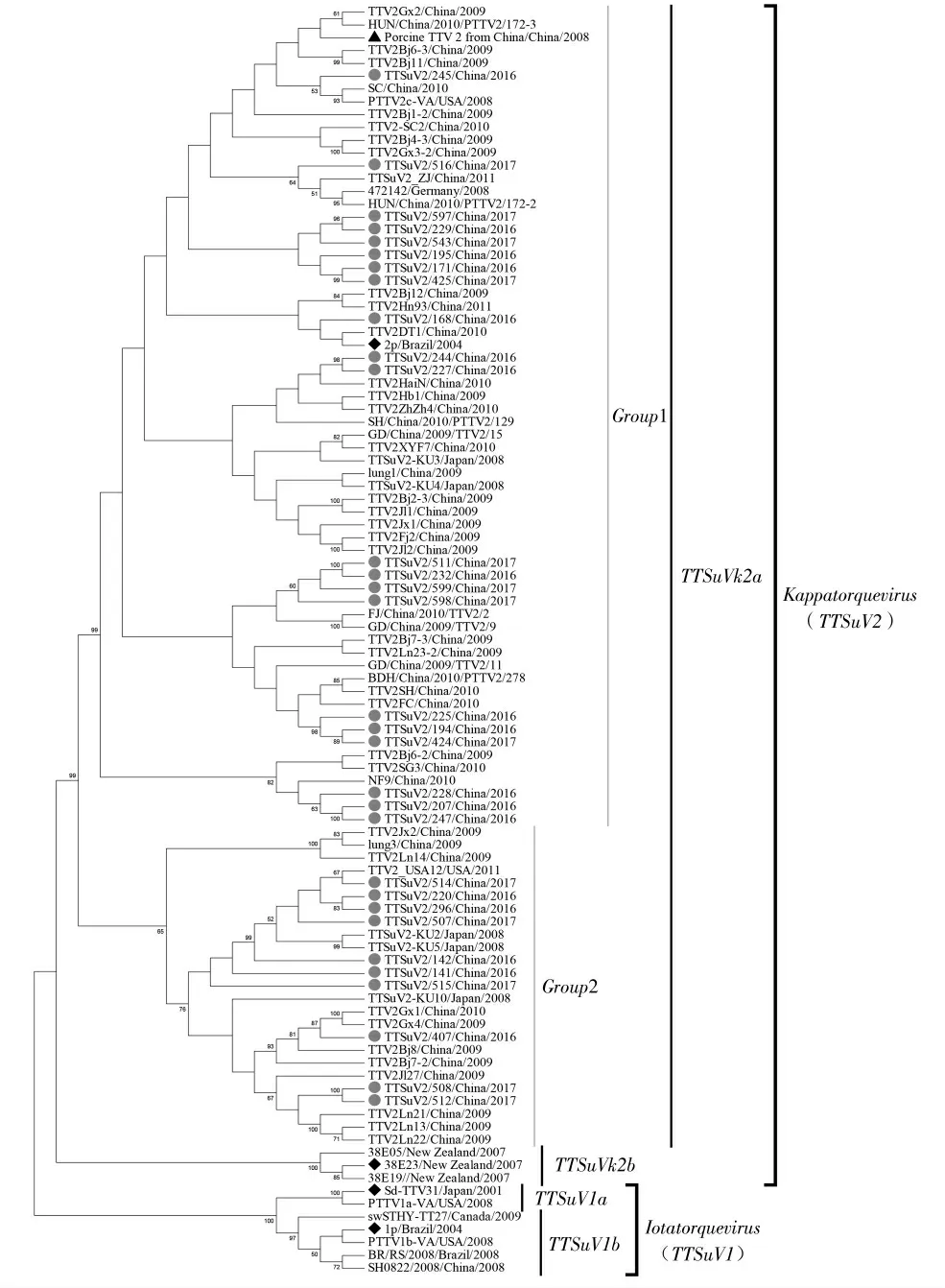
图4 TTSuV2基因组高变区系统进化树Fig.4 Phylogenetic analysis of TTSuV2 strains using genomic hypervariable region
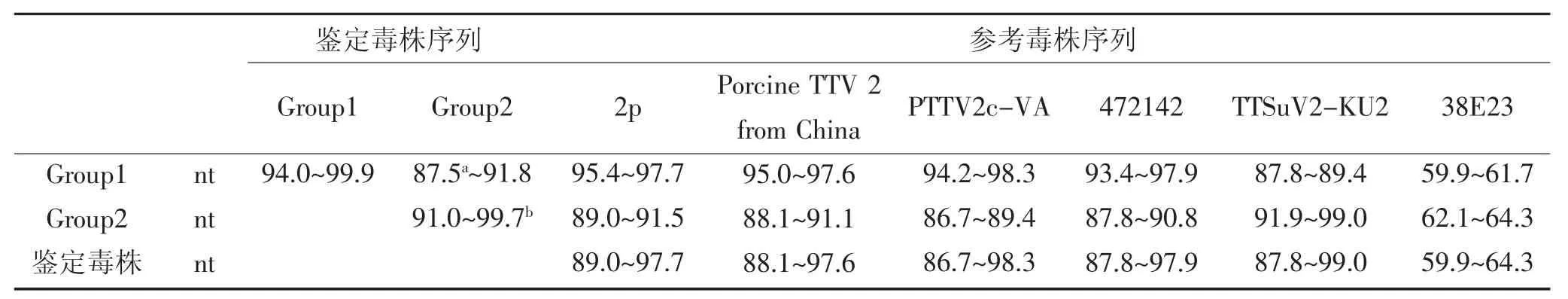
表6 TTSuV2基因组高变区核苷酸同源性分析(%)Table 6 Nucleotide homology analysis of TTSuV2 genomic hypervariable region
2.5 目的基因核苷酸与氨基酸序列比对
UTR基因序列比对结果显示:所有鉴定毒株的核苷酸突变位点主要集中在第51~230 nt,而其前50 nt及靠近ORF2的核苷酸序列比较保守,且所有毒株的TATA box基序均十分保守,未出现突变(图5)。

图5 UTR基因序列比对Fig.5 Alignment of UTR gene sequences
ORF2蛋白序列比对结果显示:所有鉴定毒株在第1~17 aa、22~34 aa、52~65 aa均比较保守,而在第39~44 aa、47~49 aa变异程度较大,所有鉴定毒株在蛋白酪氨酸磷酸酶基序的关键位点也比较保守,只有鉴定毒株TTSuV2/171出现一处突变,H25→Q25(图6)。Group1中鉴定毒株与国内代表性Porcine TTV 2 from China相比,氨基酸序列相似度很高;而与原型毒株2p相比主要出现4处氨基酸一致性突变,分别为S18→G18/D18/A18、E39→G39、H42→Q42/E42/R42、G43→E43。此外,鉴定毒株TTSuV2/207、TTSuV2/228、TTSuV2/247还存在1处与其他鉴定毒株不同的氨基酸突变,TGG47-49→HTD47-49。相比Group1,Group2中鉴定毒株的变异位点更多,也主要集中在39~49 aa;与原型毒株2p相比,其主要存在12处氨基酸一致性突变,分别为S18→D18、D21→N21/E21、D36→A36、D40→G40、R41→P41、G43→A43、D44→G44、T47→D47/V47/E47、G49→D49、D51→S51/G51/P51、A66→I66/T66、Q67→T67/I67;此外,所有Group2中鉴定毒株在第65~66 aa之间均插入一个D。

图6 ORF2蛋白基因序列比对Fig.6 Alignment of ORF2 protein sequences
部分ORF1蛋白序列比对结果显示:所有鉴定毒株在1~18 aa、35~38 aa、41~46 aa、50~58 aa、70~83 aa均比较保守,而在第19~23 aa、28~34 aa、39~40 aa、47~49 aa、59~63 aa变异程度较大(图7)。Group1中鉴定毒株与国内代表性Porcine TTV 2 from China相比,氨基酸序列相似度很高;而与原型毒株2p相比主要出现3处氨基酸一致性突变,分别为T39→A39/P39、K46→R46/G46、F54→N54。此外,鉴定毒株TTSuV2/207、TTSuV2/228、TTSuV2/247还存在2处与其他鉴定毒株不同的氨基酸突变,T14→R14、HR19-20→TH19-20。相比Group1,Group2中鉴定毒株的变异位点更多,与原型毒株2p相比,其主要存在14处氨基酸一致性突变,分别为H18→R18、R23→K23、R29→W29、Y30→H30、A33→P33、P34→T34/Y34、T39→H39/Y39、K47→R47、SR57-58→TI57-58、T60→N60、L62→Y62/F62、E64→K64、F66→I66、F75→L75;此外,所有Group2中鉴定毒株均在第38~39 aa之间均插入一个Y/R;除TTSuV2/141外,其余9株毒株均在第45~46 aa之间均插入一个V/I。
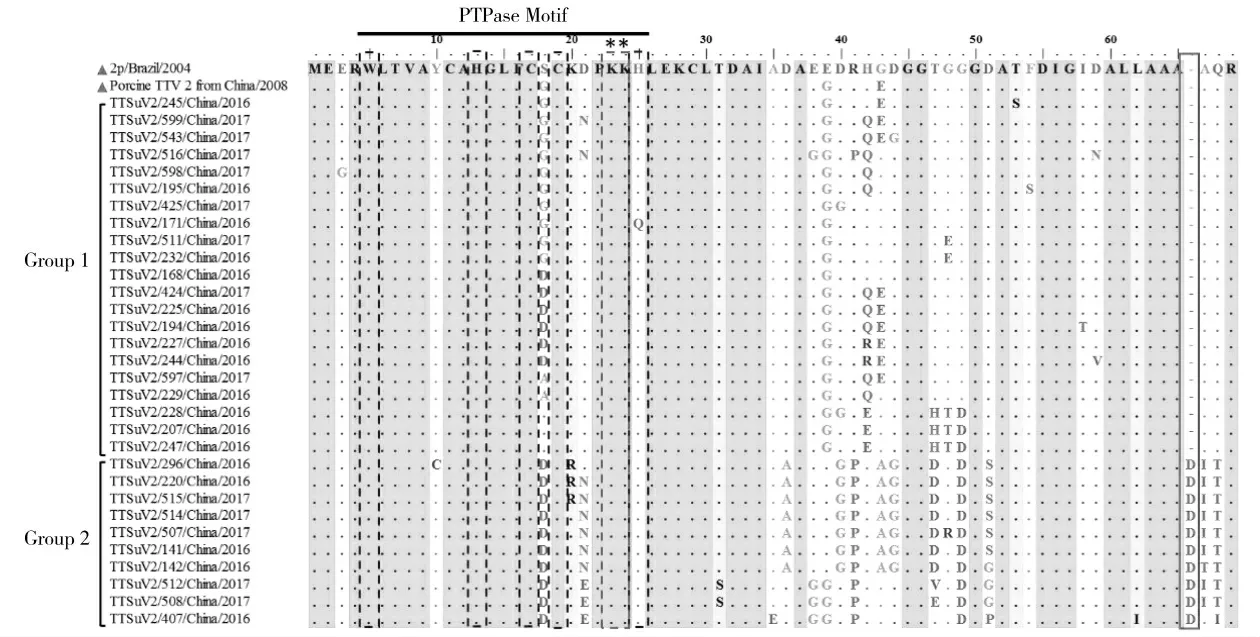
图7 ORF2蛋白基因序列比对Fig.7 Alignment of ORF2 protein sequences
2.6 重组分析
选取国内外参考毒株与试验鉴定毒株进行重组分析,结果显示共存在1个潜在的重组事件,事件仅包含潜在重组毒株TTSuV2/515,属于Group2(图8、表7)。其主要亲本毒株SH属于Group1,而次要亲本毒株TTV2_USA12属于Group2。BootScan检验和SimPlot序列相似性分析显示,潜在重组毒株TTSuV2/515毒株目的基因序列的前310 nt与上海野毒株SH序列高度相似,而后400 nt则与美国流行毒株TTV2_USA12高度相似。
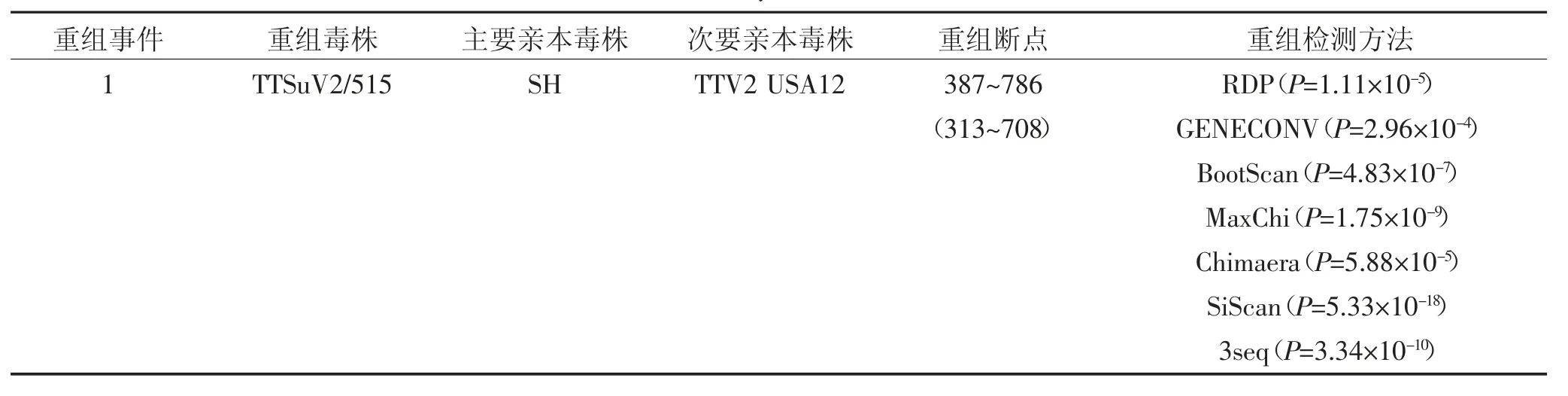
表7 TTSuV2鉴定毒株重组分析Table7 Recombination analysis of identified TTSuV2 strains

图8 BootScan和SimPlot检验Fig.8 BootScan and SimPlot verification
2.7 选择压力分析
ORF2基因进行选择压力分析结果显示,共检测到3个密码子位点受到正选择压力,与参考毒株相比均属于变异性较高的位点。共检测到4个密码子位点受到负选择压力,其中前3个密码子位点在参考毒株与所有鉴定毒株中均十分保守。此外,分析鉴定毒株ORF2基因的平均ω值为0.504,表明研究鉴定毒株的ORF2基因处于选择负压力(表8、表9)。
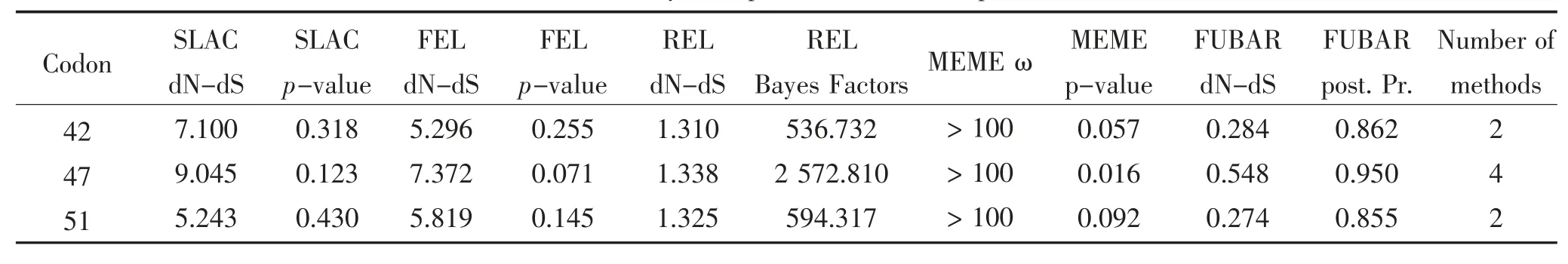
表8 正选择压力分析Table 8 Analysis of positive selection of pressure
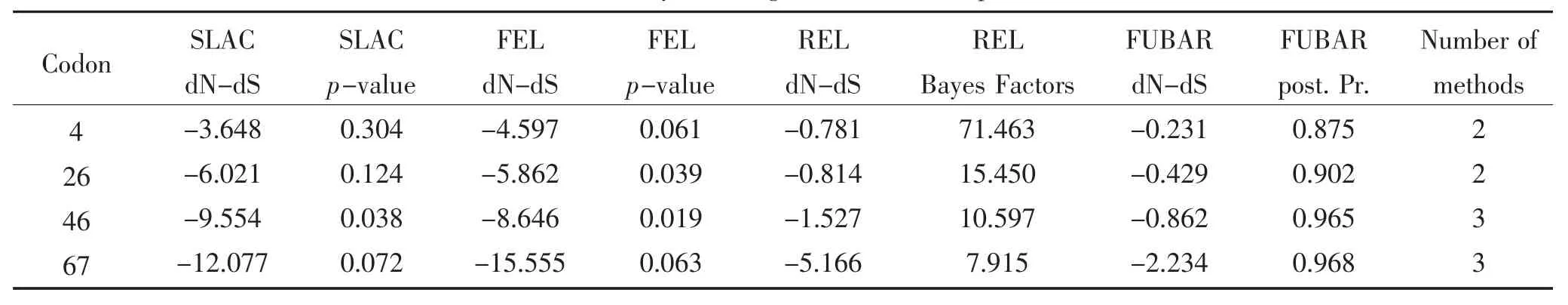
表9 负选择压力分析Table 9 Analysis of negative selection of pressure
3 讨论
2009年,王礞礞等[7]采用巢式PCR方法对TTSuV进行检测;研究借鉴此检测方法对2016-2017年中国7个地区TTSuV2的流行情况进行调查,结果与国内外学者对同种类型样品TTSuV2的检测结果存在一定差异,如:Opriessnig等[15]通过多重实时定量PCR方法对美国不同猪场腹泻猪的88份粪便样品进行多种腹泻病原的检测,结果显示TTSuV2的阳性率为48.9%。而将研究结果与近3年国内各地区学者报道TTSuV2的感染率相比,也存在一定的差异,如:2016年,王伟松等[16]通过多重巢式PCR方法对江西省11个县市采集的169份血清与组织样品进行检测,结果显示TTSuV总阳性率为50.3%,而其中TTSuV2的阳性率为40.83%。综合以上结果可知,TTSuV2在粪便中的检出率普遍低于血清与组织样品,这主要与TTSuV在各组织脏器中分布不同;此外,不同的检测方法、样本数量及来源也会导致阳性率的差异。此外,研究及国内许多研究者的检测结果也表明目前TTSuV2在我国的流行情况比较复杂,因此,TTSuV2变异情况及其与腹泻疾病的关联仍有待进一步深入研究。
系统进化树结果显示,研究所有TTSuV鉴定毒株均属于Kappatorquevirus属的TTSuV k2a基因型,其阳性率为33.7%(31/92),未发现TTSuV k2b基因型毒株。鉴定毒株的组群分布显示所有鉴定毒株被划分为2个群(Group1-2),结合鉴定毒株信息表分析发现,TTSuV2鉴定毒株的组群分布与其地域来源分布并不统一,许多来源相同的毒株却被划分到不同的组群。这表明研究鉴定毒株的组群分布无明显的地域性规律,各地区存在不同类型毒株的混合感染。这与梅淼[17]、张志成等[18]的研究结果相似。
基于全长目的基因与完整ORF2基因核苷酸同源性分析结果显示,研究TTSuV鉴定毒株自身相比,核苷酸同源性分别为87.5%~99.7%和84.2%~100%;与各国TTSuV k2a基因型代表性毒株的核苷酸同源性比较接近,均在86.7%~99.0%和85.0%~99.6%之间;而与TTSuV k2b基因型代表性毒株38E23的核苷酸与氨基酸同源性很低,仅为59.9%~64.3%和37.3%~41.8%。这与Cadar[25]、Liu[20]、Cornelissen-Keijsers等[9]研究结果相类似。其中,Group1中鉴定毒株自身相比,核苷酸同源性差异度低于Group2,且与参考毒株PTTV2c-VA相比核苷酸同源性最高,与TTSuV2-KU2的核苷酸同源性最低,而Group2中鉴定毒株则与之相反。这表明Group2中鉴定毒株的核苷酸与氨基酸变异程度更大,毒株间序列差异也更大。
UTR核苷酸序列比对结果显示:所有鉴定毒株核苷酸突变位点主要集中在51~230 nt,而其两端的核苷酸区域比较保守。研究鉴定毒株与原型毒株2p相比,存在多处核苷酸插入/缺失突变,其中大部分鉴定毒株只存在1~2处插入/缺失突变,但Group2中鉴定毒株除TTSuV2/515之外,均在第121~122 nt之间存在连续11 nt的插入,这与温立斌等[21]研究结果相类似。此外,研究还发现一些组群特征性的核苷酸突变,由于TTSuV的UTR包含重要的转录调控元件,这些核苷酸的突变不仅会影响其基因组的大小,还有可能改变TTSuV的转录效率。
ORF2蛋白序列比对结果显示:所有鉴定毒株均在第39~49 aa变异程度较大,而Group2中鉴定毒株的ORF2蛋白序列比Group1鉴定毒株变异程度更大,突变位点也更多。目前,人们对于ORF2蛋白结构与功能还并不了解[23],因此以上这些突变是否会影响TTSuV的ORF2蛋白的活性与功能,还需要进行更深入的研究。
部分ORF1蛋白序列比对结果显示:研究鉴定毒株均在第19~50 aa变异程度较大,而之前有研究表明在TTSuV2毒株ORF1蛋白N末端,尤其是前50 aa是该蛋白的亲水区,也高抗原性区域,因此该区域的突变可能有利于TTSuV逃避宿主的免疫应答[8,22]。
研究表明:TTSuV的平均遗传进化率为5.29~5.51×10-4site/y,且TTSuV的核苷酸替代率远高于典型的DNA病毒;而与RNA病毒的水平相当[24]。研究对31株鉴定毒株进行重组分析,结果显示共存在1株潜在重组毒株TTSuV2/515,属于Group2组群。其主要亲本毒株SH属于Group1,而次要亲本毒株TTV2_USA12属于Group2,重组断点位于UTR。这与之前同源性分析及序列比对结果推测相符,也与之前Cadar等[25]研究结果相类似。以上研究结果表明基因重组是TTSuV快速变异的重要方式之一。
2011年,Cortey等[24]基于不同基因型TTSuV毒株全基因组研究其遗传变异和系统发育特点,结果显示TTSuV2毒株的ORF2基因中14个密码子位点受到正选择压力,其主要集中在第40~69 aa;9个密码子位点受到负选择压力,其主要集中在第20~40 aa,ω值约为0.269。随后,Cadar等[19]在TTSuV2型毒株中也鉴定出14个码子位点受到正选择压力,也主要集中在ORF2基因的C末端,TTSuV2-2a平均ω值为0.87,TTSuV1-2b平均ω值为0.51。与以上研究结果相比,鉴定毒株的ORF2基因中3个受到正选择压力的密码子位点也均位于ORF2基因的C末端,而研究检测到4个受到负选择压力的密码子位点则位于ORF2基因的两端。此外,鉴定毒株ORF2基因的平均ω值为0.504,高于Cortey等研究结果而低于Cadar等研究结果。
4 结论
2016-2017年中国7个地区均存在TTSuV2型毒株的流行,平均感染率为28.31%,且许多地区存在不同组群毒株混合感染的情况。
TTSuV发生较大程度的变异,大部分流行毒株的基因序列中存在不同程度的插入/缺失突变,甚至还存在基因重组现象。

