奶牛乳房炎肠杆菌科主要病原菌OmpA理化特性、同源性及抗原表位生物信息学分析
王然,范钊玮,马骏,段旭阳,王欣,刘硕,周玉龙,朱战波,崔玉东,
(1.黑龙江八一农垦大学动物科技学院,大庆 163319;2.黑龙江八一农垦大学生命科学技术学院)
奶牛乳房炎是严重影响奶牛养殖业和奶业健康发展的重要疾病,临床上由革兰氏阴性肠杆菌科细菌引起的感染常常占有较大比例[1-3]。引起奶牛乳房炎的肠杆菌科细菌中,除了常见的埃希氏大肠杆菌(Escherichia coli),还有克雷伯氏菌[4]、沙门氏菌[5]、志贺氏菌[6]、沙雷氏菌[7-8]、阴沟肠杆菌[9]、柠檬酸杆菌[10]、变形杆菌[11-12]等。随着临床上长期、大量使用抗生素防治奶牛乳房炎,这些病原菌逐渐产生了耐药性和多耐药性,导致抗生素疗效甚微。进而,研究和使用疫苗预防大肠杆菌性乳房炎成为必然的选择。自上世纪80年代末,人们开始研究和使用疫苗预防埃希氏大肠杆菌引起的奶牛乳房炎,但尚未获得满意的临床可用疫苗[13-14]。近年来对E.coli外膜蛋白A(OmpA)抗原进行免疫试验研究证实[15-16],OmpA能够诱导小鼠产生良好的免疫应答和抗E.coli感染免疫保护作用,同时也能一定程度地抵抗志贺氏菌和克雷伯氏菌攻击,展现了良好的开发和应用前景。
OmpA是存在于革兰氏阴性菌外膜上的一种蛋白质,对维持外膜结构完整性发挥重要作用,也是噬菌体感染的受体和吸收大肠杆菌素K和L的必需结构[17]。但对引起奶牛乳房炎的肠杆菌科细菌的OmpA同源性、抗原性等尚缺乏认识。研究拟对引起奶牛乳房炎的肠杆菌科细菌OmpA的理化性质、同源性和抗原表位进行生物信息学分析,为进一步研究OmpA诱导的交叉免疫保护作用和建立OmpA免疫牛血清抗体检测方法提供参考。
1 材料与方法
1.1 试验用菌株
试验用菌株包括分离于奶牛乳房炎病牛乳汁的E.coli2002-1、SY200105和308-2株,分离于犊牛临床腹泻粪便样品的E.coliN150601、JS160801和SH16041株,以上菌株均由实验室鉴定保存;分离于病牛的肺炎克雷伯氏菌KPDN202010株来自于东北农业大学动物传染病学实验室;牛源沙门氏菌BNCC186363株购于北京北纳创联生物技术研究院;福氏志贺氏菌CMCC(B)51572株和粘质沙雷氏菌CMCC41002株购自广东环凯微生物科技有限公司。
1.2 菌种复苏
试验用上述菌株用接种环划线接种于LB琼脂平板上,于37℃恒温培养箱培养12 h以上,检查菌落形态特性和一致性,再挑取单菌落接种至LB液体培养基中,于37℃恒温摇床180 r·min-1培养过夜,然后对细菌进行形态染色和主要生化特性检查。
1.3 OmpA基因扩增及测序
为了扩增OmpA基因,使用Primer 5.0设计PCR扩增引物。根据GenBank中有关菌株的基因序列(NC_000913.3)设计的上游引物命名为OMPA-F,下游引物为OMPA-R;对于肺炎克雷伯氏菌(参考WP_148865386.1)和粘质沙雷氏菌(参考WP_062792 903.1),下游引物分别为Kle-R和Ser-R,各引物序列详见表1。反应体系与反应条件参照参考文献[15]。扩增产物经1%琼脂糖凝胶电泳鉴定后,送吉林库美生物科技有限公司进行测序。

表1 OmpA基因PCR扩增引物Table 1 Primers used for OmpA gene amplification by PCR
1.4 参考菌株OmpA基因序列获取
研究还从GenBank数据库中选取19株细菌的OmpA基因序列作为参考(见表2),包括E.coli2株、肺炎克雷伯氏菌2株、牛源沙门氏菌2株、福氏志贺氏菌2株、粘质沙雷氏菌2株、阴沟肠杆菌3株、弗氏柠檬酸杆菌3株、奇异变形杆菌3株。
1.5 OmpA蛋白理化性质分析
使 用ExPASy(https://www.expasy.org/)的Prot-Param功能分析OmpA蛋白的理化性质、ProtScale功能分析OmpA蛋白的亲疏水性;使用DNAstar软件中Protean功能分析OmpA蛋白的抗原性。

表2 19株参考菌株OmpA基因序列Table 2 OmpA gene sequences of 19 reference strains from GenBank
1.6 OmpA蛋白二级结构与三级结构分析
使用PRABI(http://www.prabi.fr/)的HNN二级结构预测方法对OmpA蛋白的二级结构进行分析;使用SWISS-MODEL建模服务器(https://swissmodel.expasy.org/)对OmpA蛋白的三级结构进行建模。
1.7 OmpA遗传进化分析与序列比对
使用BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)对各菌株的OmpA氨基酸序列进行搜索比对,分析保守性;使用DNAstar软件中MegAlign功能对OmpA蛋白进行同源性分析;使用Geneious Prime软件对各菌株OmpA蛋白的氨基酸序列进行比对。
1.8 OmpA蛋白的表位预测
使用IEDB数据库(http://www.iedb.org/)与BepiPred1.0(http://www.cbs.dtu.dk/services/BepiPred-1.0/)预测OmpA蛋白的线性B细胞表位。使用NetMHCIIpan 4.0(http://www.cbs.dtu.dk/services/NetMHCIIpan/)与IEDB数据库预测OmpA蛋白的CD4+T细胞表位。在B细胞预测结果中,构成某区域的氨基酸评分越高,该区域成为B细胞表位的可能性越大。
2 结果与分析
2.1 琼脂糖凝胶电泳结果
将OmpA基因的PCR扩增产物以1%琼脂糖凝胶进行电泳,在1 000 bp之上的位置可见一条清晰的目的条带(见图1),与预期的基因片段大小吻合,但各菌株间碱基数略有差异。剩余的PCR扩增产物用于测序。
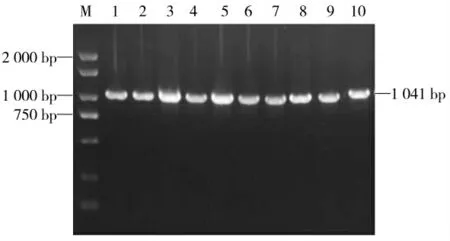
图1 PCR扩增产物琼脂糖凝胶电泳结果Fig.1 Agarose gel electrophoresis results of the amplified products by PCR
2.2 OmpA蛋白理化性质
E.coli、肺炎克雷伯氏菌、牛源沙门氏菌、福氏志贺氏菌、粘质沙雷氏菌、阴沟肠杆菌、弗氏柠檬酸菌、奇异变形杆菌的OmpA蛋白分别由346、356、350、359、348、351、352、358个氨基酸组成;等电点分别为5.43、5.72、5.18、5.17、7.04、5.47、5.46、5.66;不稳定系数分别为22.47、23.15、29.15、23.72、17.80、28.01、27.12、34.13,全部小于40,判定为稳定蛋白;亲水性平均值分别为-0.399、-0.395、-0.425、-0.376、-0.387、-0.331、-0.379、-0.331,全部位于-2~2之间,表明各菌株的OmpA均为亲水性蛋白;抗原性指数峰值越大则该部分抗原性越强,各菌株的OmpA均具有良好的抗原性(详见图2)。
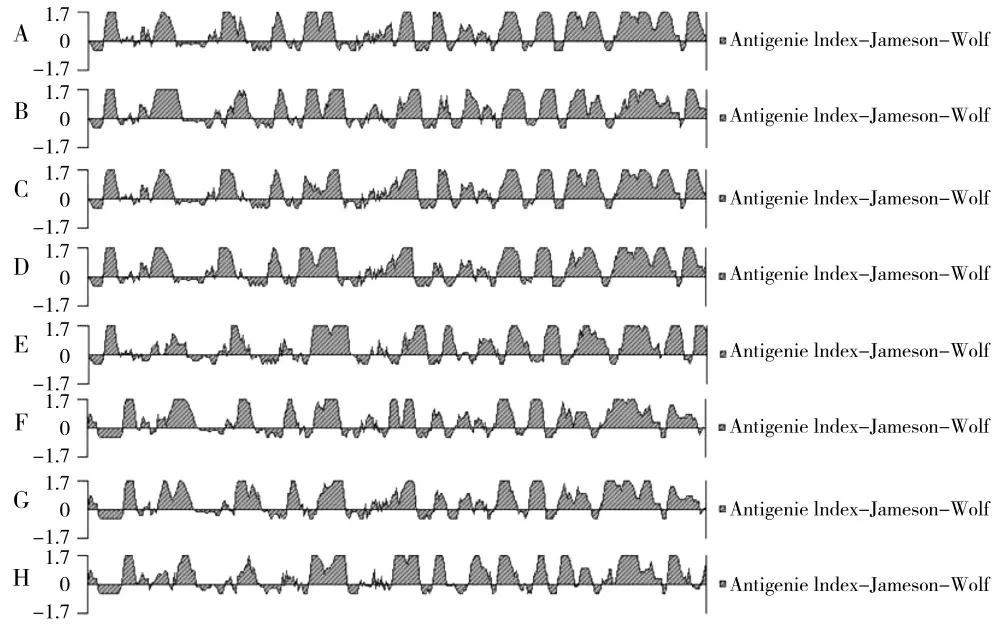
图2 OmpA蛋白抗原性指数Fig.2 Index of OmpA protein antigenicity
2.3 OmpA蛋白二级结构与三级结构分析结果
在E.coli、肺炎克雷伯氏菌、牛源沙门氏菌、福氏志贺氏菌、粘质沙雷氏菌、阴沟肠杆菌、弗氏柠檬酸菌、奇异变形杆菌的OmpA蛋白二级结构中,无规则卷曲各占61.80%、59.46%、62.20%、62.65%、55.12%、56.98%、56.82%、54.47%;α螺 旋 各 占22.05%、27.33%、21.65%、21.91%、31.63%、31.05%、28.98%、29.33%;延伸主链各占16.15%、13.21%、21.65%、21.91%、13.25%、11.97%、14.20%、16.20%。由此可见,OmpA蛋白的无规则卷曲占比较大,更易形成细胞表位。在OmpA蛋白三级结构预测中,同属内各菌株间OmpA三级结构呈现高度一致性,各属细菌OmpA与数据库中OmpA三级结构模型的一致性分别 为99.38%、86.38%、93.50%、96.56%、77.66%、88.73%、89.60%、71.30%。
2.4 OmpA同源性分析结果
8株E.coli的OmpA同源性为96.8%~100%,3株肺炎克雷伯氏菌OmpA同源性为96.1%~98.3%,3株牛源沙门氏菌OmpA同源性为99.4%~99.7%,3株福氏志贺氏菌OmpA同源性为100%,3株粘质沙雷氏菌OmpA同源性为99.7%,3株阴沟肠杆菌OmpA同源性为97.2%~99.4%,3株弗氏柠檬酸菌OmpA同源性为100%,3株奇异变形杆菌OmpA同源性为99.4%~100%。
各属间细菌OmpA氨基酸序列同源性为99.7%~69.3%。E.coli与福氏志贺氏菌、沙门氏菌高度同源,分别为97.2%和94.1%,而致腹泻E.coliJS160801与福 氏 志 贺 氏 菌CMCC(B)51572、AIL34962.1、QLG52127.1的同源性比其他E.coli株更高,均为99.1%。E.coli与其它各属细菌OmpA氨基酸序列同源性详见表3。
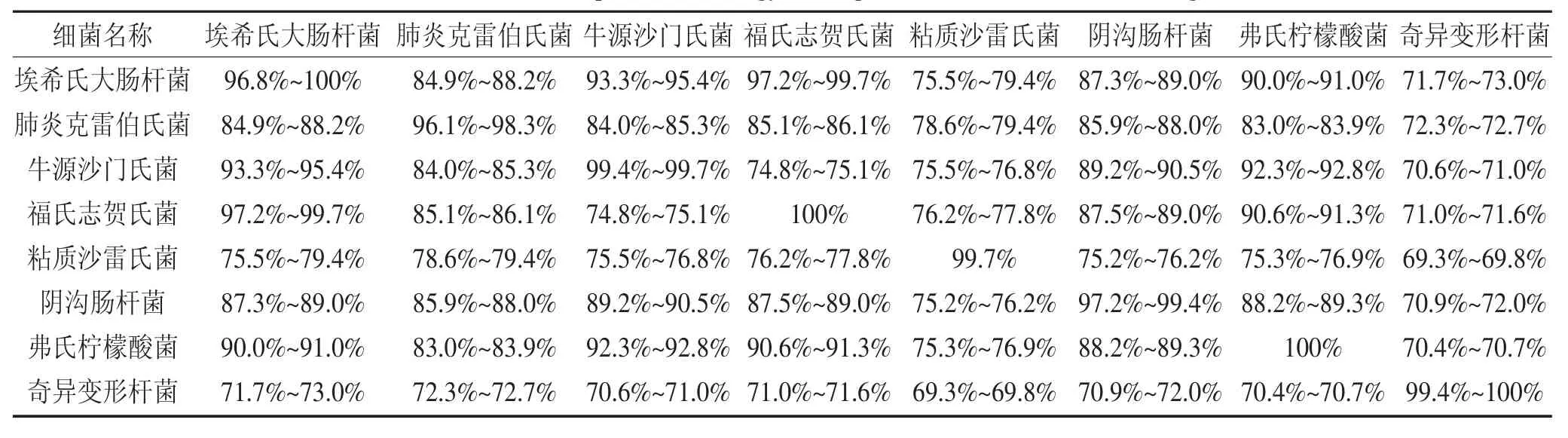
表3 各属间细菌OmpA氨基酸序列同源性Table 3 Amino acid sequence homology of OmpA from bacteria of different genus
将29株细菌OmpA氨基酸序列进行对比分析显示,氨基酸变异的区域主要集中在40~55 aa、84~93 aa、126~134 aa、143~153 aa、165~176 aa、192~205 aa、237~249 aa、266~275 aa、292~304 aa、313~320 aa十个区域,其中前3个和第5个氨基酸突变区域分别位于膜外4个Loop上;第4个和后5个氨基酸突变区域位于跨膜区域上。不同菌株的4个Loop区域存在着大量氨基酸变异,第1个Loop(Loop1)和第3个Loop(Loop3)区域比其他区域变异多,而各Loop两端附近的跨膜区域往往高度保守。结果详见图3。

图3 OmpA氨基酸序列对比分析Fig.3 Amino acid sequence alignment of OmpA from strains
2.5 OmpA的CD4+T细胞表位和线性B细胞表位预测结果
根据预测结果分析,各细菌OmpA表位预测的重叠部分存在共同表位的可能性大。由此得出,各属细菌OmpA上存在1个共同的CD4+T细胞优势表位,表位的具体位置和氨基酸序列详见表4。表位由9个氨基酸组成且高度保守,表位氨基酸序列中仅6个别菌存在第3位的I突变为V、第9位的A突变为S。
各属细菌存在OmpA上存在6个共同的线性B细胞优势表位,表位1、2、3和4位于膜外的Loop1、Loop2、Loop3和Loop4上,表位5和6位于跨膜区域上,且各表位都存在着不同程度的变异,结果详见表 5和表6。

表4 OmpA的CD4+T细胞表位预测结果Table 4 Prediction results of CD4+T cell epitopes in OmpA

表5 OmpA的线性B细胞表位预测结果Table 5 Prediction results of linear B cell epitopes in OmpA
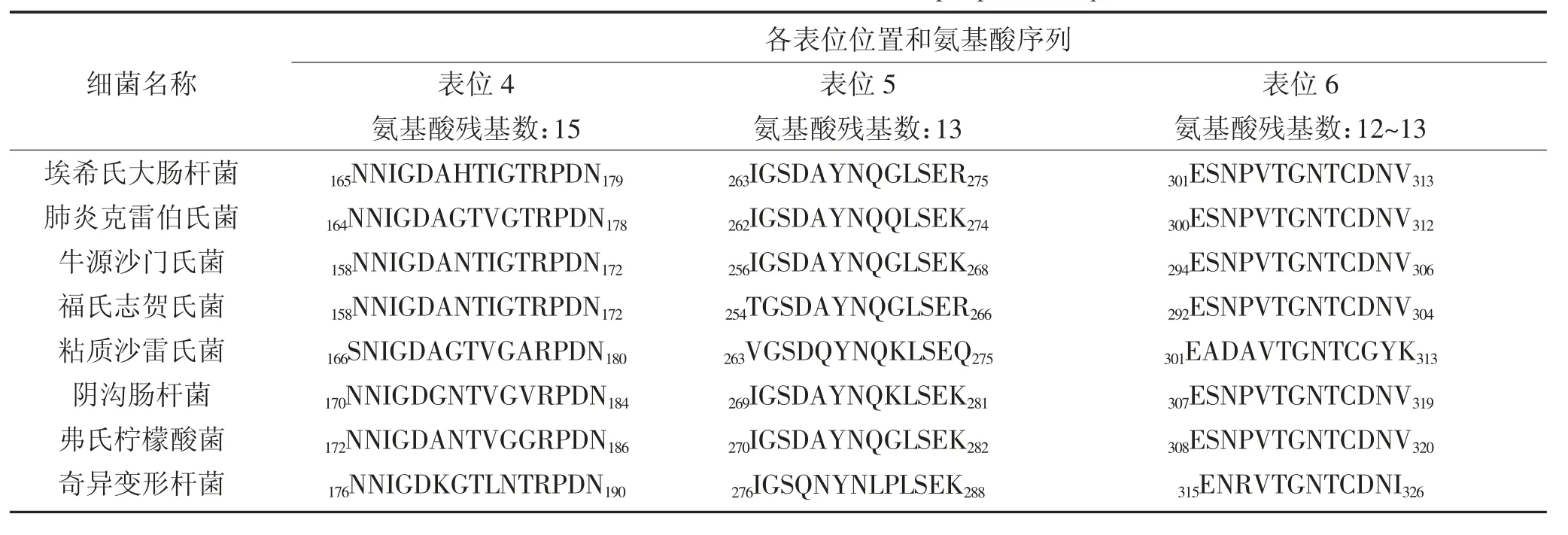
表6 OmpA的线性B细胞表位预测结果Table 6 Prediction results of linear B cell epitopes in OmpA
3 讨论
研究根据相关报道,选择了引起奶牛乳房炎的肠杆菌科8个菌属的29株细菌OmpA进行了生物信息学分析。为了使分析数据具有代表性和说服力,每个菌属至少选取3株细菌的OmpA序列进行比较和分析。
通过对各菌株OmpA的理化性质、蛋白质二三级结构、氨基酸同源性、CD4+T细胞表位以及线性B细胞表位等初步分析表明,蛋白理化性质稳定,属于亲水性蛋白,具有良好抗原性;属内菌株OmpA氨基酸序列高度同源,属间菌株OmpA同源性在99.7%~69.3%之间;各菌属菌株都存在1个保守的共同CD4+T细胞表位,6个不同程度变异的共同线性B细胞表位。
在抗原诱导机体产生特异性免疫应答过程中,抗原提成细胞通过提呈CD4+T细胞表位来刺激CD4+T细胞活化,并分化成各种辅助性T细胞,进而发挥调节B细胞产生保护性抗体、促使CD8+T细胞活化和增强吞噬细胞功能等作用[18]。因此,对OmpA的共同CD4+T细胞表位进行预测分析,在各菌OmpA分子上存在1个共同CD4+T细胞表位,由9个氨基酸组成,但存在两个氨基酸变异,即I突变为V和A突变为S。I和V两种氨基酸均为支链、非极性氨基酸,pH均为5.5~6.5,但I比V多出一个-CH2-,由此分析它们之间的变异可能不会影响表位的性质和功能。A与S两种氨基酸均为3碳氨基酸,A的pH5.5~7.0,非极性氨基酸;S的pH5.5~6.5,但S带-OH,为不带电荷的极性氨基酸,实验室在研究CD4+T细胞表位时,曾将S突变为A,表位的性质和功能没有明显变化,仅表现出诱导CD4+T细胞产生的IL-17水平略有降低[19]。可以看出,CD4+T细胞表位保守性良好,各属细菌间可能会诱导CD4+T细胞交叉免疫应答,且与交叉免疫保护作用密切相关[20]。
B细胞表位是抗原分子中被B细胞受体和抗体特异性识别并结合的部位。对OmpA的线性B细胞表位预测分析显示,OmpA上存在6个线性B细胞表位,从OmpA氨基端到羧基端依次称其为表位1、2、3、4、5和6,其中前4个表位分别位于OmpA的Loop1、Loop2、Loop3和Loop4上,表位5和6则位于跨膜的区域上。伍娜娜等[21]对OmpA生物信息学分析后,预测存在4个线性B细胞表位,与预测的表位1、2、3、6一致。进一步比较不同种属细菌的B细胞表位看出,表位5和6的氨基酸序列保守性最高,多个菌属细菌间表位序列存在2个以下的氨基酸变异,推测多能产生抗体交叉反应;表位2和4在多个菌属细菌间存在2~3个氨基酸变异,而表位1和3在多个菌属细菌间存在3个以上的氨基酸变异,变异较多的可能会影响抗体反应性。因此推测,在B细胞表位氨基酸变异少的菌属细菌间,如E.coli与志贺氏菌、沙门氏菌、克雷伯氏菌间可能会发生抗体交叉反应。呼锐[14]研究证实,用E.coli OmpA免疫小鼠后,小鼠可一定程度抵抗志贺氏菌和克雷伯氏菌攻击,说明它们存在着交叉免疫保护作用,进一步讲很可能存在抗体交叉反应。当然,各表位诱导抗体交叉反应、提供交叉免疫保护的确切结论尚需要进一步的直接试验证据。
上述各菌属细菌的OmpA同源性较高,免疫原性良好,且存在保守的共同CD4+T细胞表位和能够诱导抗体交叉反应的B细胞表位,为研究开发OmpA作为预防由肠杆菌科不同细菌引起的奶牛大肠杆菌性乳房炎候选疫苗带来信心,也为建立相关的血清学检测方法提供参考。因此,研究具有重要的实际意义和应用价值。

