玉米农艺性状配合力全基因组关联分析和预测
马 娟 刘京宝 朱卫红 黄 璐 宇 婷 乔江方
(河南省农业科学院粮食作物研究所,河南 郑州 450002)
玉米(Zea maysL.)是杂种优势利用最成功的作物之一,主要体现在单交种的生产和利用上[1]。在玉米杂交育种中,选育高配合力的亲本材料是培育高产杂交种的先决条件[2]。一般配合力(general combining ability,GCA)是指一个自交系与其他自交系组配后代在某个性状的平均表现,是评价亲本自交系利用价值的一个重要指标[1,3]。因此解析GCA 的遗传机理对玉米杂交种产量的提高具有重要意义。
GCA 和农艺性状均是多基因控制的数量性状,可以利用连锁定位和关联分析方法开展研究。Qi等[4]利用75 份玉米导入系材料和4 个测验种定位到75 个控制产量及相关性状GCA 的数量性状位点(quantitative trait loci,QTL),其中穗行数GCA的显著QTL有6个。Lu等[5]以328 个玉米重组自交系和2 个测验种组配的656个杂交种为材料,利用复合区间作图方法挖掘64 个控制产量等性状GCA 的关键位点。Liu 等[6]利用1 个重组自交系群体和4个测验种组配的杂交群体鉴定41个控制玉米穗行数、行粒数等性状GCA 的关键位点和28个热点区域。李婷等[3]利用EMMAX(efficient mixedmodel association expedited)算法对246 份玉米杂交组合的籽粒相关性状配合力进行了关联分析,检测到9、7 和5 个单核苷酸多态性(singe nucleotide polymorphism,SNPs)分别与粒长、粒宽和粒厚GCA 显著关联,单个位点解释GCA表型变异率为0.02%~25.52%。
尽管已有研究者挖掘到一些控制GCA 的关键位点,但是这些位点的遗传利用尚缺乏相关的分子改良策略。基因组选择(genomic selection,GS)是利用训练群体覆盖全基因组的分子标记和表型数据建立数学模型,从而对基因型已知的候选群体进行表型预测的一种分子育种技术。GS已广泛应用于预测玉米、水稻和小麦等作物亲本和杂交种产量等表现,为GS 辅助育种技术的应用提供了重要参考。王欣等[7]基于不完全双列杂交设计(North Carolina II,NCII)的水稻数据集,研究了单株产量等性状GCA的预测力,发现单株产量等8个性状的预测准确性介于0.39~0.74。Zhang等[8]利用32个玉米自交系和9 个测验种组配的3 组测交群体为材料对产量GCA 开展预测研究,发现仅利用自交系的信息对产量GCA 的预测精度较低,为-0.14~0.13,当加入测验种的信息时,产量GCA 的预测准确性可提高到0.51~0.65。
穗行数是玉米产量的重要构成因子之一,籽粒性状例如粒长、粒宽与玉米品质及产量密切相关,解析其GCA 遗传机理对玉米杂交种产量和品质的遗传改良具有重要意义。虽然穗行数和籽粒性状以及其分子遗传研究基础已有相关报道,但目前关于穗行数和籽粒性状GCA 全基因组关联分析(genome-wide association study,GWAS)的研究较少,而且其基因组预测分析的研究仍鲜见报道。鉴于此,本研究选用123份玉米自交系和8个测验种按照NCII 遗传交配设计获得537 份杂交组合为材料,采用7 种多位点GWAS(multi-locus GWAS,MGWAS)方法挖掘穗行数、粒长、粒宽GCA显著关联位点,并研究不同显著关联位点作为固定效应对提高3个性状GCA预测精度的影响,旨在为基因功能验证以及关键位点的基因组选择辅助育种提供重要的基因信息和技术指导。
1 材料与方法
1.1 试验材料、试验设计和配合力分析
以123份玉米自选系和8个测验种(农系531、昌7-2、20H1419、M189、M119、S110T、L119A 和PH4CV)采用NCII遗传交配设计组配537个F1杂交种。537个杂交种采用随机完全区组试验设计,于2021 年种植在河南省新乡和周口试验田,2次重复。新乡和周口环境行长分别是4.0和3.3 m,株距和行距均分别是0.22和0.60 m。收获后,每份材料选取2 个代表性果穗人工测量穗行数。利用玉米果穗/籽粒考种流水线仪器(国家农业信息化工程技术研究中心)测量约20 粒籽粒的粒长和粒宽,获得单粒粒长和粒宽用于分析。利用R 语言lme4包的lmer 函数计算2 个环境的GCA 效应值(公式1),并将2 个环境数据联合计算综合环境的GCA 效应值(公式2),均用于后续分析:
其中,y、μ、B、E、GCAL、GCAT、SCA以及ε分别表示杂交种表型、均值、区组、环境、自交系GCA、测验种GCA、特殊配合力以及误差效应。GCAL×E、GCAT×E和SCA×E分别表示自交系GCA 与环境互作、测验种GCA 与环境互作和特殊配合力与环境互作效应。
根据公式2,利用R 语言stats 包aov 函数将新乡和周口环境数据联合进行多环境方差分析。不同环境3个性状GCA 相关性分析(Pearson)和显著性检验利用R语言GGally包计算和展示。
GCA遗传力的计算公式如下:

1.2 基因型检测和分析
131份玉米自交系(123份自交系和8个测验种)叶片的DNA 采用CTAB 方法提取。利用玉米5.5K 液相育种芯片(河南省农业科学院粮食作物研究所)根据液相探针杂交原理进行靶向测序基因型鉴定,测序平台为NovaSeq 6000 (Illumina,美国)。利用BWA(v0.7.17)软件将过滤的reads 与玉米B73 第四版参考基因组(http://www.gramene.org/)比对。利用GATK (v4.1.2.0)软件检测原始变异位点33 971个。经过最小等位基因频率<0.05,缺失率>10%和杂合率>1%的过滤,获得11 734个高质量单核苷酸多态性(single nucleotide polymor hism,SNP)用于后续分析。
1.3 多位点全基因组关联分析和候选基因挖掘
利用7种MGWAS方法,即BLINK(bayesian information and linkage-disequilibrium iteratively nested keyway)[9]、FarmCPU(fixed and random model circulating probability unification)[10]、mrMLM(multi-locus random-SNP-effect mixed linear model)[11]、FASTmrMLM[12]、FASTmrEMMA(FAST multi-locus random-SNP-effect EMMA)[13]、pLARmEB(polygene-background-control-based least angle regression plus empirical Bayes)[14]和ISIS EM-BLASSO(iterative sure independence screening EM-Bayesian least absolute shrinkage and selection operator)[15]对3个环境穗行数和籽粒性状GCA进行关联分析。7种方法均考虑群体结构Q值和亲缘关系K值。群体结构Q值由Structure v2.3.4[16]计算,其中亚群数为1~10,length of burn-in period为5 000,蒙特卡罗重复个数为50 000,每个亚群数迭代次数为3。利用Structure Harvester(v0.6.94)确定最佳的亚群数为6。3次重复的Q值由CLUMPP(v1.1.26)软件的FullSearch算法 确 定[17]。亲 缘 关 系K 值 由TASSEL v5.0 软 件 的Centered_IBS 方法计算[18]。BLINK 和FarmCPU 利用R语言GAPIT 包计算[19],其他5 种方法均利用R 语言mrMLM包计算[20]。显著临界值设置为P=0.01/11 734=8.52×10-7。利用SnpEff[21]根据显著SNPs 的位置挖掘候选基因,参数按默认设置。
1.4 基因组预测分析
采用贝叶斯A(Bayes A)[22]、Bayes C[23]、Bayesian LASSO[24]、基因组最佳线性无偏预测(genomic best linear unbiased prediction,GBLUP)[25]和再生核希尔伯特空间(reproducing kernel Hilbert space,RKHS)[26]对3个环境穗行数、粒长、粒宽GCA 进行预测分析。5种方法中分子标记的效应均设置为随机效应即随机效应模型。根据不同MGWAS 方法挖掘的关联位点信息,将显著SNPs 作为固定效应,其余分子标记作为随机效应(固定效应模型),用来研究MGWAS 先验信息对GCA 预测精度的影响。固定效应模型仅考虑GBLUP和RKHS。交叉验证均采用10 倍交叉验证方式,重复100 次。利用100 次验证群体中基因组估计育种值与表型值的相关系数均值作为预测准确性。所有模型均在R 语言BGLR 包中实现[27],其中RKHS 采用核平均模型,宽带参数h为0.1、0.5 和2.5。所有模型和方案迭代次数为12 000 次,预烧为3 000 次,其他参数按默认设置。
2 结果与分析
2.1 一般配合力效应表现统计结果
新乡、周口和综合环境穗行数和籽粒性状GCA 效应值的描述性统计见图1。以综合环境为例,穗行数、粒长和粒宽GCA效应值分别介于-2.85~2.62、-0.086~0.081和-0.062~0.069(图1-A、B)。8个测验种在3个性状中表现不一:对于穗行数,测验种中正向GCA 最高的是20H1419,为1.95~2.44,其次是昌7-2(0.77~1.03),M189 和PH4CV 的配合力负向最高,为-1.39~-1.25(图1-C);粒长正向和负向GCA表现最好的测验种分别是农系531 和20H1419,而粒宽正向GCA 效应值最高的是M119,负向最高的是20H1419(图1-D)。

图1 不同环境穗行数、粒长和粒宽一般配合力效应值统计Fig.1 Statistics of general combining ability effect values of kernel row number,kernel length,kernel width in different environments
穗行数与粒长之间的相关性较小,而粒宽与穗行数表现为高度负相关,相关系数介于-0.80~-0.61。3个性状中,2 个种植环境GCA 的相关系数较高且达到极显著为水平,相关系数为0.59~0.78,且均与综合环境高度极显著正相关(r=0.87~0.95)(图2)。方差分析表明,除了区组外,环境、GCA、特殊配合力以及配合力与环境互作效应均达到显著或极显著水平(表1)。3 个性状GCA 的遗传力较高(0.88~0.95),其中穗行数GCA 的遗传力最高,为0.95,表明3 个性状GCA 主要受遗传因素影响。
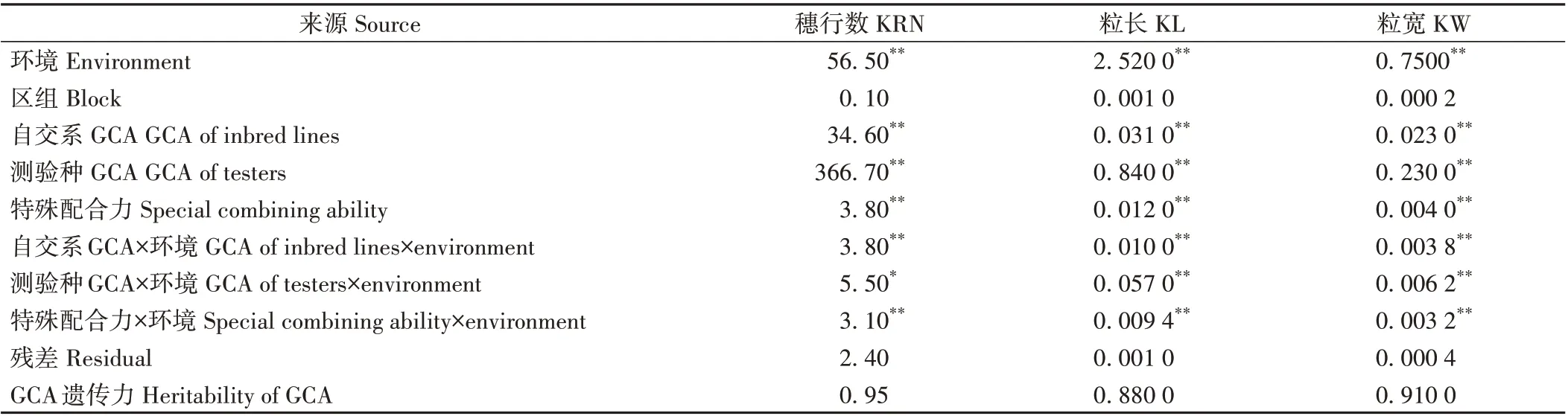
表1 3个性状多环境方差分析及其遗传力Table 1 Multi-environment analysis of variance and heritability for three traits
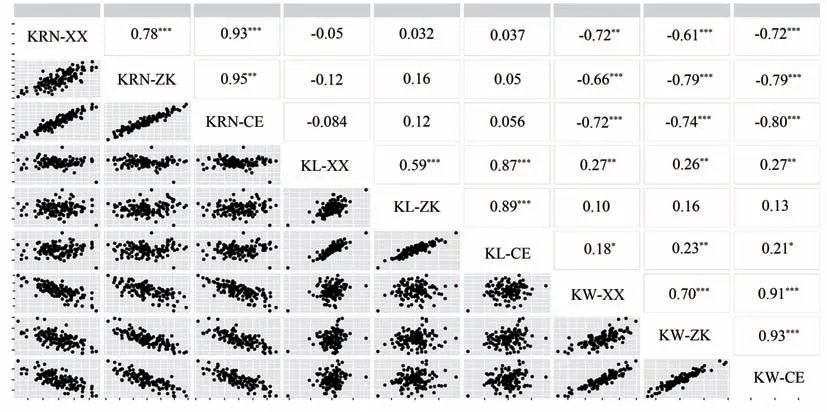
图2 不同环境3个性状相关性及显著性Fig.2 Correlations among three traits in different enviornments and significant levels
2.2 3个性状一般配合力显著关联位点和候选基因
通过7种MGWAS方法对3个环境穗行数、粒长、粒宽GCA进行全基因组位点扫描发现,穗行数、粒长、粒宽分别检测到23、8和15个显著关联SNPs(P<8.52×10-7)。穗行数利用mrMLM检测的位点最多,粒长和粒宽均利用mrMLM 和pLARmEB 检测的位点最多(电子附表1、图3)。共有10个位点被2~5种MGWAS方法同时检测到,其中穗行数、粒长和粒宽分别有5、1和4个(图3-A~C)。共有8个位点被至少2个环境重复检测到(图3-D~F)。对于穗行数GCA,5_219239213被3个环境均检测到,5个为新乡和综合环境共定位的位点。2_197716855 和2_71037706分别为粒长和粒宽GCA 环境稳定的显著位点。本研究还发现6个SNPs即1_43440622、2_69742504、2_71037706、2_197716855、5_219239213 和8_134634317为环境稳定和MGWAS 方法稳定重叠的位点(电子附表1),这些SNPs 可能是控制穗行数和籽粒性状GCA效应的重要位点。尽管3个性状GCA之间表现出一定的相关性,但在基因组水平没有发现一因多效位点。
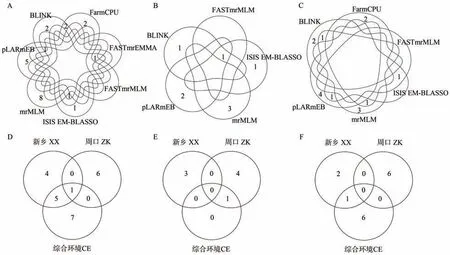
图3 MGWAS方法稳定和环境稳定的位点Fig.3 MGWAS method-stable and environment-stable loci
利用SnpEff 软件在46 个显著位点内共挖掘候选基因59个(电子附表1)。6个环境稳定和MGWAS方法稳定重叠位点挖掘的候选基因见表2,其中胚胎败育基因EMB12(embryo defective12)和类三角形四肽重复超家族蛋白基因(Zm00001d010946)等是穗行数GCA重要的候选基因,Zm00001d006084(类尿苷激酶蛋白2,UKL2)和Zm00001d003945分别是粒长和粒宽GCA重要的候选基因。

表2 MGWAS方法稳定和环境稳定重叠位点挖掘的候选基因Table 2 Candidate genes for MGWAS method-stable and environment-stable overlapped loci
2.3 不同随机效应模型的预测准确性比较
通过对穗行数和籽粒性状GCA效应值进行基因组预测分析发现,5种随机效应模型对穗行数和粒宽GCA取得较高的预测准确性,分别为0.62~0.74 和0.63~0.73,而粒长GCA 基因组预测精度较低,为0.28~0.45(图4)。3个性状均利用综合环境GCA效应值获得最高的预测准确性。对于穗行数和粒宽GCA效应值,多数情况下,RKHS相比其他4种方法略具优势。对于粒长GCA,RKHS在周口和综合环境表现出较大优势,相比其他4种参数模型,将预测准确性提高了16.63%~31.34%。
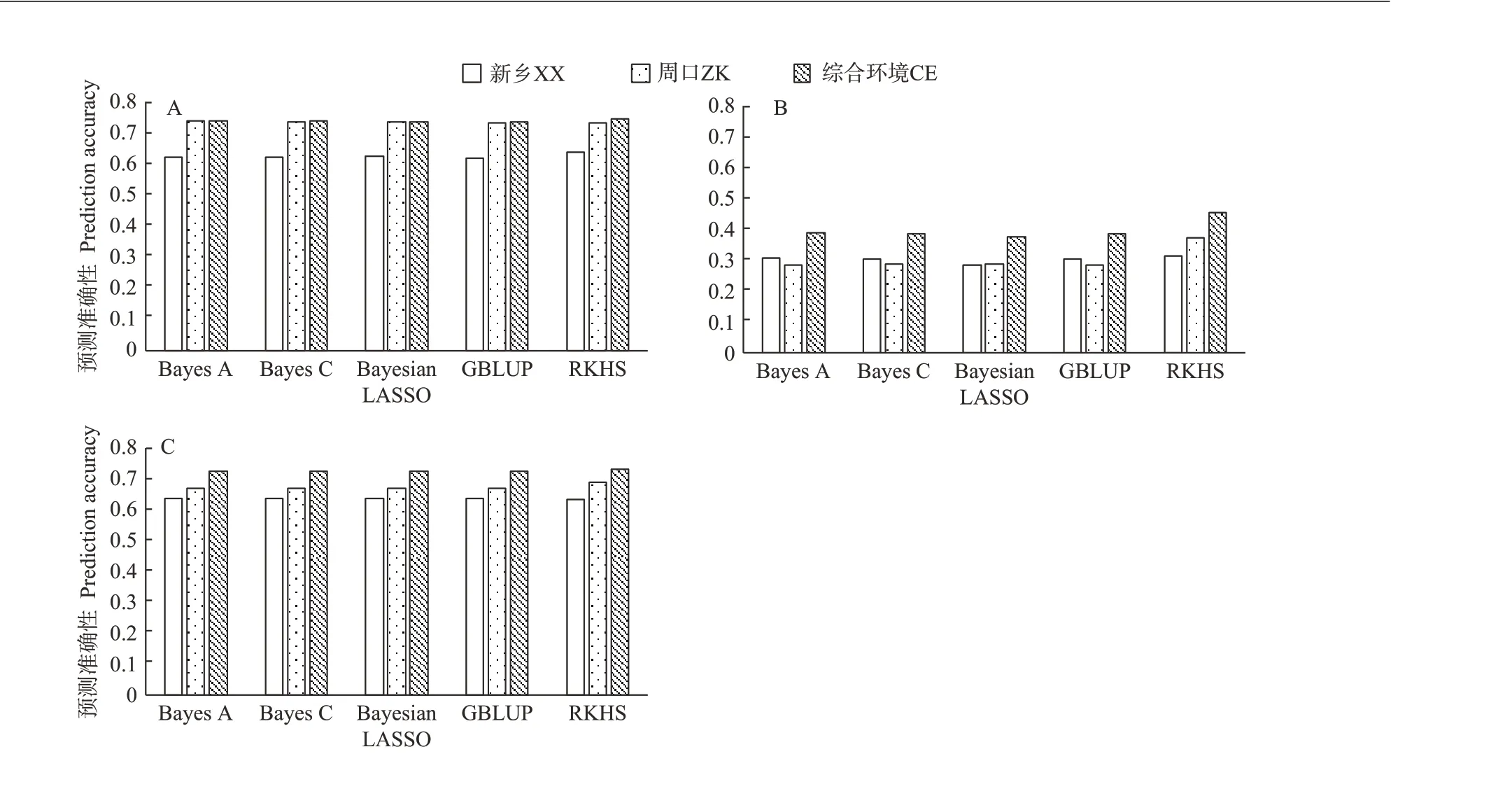
图4 5种随机效应模型对3个性状一般配合力的预测准确性Fig.4 Prediction accuracy of five random effect models for general combining ability of three traits
2.4 多位点全基因组关联分析先验信息对预测准确性的影响
率为27.58%~49.81%。在周口环境,多数情况下MGWAS先验信息加入能够提高3 个性状GCA 效应值的预测力,粒长的提高率较高,为17.65%~83.05%,而穗行数和粒宽的提高率为0.66%~12.53%。相比随机效应模型,综合环境中固定效应模型均可进一步提高穗行数和粒宽GCA 效应值的基因组预测力,提高率为2.24%~12.46%。但对于粒长,将综合环境FASTmrMLM挖掘的1 个显著位点作为固定效应会提高预测精度(9.26%~10.88%),其他2 种MGWAS 方法先验信息与预测方法的整合不会改变或略降低预测准确性。总体而言,将MGWAS 挖掘的显著关联位点作为固定效应,并加入GS 方法,是一种提高穗行数和籽粒性状GCA预测精度的有效途径。
3 讨论
3.1 MGWAS解析GCA变异位点的优势
GWAS 能够检测覆盖全基因组的变异位点,是解析数量性状遗传结构的一种统计分析方法。2006 年,Yu 等[28]首次提出整合亲缘关系K 矩阵和群体结构Q矩阵的混合线性模型方法,用来拟合基因型和表型的关系。为了解决混合线性模型方法存在的混杂问题和统计功效低等问题,多种单位点模型和MGWAS 模型如BLINK、FarmCPU、mrMLM 等相继提出,并已广泛应用于玉米等作物重要性状的遗传研究中。本研究利用7 种MGWAS 方法对穗行数和2 个籽粒性状GCA 效应进行了关联分析,共鉴定得到46个显著关联SNPs,其中mrMLM方法的检测位点最多。An等[29]利用6种MGWAS对穗行数进行定位研究发现,mrMLM 在4 个环境中的检测功效均最高。玉米雄穗分枝数的MGWAS 研究也得到了相同的结论[30]。本研究中,仅10个位点被多种MGWAS方法重复检测到,多数位点表现为方法特异性。但对于穗行数本身,An 等[29]发现40%~62%的显著位点至少被2 种MGWAS 方法重复检测到。Zhou 等[31]对成熟期籽粒含水量进行GWAS 分析时,也发现方法相同的位点具有较高的占比(约45%)。本研究中,8 个位点被至少2 个环境重复检测到,是控制穗行数和籽粒性状GCA 环境稳定的SNPs。其中6 个SNPs 不仅被多种MGWAS 方法重复检测到,还是环境稳定的位点。因此,利用多种MGWAS 方法有助于挖掘GCA 稳定的变异位点。
3.2 不同定位研究结果比较
本研究发现,15个穗行数GCA显著位点与已报道的穗行数GCA、性状本身、产量QTL 和Meta-QTL(MQTL)存在重叠(电子附表1)。其中,位点4_203093326位于3个控制穗行数GCA QTL[6]以及穗行数和产量MQTL 区间内(MQTL69)[32]。位于2号染色体上的4个显著位点(2_153730555、2_159709723、2_159709745和2_225337449)被发现与穗行数QTL以及产量、穗部等性状MQTL区间存在重叠[32-35]。其中2_153730555 是MGWAS 方法稳定的一个控制穗行数GCA的主效位点。4个环境稳定和MGWAS方法稳定的SNPs均发现位于前人定位的产量QTL或MQTL区间内(电子附表1),表明这4个SNPs可能是控制穗行数GCA效应的重要位点。
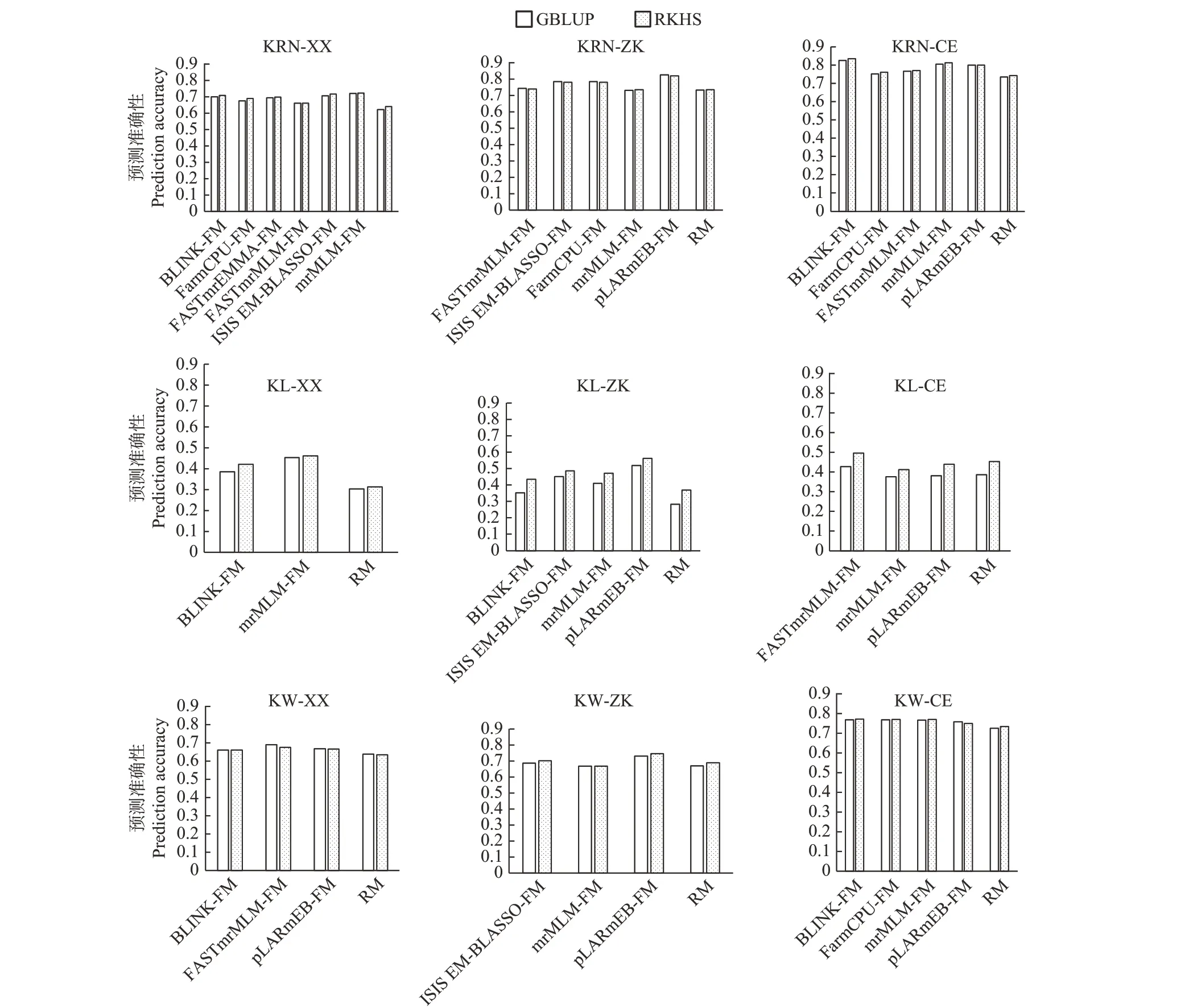
图5 GBLUP和RKHS整合MGWAS显著位点的预测准确性Fig.5 Prediction accuracy of the integration of GBLUP and RKHS with significant loci from MGWAS
19个籽粒性状GCA位于前人定位的籽粒性状GCA、百粒重和产量等性状QTL或MQTL区间内。粒宽GCA显著位点6_144964691 不仅位于一个粒宽GCA QTL(qKW6-4)的置信区间内[5],还与控制百粒重和单穗粒重QTL区间存在重叠[34,36]。3_19172706也是粒宽GCA显著关联位点,被发现位于Lu 等[5]挖掘的一个对粒宽和百粒重GCA具有一因多效的QTL区间内。1_268584002是一个控制粒长GCA的主效SNP,与前人鉴定的粒长、粒宽及其GCA、百粒重以及单穗粒重QTL 位置存在重叠[5,34,36-37]。粒长关联位点4_186588788 不仅与Lu 等[5]和Liu 等[36]定位的粒宽及粒宽GCA QTL 区间重叠,还与一个控制籽粒性状、产量及穗部性状MQTL 存在重叠[33]。2_197716855 和2_71037706 分别是控制粒长和粒宽GCA 的主效位点,不仅被多个MGWAS 重复检测到,还被2个环境同时检测到。位点2_197716855位于Lu 等[5]和Liu 等[36]鉴定的粒宽及产量QTL 的置信区间内,而2_71037706 位于Chen 等[33]利用元分析整合鉴定的一个控制籽粒性状MQTL区间内。粒宽GCA位点2_209342026 和6_90473152 至 少 被2 种MGWAS 方 法同时检测到,发现与Liu 等[36]和Zhang 等[37]定位的2个影响粒宽的数量性状位点存在重叠。
3.3 一般配合力候选基因功能挖掘
MGWAS方法稳定和环境稳定重叠位点5_219239213位于一个控制玉米产量QTL的区间内[5](电子附表1),该位点对穗行数GCA效应的平均变异解释率为12.57%,是一个主效位点,其预测的基因为EMB12。该基因编码质体翻译起始因子3 在玉米胚胎发生发育过程中起着关键作用[38]。8_134634317 也是一个MGWAS 方法稳定和环境稳定重叠的位点,其挖掘的候选基因Zm00001d010946是一类四肽重复序列(tetratricopeptide repeat domain,TPR)蛋白(表2)。TPR 蛋白在拟南芥根发育、叶绿体早期发育和生长素极性运输中起着重要作用[39-40]。Flo2(FLOURY ENDOSPERM2)是一个具有TPR基序且保守的基因,在调控水稻和小麦粒重和淀粉品质中发挥重要作用[41-42]。此外,三角状五肽重复蛋白基因PPR(pentatricopeptide repeat-containing protein)和抗坏血酸盐过氧化物酶基因APX2基因(ascorbate peroxidase2)也是可能控制穗行数GCA 的重要基因(附表1),其中APX2对应的SNP与Zhang等[35]定位的一个穗行数QTL 位置重叠。水稻育性恢复基因Rf4、玉米雄穗分枝数的一个主效QTL以及玉米种子发育关键基因Dek39均编码PPR 基因[43-45]。OsAPX2的功能缺失会影响水稻(Oryza sativaL.)幼苗的生长发育,导致水稻幼苗半矮化、叶片黄绿色和种子不育[46]。但这些基因调控穗行数GCA 的遗传机理尚不清楚,其调控机理有待于进一步挖掘和验证。
通过对籽粒显著关联位点候选基因预测,4 个转录因子基因即C3H7(C3H-transcription factor 317)、HB81(homeobox-transcription factor 81)、E2F11(E2FDP-transcription factor 211)和E2F17以及钾离子高亲和性转运蛋白基因HAK25(potassium high-affinity transporter25)、核糖体蛋白基因RPL23(60S ribosomal protein L23-like)和 质 膜ATP 酶 基 因PM4(plasma membrane ATPase4)等可能是调控籽粒性状GCA 的关键基因(附表1)。DiZF-C3H1是一个锌指结构转录因子,其过表达会导致拟南芥转基因植株根系发育迟缓、花期延迟、花器官异常和角果变形[47]。转录因子VvHB58通过多种激素信号通路影响葡萄种子和果实的发育,其在转基因番茄中的过表达会导致顶端优势丧失、果实大小和种子数量减少以及胚乳细胞变大[48]。E2F 转录因子家族在玉米种子萌发发育过程中起着重要作用[49]。敲除OsHAK26或质膜定位的H+-K+转运体基因OsHAK1或OsHAK5,均可引起水稻花粉花药畸变、花粉粒数减少以及花粉粒萌发率降低,而且体外试验结果表明,外源K+浓度对oshak5花粉萌发率有显著影响[50]。此外,粒宽GCA 候选基因Zm00001d046560 被注释为质膜ATP酶PM4。该基因家族通过参与生长素介导的细胞延长过程进而影响小麦胚乳发育[51]。核糖体蛋白在玉米籽粒发育中起着重要作用。Wang 等[52]通过对转录组数据进行共表达网络分析,发现38个核糖体蛋白基因为籽粒性状显著关联模块的核心基因。玉米Dek44编码线粒体核糖体蛋白RPL9,其突变体dek44籽粒发育迟缓、具有小粒表型[53]。
3.4 显著位点作为固定效应有助于提高一般配合力基因组预测精度
由于穗行数GCA效应的遗传力较高,即使利用5种随机效应模型也获得了较高的预测准确性(0.62~0.74)。多数研究表明,玉米自交系和杂交种穗行数性状本身具有较高的预测力,适合用来开展基因组预测[29,54-55]。粒宽和穗行数利用随机效应模型也获得了较高的预测准确性(0.63~0.73)。尽管粒长GCA 的遗传力较高,但随机模型对其预测的准确性较低(0.28~0.45)。由此可见,穗行数和粒宽GCA 预测精度主要由其遗传力决定,而粒长GCA 除了受遗传力影响外,还可能主要受性状遗传结构等因素的影响。
本研究结果表明,多数情况下,利用7 种MGWAS挖掘的显著关联位点作为固定效应加入预测方法可进一步提高穗行数和2 个籽粒性状GCA 的预测准确性。An 等[29]认为将多种MGWAS 方法挖掘的tagSNP 作为固定效应可以显著提高穗行数本身的预测力。相比随机效应模型,Ma 等[55]研究发现将4~8 个显著SNPs 作为固定效应整合到GBLUP、RKHS、Bayes A、Bayes B 和Bayes C 中可以提高玉米穗行数和穗长基因组估计育种值的准确性,但这种策略并不能提高其他性状例如单株产量、单穗重和百粒重的预测力。在玉米巢式关联群体中,整合最显著关联位点作为固定效应加入预测模型有助于提高株高、小斑病抗性和灰斑病抗性的预测精度[56]。在小麦中,整合共同关联性较强的GWAS 信号作为固定效应加入GBLUP 模型中可提高9%~10%产量的基因组预测力,而稳定性指数Pi 没有表现出优势[57]。通过模拟研究,Rice 等[58]发现绝大多数情况下固定效应作为协方差加入预测模型并不能提高预测准确性。在双亲群体中,Herter 等[59]指出将显著的QTL 作为固定效应可以提高小麦抽穗期、株高、赤霉病和叶枯病的预测准确性。在玉米BC3F5群体中,相比随机效应模型,将1~2 个QTL 作为固定效应可以提高穗行数、穗粒数、行粒数和叶夹角基因组估计育种值的准确性,而将1~5 个QTL 作为固定效应会降低穗位高的预测力[60]。
除了性状、群体类型的影响外,显著位点作为固定效应能否提高预测能力也取决于不同的预测模型和显著位点的个数。例如,玉米BC3F5群体中,Bayes A和RKHS方法整合1~5 个固定效应QTL 能够提高株高的预测力,但整合6 个固定效应QTL 会降低预测力;而Bayes C、Bayesian LASSO、Bayesian ridge regression 和GBLUP 仅整合5个固定效应QTL不会降低株高的预测准确性[60]。本研究中,GBLUP 和RKHS 利用相同MGWAS 信息在不同环境表现一致。尽管大部分鉴定的显著SNPs 作为固定效应能够有效地提高穗行数和2 个籽粒性状GCA 的预测准确性,但是各个MGWAS 方法在不同环境表现不一。这是由于不同MGWAS 检测功效不同导致了位点信息的差异。综上所述,利用多种MGWAS方法检测显著位点,不仅可以为后续基因功能验证提供可靠基因信息,还能进一步对分子标记信息进行优化,提高GCA 基因组估计育种值的准确性,为选择高配合力亲本材料提供准确的预测信息。
4 结论
利用7种MGWAS方法共鉴定46个控制玉米穗行数及籽粒性状GCA显著位点,其中6个至少被2种MGWAS方法和2 个环境重复检测到。共挖掘到59 个候选基因,其中EMB12、TPR、PPR和APX2是控制穗行数GCA的关键基因,而UKL2、E2F11、E2F17、C3H17、HB81、HAK25、RPL23和PM4是粒长和粒宽GCA 重要的候选基因。5 种随机效应模型可以有效预测玉米穗行数和粒宽GCA效应。不同MGWAS显著位点作为固定效应加入GS预测方法有利于获得更高的预测精度,尤其适用于粒长GCA的预测。

