利用全基因组重测序数据检测8个鸭品种基因组拷贝数变异
林 燕,黄 敏,李秀金,张续勐,黄运茂,田允波,伍仲平*
(1.仲恺农业工程学院动物科技学院,广州 510225;2.浙江农林大学动物科技学院·动物医学院,杭州 311300)
随着高通量测序技术的蓬勃发展,将高深度测序数据比对至参考基因组,可获得大量的变异信息用于畜禽性状分析。这些变异信息主要包括单核苷酸多态性(single nucleotide polymorphism,SNP)、插入缺失(insertion-deletion,InDel)以及拷贝数变异(copy number variation, CNV),其中CNV是常见的结构变异(structural variation, SV)。CNV是指与参考基因组(reference genome)相比,个体基因组中DNA片段从50 bp到5 Mb不等的缺失(deletions)或重复(duplications)[1-2]。由具有重叠片段的相邻CNV构成的区域则称为拷贝数变异区(copy number variation region, CNVR)。与单核苷酸多态性SNP相比,CNV覆盖的基因组区域更广阔,通过剂量效应、位置效应、缩并效应、基因融合或中断和隐性或功能多态位点的暴露等作用机制引起染色体结构变异,是导致个体基因和表型变异的重要原因之一[3]。
关于CNV的研究起先主要聚焦在人类的某些遗传缺陷疾病上,随后人们在家养动物中也发现了与表型变异相关的CNV[4-5]。例如,Moller等[6]发现KIT基因座的串联重复导致了猪显性白毛色的形成;Chen等[7]在猪5号染色体鉴别到38.7 kb大小的CNV通过影响miR-584-5p的表达阻碍了靶基因MSRB3表达从而导致猪耳变大;Zheng等[8]研究发现,AHR基因的拷贝数变异与猪产仔数有关;Wright等[9]发现,SOX5基因第一内含子的拷贝数变异导致鸡豆冠表型的形成;Yang等[10]研究发现,HOXB7和HOXB8基因的拷贝数变异与北京油鸡胡须性状的形成有关;Lin等[11]发现,SOX6基因部分区域的拷贝数变异有助于鸡的肌肉生长;Weich等[12]研究发现,KITLG基因上游6 kb序列的拷贝数增加使得家犬的被毛颜色更深、更均匀。
近年来,研究人员利用SNP芯片技术[13-14]、微阵列比较基因组杂交技术(array-based comparative genomic hybridization, aCGH)[15]和全基因组重测序技术(whole-genome sequencing, WGS)已在牛[16-17]、山羊[18-19]、绵羊[20]、猪[21]、狗[22-23]和鸡[24-25]等家养动物中开展全基因组范围内的CNVs检测。与猪、鸡等畜禽相比,鸭基因组CNVs研究相对较少。Skinner等[26]利用aCGH技术在北京鸭中鉴别到32个CNVs,其中5个为鸡、火鸡等鸟类中共享的保守CNVs;章双杰等[27]利用重测序数据检测了娄门鸭、昆山麻鸭、巢湖鸭和高邮鸭的全基因组CNVs,发现娄门鸭和昆山麻鸭中存在1 059个共有的CNVs,并推断娄门鸭与昆山麻鸭的血缘关系较近。张易[28]在润州凤头白鸭和樱桃谷鸭杂交F2群体中,利用重测序数据构建了鸭全基因组水平的CNV遗传图谱,并挖掘到2个与羽色性状相关的潜在CNV位点;Xu等[29]在北京鸭与野鸭杂交的F2分离群体中,利用全基因组CNVs开展关联分析,鉴别到6个与鸭椎骨数量变异相关的CNVs。
鉴于目前关于多品种鸭全基因组CNVs及品种特异性CNVRs的研究鲜有报道,本研究利用从美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)公共数据库中下载的包括家鸭和野鸭在内的8个品种共78个个体的全基因组重测序数据开展鸭全基因组CNVs检测,筛选不同鸭品种特有CNVRs,并鉴别与鸭重要经济性状潜在相关的CNVs,进一步丰富人们对鸭基因组CNVs的了解,为解析CNVs对鸭经济性状的影响提供前期研究基础。
1 材料与方法
1.1 全基因组重测序数据下载
本研究从NCBI公共数据库(https://www.ncbi.nlm.nih.gov)下载了包括樱桃谷鸭(Cherry Valley duck, CV, n=8)、北京鸭(Beijing duck, BD, n=8)、枫叶鸭(Maple Leaf duck, ML, n=8)、金定鸭(Jinding duck, JD, n=8)、山麻鸭(Shan Partridge duck, SP, n=8)、绍兴鸭(Shaoxing duck, SX, n=8)、高邮鸭(Gaoyou duck, GY, n=8)、绿头野鸭(Mallard, MD, n=22)等8个品种共78个个体的全基因组重测序数据,所有个体的测序深度均在6.42×(NCBI检索号:PRJNA419832)[30]。
1.2 全基因组重测序数据序列比对
获得下载的鸭全基因组重测序原始数据后,首先使用FastQC软件(https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)对原始数据进行质控过滤,然后利用BWA v0.7.17软件[31]将过滤后的测序数据与从Ensembl网站下载的北京鸭参考基因组(CAU_duck1.0)进行比对组装,最后使用GATK v.4.2.3软件[32]将组装好的文件转换成BAM文件,用于后续全基因组CNVs检测。
1.3 CNV检测和CNVR定义
本研究分别使用CNVnator[33]和CNVcaller[34]软件进行全基因组CNVs检测,二者分别采用50 bp和800 bp的窗口大小(bin size)。首先将CNVnator软件检测结果中同一品种所有个体存在至少1 bp重叠的同类型CNVs进行合并,得到每个品种的CNVRs,再与CNVcaller软件检测结果中存在至少1 bp重叠的同类型CNVRs进行合并,得到每个品种的CNVRs。为了展示CNVRs在基因组上的分布情况,利用R语言ggbio包[35]对鸭全基因组CNVRs遗传图谱进行可视化。
1.4 品种特异性CNVRs筛选
本研究将从单个品种中检测到的CNVRs定义为品种特异性CNVRs,即利用shell语言将上述得到的CNVRs进行品种间比较,筛选在其他品种中不存在的某个品种特有CNVRs,将其作为品种特异性CNVRs。
1.5 CNVRs基因注释与功能富集分析
本研究利用shell语言将各品种特异性CNVRs在参考基因组中的位置提取出来,通过与北京鸭基因组注释的GFF文件进行比对,对CNVRs所覆盖的区域进行基因注释。利用PANTHER 17.0软件(http://www.pantherdb.org)对CNVRs内的基因进行基因本体Gene Ontology(GO)富集分析。
2 结 果
2.1 全基因组CNVRs检测
本研究分别使用CNVnator和CNVcaller软件对8个鸭品种共78个个体进行全基因组常染色体CNVs检测。将两者结果合并成CNVRs后,在8个鸭品种共检测到7 550个CNVRs,总长度为16 111.2 kb,其中重复型7 098个,缺失型452个,平均长度为2 134 bp,覆盖了鸭基因组(常染色体)的1.51%(表1),各品种CNVRs在整个鸭基因组的覆盖度从0.15%到0.26%不等。在8个品种中,高邮鸭的CNVRs数量最多,达1 345个,其次为具有1 127个的山麻鸭,而绿头野鸭具有的CNVRs数量最少,但具有数量最多的227个缺失型CNVRs,枫叶鸭中则未检测到缺失型CNVRs。根据8个品种的CNVRs检测结果绘制了鸭常染色体基因组CNVRs分布图(图1),可以看出CNVRs在染色体上分布不均匀。

表1 8个鸭品种拷贝数变异检测结果Table 1 Descriptive statistics of copy number variant identified in 8 duck breeds

图1 CNVRs在鸭基因组上的分布Fig.1 Distribution of CNVRs on duck genome
2.2 全基因组CNVRs分布特征
从长度分布情况看,CNVRs主要在1.6~2.1 kb之间,其中有38.07%的CNVRs长度在1.6~1.7 kb之间,41.40%的CNVRs长度在2.0~2.1 kb之间(图2a)。从不同染色体上的分布情况看,CNVRs在鸭染色体上呈不均匀分布,CNVRs主要集中在1号和2号染色体上,占检测总数量的40.53%,而在17号染色体中则未检测到CNVRs(图2b)。

图2 鸭基因组CNVRs的长度和染色体分布Fig.2 CNVRs length and chromosome distribution in duck genome
2.3 品种特异性CNVRs
本研究在8个鸭品种中共筛选到4 304个只存于单个品种中的品种特异性CNVRs,其中重复型4 208个,缺失型96个,总长度为8 412 kb,占检测CNVRs总长度的52.21%。在8个鸭品种中,高邮鸭的品种特异性CNVRs数量最多(n=772),其次是樱桃谷鸭(n=621)。绿头野鸭的品种特异性CNVRs数量虽然最少(n=301),但具有最多的品种特异性缺失型CNVRs(n=56)(表2)。
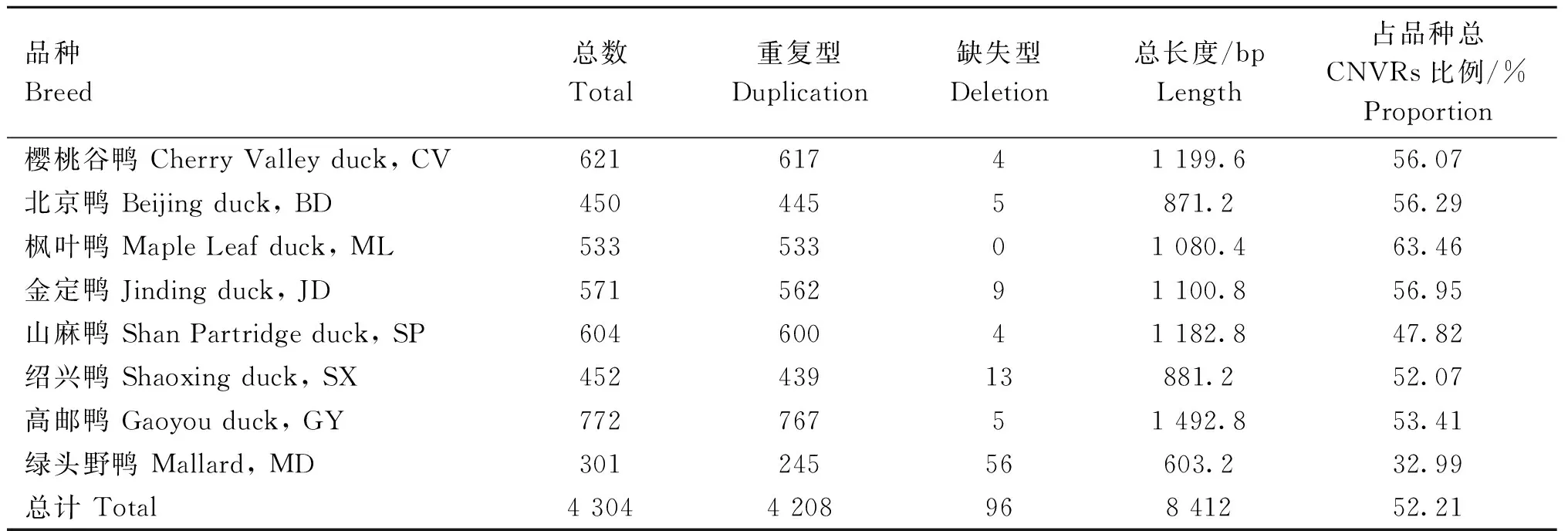
表2 8个鸭品种特异性CNVRsTable 2 The breed-specific CNVRs of 8 duck breeds
2.4 品种特异性CNVRs基因注释及GO富集分析
本研究将在8个鸭品种中筛选到的4 304个品种特异性CNVRs所覆盖的区域进行基因注释,结果显示,这些CNVRs共覆盖了1 230个注释基因,其中重复型CNVRs包含了1 183个基因,缺失型CNVRs包含了47个基因(图3)。在8个品种中,樱桃谷鸭品种特异性CNVRs包含的基因数量最多(n=305),其次为高邮鸭(n=250),绿头野鸭最少(n=111)。
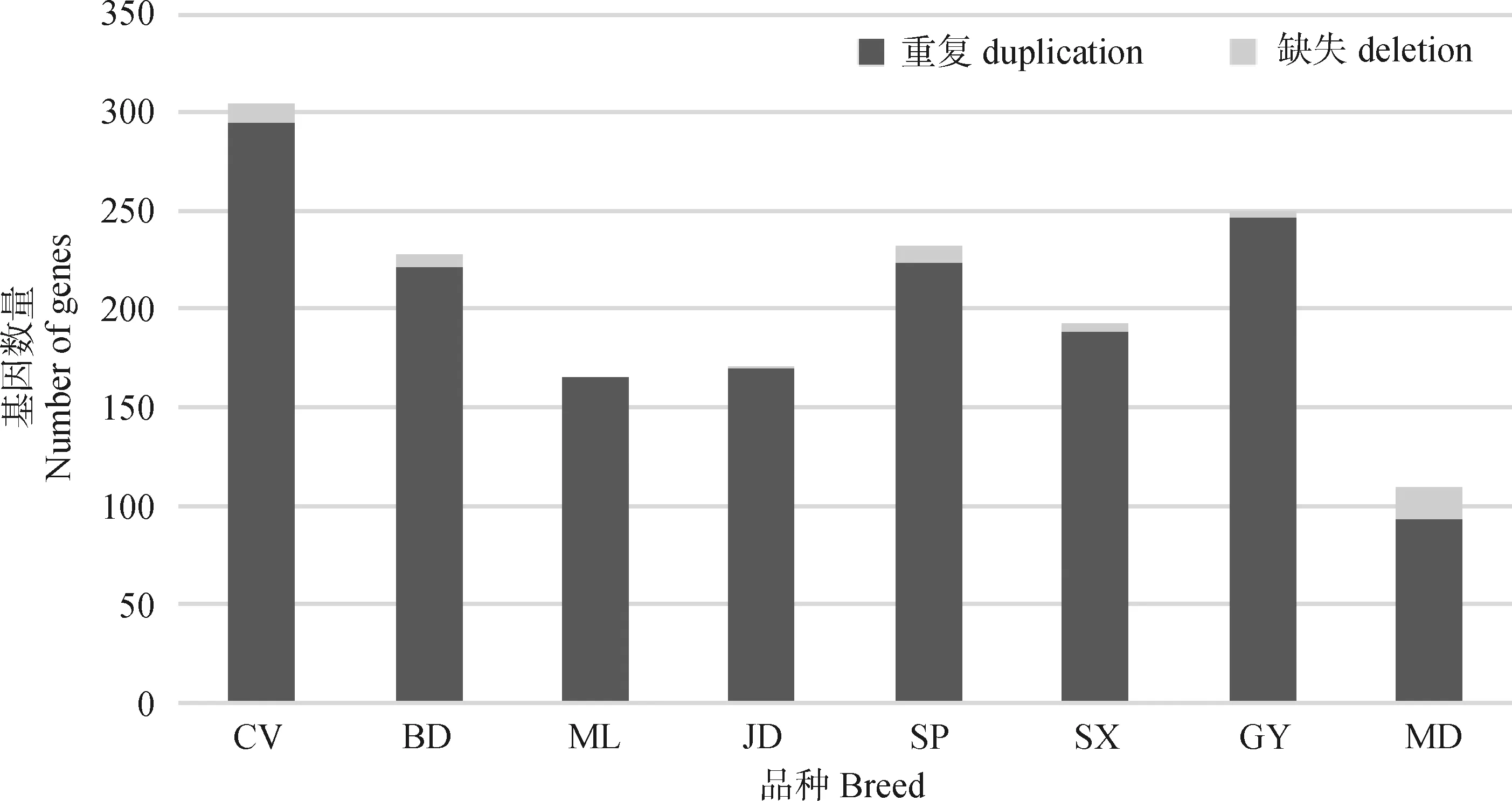
图3 品种特异性CNVRs区域中的注释基因数量Fig.3 Annotation genes in breed-specific CNVRs
为了分析这些基因的生物学功能,本研究利用PANTHER 17.0软件对筛选到的所有品种特异性CNVRs覆盖的基因进行了GO富集分析。结果显示,这些基因主要富集在细胞过程、发育过程、免疫系统过程、细胞运动、代谢过程、多细胞生物过程、对刺激的反应、信号传导及生长、繁殖等生物学功能上(图4)。

图4 品种特异性CNVRs基因GO富集分析Fig.4 GO enrichment analysis of breed-specific CNVRs genes
为鉴别与品种经济性状相关特异性CNVRs,本研究分别对8个鸭品种特异性CNVRs覆盖的基因进行了GO富集分析,共鉴别到38个与繁殖和生长相关的品种特异性CNVRs,其中,在樱桃谷鸭中鉴别到2个与繁殖相关、1个与生长相关的CNVRs;在北京鸭中鉴别到1个与繁殖相关、1个与生长相关的CNVR;在枫叶鸭中鉴别到3个与生长相关、4个与繁殖相关的CNVRs;在金定鸭中鉴别到3个与繁殖相关、6个与生长相关的CNVRs;在山麻鸭中鉴别到1个与生长相关的CNVR;在绍兴鸭中鉴别到1个与生长相关、3个与繁殖相关的CNVRs;在高邮鸭中鉴别到10个与繁殖相关的CNVRs;在绿头野鸭中鉴别到2个与繁殖相关、1个与生长相关的CNVRs。这些CNVRs共覆盖了20个与繁殖和生长相关的基因,其中与繁殖相关的基因14个,与生长相关的基因6个(表3)。
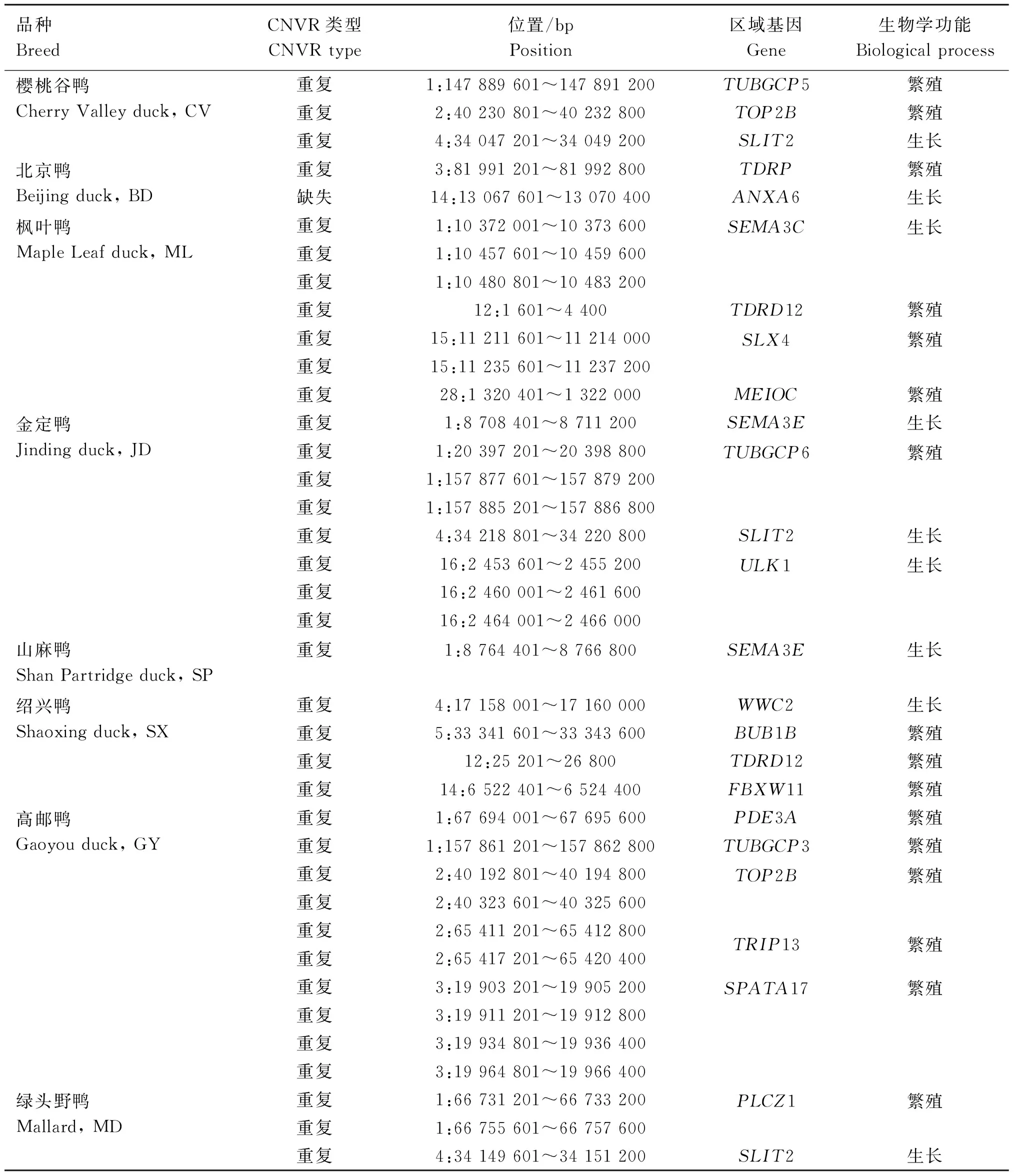
表3 8个鸭品种中与生长和繁殖潜在相关的品种特异性CNVRsTable 3 Breed-specific CNVRs potentially related to growth and reproduction in 8 duck breeds
3 讨 论
本研究利用CNVnator和CNVcaller软件,对从公共数据库下载的8个鸭品种共78个个体的全基因组重测序数据进行了全基因组拷贝数变异检测,并只保留两个软件检测结果中存在至少1 bp重叠的同类型CNVRs,旨在消除假阳性结果对试验的影响。此外,为了后续研究方便,本研究在合并CNVRs时,仅考虑只包含重复或缺失片段的CNVRs,未将同时包含重复和缺失片段的混合型CNVRs进行分析。共发现了7 550个CNVRs,总长度为16 111.2 kb,平均长度为2 134 bp,约占鸭基因组的1.51%,处于马、猪、牛和鸡中报道的0.8%~5.12%范围内[36-39]。这7 750个CNVRs长度主要集中分布在1.6~2.1 kb之间,且在常染色体上呈不均匀分布,该结果与羊、鸡等物种基因组的检测结果一致[40-41]。
家鸭驯化后经过长期的人工选择,各项生产性能较其祖先绿头野鸭均有明显的改变,并根据人类需要选育出了不同经济类型的品种,如樱桃谷鸭、北京鸭、枫叶鸭等肉用型品种,金定鸭、山麻鸭、绍兴鸭等蛋用型品种以及蛋肉兼用型品种高邮鸭[42]。在家鸭的选育过程中,与经济性状相关的遗传变异逐渐在基因组中积累,其中包括拷贝数变异。因此,本研究为探究品种特异性CNVRs是否与其经济性状有关,对各品种特异性CNVRs进行了筛选。结果在8个鸭品种中共筛选到4 304个品种特异性CNVRs,包含了1 230个注释基因。通过对注释基因开展GO富集分析,发现多数基因富集在细胞过程、发育过程、免疫系统过程等生物学功能上。此外,还有少数基因与生长和繁殖相关,而这些基因区域的拷贝数变异很可能与不同品种鸭特有的经济性状有关。
为鉴别与不同品种鸭的重要经济性状潜在相关的CNVRs,分别对各品种特异性CNVRs进行GO富集分析,结果在8个鸭品种中发现了13个与生长相关的品种特异性CNVRs,其中重复型12个,缺失型1个,这些CNVRs包含了SEMA3E、ANXA6、SEMA3C、ULK1、SLIT2、WWC2等6个基因,其中SLIT2基因已有多个研究报道与肉牛初生重、断奶重、骨重以及肉鸡体重、骨骼生长发育等生长性状相关,但未报道与该基因的CNV有关[43-47]。此外,还在8个鸭品种中发现25个与繁殖相关的重复型CNVRs,包含了BUB1B、SPATA17、MEIOC、TUBGCP5、TUBGCP3、PDE3A、TDRD12、TRIP13、TDRP、TOP2B、FBXW11、PLCZ1、SLX4、TUBGCP6等14个基因。
高邮鸭是我国三大名鸭之一,以善产双黄蛋而驰名中外。本研究在高邮鸭中筛选到10个品种特异性CNVRs,包含PDE3A、TUBGCP3、TOP2B、TRIP13、SPATA17等5个与繁殖相关的基因。大量研究表明,磷酸二酯酶3A(phosphodiesterase 3A,PDE3A)基因在哺乳动物卵母细胞发育成熟中起关键作用,该基因可通过调控环磷酸腺苷(cyclic adenosine monophosphate, cAMP)的降解从而促进卵母细胞的减数分裂并成熟[48-50]。而家禽的双黄蛋主要是由2个卵泡同时成熟并排卵进入输卵管而形成的,该过程与卵泡发育密切相关[51]。因此,推测高邮鸭PDE3A基因区域的拷贝数变异很可能与该品种产双黄蛋的特有经济性状有关。然而,目前关于PDE3A基因在家禽卵泡发育中的作用还未见文献报道,有待进一步的试验进行验证。
4 结 论
本研究对NCBI数据库中下载的8个鸭品种共78个个体的重测序数据进行全基因组拷贝数变异检测,共发现了7 550个CNVRs,总长度为16 111.2 kb。这些CNVRs在鸭基因组呈不均匀分布,覆盖了鸭基因组的1.51%。在8个鸭品种全基因组中共筛选4 304个潜在的品种特异性CNVRs,覆盖1 230个注释基因。通过基因功能GO富集分析,鉴别到38个可能与鸭生长和繁殖相关的CNVRs。这些结果对于进一步研究CNV与鸭重要经济性状的关联性有重要参考价值。

