板蓝与葡萄、漾濞槭基因组的共线性分析
肖启蒙
摘 要 板蓝(Strobilanthes cusia)具有极高的药用价值,全草可入药,是中草药南板蓝根的主要成分。为了厘清板蓝基因组的进化历程,探究板蓝的生物学功能,通过将板蓝基因组与葡萄(Vitis vinifera)、漾濞槭(Acer yangbiense)基因组进行全基因组比对,采用推断基因组同源性、同义碱基替换、Ks的核函数分析、鉴定染色体的直系和旁系同源区域、全基因组与局部比对等方法,发现板蓝在经历了核心真双子叶共享的三倍化事件后,还额外经历了一次三倍化事件,并且确定时间节点在106~120个百万年前。
关键词 板蓝;共线性;多倍化;全基因组比对;同义核苷酸替换率
中图分类号:S567.23+9 文献标志码:A DOI:10.19415/j.cnki.1673-890x.2023.15.003
板蓝(Strobilanthes cusia)是爵床科灌木状多年生草本植物,广泛分布在我国的四川、贵州、浙江和福建等地。板蓝全身皆可入药,根被称为“南板蓝根”,茎和叶经加工后可得青黛,能够治疗病毒性肝炎、流感、感冒、肺炎、炎症、疱疹、丹毒、蛇咬伤等[1],青黛是牛黄消炎丸、桂林西瓜霜等的主要原料[2-3]。其中,福建马蓝(板蓝)青黛品质最佳,是福建省大宗地道药材之一[4]。最近,高质量板蓝基因组数据可获得[5],为进一步探究板蓝基因组进化历史及生物学特征奠定了基础。
多倍化(polyploidy)是物种多样性演化的重要基因组动力,为地球上绿色植物的庞大类群的出现做出了巨大贡献,尤其在被子植物的演化过程中尤为突出[6]。物种经历多倍化,其早期基因组将以古老的多倍体形式存在,而后会出现大规模的染色体重排,以及伴随发生的重复基因丢失现象,这就导致现存物种基因组相对其祖先,已经发生了复杂的变化,因此也为研究其古老的演化历史带来了挑战。板蓝作为真双子叶植物,至少经历了3次多倍化事件,包括种子植物共享的全基因组加倍ζ(zeta)事件和被子植物共有的ε(epsilon)事件,以及最近发生的一次核心真双子叶植物共同经历的古老六倍体γ(gamma)事件。多倍化事件的鉴定及对物种进化的影响,是基因组研究的一个热点,其中对于古老时期、且在一个较短的时间里,发生的潜在的由多倍化导致的基因组激变事件的鉴定是一个难点。地球上真双子叶植物出现距今有约1.5亿年[7-9],早期分化关键节点几乎是在同一历史时期,对于板蓝而言,其从核心双子叶中分化出来的时间较早且分化较快,受到辐射性演化的影响,因而增加了这一分化前后的全基因组加倍事件的分析难度。
本研究通过使用前人开发的方法[10],将板蓝基因组与两个优良的参考基因组——葡萄(Vitis vinifera)和漾濞槭(Acer yangbiense)进行共线性分析和全基因组比对分析,旨在厘清板蓝基因组的进化历程,探究板蓝的生物学功能。
1 材料与方法
1.1 植物基因组数据
从不同的网站下载了每个基因组项目的基因组序列和注释文件。葡萄的基因组数据可以在NCBI上找到(https://www.ncbi.nlm.nih.gov/),漾濞槭的基因组信息可以在GenBank找到(http://www.ncbi.nlm.nih.gov/genbank/),板蓝的基因组数据可以在CNGB Sequence Archive (CNSA) 找到(https://ftp.cngb.org/pub/CNSA/data3/CNP0001632/CNS0343944/CNA0019301/)。
1.2 推斷基因组同源性
基因共线性表明基因组之间的基因和基因顺序在一定程度上是保守的,这可以反映共同祖先的同源染色体结构;这对于理解基因组变化,特别是推断复杂植物基因组的进化至关重要。通过BLAST对蛋白质序列进行比较[11],E值<1e-5,得分>100。随后,来自BLAST的同源基因信息被用作ColinerScan软件[12]的输入,为了定位同源基因对,笔者将同源基因之间的最大间隙分别设置为10、20、40、50、60、80、100和200个干预基因,并通过使用这些信息获得同源基因点图[13-14]。最后,当阈值设置为50时,发现了最清晰的共线性片段,并确定出旁系同源和直系同源基因。
1.3 同义碱基替换
使用Nei-Gojobori方法[15],通过使用Bioperl统计模块,估计同义位点(Ks)上的同义核苷酸替换。然后,对齐多个基因CDS,并使用ClustalW v2.0设置默认参数[16]。
1.4 Ks的核函数分析
使用核函数来分析基因组内和基因组之间的同基因同源物的Ks分布。Ks分布被认为是正态分布的混合。MATLAB v9.2.0[17]用于估计每个Ks列表的密度并获得密度分布曲线,而核平滑密度函数的宽度参数设置为0.05。通过在拟合工具箱工具中执行高斯函数来拟合曲线,评估拟合优度的参数R2通常设置为不小于95%。本文使用Ks分布曲线的最大似然估计μ(Ks峰值)。
1.5 鉴定染色体的直系和旁系同源区域
通过比较与物种分化和特定多倍体事件相关的同源区域的Ks值,确定了同源和旁系的染色体区域。
1.6 全基因组与局部比对
基于多基因组比对关系,使用McScanX的jcvi模型展示笔者在参考基因组和板蓝基因组之间选择的同源基因深度比关系的信息[18]。
2 结果与分析
2.1 共线性基因及Ks分布表征
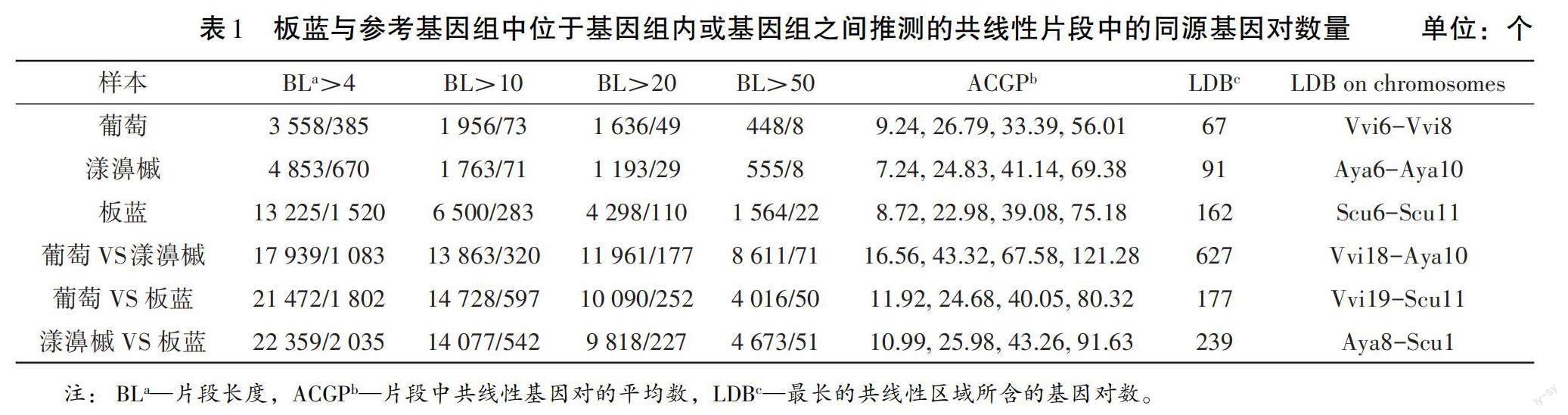
为了探究板蓝的多倍化历程,笔者比较了板蓝基因组与葡萄、漾濞槭基因组间的共线性关系(见表1)。在葡萄和漾濞槭基因组中,当共线性区域所包含的共线性基因对大于4对时,分别鉴定出了位于385和670个同源共线性区域上的3 558和4 853对共线性基因对。然而,在相同条件下,在板蓝基因组内鉴定出13 225对共线性基因对位于1 520个同源区域上,这大约是葡萄和漾濞槭基因组内共线性基因对的3倍左右。随后,笔者对基因组间也进行了比较,发现板蓝与葡萄、漾濞槭间分别存在50、51个长片段(连续基因对数量大于50对),包括了4 016和4 673对共线性基因对,值得注意的是,在葡萄与漾濞槭基因组间存在8 611对共线性基因对,这大约是板蓝与两个参考基因组间的2倍。这些基因组比较表明,板蓝基因组中有更多的区块,并且它们更小,这表明板蓝可能在ECH后经历了进一步的多倍化事件。
笔者对共线性基因对所处的同源区域进行了同义核苷酸替换分析,计算了多倍化事件和物种分化所对应的Ks峰,并进一步推断了进化关键事件发生的时间节点(见封三图1)。葡萄、漾濞槭和板蓝基因组中的共线性基因对分别在0.978 9、1.251和1.09处存在峰值,并且板蓝基因组存在一个更小的峰,约为0.929 8,因此笔者推测前面三个峰对应核心真双子叶共享的多倍化事件(core eudicots hexaploidy, ECH),板蓝的较小峰对应板蓝额外经历的多倍化事件。此外,笔者发现板蓝与葡萄、漾濞槭分化的峰分别为1.31和1.43,均大于葡萄与漾濞槭分化的峰(0.946 9),表明板蓝先于葡萄、漾濞槭分化,随后葡萄和漾濞槭再分开。进一步地,笔者对板蓝及参考基因组进化过程中的关键进化事件进行时间节点的推断,利用之前研究中确定的ECH事件发生在大约115~130个百万年前[9],笔者推算出板蓝大约在111~126个百万年前与葡萄、漾濞槭分支发生分化,随后在约106~120个百万年前经历了额外的多倍化事件,最后葡萄与漾濞槭在85~96个百万年前发生分化。从上述结果中,笔者可以看到,在ECH事件后,板蓝基因组在相当短的时间内经历了多倍化及分化事件,这会增大多倍化事件的鉴定难度,是板蓝基因组分析中的巨大挑战。
2.2 确定板蓝额外多倍化事件为三倍化
为了揭示板蓝基因组的多倍化历程,笔者对葡萄和板蓝基因组进行比较,发现板蓝的9个直系染色体区域与葡萄3个由ECH事件产生的旁系染色体区域相匹配。比如,葡萄基因组中由ECH事件产生的Vvi4、Vvi9、Vvi11与板蓝基因组中的9个旁系染色体区域Scu2、Scu9、Scu10、Scu5、Scu15、Scu16、Scu3、Scu6、Scu8相匹配(见封三图2A)。当用漾濞槭基因组与板蓝基因组进行比较时,也可观察到类似的结果(图2B)。这些基因组间共线性分析表明,葡萄与板蓝,漾濞槭与板蓝间的直系同源共线性深度比皆为3∶9。进一步地,对板蓝基因组内也进行全基因组比较,发现其自身存在两个旁系同源区域,比如,Scu3和Scu4、Scu6为旁系同源区域(图2C)。綜合基因组间和基因组内比较结果,表明板蓝基因组应该在ECH事件后经历了一次额外的三倍化事件。参考基因组与板蓝间的进化关系如图2D所示。
2.3 多重基因组比对与共线性列表的构建
在前面鉴定出的直系同源与旁系同源区域的基础上,笔者可以区分出多倍化事件相关的共线性区域。葡萄、漾濞槭和板蓝间分化的直系同源关系如封三图3A所示。比如,葡萄的18号染色体有一个直系同源区域位于漾濞槭的10号染色体上。有3个直系同源区域分别位于板蓝的3、14和16号染色体上(图3B)。同时,由于多倍化后会存在大量的基因丢失,所以在板蓝和两个参考基因组之间的微共线性同源区域的基因清楚地表明,存在一些基因没有显示出预期的共线性深度同源比(见图3B)。
此外,基于三个比较基因组中同源基因的位置,笔者构建了一个分层次和事件相关的多基因组比对表。在这里,以葡萄为参考基因组,笔者将葡萄的所有基因ID填充到第一列,然后根据多重比对推断出的基因组合成,逐列逐种添加葡萄、漾濞槭、板蓝的基因标识符。假设没有基因丢失,来自ECH事件的葡萄中的三个旁系基因中的每一个都分别在漾濞槭和板蓝中有一个和三个同源基因。因此,多基因组比对表包括15(3+3+9)列,其中包含三列葡萄基因、三列漾濞槭基因和九列板蓝基因。当一个基因在预期位置缺失时,通常是由于基因丢失、易位或可能的组装不充分,笔者在相应的位置输入一个点来表示缺失的基因,利用构建的列表进行多基因组比对(图3C)。该表总结了多重基因组和事件相关比对的结果,反映了递归多倍体化导致的三倍同源性,为爵床科生物信息学的进一步深入探究提供了重要的基因组学资源。
3 讨论与结论
递归多倍体化在植物基因组中频繁发生,为基因组功能创新提供了巨大的机会[19-22]。多倍体化后,植物基因组常常伴随着基因丢失和染色体重排,这使得基因组更加复杂,在识别多倍化事件时也会产生巨大的挑战[23]。比如,对最好匹配片段的选择存在差异[24],多倍体后进化速度加快,导致与最近多倍体事件相关的同源区块的Ks值与ECH事件的Ks值无法区分[9]等。在之前对板蓝基因组的分析中,并没有提到板蓝在进化过程中经历了全基因组多倍化事件[5]。本文对板蓝与参考基因组进行了深入的全基因组比对分析,发现葡萄与漾濞槭,葡萄与板蓝的直系同源比分别为1∶1,1∶3,表明板蓝基因组在ECH事件后又经历了一次额外的三倍化事件。这不仅可以成为探究爵床科植物进化历程的模式,也因笔者对板蓝基因组构建的多重基因组比对列表,而能为后续爵床科植物基因组学研究打下坚实的基础。此外,本研究为进一步加强板蓝育种工作,提高板蓝产量提供了理论基础。
参考文献:
[1] GU W, ZHANG Y, HAO X J, et al. Indole alkaloid glycosides from the aerial parts of Strobilanthes cusia[J]. Journal of natural products, 2014, 77(12): 2590-2594.
[2] 胡永乐,宁书菊,叶齐,等.流式细胞术测定马蓝基因组大小[J].中成药,2023,45(2):634-636.
[3] 郭志英,李卿,吴循循,等.马蓝WRKY转录因子家族生物信息学及表达特征分析[J].药学学报,2022,57(9):2864-2875.
[4] 刘小英,彭建平,林志敏.建青黛产业现状、存在问题及发展对策[J].福建农业科技,2016(3):59-62.
[5] HU Y, MA D, NING S, et al. High-Quality Genome of the Medicinal Plant Strobilanthes cusia Provides Insights Into the Biosynthesis of Indole Alkaloids[J]. Frontiers in plant science, 2021, 12: 742420.
[6] SOLTIS P S, SOLTIS D E. Ancient WGD events as drivers of key innovations in angiosperms[J]. Current opinion in plant biology, 2016, 30: 159-165.
[7] MING R, VANBUREN R, LIU Y, et al. Genome of the long-living sacred lotus (Nelumbo nucifera Gaertn.)[J]. Genome biology, 2013, 14(5): R41.
[8] MOORE M J, SOLTIS P S, BELL C D, et al. Phylogenetic analysis of 83 plastid genes further resolves the early diversification of eudicots[J]. Proc Natl Acad Sci U S A, 2010, 107(10): 4623-4628.
[9] JIAO Y, WICKETT N J, AYYAMPALAYAM S, et al. Ancestral polyploidy in seed plants and angiosperms[J]. Nature, 2011, 473(7345): 97-100.
[10] WANG J, SUN P, LI Y, et al. An Overlooked Paleotetraploidization in Cucurbitaceae[J]. Molecular biology and evolution, 2018, 35(1): 16-26.
[11] CAMACHO C, COULOURIS G, AVAGYAN V, et al. BLAST+: architecture and applications[J]. BMC bioinformatics, 2009, 10: 421.
[12] WANG X, SHI X, LI Z, et al. Statistical inference of chromosomal homology based on gene colinearity and applications to Arabidopsis and rice[J]. BMC bioinformatics, 2006, 7: 447.
[13] WANG X, WANG J, JIN D, et al. Genome Alignment Spanning Major Poaceae Lineages Reveals Heterogeneous Evolutionary Rates and Alters Inferred Dates for Key Evolutionary Events[J]. Molecular plant, 2015, 8(6): 885-898.
[14] WANG X, GUO H, WANG J, et al. Comparative genomic de-convolution of the cotton genome revealed a decaploid ancestor and widespread chromosomal fractionation[J]. The New phytologist, 2016, 209(3): 1252-1263.
[15] NEI M, GOJOBORI T. Simple methods for estimating the numbers of synonymous and nonsynonymous nucleotide substitutions[J]. Molecular biology and evolution, 1986, 3(5): 418-426.
[16] THOMPSON J D, GIBSON T J, HIGGINS D G. Multiple sequence alignment using ClustalW and ClustalX[J]. Current protocols in bioinformatics, 2003(1): Unit 2.3.
[17] CICHOSZ S L, JENSEN M H, LARSEN T K, et al. A Matlab Tool for Organizing and Analyzing NHANES Data[J]. Studies in health technology and informatics, 2020, 270: 1179-1180.
[18] WANG Y, TANG H, DEBARRY J D, et al. MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic acids research, 2012, 40(7): e49.
[19] VAN DE PEER Y, MIZRACHI E, MARCHAL K. The evolutionary significance of polyploidy[J]. Nature reviews Genetics, 2017, 18(7): 411-424.
[20] JIAO Y, LEEBENS-MACK J, AYYAMPALAYAM S, et al. A genome triplication associated with early diversification of the core eudicots[J]. Genome biology, 2012, 13(1): 1-14.
[21] SOLTIS P S, MARCHANT D B, VAN DE PEER Y, et al. Polyploidy and genome evolution in plants[J]. Current opinion in genetics & development, 2015, 35: 119-125.
[22] LANDIS J B, SOLTIS D E, LI Z, et al. Impact of whole-genome duplication events on diversification rates in angiosperms[J]. American journal of botany, 2018, 105(3): 348-363.
[23] PATERSON A H, BOWERS J E, CHAPMAN B A. Ancient polyploidization predating divergence of the cereals, and its consequences for comparative genomics[J]. Proc Natl Acad Sci U S A, 2004, 101(26): 9903-9908.
[24] WANG J, SUN P, LI Y, et al. Hierarchically Aligning 10 Legume Genomes Establishes a Family-Level Genomics Platform[J]. Plant physiology, 2017, 174(1): 284-300.
(責任编辑:丁志祥)
——以多重共线性内容为例

