万古霉素耐药基因研究进展及纳米技术在提高万古霉素抗菌活性中的应用
侴金江,顾觉奋
万古霉素耐药基因研究进展及纳米技术在提高万古霉素抗菌活性中的应用
侴金江,顾觉奋
210009 南京,中国药科大学生命科学与技术学院
万古霉素(vancomycin)是用于治疗由革兰阳性菌引起的严重感染疾病的抗生素。万古霉素能够与肽聚糖合成前体 lipoII中的 D-Ala-D-Ala 末端结合,从而抑制转肽反应来阻断高分子肽聚糖的合成,造成细菌因细胞壁缺陷破裂而死亡[1]。自 1986 年首次分离得到具有万古霉素抗性的肠球菌菌株后,万古霉素抗性在肠球菌属中广泛传播。目前关于万古霉素耐药基因的研究已经取得了一定的成果。1990 年,发现A 基因能诱导生成相应的抗性蛋白A,其具有 D-Ala-D-Lac 连接酶活性。而后又陆续发现了H 和X 等基因,且细胞壁合成途径的改变需要有H、A、X 等基因的参与[2]。截止目前,有多种万古霉素耐药基因已被发现并鉴定了部分耐药基因在基因簇中的位置和编码产物,汇总于表 1。针对万古霉素的耐药越来越普遍的现象,如何增强万古霉素的抗菌活性就显得尤为重要。目前以纳米颗粒装载万古霉素的给药方式显示出了巨大的优势[3]。本文将就最近几年有关万古霉素耐药基因的研究以及提高其抗菌活性的研究进行综述。
1 形成万古霉素耐药性的机制
目前有两种主流假说解释万古霉素耐药性的形成机制。第一种也是最常见的一种机制是肽聚糖合成前体末端上的 D-Ala-D-Ala 转变为 D-Ala-D-Lac 或者 D-Ala-D-Ser,并伴随相关多肽酶介导的 D-Ala-D-Ala 清除。研究指出,D-Ala-D-Lac 和 D-Ala-D-Ser 结合万古霉素的亲和力比 D-Ala-D-Ala 分别低1000 倍和 6 倍[1, 4],导致万古霉素不能与相应部位结合进而裂解细胞。编码万古霉素抗性的相关酶基因位于具有多顺反子性的操纵子上,具体有编码 D-Ala-D-Lac 和 D-Ala-D-Ser 连接酶的A、B、C、D、E、G 和L 基因;编码丙酮酸脱氢酶的H 等基因;编码苏氨酸解旋酶的T 和TG等基因;编码D,D-二肽酶的X 和XB等基因;编码 D,D-羧肽酶的Y、YB和YD基因。这一机制被视为产糖肽类抗生素放线菌的自我保护方式[1]。
第二种较为重要的机制认为耐药性是由内源的二组分调节系统(two-component signal transduction systems,TCS)突变而引起的,而A、B、D 基因簇均含有 TCS。研究指出能够通过此机制获得耐药性,并且 TCS 控制正常细胞壁代谢和调控细胞壁损伤时的细胞应答[1]。
2 万古霉素耐药基因研究进展
目前针对万古霉素耐药基因已经进行了很多的研究,A、B、C 等基因早已被发现并通过测序了解其在基因簇中的具体位置,此外许多研究也证明了这些基因与万古霉素耐药性的产生有关。以下将关注其中一些基因的最新研究进展。

表 1 肠球菌对糖肽类的耐药水平[5-6]
2.1 vanA
A 基因簇是 Tn1546 转座子的一部分,其中包含了R-S 二组分调控系统序列。最近,de Niederhäusern等[7]研究指出,耐万古霉素的金黄色葡萄球菌(vancomycin resistant,VRSA)的万古霉素抗性可由与之共感染的耐万古霉素肠球菌(vancomycin resistant,VRE)中包含A 基因的 Tn1546 转座子转移而来。该研究分别选取了 5 种 VRE(VanrRifs)和 5 种对利福平耐药的金黄色葡萄球突变菌株(VansRifr)进行对应的菌株接合转移,结果EMM09 和STM359 组与EMB04 和STM17 组接合转移成功。两种成功的接合转移子对万古霉素的 MIC 值为 32 μg/ml,与 EMM09 和 EMB04(MIC > 128 μg/ml)相比,MIC 偏小,但比亲本高。有两种可能的原因导致了接合转移子的万古霉素抗性低于相应的供体菌,一是遗传的不稳定性,二是接合转移后抗性产生的延迟。但也有研究表明,万古霉素抗性的消失并不完全依赖于含有A 基因的 Tn1546 转座子的缺失[8]。
2.2 vanV
Ribeiro 等[9]在粪肠球菌中鉴定了一种新的基因属于B 基因簇。在XB基因的 3' 端存在2292 的开放阅读框(open reading frame,ORF),2292 处于XB基因的下游,用 Fold-change 值衡量基因差异表达,其值为 26,与其他抗性基因处于同一诱导水平,该结果显示2292 是B 操纵子的一部分。此外,RT-PCR 和 Northern blot 结果显示,2292 和B 操纵子共转录并共用一个启动子 PYB,并将其命名为V 基因。然而,该研究发现在其他 7 种和 24 种中,仅有两株细菌能够产生预期的相似复制子。万古霉素对 V583 菌株以及V 基因缺失的 V583 菌株的 MIC 值表明,V 对万古霉素抗性的形成没有作用。V 基因是B 基因簇的一部分,与YBWHBBXB成线性排列(图 1)且不参与形成万古霉素抗性。
2.3 vanG
Peltier 等[10]研究发现,的G 样基因簇有 5 个 ORFs,分别是调控区(R 和S)和效应区(G、XY 和T)。630 菌株的基因组序列与肠球菌的G 基因簇有很高的相似性,但630 菌株对万古霉素敏感。该基因座有 6153 个碱基,在基因座的 5' 端有同源的RG和SG基因组成的二组分调控系统,在SG基因下游 51 nt 处有一茎环结构与依赖 Rho 因子的终止过程有关。抗性区域组成为G、XYG和TG样基因。G 基因簇的结构组成表明该基因簇范围因两个茎环结构而扩展(图 2)。经实时荧光定量 PCR 检测,XYG样基因和TG基因是共转录的。该研究也证明了G 样基因簇的存在与否对细胞壁的组成并没有影响,因此具有G 样基因簇的菌株依然对万古霉素敏感。
2.4 vanN
Lebreton 等[4]发现UCN71 菌株具有万古霉素抗性却不含有已知的抗性基因,并将新基因命名为N。他们对 UCN17 的总 DNA 克隆并测序表明N 与L 有 65% 的一致性,N 基因编码的连接酶与C、E、G、L 编码的连接酶有 41% ~ 65% 的相似度,所有存在于 D-Ala-D-Ser 连接酶的结构域也存在于N 中。结构分析显示,N 操纵子具有 5 个 ORFs,即N、XYN、TN、RN和SN,其中TN和TC具有 52% 的相似性,RN和SN二组分调节系统也和其他基因相似。RACE-PCR 测定N 的启动子 PN与C 和E的相似。UCN71 能合成等量 Ala 和 Ser 为末端的前体。N 是第一个被发现存在于 D-Ala-D-Ser 型万古霉素抗性菌株中的基因。
2.5 vanM
Xu 等[11]从具有万古霉素抗性的Efm-HS0661 菌株中发现了一种新的基因簇即M。基因序列分析显示,M 的长度为 1032 bp,编码一个 343 个氨基酸的蛋白质。同时,M 基因簇与A、B、D 和F 等基因簇具有较高相似度。该基因簇含有 7 个 ORFs,根据其与A、B、D 等基因簇编码蛋白的氨基酸序列相似性程度,将其中的 6 个 ORF 分别命名为RM、SM、YM、HM、M 和XM。RM-SM与A 基因簇中的R-S 二组分调节系统序列相似,而YM与A 基因簇中的Y 基因序列相似,HM则与A、B、D 等基因簇中的H、HB、HD基因序列相似,类似地,XM与A、B、D 等基因簇中的XA、XB、XD基因序列相似,以上相似度在 58.8% ~ 85.7% 间浮动。LC-MS 结果表明,由M 基因簇产生的万古霉素抵抗是由于肽聚糖前体末端的组成变为了 D-Ala-D-Lac。
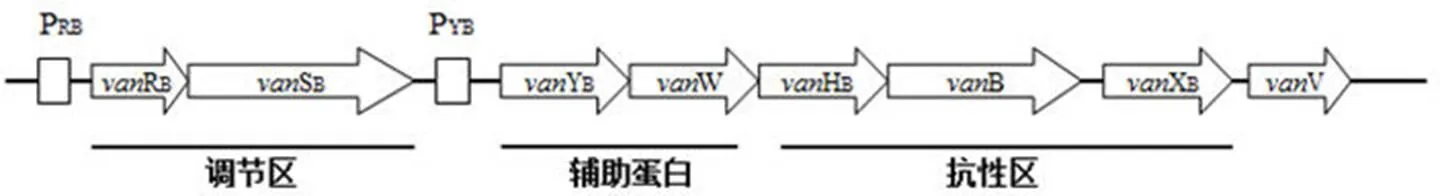
图 1 具有新基因vanV 的vanB 操纵子结构[9](在vanV 基因的上游和PYB启动子的下游之间没有其他启动子区域)
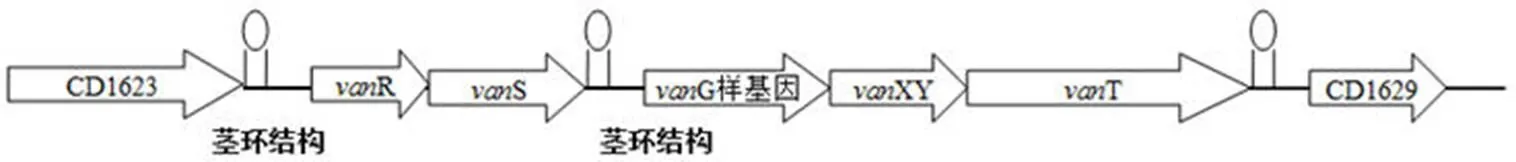
图2 vanG 样基因簇结构[10]
2.6 vanR-vanS 二组分调节系统
R-S 二组分调节系统的激活是诱导细菌产生万古霉素抗性所必须的,二组分调控系统由R 和S 两基因构成,R 基因可以对刺激进行应答调控,而S 则编码一种应激蛋白,通常R 和S 基因在染色体上与H、A、B 基因相邻。Kwun 等[2]以的抗性系统为模型进行体内实验表明,在B 型的抗性系统中,S 感应激酶的激活依赖于和 D-Ala-D-Ala 形成复合物。目前有两种模型假说解释识别S应激蛋白特异性配体的本质:第一种模型是直接诱导,认为感应激酶是与抗生素在感应区直接结合而激活的;第二种模型是间接诱导,认为感应激酶是与细胞壁代谢物结合的,而这些代谢物是在细胞壁合成和与药物反应过程中产生的。在Koteva 等[12]研究中,利用一种合成的光亲和探针 VPP 证实了第一种模型为正确解释。
3 提高万古霉素抗菌活性的相关研究进展
目前多药耐药菌株的出现引起了研究人员的高度关注,而纳米颗粒偶联万古霉素在应对万古霉素耐药问题中显示出独特的优势。
Fayaz 等[13]利用万古霉素偶联金纳米颗粒(vancomycin bound gold nanoparticles,VBGNP)对抗VRSA 取得了一定的成果。在实验中,他们利用真菌进行金纳米颗粒的生物合成,与万古霉素偶联后获得了对 VRSA、万古霉素敏感金黄色葡萄球菌(vancomycin sensitive,VSSA)和大肠杆菌的抗菌活性。结果表明,VBGNP 对于 VRSA 表现良好的体外抗菌活性。原因可能是 VBGNP 对于细胞壁前体末端可以无特异性地结合,从而导致 VRSA 的细胞壁合成受阻进而细胞裂解。透射电子显微镜照片明显看出 VBGNP 可以轻易穿过大肠杆菌细胞内膜和外膜,破坏脂多糖层的稳定性,而后 VBGNP 上的万古霉素部分就与相应的位点结合导致细胞裂解。
Chakraborty 等[14]将万古霉素装载到羧甲基壳聚糖-2, 2'-(乙烯基二氧)二乙胺-叶酸颗粒上制备成具有万古霉素偶联的纳米颗粒(nanoconjugated vancomycin,NV)。实验中测定了 NV 的万古霉素溶出率最高可在 8 h 时达到 95.24%。实验中 NV 比万古霉素显示出对 VRSA 更为强大的抗菌活性,实验结果显示,NV 和万古霉素对 VSSA 的 MIC 值分别为 1 和 2 μg/ml,对 VRSA 的 MIC 值分别为 2 和 64 μg/ml;NV 和万古霉素对 VSSA 的 MBC 值分别为 1 和 4 μg/ml;对 VRSA 的 MBC 值分别为 2 和 512 μg/ml。此外,在 NV 给药组中,VRSA 的细胞膜悬液的光密度值显著低于对照组和万古霉素给药组,提示 NV 使得 VRSA 的细胞膜厚度降低。在存活率方面,NV 给药组中,VRSA 的存活率为 35.11%,显著低于万古霉素组的 95.73%。原子力显微镜(AFM)图像显示,NV 能明显使 VRSA 的形态变化和结构破坏,在 NV 给药组中,VRSA 的细胞壁能明显看见被破坏,进而细胞裂解造成细菌死亡。透射电子显微镜图像显示,在 NV 给药组中,VRSA 细胞壁厚度为(24.34 ± 4.34)nm,明显小于万古霉素组的(54.92 ± 5.12)nm。此外,NV 能明显增加 VRSA 的 Na-K-ATP 酶的活性,Na-K-ATP 酶的活性增强使 K+的释放增加并促进胞内在 260 nm 处有吸收的物质分解从而使细胞裂解增强。
4 总结与展望
多年来,抗生素被用于抑制和杀死细菌和其他微生物,但是目前微生物的耐药性已经十分普遍,从而降低了这些抗菌药物的有效性。万古霉素本是作为在其他抗生素无效时使用的最后一道防线药物,却早已在20 世纪80 年代就出现了耐药菌株。因此,研究万古霉素耐药性产生的原因并解决耐药性问题显得十分必要。目前,关于万古霉素耐药基因的研究进展十分迅速,其中如何加强万古霉素的抗菌活性是研究人员关注的焦点之一。纳米技术以其自身独特的性质在提高抗菌活性方面有着十分广泛的用途,目前研究较多的纳米技术包括释放 NO 的纳米颗粒、壳聚糖纳米颗粒以及金属纳米颗粒[3]等。这些研究将为纳米技术及材料应用在临床上以提升万古霉素抗菌活性打下坚实基础。
[1] Bisicchia P, Bui NK, Aldrige C, et al. Acquisition of vanB-type vancomycin resistance by Bacillus subtilis: the impact on gene expression, cell wall composition and morphology. Mol Microbiol, 2011, 81(1):157-178.
[2] Kwun MJ, Novotna G, Hesketh AR, et al. In vivo studies suggest that induction of vanS-dependent vancomycin resistance requires binding of the drug to D-Ala-D-Ala termini in the peptidoglycan cell wall. Antimicrob Agents Chemother, 2013, 57(9):4470-4480.
[3] Pelgrift RY, Friedman AJ. Nanotechnology as a therapeutic tool to combat microbial resistance. Adv Drug Deliv Rev, 2013, 65(13-14): 1803-1815.
[4] Lebreton F, Depardieu F, Bourdon N, et al. D-Ala-d-Ser vanN-type transferable vancomycin resistance in Enterococcus faecium. Antimicrob Agents Chemother, 2011, 55(10):4606-4612.
[5] Johnson JK, Cashara D. Current rapid screening methods for gastrointestinal colonization of vancomycin resistant Enterococci. Clin Microbl Newsl, 2013, 35(6):45-51.
[6] Chancey TS, Zähner D, Stephens DS. Acquired inducible antimicrobial resistance in Gram-positive bacteria. Future Microbl, 2012, 7(8):959-978.
[7] de Niederhäusern S, Bondi M, Messi P, et al. Vancomycin-resistance transferability from vanA Enterococci to Staphylococcus aureus. Curr Microbl, 2011, 62(5):1363-1367.
[8] Choi HJ, Nam D, Peck KR, et al. Loss of vancomycin resisitance not completely dependent on the Tn1546 element in Enterococcus faecium isolates. Diagn Microbiol Infect Dis, 2011, 69(1):105-110.
[9] Ribeiro T, Santos S, Marques MI, et al. Identification of a new gene, vanV, in vanB operons of Enterococcus faecalis. Int J Antimicrob Agents, 2011, 37(6):554-557.
[10] Peltier J, Courtin P, El Meouche I, et al. Genomic and expression analysis of the vanG-like gene cluster of Clostridium difficile. Microbiology, 2013, 159(Pt 7):1510-1520.
[11] Xu X, Lin D, Yan G, et al. vanM, a new glycopeptides resisitance gene cluster found in Enterococcus faecium. Antimicrob Agents Chemother, 2010, 54(11):4643-4647.
[12] Koteva K, Hong HJ, Wang XD, et al. A vancomycin photoprobe identifies the histidine kinase vanSsc as a vancomycin receptor. Nat Chem Biol, 2010, 6(5):327-329.
[13] Fayaz AM, Girilal M, Mahdy SA, et al. Vancomycin bound biogenic gold nanoparticles: a different perspective for development of anti VRSA agents. Proc Biochemistry, 2011, 46(3):636-641.
[14] Chakraborty SP, Sahu SK, Pramanik P, et al. In vitro antimicrobial activity of nanoconjugated vancomycin against drug resistance Staphylococcus aureus. Int J Pharm, 2012, 436(1-2):659-676.
顾觉奋,Email:yqyan1@126.com
2014-02-08
10.3969/cmba.j.issn.1673-713X.2014.05.009

