猪圆环病毒2型Cap蛋白B细胞表位的预测及鉴定
周景明, 宋瑞雪, 刘红亮, 张秋丽, 祁艳华, 张改平, 王爱萍
(郑州大学 生命科学学院 河南 郑州 450001)
猪圆环病毒2型Cap蛋白B细胞表位的预测及鉴定
周景明, 宋瑞雪, 刘红亮, 张秋丽, 祁艳华, 张改平, 王爱萍
(郑州大学 生命科学学院 河南 郑州 450001)
利用生物信息学技术和原核表达系统,筛选猪圆环病毒2型(PCV2)核衣壳蛋白(Cap)的B细胞表位,为进一步揭示PCV2的免疫调控机制提供研究依据.运用生物信息学软件预测PCV2 Cap蛋白的亲水性、表面可及性、蛋白柔韧性、抗原性和B细胞线性表位;根据预测结果,初步确定Cap蛋白潜在的B细胞线性表位主要位于58~66、79~91、151~162、174~189、204~213和221~233位氨基酸.设计特异性引物,扩增Cap蛋白潜在表位区域基因,并克隆到pET32a(+)原核表达载体上,转化至表达菌BL21(DE3)plysS中,在IPTG诱导下表达目的蛋白;利用PCV2阳性血清鉴定Cap蛋白的B细胞线性表位,筛选出3 个能与血清结合的肽段,即Cap77-95、Cap174-189和Cap221-233;对获得的肽段进行在线同源比对,发现3 个片段均为PCV2 Cap蛋白的特有序列,有可能成为PCV2的特异性表位.
猪圆环病毒2型; Cap蛋白; 原核表达; 表位
0 引言
猪圆环病毒(porcine circovirus, PCV)是单链环状无囊膜DNA病毒,该病毒粒子呈二十面体对称结构[1],是已知的最小动物病毒之一[2]. 根据其基因组成、抗原性和致病性的不同,将其划分成PCV1和PCV2两个类型,其中PCV2是断奶仔猪多系统衰竭综合征(post-weaning multisystemic wasting syndrome, PMWS)的主要病源,同时会引发猪皮炎与肾炎综合征(porcine dermatitis and nephropathy syndrome, PDNS)、猪呼吸道综合征(porcine respiratory disease complex, PRDC)以及繁殖障碍等多种疾病,使养猪产业受到重大经济损失[3].
PCV2有11个大小不同的开放阅读框ORFs,其中ORF1和ORF2是主要的开放阅读框,ORF1编码的Rep蛋白与病毒的复制起始有关,ORF2编码的Cap蛋白为病毒的核衣壳蛋白,该蛋白与病毒和宿主细胞的结合有关,具有较好的抗原性和免疫原性[4],对Cap蛋白的研究有利于深入了解该病毒的特性.
PCV2目前被划分成PCV2a、PCV2b、PCV2c、PCV2d和PCV2e这5种基因亚型,其中PCV2b型的毒性最强,并且在国内的流行范围最广[5],碱基的突变可能是疾病流行的又一原因,保守性表位的研究对PCV2的检测及疫苗的研制至关重要.本研究主要是在生物信息学分析的基础上对预测得到的潜在表位区域基因进行扩增,并通过原核表达系统获得目的蛋白,再利用血清鉴定出具有反应性的蛋白肽段,为PCV2的检测及基因表位疫苗的制备提供依据.
1 材料和方法
1.1 材料
PCV2 KF株(GenBank No. ACE95718.1)、猪圆环病毒阳性血清由本实验室保存;IPTG、氯霉素和氨苄青霉素购自Solarbio公司;限制性核酸内切酶、T4 DNA连接酶购自Promega公司;HRP标记羊抗猪、羊抗鼠IgG购自Abbkine公司;ECL发光液购自Beyotime公司;抗His单克隆抗体购自郑州益康生物公司.
1.2 Cap蛋白结构特性及潜在表位预测
利用生物信息学软件DNAstar中Protean模块预测Cap蛋白的结构特性,其中采用Gamier-Robson法和Chou-Fasman法预测蛋白质二级结构;利用Kyte-Doolittle法、Emini法、Karplus-Sehulz法和Jameson-Wolf法预测蛋白的亲水区、表面可及性、蛋白质骨架的柔韧性以及抗原指数较高的区段;同时利用网络服务器ABCpred[6](http://www.imtech.res.in/rag hava/abcpred/)、Bepipred 1.0[7](http://www.cbs.dtu.dk/services/BepiPred/)预测Cap蛋白的B细胞线性表位,ABCpred设定阈值为0.85,表位长度为16 个氨基酸.综合分析蛋白表面可及性、抗原性、二级结构等多种预测结果,确定可能的表位区域.
1.3 重组质粒的构建
根据预测结果缩小抗原表位区段,设计蛋白表达方案.利用Primer 5.0设计引物(表1),分别引入BamHΙ和HindⅢ酶切位点.以本实验室前期构建的pET28a-ORF2质粒为模板,PCR扩增基因片段,反应程序为:95 ℃预变性5 min;94 ℃变性30 s,62 ℃退火30 s,72 ℃延伸90 s,32 个循环;72 ℃延伸10 min,乙醇沉淀法纯化回收PCR产物.将目的基因片段和pET32a(+)表达载体分别进行双酶切反应,回收酶切产物并用T4连接酶16 ℃连接过夜,次日转化至E.coliDH5α感受态细胞中,构建含有His标签的重组蛋白表达载体,提取质粒经PCR鉴定后送至生工生物工程有限公司进行测序.

表1 引物序列及位置
注:划线部分为酶切位点.
1.4 蛋白原核表达及Western-blot鉴定
将重组载体质粒转化至BL21(DE3)plysS感受态细胞中.挑取克隆置于含有氨苄青霉素、氯霉素的液体LB培养基中,37 ℃振荡培养过夜,次日将过夜菌按1∶100比例加至含有双抗的培养基中,37 ℃振荡培养,当OD450值达到0.4~0.6时,加入1 mmol/L IPTG,16 ℃诱导10 h,收集菌体,经SDS-聚丙烯酰胺凝胶电泳分析后转至PVDF膜上,2.5%脱脂奶室温封闭1 h,抗His单克隆抗体(1∶2 000)4 ℃过夜,次日室温孵育1 h,PBST洗3次,每次10 min,羊抗鼠HRP-IgG(1∶4 000)作为二抗,室温孵育1 h,PBST洗3次,每次10 min,用ECL发光液进行显色.
1.5 优势表位分析
融合蛋白经鉴定后进行Western-blot分析[8],用PCV2阳性血清(1∶100)作为一抗,羊抗猪HRP-IgG(1∶2 000)作为二抗,筛选能与阳性血清特异性结合的肽段作为优势表位.
2 结果与分析
2.1 PCV2 KF株同源树的建立
构建KF株Cap蛋白氨基酸序列同源树,结果如图1所示,该病毒株的Cap蛋白序列与PCV2b型具有较高的同源性,因此判定该病毒株为PCV2b亚型.

图1 PCV2 KF株氨基酸的同源树Fig.1 Homology-tree of PCV2 KF
2.2 Cap蛋白结构特性分析及表位预测
利用ExPASy(http://web.expasy.org/protparam/)预测该蛋白的基本性质,Cap蛋白基因编码233个氨基酸,其相对分子质量为27.85 kD(如图2).综合Protean模块、ABCpred和Bepipred1.0预测结果显示,在58~66、79~91、151~162、174~189、204~213和221~233区域内易形成转角和无规则结构,同时其亲水性较强、抗原指数较高且表面可及性较好,可将此区域作为潜在的B细胞表位.

图2 PCV2 Cap蛋白结构特性Fig.2 The structure properties of PCV2 Cap protein
2.3 重组载体的鉴定及测序
重组质粒的PCR产物经3%的琼脂糖凝胶电泳分析显示(图3),在相应位置有明显的DNA条带,大小分别约为44 bp、71 bp、50 bp、62 bp、50 bp、53 bp.质粒测序结果分析显示,目的片段插入位置及读码框均正确无误,同时与GenBank公布的基因序列进行比对发现(如图4),目的片段碱基序列未发生突变,说明成功构建了目的基因的原核表达质粒.
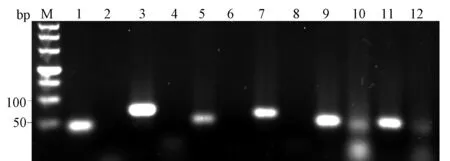
1: Cap56-67基因片段; 3: Cap77-95基因片段; 5: Cap151-162基因片段; 7: Cap174-189基因片段; 9: Cap204-215基因片段;11: Cap221-233基因片段; 2、4、6、8、10、12: 阴性对照; M: marker图3 质粒PCR鉴定结果Fig.3 PCR identification of plasmid

图4 质粒测序图谱Fig.4 The map of plasmid sequence
2.4 Cap截短蛋白表达及Western-blot鉴定
将重组表达载体质粒pET32a-(Cap56-67、Cap77-95、Cap151-162、Cap174-189、Cap204-215、Cap221-233)转化到表达菌BL21(DE3)plysS后通过IPTG诱导蛋白表达,并收集菌体进行SDS-PAGE分析,结果如图5A所示,重组表达菌诱导后在预期位置均出现融合蛋白条带,分别约为21.3 kD、22.8 kD、21.5 kD、22.0 kD、21.5 kD、21.6 kD.Western-blot分析显示(图5B),表达的融合蛋白在对应位置有明显的印迹条带,大小与理论值相符.以上结果表明,融合蛋白均得到正确表达.

A: IPTG诱导表达融合蛋白的SDS-PAGE分析. 1: pET32a-Cap56-67-BL21; 2: pET32a-Cap77-95-BL21; 3: pET32a-Cap151-162-BL21; 4: pET32a-Cap174-189-BL21; 5. pET32a-Cap204-215-BL21;6: pET32a-Cap221-233-BL21; 7: pET32a-BL21; M: markerB: 蛋白Western-blot鉴定.1: pET32a-Cap56-67-BL21; 2: pET32a-Cap77-95-BL21; 3: pET32a-Cap174-189-BL21;4: pET32a-Cap204-215-BL21; 5: pET32a-Cap221-233-BL21; 6: pET32a-BL21;7: pET32a-Cap151-162-BL21; 8: pET32a-BL21; M: marker图5 融合蛋白的分析鉴定Fig.5 Analysis and identification of fusion proteins
2.5 优势表位的鉴定
利用PCV2阳性血清对表达的融合蛋白进行Western-blot分析显示(图6),pET32a-Cap77-95-BL21、pET32a-Cap174-189-BL21、pET32a-Cap221-233-BL21表达的蛋白可以与阳性血清发生特异性反应.

1: pET32a-Cap56-67-BL21; 2: pET32a-Cap77-95-BL21; 3: pET32a-Cap151-162-BL21; 4:pET32a-Cap174-189-BL21;5: pET32a-Cap204-215-BL21; 6: pET32a-Cap221-233-BL21; 7: pET32a-BL21; M: marker图6 蛋白表达Western-blot分析Fig.6 Western-blot analysis of protein
3 讨论
PCV2型病毒广泛存在于世界范围内,大多数养猪场均感染过PCV2病毒.目前,该病毒的预防手段主要是疫苗接种,但现有的商品化疫苗大多是根据PCV2a型病毒研制的,虽然对PCV2b型具有部分交叉保护的作用,但不能明显抑制PCV2b型病毒的感染[9].PCV2b作为当前主要的流行性亚型,对其表位的分析有利于抗原表位疫苗的研制.
目前已有多种表位鉴定的研究方法,如噬菌体展示肽库技术、肽探针扫描技术和生物信息学软件预测等技术.其中生物信息学预测技术因其简单、快速易行,已经被越来越多的研究者使用,该技术主要是通过预测蛋白的二级结构、亲水性、表面可及性等来确定表位的潜在区域.抗原表位与蛋白质的二级结构有关,α-螺旋和β-折叠的化学键键能较高,形态规则不易改变,具有稳定蛋白质骨架的作用,无规则卷曲和β-转角一般是凸出结构,会发生扭曲,易与抗体结合,故成为抗原表位的机率较大[10].蛋白质的亲水性、骨架柔韧性、表面可及性等与抗原表位的形成具有密切联系[11-12],亲水性高的区域,暴露在蛋白表面的可能性较大;柔韧性较强,与抗体分子嵌合机率较高,有利于形成抗原表位;表面可及性反映了溶剂分子接触氨基酸的可能性,预测值大于1的区域易形成抗原表位;抗原性指数可以直接反映出氨基酸抗原性的强度.多种方法结合的策略可提高表位预测的准确性.
本研究通过构建同源树确定PCV2 KF株为PCV2b亚型,同时利用生物信息学软件预测该病毒株的潜在B细胞表位区域,然后采用原核表达方法获得目的蛋白并进行Western-blot分析,其中Cap77-95、Cap174-189和Cap221-233的表达蛋白能与阳性血清发生特异性结合.
利用NCBI中的Protein Blast对所得的序列区域进行在线同源性分析,从而鉴定优势表位的特异性.同源性分析表明,Cap77-95、Cap174-189和Cap221-233的氨基酸序列为PCV2型Cap蛋白的特有序列,因此可能成为Cap蛋白特异的优势表位.将所得序列与IEDB数据库中已发表的B细胞表位进行比较显示,Cap174-189和Cap221-233氨基酸序列与文献[13]所报道的表位基本符合,肽链Cap77-95与文献[14]利用单克隆精确鉴定的表位有部分重叠,但是不同的基因型,其抗原性存在一定的差异[15].
Cap蛋白作为PCV2的主要保护性抗原,对重组抗原和表位疫苗等的研制至关重要.本研究利用软件预测与实验相结合的方法对Cap蛋白的B细胞线性表位进行分析和鉴定,筛选出3 个可以与阳性血清特异结合的片段,即Cap77-95、Cap174-189和Cap221-233,为猪圆环病毒的检测以及表位疫苗的制备奠定了基础.
[1] KHAYAT R, BRUNN N, SPEIR J A, et al. The 2.3-angstrom structure of porcine circovirus 2[J]. Journal of virology, 2011, 85(15): 7856-7862.
[2] SEGALÉS J, KEKARAINEN T, CORTEY M. The natural history of porcine circovirus type 2: from an inoffensive virus to a devastating swine disease?[J]. Veterinary microbiology, 2013, 165(1): 13-20.
[3] 王爱萍, 李鹏飞, 周景明,等. 猪圆环病毒Ⅱ型抗体检测ELISA试剂盒的制备[J].郑州大学学报(理学版), 2015,47(3):92-98.
[4] CHEUNG A K, GREENLEE J J. Identification of an amino acid domain encoded by the capsid gene of porcine circovirus type 2 that modulates intracellular viral protein distribution during replication[J]. Virus research, 2010, 155(1): 358-362.
[5] ALLAN G, KRAKOWKA S, ELLIS J, et al. Discovery and evolving history of two genetically related but phenotypically different viruses, porcine circoviruses 1 and 2[J]. Virus research, 2012, 164(1/2): 4-9.
[6] SAHA S, RAGHAVA G P. Prediction of continuous B-cell epitopes in an antigen using recurrent neural network[J]. Proteins-structure function & bioinformatics, 2006, 65(1): 40-48.
[7] LARSEN J E P, LUND O, NIELSEN M. Improved method for predicting linear B-cell epitopes[J]. Immunome research, 2006, 2(1):1-7.
[8] LI Y, JIA Y, WEN K, et al. Mapping B-cell linear epitopes of NS3 protein of bovine viral diarrhea virus[J]. Veterinary immunology & immunopathology, 2012, 151(3/4): 331-336.
[9] 赵晨辰,王敏,刘长辉,等.猪圆环病毒2型分子流行病学及疫苗研究进展[J].中国兽药杂志,2015,49(2): 63-69.
[10]高巧艳,李燕,周密,等.人白细胞介素37的二级结构及B细胞表位预测[J].中国免疫学志,2014,30(5): 648-650.
[11]刘银凤,张莉,李海龙,等.淋病奈瑟菌外膜蛋白PorB二级结构及其B细胞和T细胞表位分析[J].中国病原生物学杂志,2015,10(3): 215-219.
[12]MOHABATKAR H. Prediction of epitopes and structural properties of Iranian HPV-16 E6 by bioinformatics methods[J]. Asian pacific journal of cancer prevention, 2007, 8(4): 602-606.
[13]SHANG S B, JIN Y L, JIANG X, et al. Fine mapping of antigenic epitopes on capsid proteins of porcine circovirus, and antigenic phenotype of porcine circovirus type 2[J]. Molecular immunology, 2009, 46(3): 327-334.
[14]张璐.猪圆环病毒2型Cap蛋白B细胞线性抗原表位鉴定[D]. 哈尔滨:东北农业大学,2014.
[15]KOCH K S, LEFFERT H L. Ectopic expression of CD74 in Ikkβ-deleted mouse hepatocytes[J]. Acta histochemica, 2011, 113(4): 428-435.
(责任编辑:王浩毅)
Prediction and Identification of B Cell Epitopes of the PCV2 Cap Protein
ZHOU Jingming, SONG Ruixue, LIU Hongliang, ZHANG Qiuli, QI Yanhua, ZHANG Gaiping, WANG Aiping
(SchoolofLifeSciences,ZhengzhouUniversity,Zhengzhou450001China)
Screening B cell epitopes of PCV2 Cap by bioinformatics techniques and prokaryotic expression systems, could provide a basis to further reveal the immune regulation mechanism of PCV2. The hydrophilicity, flexibility, accessibility, antigenicity and B-cell liner epitopes of PCV2 Cap were predicted by bioinformatics methods. There were 6 potential B-cell linear epitopes in Cap. They might locate in the regions of 58-66, 79-91, 151-162, 174-189, 204-213 and 221-233 aa. The potential epitopes gene were amplified by PCR using specific primers and cloned into the expression vector pET32a(+). E coli BL21(DE3)plysS cells were transformed with the recombinant plasmid; and the target proteins were expressed by induction using IPTG; B-cell linear epitopes of Cap protein were identified by detecting the reactivity of the target proteins with PCV2 positive serum. Three kinds of protein peptides (Cap77-95, Cap174-189,Cap221-233) with reactogenicity were selected. Analysis of homology indicated that three segments were unique to Cap which might be the specific epitopes of PCV2.
porcine circovirus type 2; Cap protein; prokaryotic expression; epitopes
2017-01-20
国家重点研发计划项目(2016YFD0500704);郑州市科技创新团队项目(131PCXTD622).
周景明(1972—),男,河南新野人,副教授,主要从事分子免疫学和免疫学检测技术研究,E-mail: zhoujingming@zzu.edu.cn;通信作者:王爱萍(1970—),女,河南唐河人,教授,主要从事分子免疫学和免疫学检测技术研究,E-mail: pingaw@zzu.edu.cn.
S858.28
A
1671-6841(2017)03-0111-06
10.13705/j.issn.1671-6841.2017014
——一道江苏高考题的奥秘解读和拓展

