猪流行性腹泻病毒Nsp5基因的原核表达及生物信息学分析
刘正奎,吴 瑗,陈 琳,王 磊,牟泓烨,祝徐航,王晓杜,*
(1.浙江农林大学 动物科技学院,浙江 杭州 311300; 2.金华职业技术学院 农业生物工程学院,浙江 金华321007)
猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)常感染3~10日龄仔猪,可引起该猪群发生猪流行性腹泻(porcine epidemic diarrhea,PED),临床症状以急性肠炎和脱水致死为特征的高度传染性疾病。PEDV感染猪群,仔猪发病率和死亡率都很高,一年四季均可发病,自2011年以来,新的变异毒株开始在各地纷纷流行[1-3],现已出现在世界各个国家和地区[4-6],给养猪业带来严重的经济损失[7-8]。
猪流行性腹泻病毒属于冠状病毒科冠状病毒属α型冠状病毒,其复制酶蛋白是由ORF1a和ORF1b基因编码的PP1a和PP1ab前体蛋白,经病毒编码蛋白酶加工而成,形成至少16个非结构蛋白(nonstructural proteins,NSPs),从PP1a或PP1ab的氨基端起依次为Nsp1-16,这些蛋白在病毒的转录、复制、抗宿主细胞防御机制过程中发挥重要功能[9-10]。其中Nsp5被称为3C样蛋白酶[11],是一种富含组氨酸和半胱氨酸的蛋白酶[12],该蛋白酶具有切割活性,可以将多聚前体蛋白PP1ab切割为成熟的非结构蛋白Nsp4-16[11]。除了具有切割活性外,还发现Nsp5是一种干扰素的拮抗分子[13],同时在病毒复制过程中,对N蛋白进行加工修饰,调节PEDV对宿主细胞的适应性[14]。因此,研究PEDV的Nsp5蛋白结构及相关功能对了解该病毒的致病机理,开发新型抗病毒药物具有重要意义。本研究通过克隆Nsp5基因,构建原核表达载体,在大肠埃希菌进行表达,同时利用生物信息学软件进行结构预测分析,为解析Nsp5的功能提供生物材料和数据支持。
1 材料与方法
1.1 试验材料
PEDV/LY/2014/04分离株来自临床腹泻样本,由本实验室分离得到,保存于浙江农林大学动物预防医学与公共卫生实验室;pET-28a(+)载体由本实验室保存。
1.2 主要试剂
E.coliDH5α和BL21(DE3)感受态细胞购自天根生化科技(北京)有限公司;T4连接酶、限制性内切酶SalI和EcoRⅠ购自NEB公司;质粒小提取试剂盒、DNA胶回收试剂盒购自天根生化科技(北京)有限公司;KOD plus Neo购自TOYOBO公司。
1.3 试验方法
1.3.1 引物设计与合成
依据猪流行性腹泻病毒CV777疫苗株全基因组序列(GenBank:KT323979),利用序列比对方法,找到编码Nsp5的基因,使用DNAStar中的Primer Select软件进行Nsp5基因片段的引物设计。设计一对扩增Nsp5基因的特异性引物,上下游引物两端添加EcoRⅠ和SalⅠ酶切位点,扩增片段大小为616 bp。引物由上海生工生物工程有限公司合成,如表1。
1.3.2Nsp5基因扩增
采用TRIZOL法提取PEDV/LY/2014/04分离株的RNA,随机引物反转录得到其cDNA,以此cDNA为模板进行目的片段Nsp5的PCR扩增。PCR扩增体系:rTaqMix 10 μL,dd H2O 6 μL,上下游引物各1 μL,cDNA模板2 μL,共20μL。PCR扩增条件:预变性94 ℃ 3 min;94 ℃变性45 s,65 ℃退火30 s,72 ℃延伸1 min,共35个循环,72 ℃延伸10 min。PCR产物用1%琼脂糖凝胶进行电泳检测。
表1Nsp5基因扩增引物及鉴定引物
Table1Nsp5 gene amplification primers and identification primers

引物名称Primer name引物序列Primer sequence(5’-3’)目的片段大小Product size/bpNsp5-FwdCCGGAATTCATGTTGCAGAGTGTTGCATCTACTTATGTAG616Nsp5-RevGACGTCGACTCACTGGAGCTTAACAATACGCTCACNsp5-FwdCCGGAATTCATGTTGCAGAGTGTTGCATCTACTTATGTAG705T7-TerminalGCTAGTTATTGCTCAGCGG
1.3.3 重组质粒pET-28a(+)-Nsp5构建
利用胶回收试剂盒将Nsp5扩增片段纯化回收,利用EcoRⅠ和SalⅠ进行双酶切,同时用EcoRⅠ和SalⅠ双酶切pET-28a(+)载体,酶切结束电泳后切胶回收,两种酶切产物按照适宜比例,利用T4连接酶在16 ℃过夜连接。连接产物经热击法转化至E.coliDH5α感受态细胞,涂布卡那霉素的平板,37 ℃过夜培养,挑取单个菌落进行菌落PCR鉴定。鉴定呈阳性的菌落经扩大培养后,质粒小提试剂盒提取质粒,再进行EcoRⅠ和SalⅠ双酶切鉴定,酶切鉴定正确的克隆送交上海生工生物有限公司进行测定验证。测序验证正确的重组质粒命名pET-28a(+)-Nsp5。
1.3.4 His-Nsp5重组蛋白的诱导表达
将1.3.3节构建正确的pET-28a(+)-Nsp5重组质粒转化至E.coliBL21(DE3)感受态细胞,涂布于含卡那霉素抗性的LB平板,37 ℃过夜培养,分别挑选3~5个单菌落于LB液体培养基,37 ℃、220 r·min-1培养12~16 h,以此为种子液。种子液按照体积比1∶100转接液体LB培养基里,当菌液的D600值在0.5~0.6时,取1 mL菌液作为诱导前样品,加入终浓度为1 mmoL·L-1的IPTG,在37 ℃、200 r·min-1进行诱导表达,诱导6 h后取1 mL菌液作为诱导后样品,所取样品处理后进行SDS-PAGE电泳,考马斯亮蓝染色后分析重组Nsp5蛋白表达情况。
1.3.5 His-Nsp5重组蛋白的纯化
小量表达验证成功的克隆,扩大培养后进行大量诱导,收集大量诱导表达菌体后,用PBS缓冲液重悬,冰浴超声破碎30 min,离心收集包涵体沉淀。包涵体沉淀经尿素溶解后,在4 ℃、12 000 r·min-1离心25 min,上清移至新的离心管;将上清与平衡过的镍柱在4 ℃结合3~4 h,待结合液流出,加入30 mL 50 mmoL·L-1咪唑洗除杂蛋白,然后缓慢持续地加入5 mL 400 mmoL·L-1咪唑洗脱目的蛋白,收集洗脱下来的目的蛋白,酶标仪测定蛋白浓度,纯化得到的蛋白加入等体积100%甘油,保存于-80 ℃备用。纯化后目的蛋白进行SDS-PAGE电泳,分析His band Ni+纯化后重组蛋白的纯度和表达量。
1.3.6 Nsp5蛋白质的生物信息学分析
Vector NTI Advance 软件对Nsp5蛋白质的化学性质进行理论分析(包括分子质量、等电点以及氨基酸组成等),ProtScale(http://web.Expasy.org/protscale/)分析Nsp5蛋白质的亲水性与疏水性,ABCpred服务器在线分析了Nsp5蛋白的B细胞潜在抗原表位,SOPMA服务器在线分析了Nsp5蛋白质二级结构预测,Phyre2.0(http://www.sbg.bio.ic.ac.uk/ phyre2/html/page.cgi?id=index)预测分析Nsp5蛋白质的3D结构,Raswin软件初步预测了Nsp5蛋白质的三级结构。
2 结果与分析
2.1 Nsp5基因的PCR扩增及重组表达载体的PCR鉴定
PCR扩增产物经1%琼脂糖凝胶电泳显示,大小约616 bp,与预期大小相符(图1)。将Nsp5的PCR产物和载体pET-28a(+)用限制性内切酶EcoRⅠ和SalⅠ进行双酶切之后,经T4连接酶连接,热击转化至DH5α感受态细胞中,得到重组载体pET-28a(+)-Nsp5。通过PCR的方法,鉴定引物为目的基因的上游引物Nsp5-Fwd和载体pET-28a(+)的T7-Terminal引物(表1),同时通过双酶切(EcoRⅠ和SalⅠ)鉴定的方法,筛选并验证得到阳性克隆(图2)。阳性克隆送生物公司测序,序列比对结果表明,该序列与GenBank的PEDV毒株(KM213245)同源性高达99%,且编码框插入正确。
2.2 重组蛋白的诱导表达及鉴定
将重组质粒pET-28a(+)-Nsp5转化至E.coliBL21(DE3)感受态细胞中,经IPTG诱导,SDS-PAGE分析结果显示,在诱导后样品泳道出现了特异性蛋白表达,其分子质量约为22 ku,与预期大小相符(图3)。针对诱导后的包涵体沉淀,采用His band Ni+柱进行纯化,所得样品进行SDS-PAGE检测,结果获得纯度达到70%以上的重组蛋白(His-Nsp5)。

M,DL 2000 bp DNA标准分子量;1,阴性对照;2,Nsp5基因PCR扩增产物。M, DL 2000 bp DNA marker; Lane 1, Negative control; Lane 2, Nsp5 gene PCR products.图1 Nsp5基因PCR扩增结果Fig.1 Agarose gel electrophoresis of Nsp5 PCR products

M,DL 2000 bp DNA 标准分子量;1,pET-28a(+)-Nsp5经EcoRⅠ和SalⅠ双酶切产物。M, DL 2000 bp DNA marker; Lane 1, pET-28a(+)-Nsp5 digested by EcoRⅠ and SalⅠ.图2 pET-28a(+)-Nsp5双酶切鉴定Fig.2 Identification of recombinant pET-28a(+)-Nsp5 plasmid by double enzyme digestion
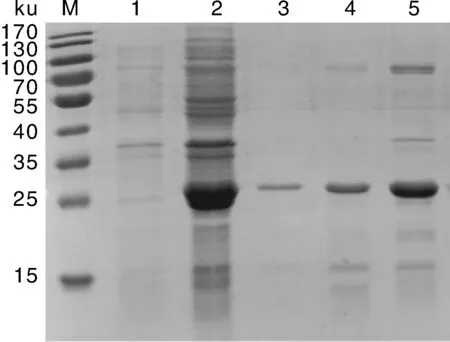
M,低分子量标准蛋白质Marker;1,pET-28a(+)-Nsp5未经IPTG诱导全菌样品;2,pET-28a(+)-Nsp5经IPTG诱导后全菌样品;3-5,pET-28a(+)-Nsp5包涵体纯化后蛋白浓度由低到高的样品。M, Low molecular weight standard protein marker; Lane 1, Non-induced pET-28a(+)-Nsp5; Lane 2: IPTG induced pET-28a(+)-Nsp5; Lane 3-5, Different concentrations of purified protein.图3 Nsp5蛋白表达产物SDS-PAGE电泳Fig.3 SDS-PAGE analysis of Nsp5 protein expression
2.3 猪流行性腹泻病毒Nsp5蛋白质的生物信息学分析
2.3.1 理化性质
猪流行性腹泻病毒的Nsp5的基因含有1个由591 bp组成的开放阅读框,编码196个氨基酸残基组成的多肽,其中A+T=55.50%、C+G=44.50%,蛋白质分子质量的理论值为21 820.07 u,理论等电点(pI)为8.734,在正常生理状态下,该蛋白带正电荷;原子组成是C954H1553N275O291S9;正电荷残基共有(Lys+Arg)24个,负电荷残基(Asp+Glu)21个。该蛋白质由16种氨基酸组成,其中Val、Ala、Ser、Arg的含量相对较多,Cys、Phe、Trp相对较少。不稳定系数(instability index)为42.90,表明这个蛋白质性质不稳定。脂肪系数为95.97,总平均亲水性为-0.214。亲水性分析结果表明该蛋白质是亲水蛋白质(图4)。
2.3.2 Nsp5蛋白质的高级结构预测
SOPMA服务器预测结果显示,该蛋白质二级结构骨架(图5)组成包含:α-螺旋(h)占55.61%、β-折叠(t)占7.65%、无规则卷曲(c)占20.92%、延伸链(e)占15.82%。利用Phyre2.0蛋白质结构预测服务器(使用Intensive选项)对Nsp5白质进行三级结构预测,结果显示,所得结构有较多的α-螺旋和无规则卷曲(图6),结合图5的结果,三级结构预测和二级结构预测基本一致。
2.3.3 Nsp5蛋白质B细胞表位预测
应用ABCpred服务器对Nsp5蛋白的B细胞潜在抗原表位进行预测(阈值设定为0.51,窗口大小设定为16),结果如表2: Nsp5蛋白共有15个潜在的B细胞抗原表位,其中41—56位aa得分最高(结果按得分降序排列,得分越高则意味着是目的肽段的可能性越大),PEDV感染猪群的血清中可能存在特异性抗体。
3 讨论
作为冠状病毒复制启始的主要蛋白酶,Nsp5不仅在病毒非结构蛋白加工方面发挥重要作用,而且参与酶解宿主蛋白[15]、RNA结合以及病毒复制等过程[16-17],Zhu等[18]报道猪流行性腹泻病毒3C样蛋白酶通过切割NEMO调节其干扰素β拮抗作用,因此一直被认为是开发抗冠状病毒药物的有效靶标[19]。本研究通过利用pET-28a(+)原核表达系统,成功构建并在E.coliBL21(DE3)中诱导表达了PEDV/LY/2014/04分离株的重组His-Nsp5蛋白,利用生物信息学软件分析该蛋白,为今后该病的诊断技术和免疫机制研究提供重要的基础信息。

图4 Nsp5蛋白质的亲水性与疏水性分析Fig.4 Hydrophilicity and hydrophobicity analysis of Nsp5 protein
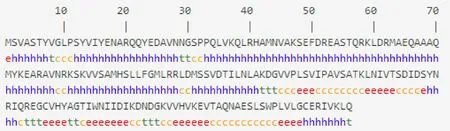
图5 Nsp5蛋白质二级结构模拟图Fig.5 Simulated secondary structure of protein Nsp5
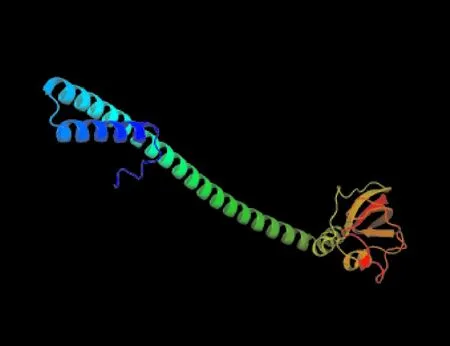
图6 Nsp5蛋白质3D构象预测Fig.6 Prediction of the 3D structure of protein Nsp5
表2PEDV/LY/2014/04分离株Nsp5的B细胞表位预测
Table2Prediction of B cell epitopes of PEDV/LY/2014/04 isolateNsp5

序号No.B细胞抗原表位潜在序列B cell epitope potential sequence位置Position/aa得分Score1HAMNVAKSEFDREAST41-560.952NARQQYEDAVNNGSPP18-330.863IWNIIDIKDNDGKVVH154-1690.854VGLPSYVIYENARQQY8-230.835TSDIDSYNRIQREGCV133-1480.826AAAQMYKEARAVNRKS67-820.817VVHVKEVTAQNAESLS167-1820.818DMSSVDTILNLAKDGV100-1150.819NRIQREGCVHYAGTIW140-1550.7810IKDNDGKVVHVKEVTA160-1750.7611VVSAMHSLLFGMLRRL84-990.7312DREASTQRKLDRMAEQ51-660.7313AESLSWPLVLGCERIV178-1930.7014DGVVPLSVIPAVSATK113-1280.6915GSPPQLVKQLRHAMNV30-450.58
运用结构基因组学研究方法,基因克隆、蛋白质纯化、晶体的筛选和晶体衍射数据的收集,构建出病毒晶体结构,是研究PEDV 3C样蛋白酶结构的关键技术。St. John等[12]通过X射线晶体衍射的技术分析了3C样蛋白酶的空间结构,3C样蛋白酶在空间群P212121中结晶为不对称单元的二聚体。如人类冠状病毒229E 3C样蛋白酶[20]和SARS 3C样蛋白酶[21]所报道的一样,PEDV 3C样蛋白酶是在每个单体中含有三个结构域的同型二聚体,含有由残基Cys144和His41形成的二元催化体系,其活性位点位于结构域Ⅰ和Ⅱ之间的裂缝中,结构域Ⅲ参与单体二聚化,其最终负责形成蛋白酶的活性[22]。正是因为研究者解析了该蛋白的结构,发展了许多3C样蛋白酶抑制剂,很多抑制剂能有效抑制冠状病毒的复制[19,23]。
PEDV的Nsp5基因序列比对分析表明,Nsp5基因是PEDV基因组中比较保守的区段,保证了其功能正确实施。PEDV的Nsp5分子质量理论值为21 820.07 u,理论等电点(pI)为8.734;具有多个高亲水性区域,属于亲水蛋白质;SOPMA服务器(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)是常用的蛋白质预测二级结构在线工具,该工具可以预测蛋白二级结构、蛋白质序列中的复杂基序、构建蛋白质的3D模型,Nsp5则主要由α-螺旋和无规则卷曲构成;Phyre2.0蛋白质结构预测服务器(http://www.sbg.bio.ic.ac.uk/ phyre2/html/page.cgi?id=index)主要用于蛋白质的3D结构和高级结构,Nsp5则主要由螺旋结构构成。ABCpred服务器(http://crdd.osdd.net/raghava/abcpred/)是采用机器学习算法,依赖于Bcipep数据库,对固定长度的多肽进行B细胞表位预测,Nsp5蛋白共有15个潜在的B细胞抗原表位,原核表达产物可作为设计新型疫苗的靶标,也可作为检测PEDV病毒抗体的ELISA检测方法的包被抗原,可以判定是否存在PEDV的感染。
本研究开展了Nsp5蛋白的原核表达,对其理化性质、B细胞抗原表位、高级结构预测方面进行了生物信息学分析,为Nsp5蛋白的研究提供了基本数据资料,可为后续深入探讨Nsp5蛋白在猪流行性腹泻病毒复制过程中的作用机理研究奠定一定的基础,也为进一步开展PEDV的诊断技术及免疫学研究奠定了基础。

