电子克隆获取甜菜富亮氨酸类受体蛋白激酶基因BvLRR-RPK2;1完整编码区
王希 ,陈丽 ,赵春雷
(1.中国农业科学院甜菜研究所/黑龙江大学农作物研究院,哈尔滨150080;2.黑龙江省普通高等学校甜菜遗传育种重点实验室,哈尔滨150080;3.中国农业科学院北方糖料作物资源与利用重点开放实验室,哈尔滨150080)
0 引言
基因资源是一切基因功能研究乃至一切代谢过程分子机理研究的必要前提,而我国甜菜基因克隆的研究基础薄弱且起步较晚,得到克隆的基因资源种类和数量都相当有限[1-3],且集中于报道相对较多、功能比较确定的基因。
受体蛋白激酶(Receptor protein kinase,RPK)是一大类跨膜蛋白的总称,因其与细胞外信号结合且具有激酶活性而得名。在植物中,由于大多数该类激酶的受体活性尚未经证实,因此也被称为类受体蛋白激酶(Receptor-like protein kinase,RLK)[4-5],首次报道见于1990年[6]。植物的类受体蛋白激酶通常属于丝/苏氨酸蛋白激酶,位于细胞膜上[7],也有极少数定位于细胞质中[8]。植物RPK/RLK又可按胞外区的一级结构分为多种类型,其中,很大一部分为富含亮氨酸的RPK/RLK(Leucine-rich repeat receptor-like kinase,LRR-RLK)[9-10],称为富亮氨酸类受体蛋白激酶。
如今已开展了一些关于植物LRR-RPK基因克隆以及序列、表达模式等方面的研究[11-13],也已经开始分析其生理功能[14-16]与分子功能[17-19]。研究结果表明植物的LRR-RPK可影响生长发育、生物胁迫应答、非生物胁迫应答等多种过程,基因的表达也受到多种生物与非生物环境信号的调控,且与多种信号分子以及其它激酶发生互作,从而参与多种信号途径。这些报道体现了植物LRR-RPK具有重要且多样的功能,因此,获得编码LRR-RPK的基因,用于分析相关基因功能及其所参与代谢过程的分子机理,具有重要的研究价值与前景。甜菜LRR-RPK克隆的研究尚无报道。
本研究以前期开发的一个分子标记位点为起始材料,利用多个甜菜序列数据库,通过检索、比对、定位、电子延伸等操作,对标记位点的侧翼序列进行产物功能预测与电子克隆,直至获得侧翼序列中的编码序列,再通过巢式PCR与双向测序对电子克隆结果进行验证与修正,获得可靠的基因编码区,最后利用公共数据库对所得编码区进行生物信息学分析,预测基因的编码产物,为该基因及其类似物的功能分析奠定基础。
1 材料与方法
1.1 试验材料
植物材料为甜菜品系DP02,甜菜基因组版本为1.2.2[20],DNA聚合酶EsTaq、总DNA提取试剂盒购自哈尔滨康为世纪公司,克隆载体购自北京艾德莱公司,其它酶类与主要分子生物学试剂购自哈尔滨无限峰公司,引物合成和DNA测序由上海生工生物公司完成。
1.2 试验方法
序列库批量比对使用本地blast工具(V2.2.25),下载自NCBI网站(https://blast.ncbi.nlm.nih.gov)。少数序列比对、序列在线比对搜索均使用在线blast工具(NCBI)。序列拼接和多重比对使用DNAMAN软件(V6.0)。引物设计使用Primer Premier(V5.5)。序列在线分析使用NCBI、pfam、CBS、TMpred等。总DNA提取、总RNA提取均使用吸附柱法。cDNA通过RNA逆转录获得。
2 结果与分析
2.1 标记位点的电子克隆
基因克隆起始材料为一个已转化的序列标签位点(Sequence tag site,STS),名为BvRE051,长178 bp。首先,以BvRE051序列为种子序列,利用同一品种的转录本序列库,进行第一轮电子延伸,结果见图1。

图1 BvRE051在转录本库中的电子延伸Fig.1 In silico enlongation of BvRE051 with transcript sequence library
通过标记位点在转录本库中的检索,得到了一条长度为500 bp的转录本,序列与标记位点序列有147 bp的匹配序列,序列一致性达到100%。组装标记位点与转录本序列,得到一条长531 bp的第一轮电子延伸结果(图1a)。暂将该段延伸结果称为C531。本次得到了单向延伸,延伸方向为种子序列3'端。
利用公共数据库(包括核酸数据库与甜菜基因组数据库)分别进行blastN比对检索,对C531进行验证与第二轮延伸。结果表明标记位点所在序列有可能代表着一段LRR-RPK基因。同时,发现在延伸方向上的基因组序列与转录本序列并不完全一致。其中,一致部分长343 bp。取该段序列为第二轮延伸结果,暂称为C343。
再次通过数据库比对检索,对C343进行验证与第三轮延伸,得到的匹配序列与第二轮一致,均为2条预测的mRNA序列(图2)。这2条序列均编码LRR-RPK,与C343的匹配长度均为306 bp,匹配区域序列一致性均为100%,误差打分也相等。根据以上结果可确定C343与C531代表同一段目标序列。
以上3次电子延伸结果均为单向延伸,延伸方向为种子序列3'端。由图2可见,种子序列已向3'端得到了比较可靠的电子延伸。但由于种子序列位于编码区外侧,为获得与标记位点关系最密切的编码区,还需要将序列向反方向,即种子序列的5'端进行延伸,以保证延伸的正确性。
基因编码区域通常不大于5 kb,再观察blast结果,subject序列的编码区长约3.5 kb,远小于5 kb。因此,提取基因组序列中C343所在区域上下游各约5 kb序列。对该段序列进行开放读码框(Open reading frame,ORF)分析,确定其中是否包含其它的基因编码区。结果见图3。
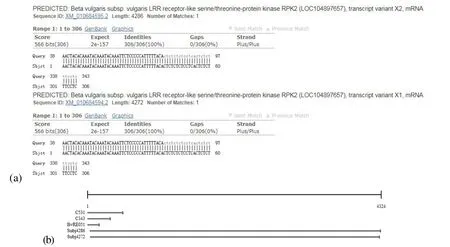
图2 C343在公共数据库中的blastN比对结果Fig.2 BlastN result of C343 in public database

图3 基因组中C343所在区域的ORF分析结果Fig.3 ORF found in genome sequence around C343
向上下游各延伸约5 kb后,仅出现了一个较完整的ORF,即图3中“ORF29”。再将ORF29的碱基序列进行blastN比对,也得到了与C343相同的结果。
将ORF29与图2a中得到的2条blast匹配序列、基因组序列、C343比对,发现匹配区域序列一致性很高。在ORF区域,两条blast结果以及基因组序列这三者完全相同,在ORF的3'端外侧约80 bp之外开始出现强烈差异。种子序列位于该ORF的5'端外侧,可能位于5'非翻译区。
根据以上延伸-ORF分析-序列比对与搜索结果,最终确定种子序列所在编码区为ORF29。该编码区长度为3 141 bp,编码产物长1 046 aa,很可能属于LRR-RPK类蛋白。
该ORF在基因组中与在mRNA中完全一致,因此,预测该目的基因在基因组中连续存在,不包含内含子区域。
2.2 电子克隆结果验证与目的片段扩增
目的序列及引物的相对位置如图4所示:根据电子克隆结果,将待验证的目的序列确定为ORF29+5'端外侧种子序列区域+3'端外侧无差异区域,总长度为3 514 bp。暂将该段序列称为T3514。尽量选取靠近T3514两端的序列设计引物,以保证其中编码区部分得到完整扩增。设计多轮巢式引物以保证扩增的特异性。

图4 待验证序列的范围及引物设计位置Fig.4 Positions of target sequence T3514 and verification primers S1,S2:senseprimers;A1-A3:antisense primers
分别以DNA和cDNA为模板,以巢式PCR对电子延伸结果进行验证。琼脂糖凝胶电泳结果(图5)表明,以2种模板进行的PCR均得到了与预期相符的目的序列。2种模板的扩增结果长度一致。
回收、克隆产物条带,取转化子进行sanger测序,结果显示所得序列长3 467 bp,延伸结果两端序列与所设计的引物序列匹配,序列与目标序列T3514有7个碱基的差异(图6)。结果符合预期目标,表明T3514得到了验证与修正。暂将该段巢式PCR产物称为N3467。利用在线工具对该序列进行ORF分析,也得到了一个完整且长度符合预期的ORF2,长3 141 bp。
将N3467中的ORF2与T3514中的ORF进行blastN、blastX比对(图6),结果表明ORF2的长度与预期相同,均为3 141 bp,编码1 046 aa的产物;二者在核苷酸序列上一致性99%,有5个碱基的差异,在氨基酸序列上一致性99%,有2个氨基酸的差异。
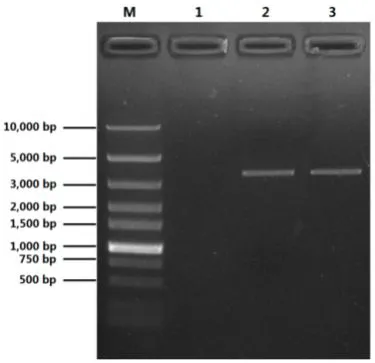
图5 电子延伸T3514的巢式PCR验证Fig.5 Verification of in silico elongated sequence T3514 by nested PCR

图6 巢式PCR产物N3467中ORF2与T3514中目标ORF的比对Fig.6 Alignments of ORF2 of N3467 from nested PCR and target ORF in T3514
再将种子序列BvRE051、验证后的延伸结果N3467分别在整个甜菜基因组序列中进行blast比对,匹配区域位于6号染色体同一区域,相对位置也与电子延伸情况一致。
以上结果表明,所得序列的ORF结构完整,编码产物及序列定位均符合预期,可以认为已获得了种子序列所在处的完整编码区。
2.3 目的片段编码产物结构与功能分析
利用在线blastN、blastX工具,在所有植物的序列库中对ORF2进行序列比对,所得序列ORF2与最高分匹配序列(两段甜菜预测LRR-RPK的mRNA)均有5个碱基、2个氨基酸的差异。除来自甜菜的预测RPK外,高分匹配序列均属于RPK/RLK2,来自藜麦(一致性84%)、菠菜(一致性84%)、糙叶山黄麻(一致性64%)等物种,序列一致性均不超过85%。
至此,通过电子延伸和常规基因克隆,获得了目的基因编码区,并推断基因产物属于LRR-RPK2类蛋白。按该基因命名习惯,将所得序列命名为BvLRR-RPK2;1。
用TMpred预测基因编码产物蛋白BvLRR-RPK2;1的跨膜结构域(图7),结果表明其中第708~730个氨基酸处有1个比较可靠的跨膜结构域,BvLRR-RPK2;1的N端位于细胞质膜外侧,C端位于质膜内侧。

图7 基因翻译产物BvLRR-RPK2;1的跨膜结构预测Fig.7 Trans-membrane structure presumption of BvLRR-RPK2;1(translated peptide)
利用Pfam在线工具对BvLRR-RPK2;1进行结构域预测(图8),并与LRR-RPK/RLK类蛋白对比,结果表明BvLRR-RPK2;1在第22~61个氨基酸与已知的亮氨酸结构域相似,第772~971个氨基酸与已知的蛋白激酶结构域相似,表明基因产物在胞外N端具有一个亮氨酸结构域,在胞内C端具有一个蛋白激酶结构域,符合RPK/RLK的结构特征。

图8 BvLRR-RPK 2;1的结构域预测Fig.8 Domains presumption of BvLRR-RPK 2;1
以上结构预测结果均与序列比对结果一致,因此可预测BvLRR-RPK2;1基因属于富亮氨酸类受体蛋白激酶类基因。
3 结论
(1)通过电子延伸与常规基因克隆,获得了一个完整的基因编码区,长3 141 bp,编码1 046 aa的产物,编码区在基因组中连续存在,无内含子。
(2)编码区与甜菜中已预测的2条编码富亮氨酸类受体蛋白激酶LRR-RPK的mRNA在ORF内部有5个碱基、2个氨基酸的差异,基因编码产物与其它物种中的LRR-RPK2类蛋白相似,且序列相似性在85%以下。将基因命名为BvLRR-RPK2;1。
(3)基因编码产物BvLRR-RPK2;1具有一个跨膜结构域,具有LRR-RPK家族特征性的亮氨酸结构域和蛋白激酶结构域。
4 讨论
本研究以一个标记位点为起始材料,结合电子克隆、序列分析、常规基因克隆,并通过多角度的结构分析与验证,再结合相似基因的研究背景,最终获得了符合预期的目的基因编码区。本研究所得基因BvLRR-RPK2;1在序列上、基因组相对位置上、产物结构上均与标记位点来源相符:基因及翻译产物与已报道LRRRPK/RLK2类基因的mRNA序列和蛋白序列有较高相似性,标记位点与编码区位于基因组同一区域且连续存在,编码区翻译产物具有LRR-RPK特征性的跨膜结构域、N端富含亮氨酸结构域、C端蛋白激酶结构域[5,21-22]。因此,可以确定该基因是一个激酶类基因,其编码产物属于富亮氨酸类受体蛋白激酶,且与所关注的标记位点有密切联系。该基因在后续研究中可应用于两方面,一是针对基因本身进行功能研究与调控机制研究,二是与起始标记结合对性状进行鉴定,并分析该基因对性状的作用。
我国的甜菜基因资源挖掘工作远远落后于大部分主要作物,这可能与甜菜的育种历程、种质资源丰富程度、性状检测难度等客观背景有关,也可能与甜菜自交不亲和、基因位点杂合度高、序列的获取易受干扰有关。为获取目的基因,本研究采用了电子克隆与常规扩增相结合的技术方案。电子克隆是基于后基因组时代序列信息大量丰富而产生的基因克隆技术,在序列延伸与验证方面都具有明显的优势。对于甜菜而言,较薄弱的研究基础限制了电子克隆可利用的序列材料,尤其是mRNA序列材料。但是,利用已公布的甜菜基因组,甜菜的基因资源挖掘有了新的可能性,即本研究所采用的技术路线:借助基因组序列进行电子克隆,随后通过生物学实验进行延伸结果验证与序列获取,同时,借助前期研究基础,对目的基因类型、功能、位置等信息进行预判,最后再利用基因组序列确定所得序列与研究目的是否相符、排除基因组其它区域相近序列的干扰。采用该方案进行基因克隆,可在一定程度上节约目的片段延伸的工作量,排除结果验证时可能的干扰,间接地降低在选择种质、选择目的基因、验证克隆结果时受到干扰的风险。
然而,该电子克隆方案仍然不适合用于所有类型基因的克隆。一方面,甜菜电子克隆可用的参考序列数目还相当有限。在本研究中,可应用的mRNA序列仅限于公共数据库中的2条预测序列与1条自主测序所得序列,电子克隆前期也出现了多种延伸结果,这些结果因参考序列的来源和质量而异,并对克隆工作造成了较大的干扰,编码区外侧的非翻译区序列也因参考序列质量不高而暂时无法克隆。另一方面,甜菜许多关键代谢过程的分子机理并不够清楚,使得目的基因的选择与验证可用的参考信息不足。本研究由于起始序列来自于前期研究,与性状有一定相关性,能够在克隆过程中,根据基因在其它物种中的研究结果,评估基因与性状的相关性,才对电子克隆结果成功地进行了判断。相应地,若前期研究基础不足且种子序列较短,则很难通过电子延伸获得可靠的结果。
此外,即使参考序列充足,电子克隆结果也必须要通过生物学实验进行验证,才能克隆出可靠的基因片段并确定基因序列。电子克隆参考序列的来源比较多样,且目的序列在品种之间的保守性也无法预料,若直接以电子克隆结果作为目的基因序列进行后续操作,序列差异很可能干扰方案的设计与实施,甚至导致载体构建等后续工作无法完成。本研究在电子克隆结果验证中,使用了标记位点来源材料,以确保植物材料中包含目的基因,并设计了巢式PCR引物,以排除非特异扩增与低保守性引物序列的干扰,最大限度地避免了以上风险,并及时修正了7 bp的序列差异。
总之,基因电子克隆的难度、工作量及结果可靠性与参考序列的质量、数量密切相关,且需要足够的前期研究基础作为辅助,并需要通过生物学实验进行验证,才能获得确定的、可靠的目的基因片段。
目前,甜菜已发表的EST、mRNA、蛋白质序列均不够丰富,基因组数据也刚刚发布并只更新了一次,还有必要获取数量更多、种类更丰富、序列更可靠、来源更多样的甜菜序列信息,以辅助甜菜的电子克隆以及其它的生物信息学分析。在甜菜序列信息更丰富或基因组序列更可靠之后,也有望借助这些序列,对已完成克隆的各个编码区,包括本研究所得的BvLRR-RPK2;1基因,进行进一步的克隆、基因结构预测、调控元件分析等研究。

