真核微藻糖转运蛋白基因的比较基因组学分析
刘亚铭, 陈 高, 秦 松, 崔玉琳, 崔红利
(1. 中国科学院 烟台海岸带研究所 海岸带生物学与生物资源利用重点实验室, 烟台 264003;2. 中国科学院大学, 北京 101418; 3. 山东省农业科学院 生物技术研究中心, 济南 250100;4. 山东省作物遗传改良与生态生理重点实验室, 济南 250100;5. 山西农业大学 分子农业与生物能源研究所, 太谷 030801)
糖类物质是地球上生物最重要、最基本的能量来源,细胞摄取糖类物质是生物体生长、代谢和稳态至关重要的一个过程。而糖类物质自身无法进入细胞内发挥作用,必须依靠转运蛋白这种“运输机器”,因此研究糖转运蛋白对了解糖的吸收、代谢方面具有重要的意义。糖转运蛋白是一类位于细胞膜上的重要载体蛋白,可以将胞外的糖类物质转运至胞内供细胞代谢利用,其广泛分布于真核微藻及高等动植物中,其中真核微藻中的糖转运蛋白属于MFS (major facilitator superfamily)转运蛋白家族[1]。依据转运糖的类型划分,真核微藻中的糖转运蛋白属于单糖转运蛋白;依据转运蛋白转运方式的划分,而真核微藻中的糖转运蛋白属于次级主动转运蛋白中的同向转运蛋白[2]。糖转运蛋白大多由400~600个氨基酸残基组成, 二级结构预测糖转运蛋白大多具有12次跨膜α-螺旋结构域,这种结构被命名为“MFS fold”, 12次α螺旋又分为2个结构域: N端结构域和C端结构域,每个结构域都由6个α-螺旋组成, 虽然2个结构域中氨基酸序列同源性很低, 但从结构上观察2个结构域呈现二次对称[2]。
糖类被植物利用的研究始于真核微藻,Tanner等证明绿藻有利用葡萄糖的能力; Sauer和Tanner(1989)发现了植物的第一个糖转运蛋白基因——源于小球藻的HUP1(H+/hexose cotransporter 1),并证明HUP1编码的蛋白为单糖-H+同向转运蛋白(monosaccharide-H+symptor)[3];对HUP1的氨基酸序列分析后发现与哺乳动物[4]、酵母[5]和细菌[6]中的单糖-H+同向转运蛋白具有很高的同源性。之后在此基础上以HUP1序列为探针,对一些高等植物的基因组文库进行了筛选, 在拟南芥和烟草中找到与HUP1高度同源的单糖转运蛋白基因[7-8]。
小球藻中的单糖转运蛋白家族分为HUP1、HUP2和HUP3。其中HUP1蛋白主要转运葡萄糖,为葡萄糖-H+同向共转运蛋白[9];HUP2蛋白主要转运半乳糖,是半乳糖-H+同向共转运蛋白[7, 10];而HUP3蛋白虽然转运葡萄糖,但转运量极少,也是葡萄糖-H+同向共转运蛋白[9]。
真核微藻属于一类古老的植物,具有复杂的进化历史和广泛的分布,是逆境生理研究理想的实验材料,目前,已经完成和正在完成基因组测序的真核微藻有28株,大量真核微藻基因组数据的释放为比较基因组学研究提供了强有力的支持。作者选取已经被功能鉴定的糖转运蛋白序列作为源序列, 利用同源比对的方法对真核微藻基因组序列进行搜索, 发现编码糖转运蛋白的基因序列广泛存在。本实验建立了微藻糖转运蛋白基因-结构-功能的框架结构,为之后探讨利用外源葡萄糖机制提供参考,同时为后期利用代谢工程手段构建优良藻株奠定基础。
1 材料与方法
从NCBI的数据库和JGI Genome Portal的数据库中下载28株真核微藻的Fasta格式的全基因组氨基酸数据库,包括Chlamydomonasreinhardtii,Chlorellasp. NC64A,Chlorellavulgaris,Coccomyxasp. C-169,Volvoxcarteri,Micromonaspusilla,Micromonassp. RCC299,Ostreococcussp. RCC809,Ostreococcustauri,Ostreococcuslucimarinus,Phaeodactylumtricornutum,Thalassiosirapseudonana,Fragilariopsiscylindrus,Aureococcusanophagefferens,Auxenochlorellaprotothecoides,Emillianiahuxleyi,Guillardiatheta,BigelowiellanatansCCMP2755,Monoraphidiumneglectum,Pseudo-nitzschiamultiseriesCLN-47,Thalassiosiraoceanic,Galdieriasulphuraria,PorphyridiumcruentumCCMP1328,Nannochloropsisgaditana,AurantiochytriumlimacinumATCC MYA-1381,Symbiodiniumminutum,Chrysochromulinasp. CCMP291,Klebsormidiumflaccidum。原始红藻(Cyanidioschyzonmerolae)的全基因组氨基酸数据从原始红藻基因组计划网页(http:merolae.biol.s.u-tokyo.ac.jp/)下载。用于本地blast搜索的糖转运蛋白基因是根据文献报道的已经进行功能验证的小球藻(chlorellakessleri)的HUP1、HUP2和HUP3(表1)。
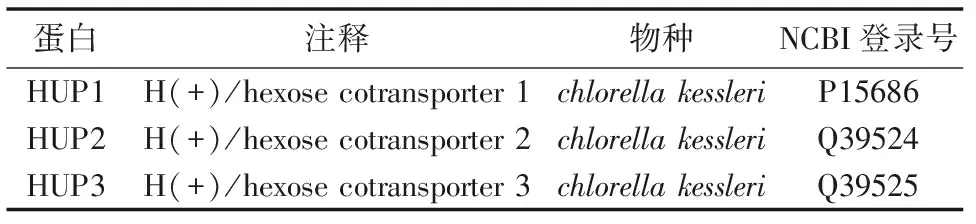
表1 作为源序列的功能已知的糖转运蛋白基因
将29株真核微藻基因组数据分别建立一个本地数据库,以HUP1、HUP2和HUP3基因作为源序列,利用BLASTP程序搜索29株真核微藻的基因组数据库。为了排除假阴性,采用HMMER程序进行目的基因的重新搜索。对两种方法获得目的基因序列合并和分析,删除重复序列。为了排除目的序列中的假阳性,采用SMART,Pfam和CDD软件进行蛋白结构域分析。
采用Clustal X2将搜索出来的目的蛋白序列进行了对比和人工调整。建树模型的选择采用Protest软件,LG模型被选择进行系统进化树的构建,采用MEGA6软件最大似然法构建系统进化树。采用Pfam, SMART及CDD等软件进行结构域分析,采用TMHMM进行跨膜区预测,亚细胞定位预测和分析采用TargetP。
2 结果与分析
2.1 真核微藻类糖转运蛋白的种类和分布
从29株真核微藻基因组中我们发现了9个HUP1,39个HUP2和22个HUP3类型的糖转运蛋白基因。HUP的种类数量在不同真核微藻基因组中的分布是不一样的。
在糖转运蛋白基因家族中,HUP1的同源基因分布于9种藻株中,包括绿藻门(C.reinhardtii,M.pusilla,C.sp. C-169,C.variabilis,C. sp. NC64A,O. sp. RCC809)、不定鞭藻门(A.anophagefferens,A.limacinumATCC MYA-1381)和丝足虫门(B.natansCCMP2755)。HUP2的同源基因广泛分布于所有的硅藻门, 不定鞭藻门及定鞭藻门中,但是在其他藻株中缺失了,包括绿藻门(C.reinhardtii,O.tauri,O. sp. RCC809)、红藻门(P.cruentumCCMP1328)、定甲藻门(S.minutum)和隐藻门(G.theta)。HUP3的同源基因仅分布于8种藻株中,包括绿藻门(C.variabilis,C. sp. NC64A,M.sp.RCC299,O.tauri,O.sp. RCC809)、红藻门(P.cruentumCCMP1328,G.sulphuraria), 硅藻门(P.multiseriesCLN-47,F.cylindrus) 、不定鞭藻门(A.limacinumATCC MYA-1381)
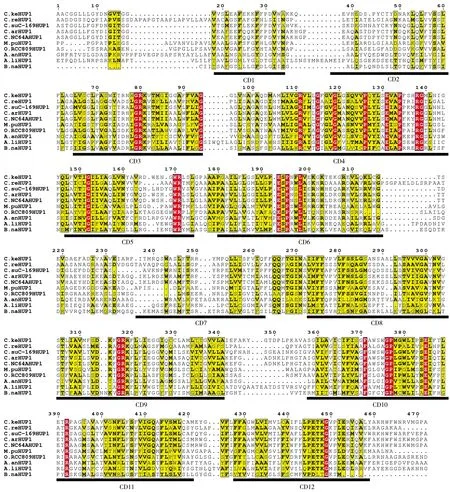
图1真核微藻中糖转运蛋白的HUP1结构域分析
Figure 1 HUP1 Domain structure of sugar transporter from algae
有些微藻基因组中发现了基因复制现象,如HUP2基因在淡水绿藻(C.subellipsoideaC-169 NIES2166,C.variabilis,C.sp.NC64A)和海洋绿藻(A.protothecoides)的基因组中有多个拷贝,说明在这个基因的进化过程中发生了物种特异性的基因复制现象。与之相类似,HUP3基因在一些藻株的基因组中具有两个或者更多拷贝,尤其是杂色藻株中,说明也存在基因复制现象。
2.2 糖转运蛋白的结构域分析
为了进一步研究真核微藻来源的糖转运蛋白,将糖转运蛋白基因进行比对分析,发现共有12个非常保守的结构域存在于所有的真核微藻来源的糖转运蛋白中(图1)。来源于不同类型藻株的HUP1具有高度相似的结构域,说明这个基因的出现早于不同类型的藻株的形成之前。HUP家族起转运作用的保守结构域存在于所有的HUP序列中,如CD1、CD3、CD6、CD8、CD9、CD10、CD11、CD12为跨膜螺旋结构TMS(transmembrane-spanner), CD2、CD4、CD9为结合糖类物质的结构SP (sugar porter) ,CD5和CD7为糖转运结构Sugar_tr(Sugar transporter),这些结构域在糖转运蛋白序列中高度相似,暗示具有共同的起源。
2.3 真核微藻糖转运蛋白基因的起源和系统进化
为了研究糖转运蛋白基因的起源和系统进化历史,来源真核微藻的 HUP1类型、HUP2类型和HUP3类型的氨基酸序列进行系统进化分析(图2)。本研究采用的真核微藻以外的糖转运蛋白基因总结见表2。
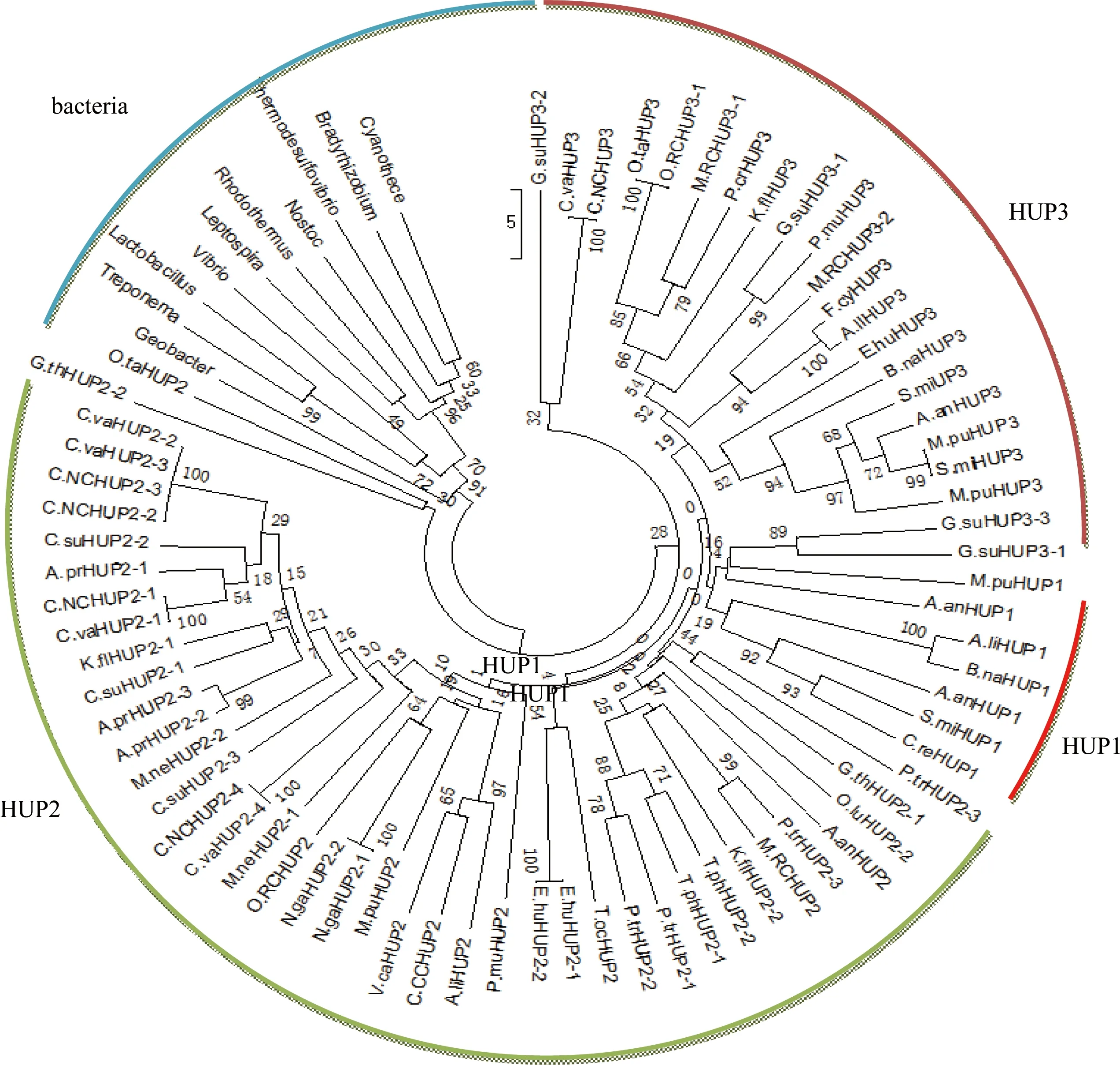
图2真核微藻糖转运蛋白的系统进化树
Figure 2 A maximum likelihood tree of our sugar transporter database and some other sugar transporter from bacteria
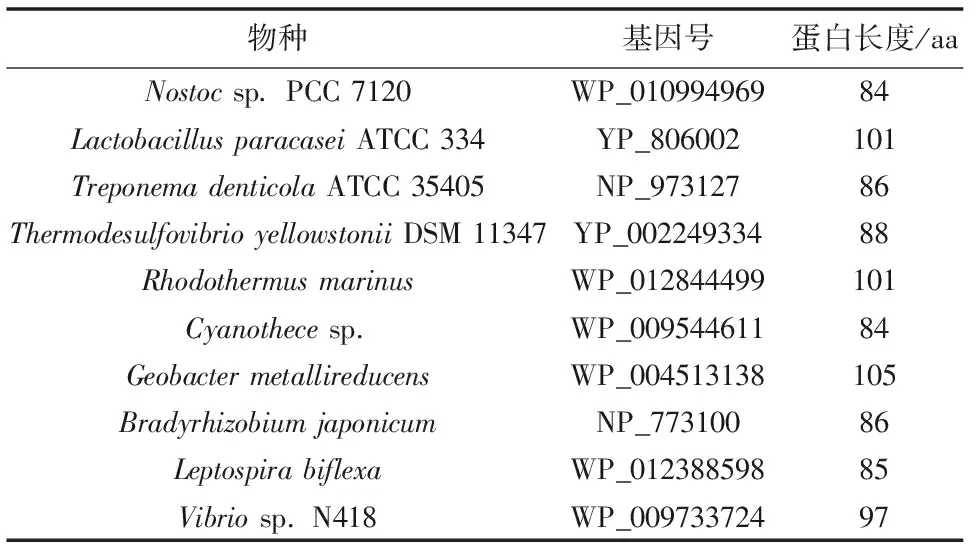
表2 系统进化树中采用的真核微藻以外生物来源的糖运蛋白基因
真核微藻的糖转运蛋白基因分成3个单系群,即为HUP1、HUP2和HUP3。来源于所有藻株编码的HUP2基因形成了单系群,这种进化方式说明HUP2基因出现是在藻株多样性形成之前。从系统进化树可以看出,与编码HUP1和HUP3的基因相比较,编码HUP2的基因是一个更为古老的基因。编码HUP2和HUP3的基因在进化上比较相近,可能是由HUP2的基因复制而来,在之后的进化过程中分化形成不同的功能。来源细菌、真核微藻的糖转运蛋白形成一个单系群(BS:91%),这种进化关系说明真核微藻的糖转运蛋白基因起源于细菌。
3 讨论
3种不同类型的糖转运蛋白(HUP1、HUP2和HUP3)在真核微藻基因组中都有发现,但是不同的藻株含有的种类和数量不一致,揭示糖转运蛋白基因在这些藻株的后续进化历史中经历了不同的过程。在真核微藻的进化过程中基因复制,丢失及基因转移是常见的方式。
预测的HUP1的同源基因仅分布于8种藻株中,包括绿藻门(C.reinhardtii,M.pusilla,O.lucimarinus,O. sp. RCC809)、不定鞭藻门(A.anophagefferens,A.limacinumATCC MYA-1381)、丝足虫门(B.natansCCMP2755)和甲藻门(S.minutum)。在真核微藻和高等植物中,HUP1类型的糖转运蛋白长久以来被认为是唯一的参与糖转运的蛋白[10]。编码 HUP1的基因已经从很多生物中进行了基因克隆和功能验证,包括莱茵衣藻[11]及大肠杆菌[12],不仅如此小球藻HUP1表达的蛋白纯化以及其体外重建已经完成[13-14]。
我们的实验发现真核微藻的糖转运蛋白基因起源于细菌起源,糖转运蛋白帮助底物完成跨膜运输的过程需要线粒体供能,这与真核微藻中的线粒体起源于细菌的理论相符合[15]。
HUP2的基因是比较古老的基因,它的出现早于现有的不同类型的真核微藻多样性形成之前。编码HUP1和HUP3的基因是通过基因复制获得,在进化过程中受到特殊自然选择压力而进行了功能分化。在进化过程中,基因复制及水平转移现象在糖转运蛋白基因进化过程中发挥重要作用。尽管预测出来的糖转运蛋白基因大多数没有进行基因克隆和功能验证,但本研究初步建立了真核微藻糖转运蛋白的序列-种类-分布-结构-起源-进化的框架结构,为进一步研究糖转运蛋白的功能奠定了基础。

