基于U-Net改 进模型的直肠肿瘤分割方法
高海军 ,曾祥银 ,潘大志 ,3,郑伯川 ,3*
(1. 西华师范大学数学与信息学院,四川南充 637009; 2. 西华师范大学计算机学院,四川南充 637009;3. 西华师范大学计算方法与应用软件研究所,四川 南充637009)
0 引言
直肠癌是指从齿状线至直肠乙状结肠交界处之间的恶性肿瘤,是消化道最常见的恶性肿瘤之一[1]。根据2019 年中国癌症统计,直肠癌占癌症发病总数的38.8%,高居第二位[1],是导致死亡的主要癌症之一。直肠癌也是全球癌症死亡率较高的常见癌症[2],每年有许多癌症患者死于直肠癌。幸运的是,直肠癌的病情通常发展缓慢,如果在直肠肿瘤发展为癌症之前发现并将其切除,则可以预防直肠癌[3]。因此,直肠肿瘤的定位与识别是直肠癌诊断和治疗的基础和关键。一般情况下,医生通过观看患者的计算机断层扫描(Computed Tomography,CT)影像图,诊断直肠部分是否有肿瘤和潜在的直肠癌,如有则标出直肠肿瘤区域以便实施手术。直肠肿瘤在CT影像图中特征不明显,需要经验丰富的医生才能准确识别并标定出肿瘤区域。如果医生经验不足或者诊断不够认真仔细都可能导致对直肠肿瘤的漏诊或者误诊。因此,有必要对直肠肿瘤自动分割技术开展算法研究。直肠肿瘤自动分割技术可以辅助医生诊断,降低医生的劳动强度,提高直肠癌诊断准确率,为患者治疗争取宝贵时间。
近年来,随着人工智能技术的发展,图像分割越来越多地采用深度学习技术,取得了优于传统分割技术的分割效果。2015 年,Ronneberger 等[4]提出的 U-Net 网络模型采用编码器-译码器结构,编码器部分通过卷积和逐级下采样提取图像层级的特征对输入图像进行编码;解码器通过卷积和逐级上采样将编码信号映射成相应的二值分割掩模,得到分割结果。受U-Net模型启发,许多研究者对U-Net网络进行了改进。文献[5]中提出了U-Net++模型,通过增加编码器和解码器之间的细粒度信息来重新设计跳跃连接;文献[6]中提出的R2U-Net 用反卷积替换U-Net 中的卷积块;文献[7]中将注意力机制融入到U-Net 模型中,可以决定编码器到解码器的跳跃连接中传播什么信息;文献[8]中使用密集卷积块和双向卷积长短期记忆模型将最有效的特性从编码器传播到解码器;文献[9]中则采用双层编码器到解码器结构,同时使用卷积自编码注意力机制,可对细粒度图像进行分割。这些基于U-Net模型的改进都在一定程度上提升了对特定图像的分割性能。
医学图像分割作为图像分割的一类,广泛采用最新的图像分割技术。受残差网络(Residual Network,ResNet)[10]的启发,文献[11]中提出了H-DenseUNet 模型用于肝脏肿瘤CT 图像的分割,解决了二维卷积不能充分利用三维空间信息、卷积计算成本高以及GPU 内存消耗大的问题。文献[12]中提出的Y-Net 可用于对直肠息肉进行分割检测。Y-Net 也是基于U-Net结构的,不同的是Y-Net比U-Net多一个编码器,其中一个编码器使用预训练好的VGG19 权值。在直肠息肉分割中,Y-Net分割效果要优于U-Net。文献[13]中提出了一种注意力机制的残差网络模块FocusNetAlpha,并将其嵌入到U-Net 结构中,形成新的网络模型。该模型能记住更多的息肉区域的图像特征,输出更好的息肉区域上下语义信息。关于图像分割和检测的深度学习回顾,请参阅文献[14-16]。
直肠肿瘤分割方法主要分为两大类:传统的分割方法[17-19]和神经网络算法[20-22]。文献[17]中提出了一种基于超像素的分割算法,但该算法无法自动分割,需要人机交互。文献[18]中提出了一种基于区域生长的直肠癌肿瘤分割算法,但该算法在分割过程中需要人为给出肿瘤区域的强度信号,整个过程也需要人机交互,而且容易造成分割过度。文献[19]中提出了一种直肠癌肿瘤水平集的分割算法,该算法对边界不敏感,当边界存在模糊情形时的分割效果不好。文献[20]中提出了基于全卷积神经网络(Fully Convolutional Network,FCN)的直肠癌肿瘤磁共振影像自动分割方法。该方法主要应用一个预训练好的ResNet50 提取特征,在网络隐藏层添加3 个边输出模块,实现图像数据的多尺度特征提取,最后融合3 个边输出模块获得最终的分割结果。文献[21]中提出了一种基于改进FCN 的肝脏CT 图像肿瘤分割方法,在FCN 中进行多尺度卷积层融合,能解决传统FCN 分割精度不佳的问题。文献[22]中提出了一种基于卷积神经网络(Convolutional Neural Network,CNN)来实现直肠肿瘤区域分割的算法,但耗时较长且无法兼顾图像的局部信息和全局信息。文献[23]中提出了一种类似2D U-Net模型分割直肠肿瘤的算法,通过利用图像的3D 信息,以5 通道张量作为网络输入,采用两阶段训练策略来提升训练效率。
虽然U-Net、Y-Net和FocusNetAlpha 模型在肝脏肿瘤分割和直肠息肉分割中取得了不错的分割效果,而且将三种模型直接用于直肠肿瘤分割时,体积大且特征明显的肿瘤块也能被较好地分割,但细小的肿瘤块分割却不理想。对于分割精度来说,以上模型对直肠肿瘤区域的分割精度还有待提升。本文针对直肠肿瘤区域分割,提出一种U-Net 改进模型,其主要改进在于在编码器的每级特征提取模块中嵌入一个同时进行上下采样的子编码模块,以提升网络对目标区域的特征表达能力。另外还通过对比实验选择最适合的优化器训练模型,通过数据扩充增加数据样本来提升模型性能。本文将改进的U-Net 模型称作带子编码块的U 型网络(U Network with Sub Coding Block,U-Net-SCB)。为了评价分割模型对小目标的分割性能,本文借鉴COCO 数据集[24]中对小目标的定义,将小于等于32个像素的直肠癌肿瘤区域看作小目标。
1 U-Net-SCB网络模型
U-Net-SCB 模型是基于 U-Net[4]的改进模型,主要改进是将每个层的编码部分用1 个子编码模块替换,以提高其对小目标的特征表达能力,从而提高对直肠肿瘤区域的分割性能。
1.1 U-Net-SCB结构
U-Net-SCB模型整体呈U型结构,如图1所示。
U-Net-SCB模型的每一层都由一对编码器和解码器构成,都有相同大小的特征图,特征图的通道数随特征图尺度变小而增大;每层解码器接受下层解码器的上采样特征和同层编码器输出特征的合并特征,合并特征再经过2 个3×3 卷积和1个1×1 卷积后上采样输出到上层解码器;每层编码器除了向同层解码器输出特征外,还向下层编码器下采样输出特征;最上层解码器输出分割结果。改进主要包括:1)在1~4 层的编码器中,每层用1 个子编码模块替换原来的多层卷积操作(见图1 中SCB1~SCB4);2)解码器部分每层比U-Net 结构多一个卷积操作。子编码块具体操作如图1 中SCB 虚线框所示,子编码模块开始经过3 个卷积层,然后分成两个分支:上分支由上采样操作,2 个卷积操作之后再作最大池化操作;下分支通过最大池化操作,2 个卷积操作之后再作上采样操作;最后合并后进行3个卷积操作。
可以看到U-Net-SCB 模型加深了编码器部分的网络深度。子编码模块通过上下采样进一步放大混合了图像中的细节特征,使模型U-Net-SCB 对直肠肿瘤的分割效果更好,特别是对小目标的分割效果更好。
1.2 损失函数
本文采用文献[12]中的损失函数。设P为算法得到的分割图中所有像素构成的集合,G为真实的标注掩模图中所有像素构成的集合。定义损失函数为:
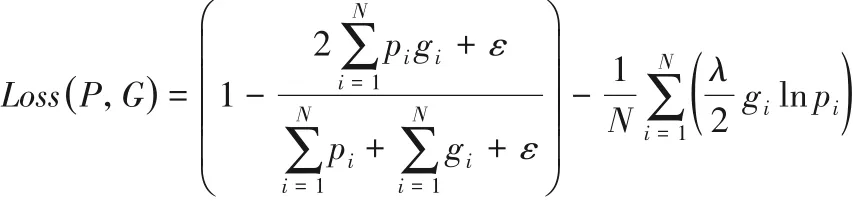
其中:第一项主要计算Dice 系数;第二项则是计算二分类交叉熵;pi∈{0,1}为算法得到的分割图中像素点i的值;gi∈ {0,1}为标注掩膜图中像素点i的值;N是分割图和标注掩模图中的像素点总个数;λ为惩罚系数;ε是光滑算子,用于避免损失函数中分母为0。
2 实验与分析
本文的模型在TensorFlow 和Keras 框架下实现,硬件环境 :CPU 为 Inter Xeon 4114,2 个 CPU,2.20 GHz,GPU 为NVIDIA P6000。软件环境:Unbuntu18.0、TensorFlow、Keras、Python 3.7。首先通过对比实验选择最适合的优化器,然后用选定的优化器训练U-Net-SCB 得到训练模型。最后将U-Net-SCB和其他三种网络结构进行性能对比测试。
2.1 数据集
本文采用第七届“泰迪杯”数据挖掘挑战赛B 题的数据[1]。直肠肿瘤患者的影像数据可分为动脉期和门脉期两种,动脉期是患者在造影注射25~30 s 时间拍摄,而门脉期是在患者造影注射60 s 后拍摄,两者在肿瘤区域和周围的组织的强度存在差异。在实际的肿瘤分割任务中,只需分割直肠肿瘤区域,故将两种数据放在一起进行训练。由CT影像和标注的直肠肿瘤区域的掩模作为数据训练样本进行训练。由于CT 图像是连续断层扫描图像,故图像中有些含有直肠肿瘤区域,有些则没有。训练集包含1 693 张带有标注的图像,每张图像都包含直肠肿瘤区域;测试集包含104 张带有标注的图像,其中102 张图像包含直肠肿瘤区域,剩余2 张不包含。所有训练和测试图像大小统一为256 × 256,并进行归一化处理。
2.2 评价指标
为了评价算法的分割性能,实验中主要采用Dice 系数CDice、查准率P和查全率R三个指标进行定量评价[18]。指标的计算公式分别如下:
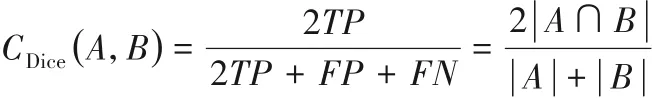
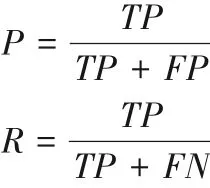
其中:A是标注的直肠肿瘤区域像素构成的集合;B是分割得到的直肠肿瘤区域像素构成的集合;TP表示被正确检测为正样本的像素数量;FP表示被错误检测为正样本的像素数量;FN表示被错误检测为负样本的像素数量。Dice 系数的取值范围是[0,1],Dice值越大说明算法分割的结果与标注的结果越接近,分割效果越好。如果查全率R过低,表示较多的直肠肿瘤区域没有被分割出来;如果查准率P太低,说明分割不够精确。
2.3 优化器选择
实验发现U-Net-SCB 模型的分割性能和优化器的选择有关,因此为了获得最适合的优化器,得到最好的分割性能,本文对比实验了 U-Net-SCB 模型分别采用 diffGrad[25]、SGDM[26]、AdaDelta[27]、AdaGrad[28]、RMSProp[29]、AMSGrad[30]和 Adam[31]优化器之后的分割性能。实验发现,当采用优化器AdaDelta、RMSProp 和 AMSGrad 时,训练 50 次之后,Dice 系数仍然低于20%,且不能收敛。U-Net-SCB 模型采用其他4 种优化器后,分别得到的性能评价指标值如表1 所示。可以看出,U-Net-SCB 模型采用Adam 优化器后,其整体性能最好,因此本文的U-Net-SCB模型采用Adam优化器进行训练。
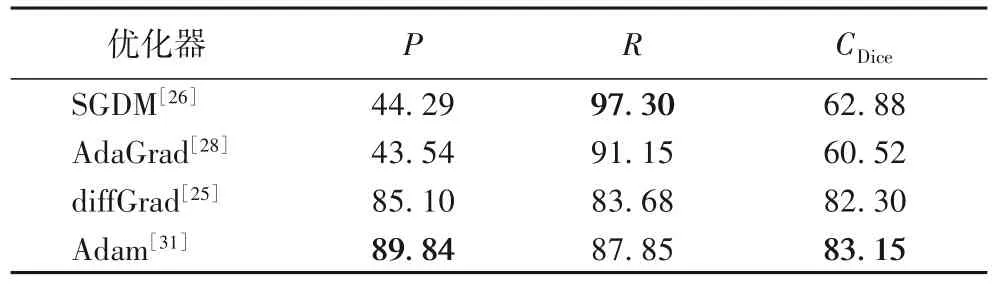
表1 不同优化器性能指标对比 单位:%Tab. 1 Comparison of performance indicators of different optimizers unit:%
2.4 数据扩充
考虑到训练集数据量有限,实验中对训练集进行数据扩充,主要包括:10°~350°范围内随机旋转图像;随机颜色扰动形成新的图像;在x方向和y方向-10~10 像素范围内随机裁剪图像。数据扩充后,训练集由原来的1 693 张扩充到8 465张。图2(a)的3 张原图经过数据扩充后得到的图2(b)~(d)所示。
为了对比数据扩充对U-Net-SCB 模型性能的影响,分别将原始训练集与数据扩充后的训练集训练U-Net-SCB,然后测试分割性能,性能对比结果如表2 所示。表2 表明,数据扩充提升了U-Net-SCB的分割性能。
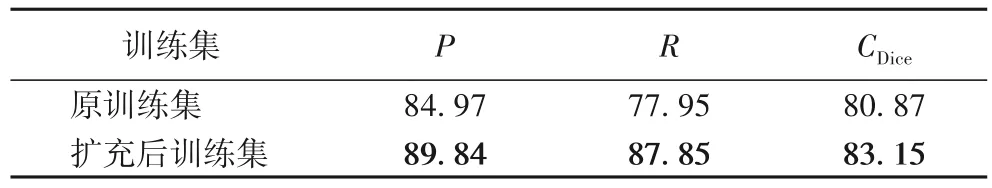
表2 数据扩充前后性能指标对比 单位:%Tab. 2 Comparison of performance indicators before and after data augmentation unit:%
2.5 模型训练
训练模型时将训练集的20%作为验证集,剩下的80%继续作为训练集,因此训练集、验证集和测试集在数据扩充前后包含的图像数量如表3,其中测试集未做任何数据扩充处理。训练时设置batchsize 为16,学习率η为0.000 01,光滑算子ε为10-4,训练50轮。
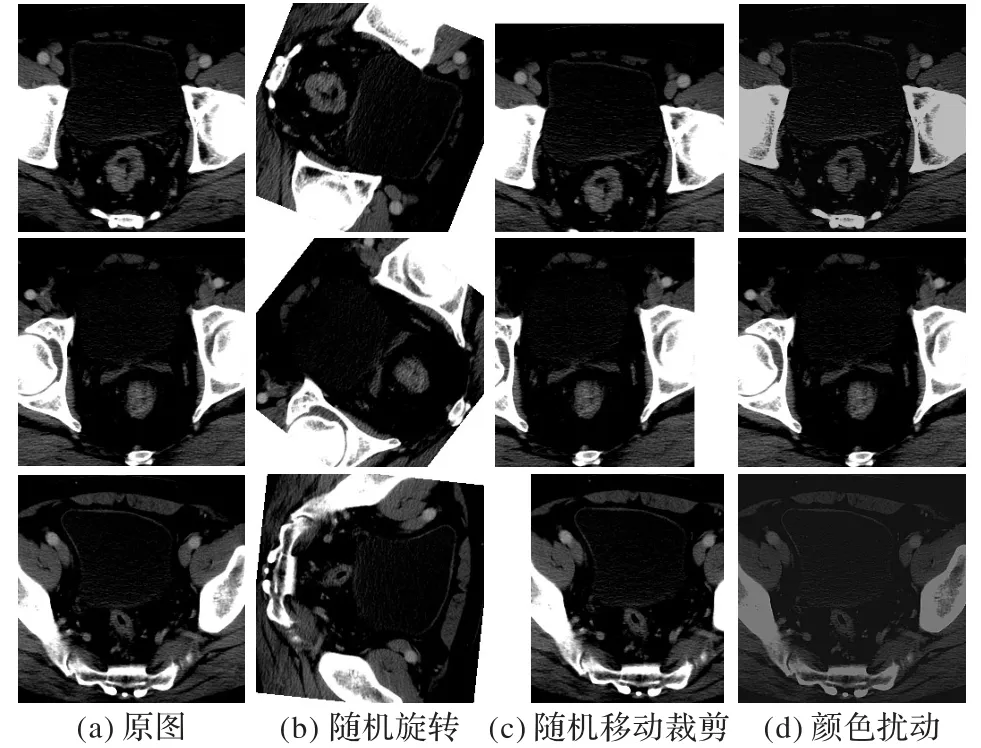
图2 数据扩充的部分结果Fig. 2 Some results of data augmentation
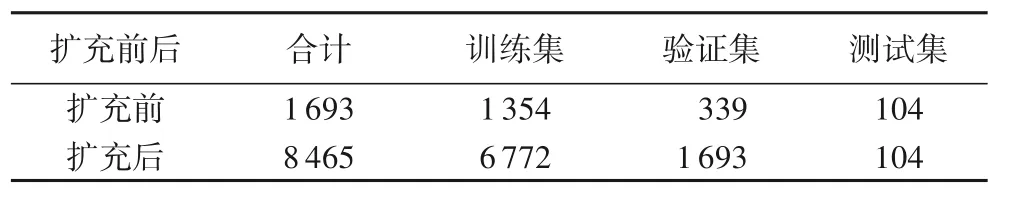
表3 训练集、验证集和测试集的大小Tab. 3 Sizes of training set,validation set and test set
数据扩充后,U-Net-SCB 模型训练时的Dice 系数随训练次数的变化曲线如图3(a)所示,训练集和验证集的损失函数随训练次数的变化曲线如图3(b)所示。可以看出,训练50轮时,损失函数已经收敛,收敛时训练集和验证集的Dice 系数都达到99%以上。
2.6 测试结果
为了验证 U-Net-SCB 的分割性能,将 U-Net-SCB 与 U-Net、Y-Net和FocusNetAlpha 三种网络模型的分割性能进行对比实验,部分结果如图4、5所示。
图4 展示的是对小目标的分割效果,可以看出U-Net-SCB对小目标的分割与标注图更接近,优于其他三种模型;图5 展示的是对直肠肿瘤分割的细节对比,红色实线框(大矩形框)部分是虚线框部分的放大图,通过放大图可以看出,相对其他三种模型,U-Net-SCB模型分割得到的直肠肿瘤区域的边缘轮廓形状与标注图更接近。另外从每张分割图下方的Dice 系数也可以看出,U-Net-SCB 模型所得分割图的Dice 系数最高,说明U-Net-SCB模型得到的肿瘤区域与标注图更接近。
实验得到的4 种网络模型的评价指标值如表4 所有目标列所示。由表4 可以看出,U-Net-SCB 模型的3 个指标值都优于其他三种模型,其中P比最好的提高了8.62个百分点,R比最好的提高了8.49个百分点,CDice比最好的提高了2.55个百分点。
为了进一步验证对小目标的分割性能,从测试集中提取包含直肠肿瘤小目标的CT图像28张,对比4种分割网络模型对这28 张图中的小目标的分割性能,对比结果如表4 中的小目标列所示。可以发现,相较其他三种模型,U-Net-SCB 模型对小目标具有更好的查准率、查全率和Dice 系数,对小目标的分割性能更突出。
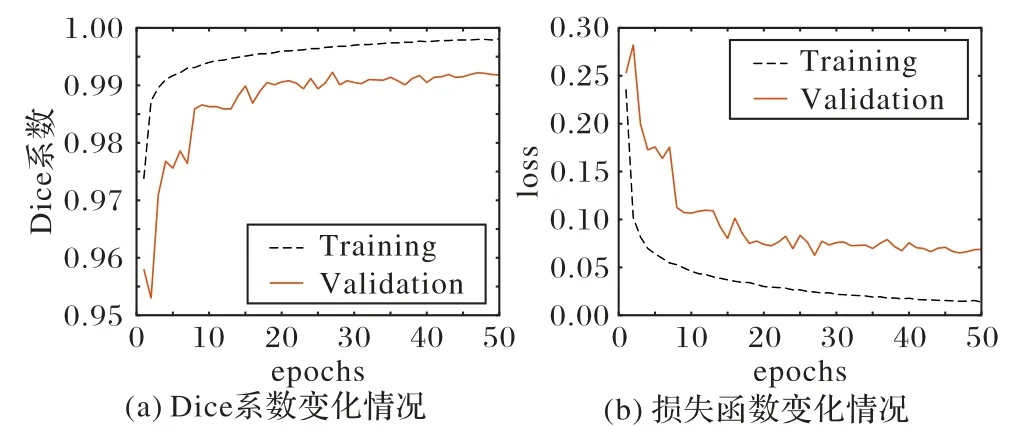
图3 Dice系数和损失函数随训练次数的变化情况Fig.3 Dice coefficient and loss function varying with epochs

图4 各对比模型的小目标分割结果Fig. 4 Segmentation results for small objects of different models
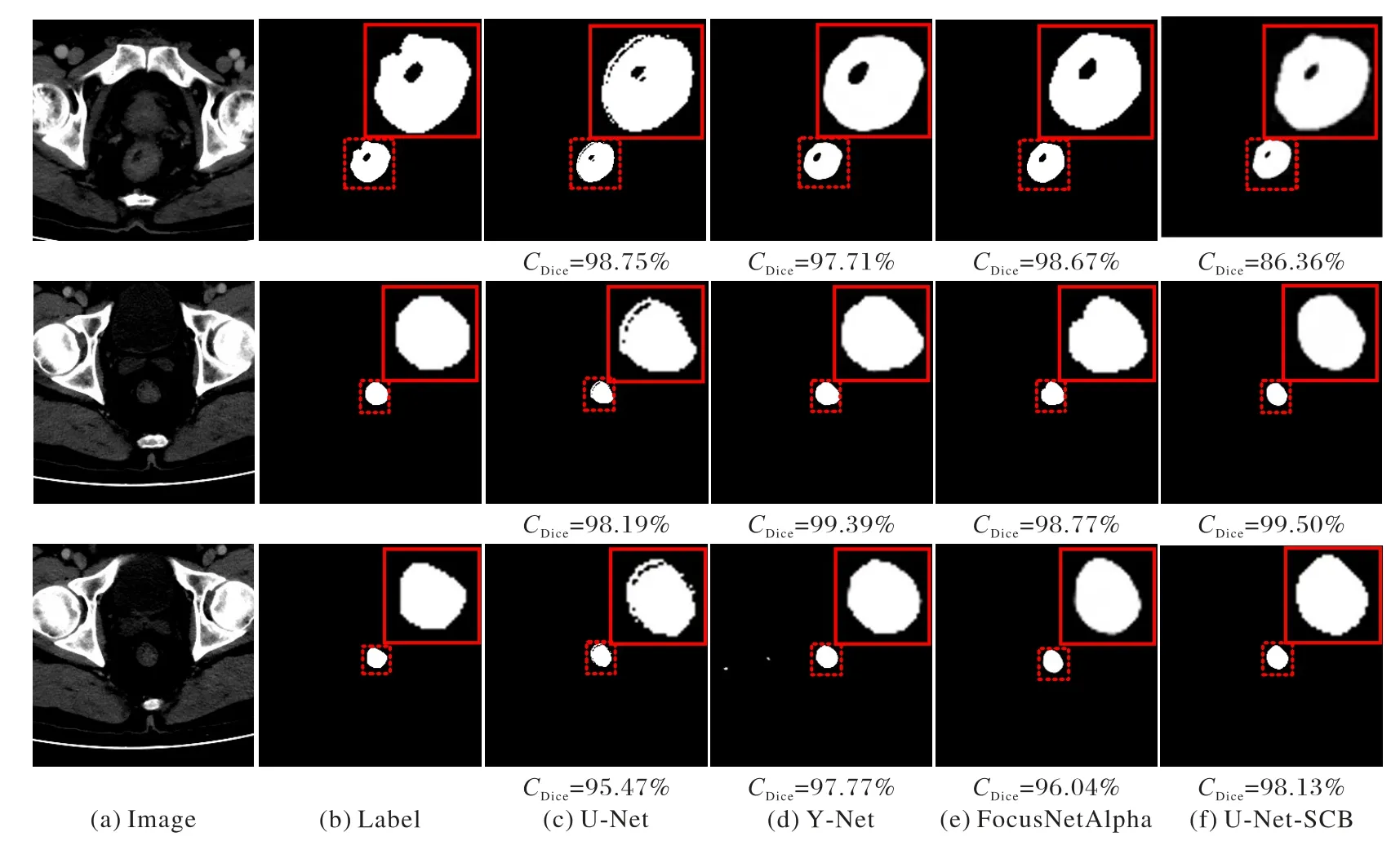
图5 各对比模型的分割细节对比Fig. 5 Segmentation details of different models
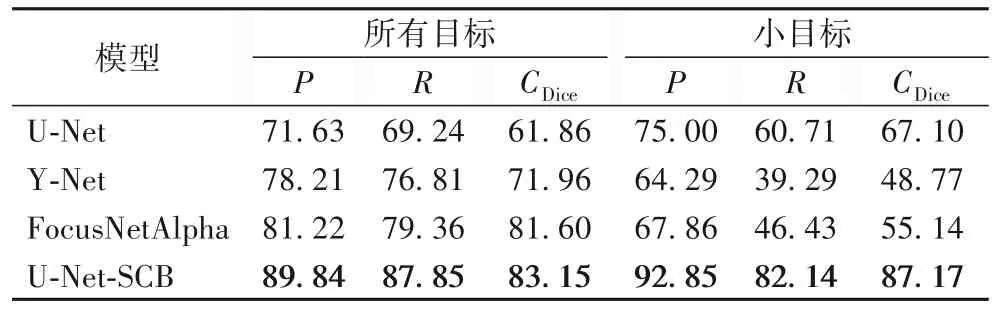
表4 不同网络针对不同目标的性能指标对比 单位:%Tab. 4 Comparison of performance indicators of different networks for different goals unit:%
3 结语
直肠癌是我国严重恶性肿瘤之一,死亡率高,而直肠肿瘤区域的精确分割往往是直肠癌诊断的关键,运用深度学习方法对直肠肿瘤区域进行精准分割,是人工智能技术在医学领域的重要应用。基于U-Net-SCB 的直肠肿瘤分割能辅助医生对直肠肿瘤进行诊断,有助于减轻医生劳动强度,减少漏诊和误诊,为患者争取宝贵的治疗时间。
——编码器

