大豆种子蛋白质组样品制备与数据分析方法
牟永莹 王道平 陈明 邱丽娟 潘映红
(中国农业科学院作物科学研究所 农作物基因资源与基因改良重大科学工程,北京 100081)
蛋白质组是连接转录组与代谢组的关键环节,通过研究不同时空的蛋白质组构成,可揭示各种复杂的生命活动过程。大豆(Glycine max)是一种在全球范围内广泛种植的豆科植物,也是世界上产量最高的油料物。自2010年大豆品种Williams基因组测序完成后,蛋白质组学分析技术在大豆生长发育、逆境胁迫研究等多个方面得到广泛应用,目前已成为深入研究大豆育种和栽培分子基础的有力工具[1-4]。
蛋白质组学分析流程主要包括样品制备、质谱分析、数据挖掘等步骤,其中,蛋白质提取和酶切等样品制备技术是决定后续质谱分析重复性的关键因素之一,液相参数设置和质谱数据分析方式也会对蛋白鉴定结果产生影响。尽管前人报道过一些大豆种子蛋白质组学相关工作[5-6],但是仍缺乏对大豆种子蛋白质组样品制备和分析技术的系统研究。
林杨杰等[7]曾采用多种缓冲液进行大豆种子蛋白初提和复提,并结合一维电泳预分离,可有效提高蛋白鉴定率,然而此方法处理步骤较繁琐,且存在胶内酶切过程中分子量大和疏水性强的肽段抽提难、酶解肽段回收率低的问题。为了提高样品制备过程中蛋白提取与酶切的效率,Park等[8]针对强疏水性膜蛋白的酶解提出了热变性方法,发现大豆种子蛋白样品经过热变性处理后,蛋白质更容易被酶解。除热变性方法外,化学变性法也是广泛使用的样品处理方法。十二烷基硫酸钠(Sodiumdodeeysuzfate,SDS)是用于细胞、组织变性和增溶的首选试剂,但SDS在溶液中很难被完全去除,对后续酶切、色谱分离和质谱分析有明显影响,因此,开发替代方法尤为重要。林勇等[9]报道脱氧胆酸钠(Sodiumdeoxyeholate,SDC)具有与SDS近似的变性作用,经酸化处理和离心后可以从样品中去除,在提高膜蛋白提取率的同时又不会明显影响后续步骤。最近,Lys-C/trypsin顺序酶切法也被用于蛋白质组学研究中,李倩等[10]利用该方法对培养的人细胞全蛋白样本进行酶解消化,肽段和蛋白质的鉴定数目均有显著提高。
除样品制备外,色谱分离和质谱谱图解析等对蛋白质组分析结果也有明显影响。在复杂蛋白分离过程中,任何梯度时间点上都可能存在大量同时洗脱的肽段,理想情况下,分析样品中的所有肽都应进行MS/MS裂解,以实现蛋白质组的完全覆盖,然而质谱仪的局限性和采集时间的限制导致大多数肽段在数据依赖采集中没有被检验,因此,提高多肽鉴定的一个途径是在质谱分析之前改进肽段的液相色谱梯度分离[11-12]。Woehlbrand等[12]通过比较不同液相色谱梯度对海洋细菌蛋白质组分析的影响,证明较长的梯度会增加鉴定的肽段和蛋白质的数量以及结果的可重复性。受质谱类型和经费的限制,一般情况下质谱谱图解析软件的选择余地较小,但有多种免费蛋白数据库可用于谱图解析。对于植物材料,Phytozome、UniProt和NCBI这3种数据库应用较广泛,Phytozome是一个专门比较植物基因组和基因家族的数据库,可提供每种植物的序列、基因结构、基因家族和基因组功能注释[13]。UniProt是国际上广泛使用的蛋白质数据库,主要包括UniProt知识库(UniProtKB)、UniProt归档库(UniParc)和UniProt参考序列集(UniRef)3部分,具有序列数据完整、资源广泛的优势,为生命科学领域提供了宝贵资源[14]。NCBI是美国的一个大型生物信息学系统,拥有多种数据库分析资源,是全球最有影响的生物学网站之一,可提供综合的核酸和蛋白数据库[15]。这些数据库的蛋白质序列以及功能注释信息的格式和更新速度各不相同,关于比较3种数据库的大豆质谱数据解析结果的文章未见报道。
大豆种子蛋白质组分析具有重要的科学意义和应用价值,但大豆种子样品制备中存在蛋白提取和酶切不彻底的问题,迫切需要优化蛋白提取和酶切方法。液相分离时长可影响质谱分析中可用的肽段检出率,从而影响可识别蛋白的数量。而且目前可应用的解谱数据库种类较多,何种数据库更适合大豆种子蛋白质组分析也有待研究。
本研究拟以大豆种子为材料,通过改进传统的尿素硫脲蛋白提取液,选择适合的酶切方法、液相梯度和数据库,初步构建一种适用于大豆种子蛋白质组质谱分析的技术体系,为深入的大豆蛋白组学研究创造条件。
1 材料与方法
1.1 材料
中黄35和十胜长叶成熟大豆种子由中国农业科学院作物科学研究所大豆基因资源挖掘与利用课题组提供。
尿素(urea)、三羟甲基氨基甲烷(tris)购于美国昂飞(Affymetrix)公司;碘乙酰胺(IAA)、硫脲(thiourea)购于GE公司;胰蛋白酶(trypsin)、赖氨酸内切酶(Lys-C)为Promega公司产品;二硫苏糖醇(DTT)、乙腈(ACN)、甲酸购于赛默飞世尔科技(Thermo Fisher);碳酸氢铵(ammonium bicarbonate)、乙二胺四乙酸(EDTA)、3-[(3-胆固醇氨丙基)二甲基氨基]-1-丙磺酸(CHAPS)购于Amresco公司。Easy-nano LC1000液相系统和Q-Exactive Plus质谱仪(赛默飞世尔科技公司,Thermo Fisher)。
1.2 方法
对大豆种子蛋白质组分析流程中的蛋白质提取、酶切、液相质谱分析和谱图解析进行优化和比较(图1)。
1.2.1 蛋白质提取 常规尿素硫脲法(Urea thiourea extraction,UT):将Natarajan等[16]与Wisniewski等[17]的方法结合用于大豆蛋白质提取,具体步骤如下:首先取适量成熟大豆种子,在液氮中研磨至粉末,分别称取50 mg种子粉置于2 mL的EP管中,加入0.3 mL 提取缓冲液(7 mol/L Urea、2 mol/L Thiourea、25 mmol/L Tris-HCl、50 mmol/L DTT和4%CHAPS),冻融3次后15 000 r/min离心20 min,转移上清液到10 K超滤管内超滤离心(15 000 r/min,20 min,下同),样品经超滤管离心后加入100 μL现配碘乙酰胺(Iodoacetamide,IAA)溶液(50 mmol/L IAA、8 mol/L Urea、100 mmol/L Tris-HCl,pH 8.1)暗置30 min,离心,用100 μL尿素缓冲液(8 mol/L Urea,H2O)洗涤超滤3次,加200 μL 50 mmol/L碳酸氢氨洗涤超滤2次,获得蛋白混合物待酶切。改进的尿素硫脲法(Modified urea thiourea extraction,mUT):为了提高蛋白提取效率,对尿素硫脲缓冲液进行改进,在原有的基础上增加EDTA和NaCl成分,具体配置方法为8 mol/L Urea、2 mol/L Thiourea、25 mmol/L Tris-HCl、50 mmol/L DTT、4% CHAPS、2 mmol/L EDTA和200 mmol/L NaCl。还原和烷基化步骤同上。
1.2.2 蛋白酶切 常规胰蛋白酶切(Trypsin digestion,TD):向超滤管的蛋白混合物中加1 μg酶,37℃反应12 h,室温,15 000 r/min离心30 min,收集超滤外管中的液体并转移至上样管中。高温辅助酶切(High temperature assisted trypsin digestion,HTTD):将超滤管置于100℃沸水中5 min,温度降为室温后加1 μg酶,37℃反应12 h,离心、收集步骤同上。
赖氨酸C端内切酶/胰蛋白酶顺序酶切(Lys-C/trypsin digestion,Lys-C/TD):蛋白混合物中加入Lys-C,37℃反应2 h后,加入1 μg酶,剩余步骤同上。脱氧胆酸钠辅助酶切法(Sodium deoxycholate assisted trypsin digestion,SDC-TD):蛋白混合物溶解在0.4%SDC,再加1 μg酶,37℃反应12 h后加10%TFA,离心去除SDC沉淀,取上清转移至上样管。
1.2.3 LC-MS分析和参数设置 使用基于质谱的非标记定量方法对大豆种子蛋白样品进行分析,液相为Easy-nLC 1000 nano液相系统,填装C18 PepMap预 柱(100 μm×20 mm,Thermo Fisher Scientific)和C18 Tip色谱分析柱(75 μm×150 mm,Thermo Fisher Scientific)流动相A为0.1%FA,ddH2O;流动相B为0.1%FA,Acetonitrile。条件为流速420 nL/min。比较3种洗脱时间和梯度的分离效果:分别为30 min(0-22 min,0-35%;22-23 min,35%-100%;23-30 min,100%)、90 min(0-8 min,4%-5%;8-35 min,5%-9%;35-76 min,9%-18%;76-84 min,18%-31%;84-85 min,31%-100%;85-90 min,100%)和120 min(0-1 min,0-3%;1-4 min,3%-4%;4-24 min,4%-6%;24-104 min,6%-19%;104-109,19%-35%;109-110 min,35%-100%;110-120 min,100%),未涉及液相分离比较步骤时,液相色谱分离采用90 min梯度洗脱。试验所用质谱仪为Orbitrap Q-Exactive,质谱的采集模式为数据依赖采集(Data dependent acquisition,DDA),一级质谱的分辨率为70 000,扫描范围为300-1 800 m/z,自动增益控制值为3e6,注入时间为50 ms。二级质谱的分辨率为17 500,自动增益控制值1e5,注入时间45 ms,碰撞能量27 NCE。二级采集的标准以前20个最强离子为母离子,再进行Orbitrap检测,每个样品进行3次质谱技术重复。
1.2.4 数据检索软件与数据库 利用Proteome Discoverer 2.1(PD)定性分析软件对质谱的原始数据进行定性分析。分别使用Phytozome、UniProt和NCBI大豆蛋白质数据库进行搜索(未涉及数据库比较步骤时,定性分析仅使用UniProt数据库)。搜索参数设定为:最多漏切位点为2、母离子质量偏差为±20 ppm、固定修饰为半胱氨酸碘乙酰胺化(Carbamidomethy/+57.021 Da)、可变修饰为甲硫氨酸(Met)氧化(Oxidation/+15.995 Da)和乙酰化(蛋白N端)。
1.2.5 分析技术体系优化和验证 在比较大豆种子蛋白质组提取、酶切、分离和解谱等技术方法的基础上,构建优化的分析技术体系,并以大豆品种十胜长叶的成熟种子为材料进行3次技术重复验证。选用UniProt数据库解析质谱谱图,使用PD和Maxquant[18]软件进行定性和定量分析。
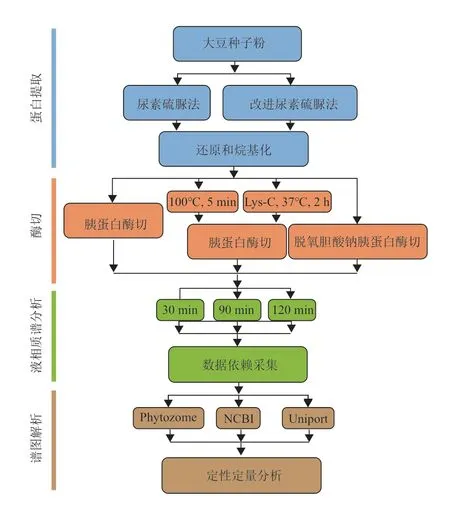
图1 大豆种子蛋白质组分析技术流程图
2 结果
2.1 蛋白质提取方法比较
使用UT和mUT法提取等量中黄35大豆种子的蛋白进行蛋白质组分析,发现两者的蛋白质数鉴定数和二级谱图数差异不显著,但mUT法得到的肽段鉴定数和肽段-谱图匹配数均显著高于UT法,且mUT法的各组数据标准差均较UT法低(图2-A)。UT法3次重复制样平均鉴定到1 228组蛋白,其中共鉴定蛋白占62%,而mUT法3次制样平均鉴定到1 334组蛋白,共鉴定蛋白占68%,共鉴定蛋白数目较UT法增加148组(图2-B)。

图2 UT法和mUT法提取大豆种子蛋白的质谱鉴定结果
2.2 蛋白酶切方法比较
分析4种酶切方法的酶解效率发现,使用Lys-C/TD法鉴定蛋白数目平均值为1 536组,显著高于其他酶切方法,而SDC-TD和HT-TD法获得的肽段和蛋白鉴定数目没有显著差异,但均比TD法显著提高,蛋白鉴定数目最大差异约15%(图3-A)。对各试验组中蛋白序列覆盖度在20%-30%、30%-40%、>40%区间的蛋白数目进行统计(图3-B)。比较鉴定蛋白的肽段序列覆盖率发现,在序列覆盖率20%-30%和30%-40%区间,Lys-C/TD法所鉴定到的蛋白数显著高于其他方法,在>40%区间HT-TD法的鉴定蛋白数和重复性均较高(图3-B)。进一步分析显示,使用Lys-C/TD法酶切时,蛋白漏切位点的比例约为25%,而其他方法的比例均大于33%(图3-C)。
2.3 液相色谱分离效果比较
在液相洗脱梯度分别为30、90和120 min的色谱分离条件下,同等上样量条件下的质谱总离子流图(TIC)如图4所示,90和120 min梯度下总离子流的分布更均匀,液相色谱分离效果更好。质谱鉴定结果(表1)显示,90和120 min梯度相比,采用120 min梯度时,肽段-谱图匹配数(PSMs)和二级图谱鉴定数(MS/MS spectrum)较90 min梯度呈现倍数增长,肽段鉴定数虽有显著差异却未成倍增长,并且2种梯度下蛋白鉴定数无显著差异。
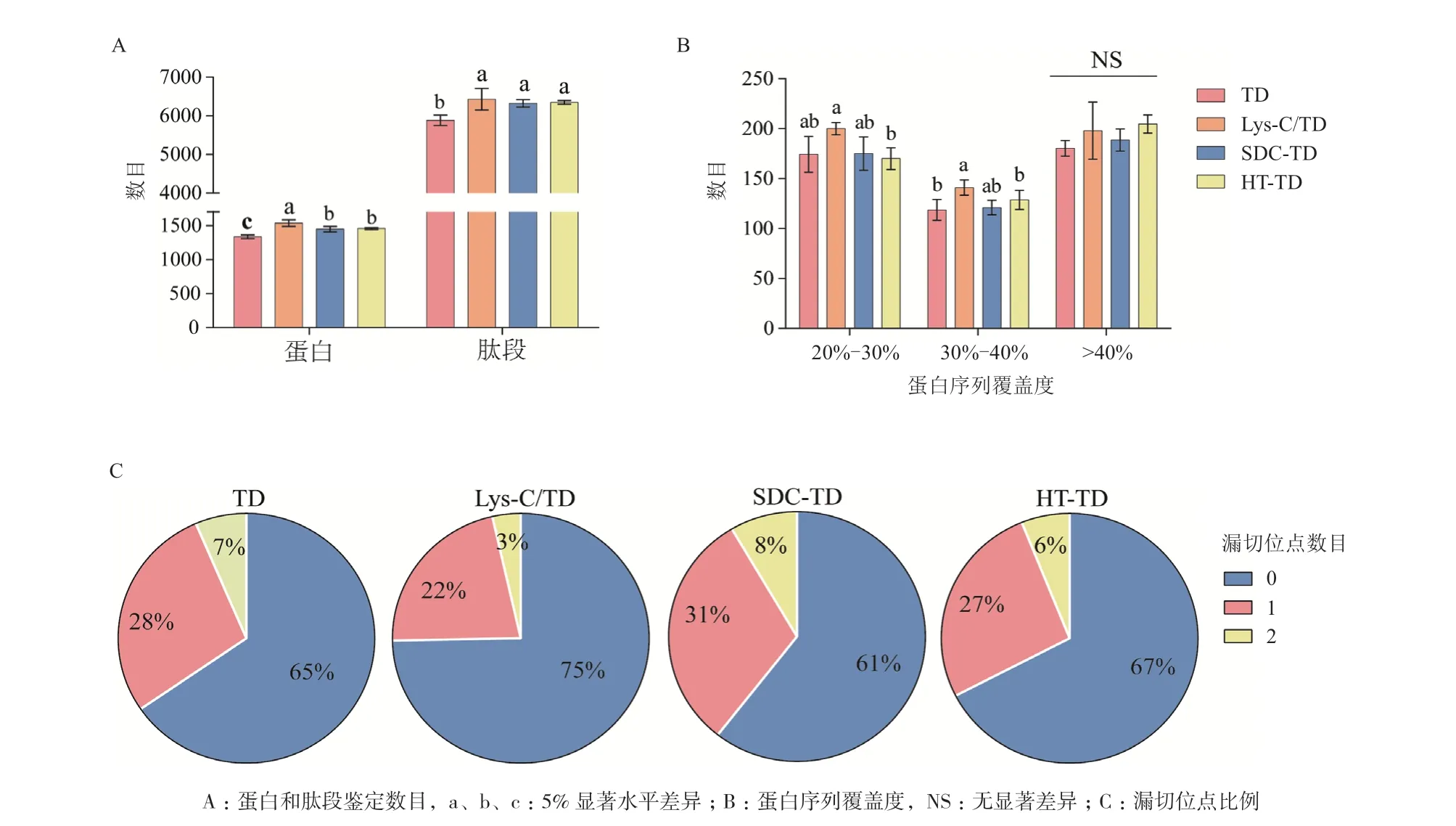
图3 TD、Lys-C/TD、SDC-TD和HT-TD 4种酶切方法的质谱鉴定结果
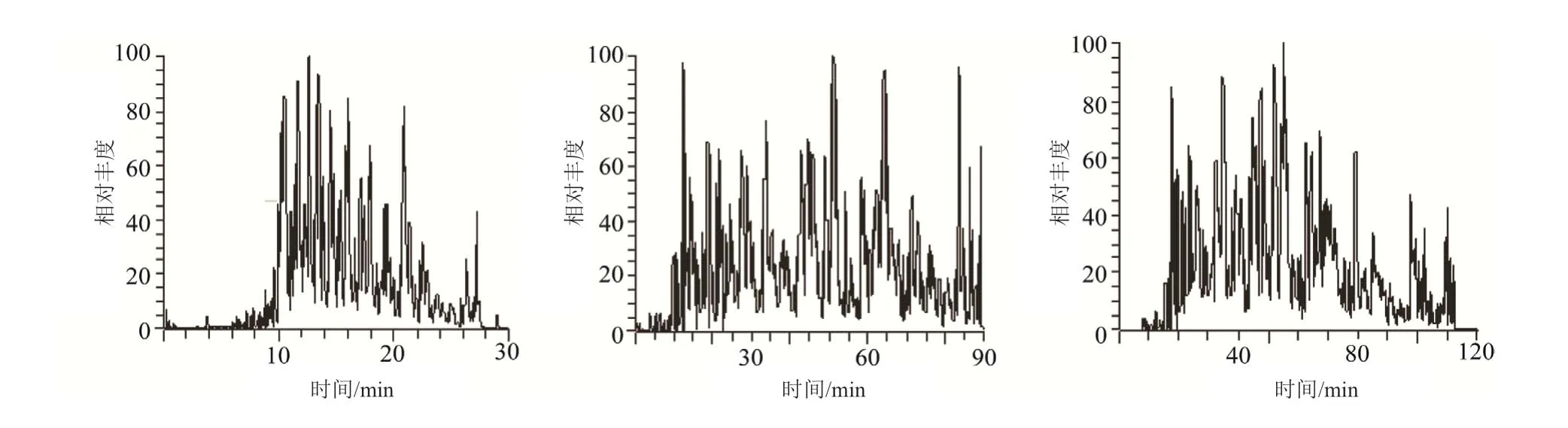
图4 三种液相梯度分离蛋白的总离子流图

表1 不同液相梯度时间蛋白分离效果比较
2.4 数据库谱图解析结果比较
以目前常用的3个蛋白质组数据库Phytozome、UniProt和NCBI为背景库,对用DDA模式采集的同批高质量质谱数据进行谱图解析,分别鉴定到1 971、1 990和1 958种大豆种子蛋白,其中,使用NCBI数据库鉴定到有功能注释的蛋白最多,占其总鉴定蛋白数的76.66%,UniProt大豆数据库得到有功能注释蛋白所占的比例最小,仅占其总鉴定蛋白数的62.16%(表2)。
2.5 蛋白提取流程可靠性验证
在前述结果分析的基础上,选取mUT蛋白提取法、Lys/C-TD顺序酶切法、90 min液相梯度和Uniport数据库构建了大豆种子蛋白质组分析技术体系。定性分析结果显示,3次样品制备重复试验共鉴定非冗余蛋白2 244组,单次最大鉴定量为6 946组肽段和1 631组蛋白,约60%的蛋白可被重复鉴定(图5-A和图5-B)。定量结果显示,3次独立样品制备重复实验中肽段和蛋白的相关性均在70%以上,且相同信号强度内定量的蛋白数目大致相同(图5-C和图5-D)。
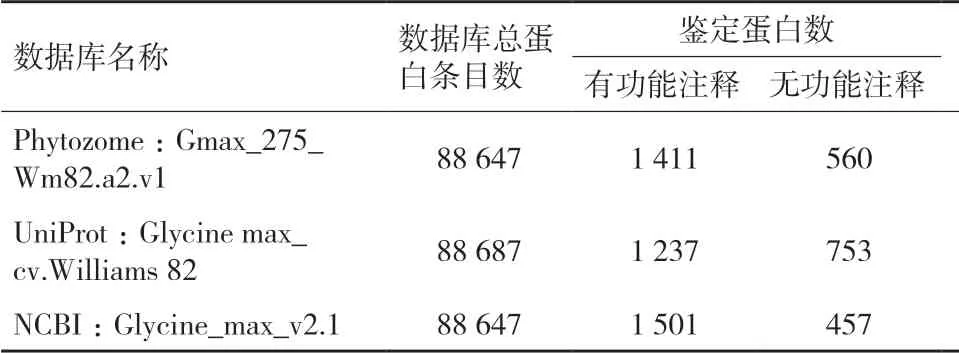
表2 不同数据库搜库结果比较
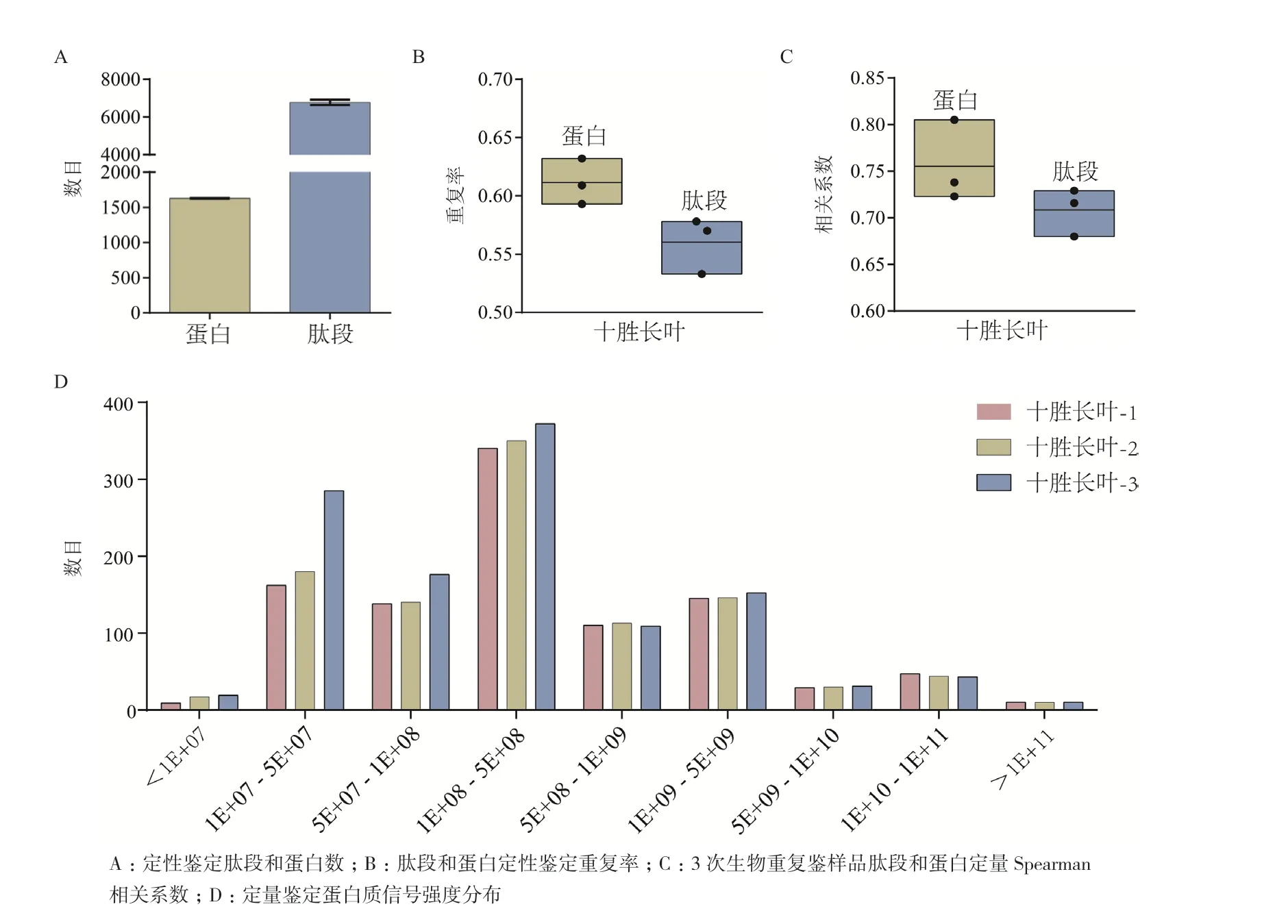
图5 十胜长叶质谱定性定量分析结果
3 讨论
3.1 使用改进尿素硫脲提取法配合Lys-C/Trypsin顺序酶切法可以显著提高蛋白鉴定数目
蛋白质组学样品制备的第一步是蛋白提取,常用的大豆蛋白质提取方法有尿素硫脲法和三氯乙酸(TCA)丙酮法[19-21]。林杨杰等[7]前期工作表明用尿素硫脲法初提大豆种子蛋白时,样品蛋白得率最高,但是此方法在提取蛋白过程中加入丙酮沉淀蛋白去除杂质,会导致部分样品损失。为了解决这个问题,本研究参考过滤辅助样品制备法(Filter-aided sample preparation,FASP)[17],使提取蛋白的除杂和酶切步骤均在超滤管内完成。目前公认利用超滤膜可以较有效地去除表面活性剂和更换蛋白裂解液,也可以分离酶切后的肽段,该方法具有操作简单,样品损失少,酶切后肽段纯度高的优势[22]。除了蛋白提取方式外,本试验对蛋白提取液也进行了改进,增加了EDTA等成分。使用原始尿素硫脲法(UT)和改进后的尿素硫脲法(mUT)的大豆种子蛋白提取结果如图2所示,2种方法鉴定的蛋白数目虽无显著差别,但是mUT法3次制样重复的共有蛋白占的比列相对于UT法明显增加,说明mUT法具有更好的重复性。这一结果或许与金属螯合剂EDTA的加入相关,Jez等[23]研究表明EDTA可以去除缓冲液中存在的痕量重金属离子。因此,在蛋白提取液中加入EDTA可以防止重金属离子与蛋白质形成不溶复合物,避免因复合物酶切不彻底对质谱鉴定产生的影响,提高了质谱鉴定结果的稳定性。
酶切是影响样品制备效果的另一个重要因素,大豆种子蛋白中存在一些结构紧凑的难酶解蛋白,单独使用胰蛋白酶酶切无法将其完全酶解,因此,需要通过一些物理和化学方法的辅助,破坏蛋白的紧凑结构,从而提高蛋白的溶解性。本研究比较了单一胰蛋白酶切(TD)、赖氨酸C端内切酶/胰蛋白酶顺序酶切(Lys-C/TD)、脱氧胆酸钠辅助酶切(SDC-TD)与高温辅助酶切(HT-TD)4种方法的酶切效果。结果显示,Lys-C/TD的蛋白鉴定数目显著高于另3种方法,其中蛋白序列覆盖度在20%-30%和30%-40%区间的蛋白增加较多,蛋白漏切位点数目低于其他方法。这与Betancourt等[24]和Hakobyan等[25]报道结果相符,Lys-C酶和Trypsin酶顺序酶切可以显著降低漏切位点的数目,提高序列覆盖度在20%-40%的蛋白鉴定数目。SDC-TD法的蛋白鉴定数目高于TD法,漏切位点数目与TD相比无明显差异。前人研究表明SDC在促进蛋白的溶解和变性的同时,还具有增强胰蛋白活性的作用[26-27],本研究结果也证实了这一观点。除此之外,本研究还表明SDC的加入对降低蛋白漏切位点无显著作用。HT-TD与SDC-TD法的蛋白鉴定数目均高于TD法并且低于Lys-C/TD法,漏切位点也与SDCTD法和TD法无明显差异,且序列覆盖度在20%-40%区间的蛋白数目相对较少,>40%区间蛋白数目比例提高,这与前人的研究结果类似[28]。
综上所述,mUT蛋白提取方法具有更强的可重复性,Lys-C/TD、SDC-TD和HT-TD酶切法与TD法相比均可以提高蛋白鉴定数目,Lys-C/TD提升效果最佳而且可以降低漏切位点。因此,mUT配合Lys-C/TD可以显著提高蛋白鉴定数目,更适用于大豆种子样品制备。
3.2 适宜的液相梯度分离时间可以有效提高蛋白鉴定数目
液相对酶切肽段的分离时长和梯度决定了每个时间间隔可用于电离和质谱分析的肽段数量。本研究确定大豆种子酶切肽段在梯度洗脱时间为30、90和120 min时的分离效果,结果表明,相对于30 min分离时长,90和120 min分离时长得到的离子峰分布更均匀,即肽段在不同梯度时间点上的分布更均匀,可减少因同一时间点洗脱肽段过于集中而造成的质谱漏检情况。蛋白鉴定结果也证实了这一点,与30 min分离相比,采用90和120 min梯度分离后的肽段和蛋白鉴定率均显著提高。Hsieh等[29]研究显示液相色谱梯度长度增加主要通过增加样品采集分析时间来影响肽的鉴定,将梯度长度从30 min增加到60或90 min会使MS/MS光谱数增加两倍以上。增加梯度长度会使肽段鉴定数目增加,但是肽段提升程度不是线性变化的,而是会随着梯度增加而逐渐减弱,这与本实验的研究结果一致。
虽然大量研究证明在液相质谱分析中,梯度洗脱时间越长,肽段和蛋白的识别率越高[11,30-31],但是增加梯度长度在提升肽段鉴定率的同时会降低样品通量。因此,在设置液相色谱分离时间梯度时,需要结合蛋白特性,在保证样品通量的前提下尽可能延长梯度时间,以实现样品的有效采集,提高蛋白鉴定率。
3.3 采用适宜的质谱解析数据库可以使后续数据分析更便捷
获得质谱数据后,要利用数据库对谱图进行解析。目前,常用的蛋白质组数据库有Phytozome、UniProt和NCBI,从表2得知,3个数据库的大豆总蛋白条目数相近,而且蛋白质定性结果显示本研究鉴定到的大豆种子蛋白数目均接近2 000组,并无明显差异,这表明对于大豆种子样品,数据库的选择似乎对最终蛋白鉴定数目无显著影响。但值得注意的是,不同的数据库有各自的蛋白编号,对于UniProt和NCBI大豆数据库的蛋白编号,PD软件在定性时可同时进行功能注释,而Phytozome数据库则需要使用其他软件再次注释。在后续分析过程中,我们也发现使用Phytozome数据库导出的蛋白编号需要通过序列比对转换为可识别编号,给后续研究增加了一些无意义的工作量,而UniProt数据库蛋白编号与多数生物信息学分析网站的兼容度最高,质谱定性和定量解析时选择UniProt数据库可以使后续数据分析更便捷。
3.4 蛋白提取与分析流程的稳定性分析
经过对大豆种子蛋白提取与分析流程的比较优化,提出了由mUT法和Lys-C/TD法提取和酶切,配合90 min色谱分离,质谱数据采用UniPort数据库解析的分析流程,并将其应用于十胜长叶成熟种子蛋白质组学分析中。结果显示,3次样品制备重复试验共鉴定非冗余蛋白2 244组,平均鉴定蛋白数量稳定在1 628组,是Xu等[32]报道的使用双向凝胶电泳方法检测的2倍以上,但是蛋白定性鉴定重复率较低,仅约60%的蛋白可被重复鉴定到,进一步的定量分析证实,重复制备样品得到的定量蛋白Spearman系数水平均值为0.75,相关性较显著,3次重复样品中重复性差的蛋白信号强度主要分布在5E+08以下,而信号强度大于5E+08的蛋白数量很稳定,这一现象可能与质谱分析时采用数据依赖采集方式相关,高丰度蛋白更容易被检测,因此出现的频率更稳定。总体而言,以上结果表明本蛋白质组分析技术体系可以提高蛋白鉴定数目,具备一定的可靠性和稳定性,更适用于大豆种子蛋白质组学分析。
4 结论
对于基于质谱的大豆种子蛋白质组学分析,采用改进后的尿素硫脲提取液配合赖氨酸C端内切酶/胰蛋白酶顺序酶切法,经90 min纳升级液相梯度分离和Q Extractive质谱分析,蛋白鉴定数目最高且定量重复性较好,研究还发现Phytozome、UniProt和NCBI数据库的大豆蛋白总条目数相近,使用以上数据库搜库时,大豆种子蛋白定性鉴定数目均接近2 000组,并且使用UniProt数据库可以使后续分析更便捷。

