基于深度学习的作物基因组学和遗传改良
辛志奇, 赵航, 汪海, 路铁刚*
1.中国农业科学院生物技术研究所,北京100081;2.中国农业大学国家玉米改良中心,北京100193
随着全球人口数量不断增加,到2050 年,全球对粮食的需求预计将比2005 年增加100%~110%[1-2]。为满足人们对农作物产品日益增长的需求,创新育种技术显得尤为重要。在漫长的农业历史中,育种主要经历了三个阶段:通过观察植株表型,选育优良自交系的传统育种;应用统计学、数量遗传学预先设计杂交育种实验,获得杂种优势的杂交育种;综合单倍体育种、分子标记育种和转基因育种的现代生物工程育种[1]。Edward S Buckler[2]总结了过去的三个时代,并提出了“育种4.0”的概念。王向峰等[1]提出了在“育种4.0”时代深度融合生命科学、信息科学和育种科学的理念。人工智能设计育种是由人工智能与育种相结合,能够给传统育种带来革命性的改变。它包括利用深度学习和机器学习把基因组学、转录组学、蛋白质组学、表观遗传学、代谢组学和表型组学的多组学数据结合,构建遗传调控网络,实现对作物表型的精准预测;深度学习指导基因编辑,实现对作物表型的控制和设计;深度学习在合成生物学的应用会使作物的设计育种更加自由高效。
1 人工智能及分支
人工智能这一概念最早在20 世纪40 年代被提出,但是受计算能力的限制,人工智能领域一直处于发展的低谷。进入21 世纪后,计算机性能的大幅提升(尤其是GPU 的发展)使得人工智能领域重新回到人们的视野。目前,人工智能已被应用于多个领域。
1.1 机器学习
人工智能领域最主要的研究方法是机器学习,机器学习按学习形式可以分为监督学习和无监督学习两种。监督学习是指在训练实例中学习输入变量数据和其因变量(或叫标签)之间的关系,然后以此在新实例中预测结果,主要应用于回归和分类问题。例如,可以用大量历史气象数据训练机器学习模型,该模型可以以过去的天气数据为预测因子,预测未来的天气。如果预测的目标变量为离散变量,则该机器学习任务称为分类问题(classification);如果预测的目标变量为连续变量,则该机器学习任务称为回归问题(regression)。在机器学习中有很多监督学习算法及应用,例如结合统计学的隐马尔可夫模型(hidden Markov model, HMM)和机器学习的支持向量机(support vector machine,SVM)可以快速准确预测和区分DNA 和RNA 结合残基的方法,这有利于进一步预测蛋白质-DNA 和蛋白质-RNA 相互作用的序列[3-4];用随机森林和支持向量机模型通过DNA 甲基化数据精确诊断癌症[5]。无监督学习是指在训练实例中输入没有因变量(或标签)的数据,又称为归纳性学习,典型的无监督学习包括降维(dimensionality reduction)和聚类(clustering),适合学习高维度数据,例如组学数据[6-7]。
1.2 深度学习
深度学习是机器学习领域的一个相对年轻的分支,已经成为机器学习领域最流行和最强大的技术之一[8]。人工神经网络以数学模型模拟神经元活动,包括输入层、隐藏层和输出层三个部分(图1),其深度神经网络用多层的隐藏层使神经网络的性能大幅提高,同时需要的计算能力和数据量也大幅提升。
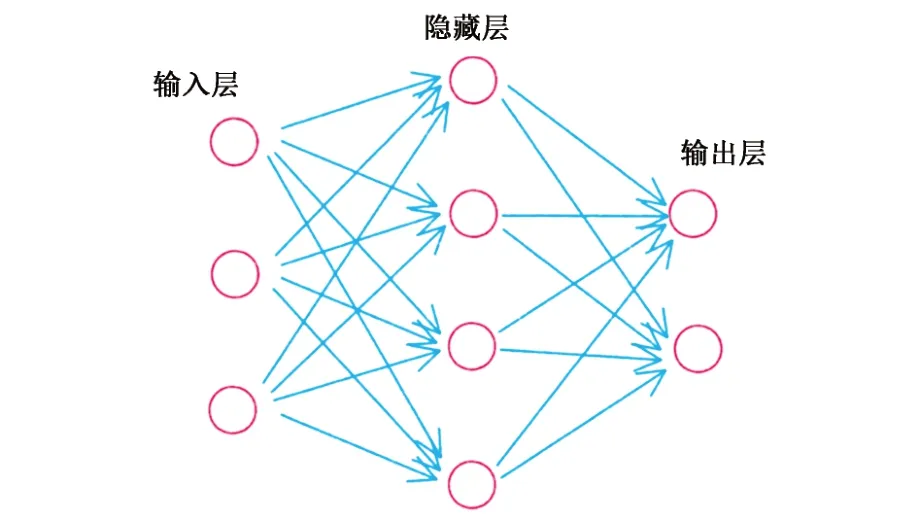
图1 人工神经网络层次Fig1 Artificial neural network
卷积神经网络(convolutional neural network,CNN)是深度神经网络的一种,也是基础的深度学习模型,用卷积这一数学计算方式提取数据中的特征信息,再经深度神经网络处理,可以大大增加神经网络的性能。卷积神经网络更擅长提取结构信息。目前已经有很多利用CNN 解决基因组学问题的例子。例如,Babak 等[4]预测DNA 和RNA与蛋白质的结合位点,Hashemifar 等[9]预测蛋白质之间相互作用;Gao 等[10]基于基因序列预测poly(A)位点;Zhou等[11]预测了人类基因组变异对基因表达调控和疾病的影响;Zhou 和Wang等[12-13]预测了非编码基因突变的影响;Jost 等[14]结合CRISPR技术实现调控基因表达量变化等。另一种监督学习模型,循环神经网络(neutral network, RNN)加入时间步(timestep)概念,使其具有记忆性和参数共享的特点,适合处理有时间信息的数据,广泛应用于自然语言处理领域。在生物学领域常被用来预测序列的功能。例如,Shen 等[16]结合RNN 和k-mer[15]预测转录因子识别位点;Li 等[17]利用CNN和RNN 从氨基酸序列预测酶的生化功能;Quang等[18]利用RNN 和CNN 预测非编码基因的功能等。值得注意的是,有报道指出,CNN 在提取特征方面更高效,而释义DNA 序列方面,来自自然语言处理领域的k-mer 方法显得比CNN 和RNN 更优秀[19]。
自编码器(auto-encoder)是深度学习中的无监督学习的重要组成部分。自编码器分为编码和解码两部分。编码部分负责将输入数据低维化处理,也可以理解为特征提取;解码部分负责将编码得到的结果恢复到原始输入的形式,它是理解复杂深度学习模型的关键,可以把数据中的关键数据提炼并展现出来,解决了深度学习模型训练过程的不可见问题。目前自编码器在图像识别、降噪、色彩化方面有广泛应用。Zhang 等[7]用自编码器整合多组学数据,有效缓解了生物领域在运用人工智能模型时出现的“少样本,高维度特征”的问题;用自编码器解码深度学习模型并结合全基因组关联分析(genome wide association study,GWAS)的技术观察到未分类的基因在深度学习模型的不同深度中被有序的分类[20]。
生成模型技术作为深度学习领域的重要分支,它既不属于监督学习也不属于无监督学习。主要包括生成式对抗网络(generative adversarial network, GAN)和变分自动编码器(encoder)两种模型。
生成式对抗网络[21]分别建立并训练生成模块和判别模块,将生成模块生产的伪数据交由判别模块判断真伪,通过这种对抗学习的方式进行训练,可以生成真实度高的数据。目前在生物医药方面已经有相关的文章报道:基于生成式对抗网络设计蛋白酶抑制剂[22];RamaNet 模型从头设计合成螺线蛋白骨架[23];基于生成式对抗网络设计合成大肠杆菌启动子序列[24]。
变分自动编码器[25]与生成式对抗网络同属生成模型家族成员,两种模型都致力于生成更接近真实的数据,但是二者的实现思路不同。变分自动编码器在结构方面与自动编码器有相似之处,也是由编码器和解码器组成(也被称作识别模型和生成模型),并且二者都是学习输入数据的潜在向量并试图重建输入数据。不同的是,变分自动编码器学习潜在向量的分布关系,在潜在空间中是连续的,再由生成模型构建输入数据;生成式对抗网络由生成器和判别器组成,生成器负责创造数据,而判别器负责评价生成器创造的数据是否能够以假乱真。Davidsen 等[26]用变分自动编码器模型生成T细胞受体的蛋白质序列。
2 深度学习在作物基因组学中的应用
目前人工智能在农业上应用的报道主要是对图像和视频进行识别,如对玉米照片进行识别和对玉米干旱胁迫下的表型进行分类[27];视频检测植物生长早期干旱胁迫[28];视频识别水稻虫害和病害[29];以拟南芥为例基于植物图像对植物表型分类[30-31]等。生物的遗传信息是沿着中心法则传递的,想对植物基因进行设计,表型精准预测就一定要对基因组、转录组、蛋白质组、表观遗传组甚至是代谢组规则有更深的认识。近年来,在基因组学领域,围绕各种分子表型发展出了一系列基于二代测序的高通量技术,如转录组技术、开放染色质分析技术、DNA-转录因子互作技术[32]等。深度学习技术可以对这些大规模数据集进行建模。
2.1 深度学习模型建立的过程
深度学习模型建立首先遇到的一个问题就是生物学数据该以何种形式输入到人工智能模型中,这个问题在基因组和转录组已经有了统一的答案。One-hot 编码方式可以高效地将基因组和转录组数据储存在电脑中作为输入数据。将基因的A、T、G、C 4 种碱基储存在一个4×N 的矩阵中,每一列只储存1 个碱基(图2),这个方法可以将N bp的基因数据输入模型。
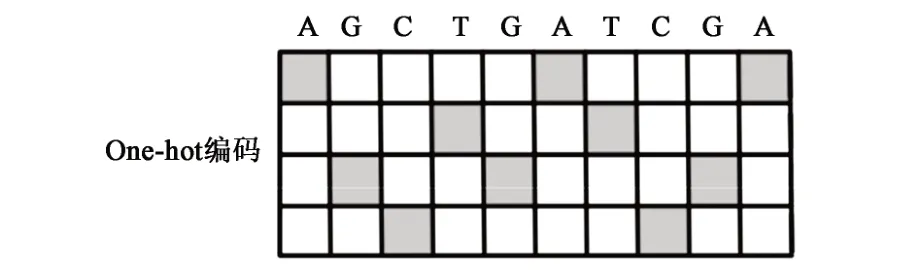
图2 One-hot编码Fig.2 One-hot encoding
当建立机器学习模型时,观测数据通常被随机分为训练集(用于训练模型)、验证集(用于确定模型结构和超参数),以及测试集(用于评估模型的性能)。这种随机划分能够避免数据间存在规律性特征而得出准确率虚高的模型。训练集/测试集的划分应尽量保持数据分布的一致性,避免混杂因素(confounder)对最终结果的影响。最常用的训练集/测试集分割方法为交叉验证法。在训练集上的准确度高于在测试集上的准确度,这种现象被称为过拟合(over-fitting)。有几种情况会导致过拟合。一个通常出现的问题是特征空间中的维度有时大大超过观测值。例如,当从基因组变体预测一个表型时,检测到的基因组单核苷酸SNP数目几乎总是超过植物基因型的数目。在这种情况下,可以使用主成分分析(principal component analysis,PCA)和自动编码器[11-12]等降维技术来减少特征的数目。然而,在处理基因组学中的问题时,过拟合有时候是隐藏的。例如,当一个基因家族的成员被划分为训练集和验证、测试集时,模型将学习家族特异性的分子特征,并高估预测准确性。
2.2 利用深度学习技术预测生物学序列
各个组学数据都有被人工智能挖掘有用信息的巨大潜力。在DNA 层次上,Umarov 等[33]利用CNN 构建了启动子的预测模型,分析了几种原核和真核生物的启动子序列特征,包括人、老鼠、植物(拟南芥)和细菌(大肠杆菌和枯草芽孢杆菌)。DanQ 是一种将CNN 和双向长短期记忆循环神经网络(BLSTM)相结合的混合框架,用于从头预测非编码区的功能。DanQ 学习了一种调节语法来改善预测准确性,并为非编码基因组区域提供了新的见解[18]。 DanQ 还结合 CNN 和 BLSTM 在序列中从头预测非编码区功能[18]。Sample 等[34]使用CNN 和遗传算法精准预测了人类5’UTR 变体对核糖体装载的影响。
在RNA 水平上,使用循环神经网络(neutral network,RNN)在人类 mRNA 和 lncRNA 序列上训练了一个门控RNN,然后用它来预测RNA 分子是否编码蛋白质[35]。使用 CNN 预测人类 5’UTR 变异对核糖体装载的影响[34]。他们将28 万个随机的5’UTR 的多聚体分析与深度学习相结合,建立了一个模型,从人类5’UTR 序列预测翻译效率。此外,DeepChrome 是一个从组蛋白修饰数据预测基因表达量的CNN,能够自动提取重要特征之间的复杂交互作用[36]。为了预测组织特异性的基因表达,研究人员将CNN 与空间特征变换和L2 正则化线性模型相结合,建立了ExPecto模型[37]。
在蛋白质水平上,为了在从头生成的肽序列中提取重要的氨基酸特征,利用CNN 方法开发了DeepNovo[38]。为了预测蛋白质的二级结构,使用了相对溶剂可及性和残基间接触映射数据训练了深度学习模型rawMSA[39]。最近,谷歌的Alpha-Fold 利用深度学习模型预测蛋白质的三级结构,其精确度远超传统机器学习方法[40]。此外,深度学习模型也用来预测蛋白质—蛋白质的相互作用。DPPI 是一种能够从蛋白质序列信息预测蛋白相互作用和蛋白二聚体的深度学习模型[41]。DEEPre 可以从蛋白质序列预测酶的类别,利用该模型可以发掘在宏基因组、工业生物技术和人类疾病中起重要功能的蛋白质[42]。
除了用各组学数据分别预测之外,Ma 等[7]将各组学数据整合,使生物学数据更立体,与表型相关的信息也会更丰富准确,同时也会有效缓解人工智能与生物学结合领域一直存在的问题,即生物学“数据特征维度高但样本少”的问题,Ma 等[7]也指出这样做的难点在于各组学数据的信息不均匀。
3 深度学习在育种4.0中的应用
作物自然群体中存在着海量的自然变异,其中能够影响作物表型的变异称为功能变异。功能变异位点的不同等位变异具有不同的表型效应,可以划分为有利等位变异和有害等位变异。作物育种很大程度上可以视为有利等位变异的富集(也可以从另一个方面看做有害等位变异的清除)。过去的30 年被概括为育种3.0 时代,在这一历史阶段,获取高通量基因型数据和表型数据的成本不断降低,同时通过关联分析和连锁分析克隆了大量控制重要农艺性状的关键位点。以此为基础,分子标记辅助选择技术、基因组预测技术在作物育种中逐渐成为常规技术。未来我们将进入一个新的育种历史阶段:育种4.0。在这一阶段,人工智能将主要从三个方面促进设计育种发展:①发掘功能变异,指导精准杂交育种。通过各生物组学数据和环境数据预测出作物的产量和表型性状,从而实现简单化精准化的预测作物复杂优良性状。②设计有利等位变异,指导基因编辑育种。从基因水平、转录水平,以人工智能模型指导基因编辑,进一步细致调控基因表达,从而改良性状。③设计具有特定功能的基因组元件,指导合成生物学。创造新的DNA 元素、基因,甚至具有某种特定功能的调控通路,并将其应用于作物育种。
目前大多数研究都聚焦于人工智能进行分类和回归的能力。Wang 等[19]的文章中提到人工智能的生成模型可以通过学习生成新的基因元件从而应用于合成生物学。生成模型技术与合成生物学结合,根据预测模型的指导,重新设计非自然的基因、蛋白质等应用已经被报道。如深度学习指导编辑gRNA实现基因表达量的调控[14];结合生成式对抗网络设计大肠杆菌基因启动子序列[24];设计蛋白质序列以拓展蛋白质空间[43];设计螺线蛋白质骨架[23];生成T细胞受体的蛋白质序列[26]等。
深度学习模型存在迁移学习的性质,即可以用某一物种训练的预测模型预测相近物种,这种性质使得生物学中单一物种训练的模型有了更广泛的用处,如小鼠基因组训练的模型可以用在人类基因组上[50],单一植物叶片胁迫表型的识别模型可以用来预测其他植物的叶片胁迫表现[44]。
4 展望
人工智能特别是深度学习出现之后,已经在多个领域掀起新的浪潮,现阶段已经在基因组学、转录组学、蛋白质组学和合成生物学等领域发挥了巨大作用,如完善基因组功能注释、挖掘新功能基因、预测植物表型、发现基因、RNA、蛋白质等物质的新分类模式,指导基因编辑。高通量技术的发展见证着植物基因组学的进步,它以较低的花费识别着多种分子表型。然而,基因组学也要求利用强大的数据挖掘工具来预测和解释这些分子表型,深度学习则可以预测任何基因组变异的分子表型效应,获得直接控制分子表型的功能变异。此外,在合成生物学中应用深度学习模型也有望创造具有理想功能的新基因。总之,深度学习在未来植物基因组学研究和作物遗传改良中将发挥中心作用,人工智能将会是未来农业发展不可或缺的一部分。
——编码器

