基于生物信息学筛选肝癌组织自噬相关基因并建立风险预测模型
杨育华 于飞 梁善雄 杜梅珍 黄天壬
摘要:目的: 利用生物信息學分析TCGA数据库中肝癌组织与癌旁组织中表达具有差异性的自噬基因并建立肝癌预后风险预测模型。 方法 从癌症基因图谱(TCGA)数据库获取肝癌患者临床信息与mRNA表达信息,利用R语言DEseq2、Limma、EdegR软件包分析374例肝癌组织及50例癌旁组织中自噬相关差异表达基因(ARGs),对三个软件包获取的结果取交集作为差异基因结果;随后对ARGs进行基因本体论(GO),京都基因与基因组百科全书(KEGG)功能富集分析。采用单因素与多因素COX回归分析建立肝癌预后风险预测模型。结果:差异分析分别获取32,35,32个自噬相关基因,对结果取交集后得到19个自噬相关差异表达基因,其中包括11个上调基因和8个下调基因; 基因富集化结果显示19个ARGs主要参与了ErbB信号通路、自噬、细胞凋亡、Hippo信号通路等;经单因素与多因素COX回归分析后确定了BIRC5与CLN3基因表达量与肝癌患者预后生存时间相关,由此建立以自噬基因为基础的多因素风险预测模型;对风险预测模型的验证表明该模型的风险评分是肝细胞癌患者预后的独立危险因素(HR=1.27,95%CI=1.15~1.41,p=0.001),该模型高风险组患者生存时间显著低于低风险组患者(p<0.001),提示该模型在肝癌患者预后生存时间预测上具有较高的准确性。 结论 本研究基于TCGA数据库构建的自噬基因相关的肝癌预后评估模型可独立预测肝癌患者预后,有助于临床医生选择精准化的肝癌预防和治疗策略。
关键词: 肝癌;自噬 ;预后模型;自噬基因
【中图分类号】R322.4+7 【文献标识码】A 【文章编号】1673-9026(2022)04--03
Objective To analyze the differentially expressed autophagy genes in hepatocellular carcinoma tissues and adjacent tissues in TCGA database and establish a prognostic risk prediction model for hepatocellular carcinoma. Methods Clinical information and mRNA expression information of HCC patients were obtained from cancer Gene Mapping (TCGA) database. Autophagy-related differentially expressed genes (ARGs) in 374 HCC tissues and 50 paracancer tissues were analyzed using R language DEseq2, Limma and EdegR software packages. The intersection of the results obtained by the three software packages was taken as differential gene results. ARGs were then analyzed by gene Ontology (GO) and Kyoto Encyclopedia of Genes and Genomes (KEGG) for functional enrichment. Univariate and multivariate COX regression analysis were used to establish the prognostic risk prediction model of liver cancer. Results: 32,35,32 genes related to autophagy were obtained by differential analysis, and 19 differentially expressed genes related to autophagy were obtained by intersection of the results, including 11 up-regulated genes and 8 down-regulated genes. The results of gene enrichment showed that 19 ARGs were mainly involved in ErbB signaling pathway, autophagy, apoptosis and Hippo signaling pathway. After univariate and multivariate COX regression analysis, it was determined that the expression levels of BIRC5 and CLN3 genes were correlated with the prognosis and survival time of HCC patients, and a multi-factor risk prediction model based on autophagy genes was established. Validation of the risk prediction model showed that the risk score of the model was an independent risk factor for the prognosis of HCC patients (HR=1.27, 95%CI= 1.15-1.41, P =0.001), and the survival time of patients in the high-risk group was significantly lower than that in the low-risk group (P <0.001). It suggests that the model has high accuracy in predicting the survival time of HCC patients. Conclusion It is suggested that the autophagy gene related hepatocellular carcinoma prognosis assessment model constructed in this study can independently predict the prognosis of HCC patients, which is helpful for clinicians to select accurate prevention and treatment strategies for HCC.
Key words:Liver cancer; Autophagy. Prognostic model; Autophagy genes
肝细胞癌(Hepatocellular carcinoma ,HCC)是全球排名第三的癌症死因,是世界范围内的主要健康问题,严重威胁人类的健康与安全。根据最新统计[1],全球大约有四亿人感染了乙型肝炎病毒(Hepatitis B virus, HBV);中国的肝癌患者大约占全球50%以上;据估计,每年在中国因肝癌死亡的病例達到42.21万,新发病例46.61万,在恶性肿瘤中的发病率与死亡率分别排在第四位和第三位[2]。目前在临床上肝癌的治疗主要依靠化疗与手术切除,但由于肿瘤耐药性,肝癌患者的预后生存率依然不乐观;因此研究寻找更有效的临床预测肝癌患者预后指标变得十分关键,有助于解决肝癌这个全球公共卫生难题。
细胞自噬是细胞自身为调节细胞代谢与细胞器更新提供原料及能量的过程,自噬参与了细胞内炎症反应、细胞死亡以及免疫在内的生理病理过程以维持细胞稳态。自噬受到自噬相关基因(Autophagy-related genes,ARGs)以及多种信号传导途径的调控。现有研究表明HBV能够诱导自噬反应,但自噬在肝癌中体现出促进与抑制的双重作用,一方面自噬具有保护肝细胞免受内外环境的应激,清除细胞内不必要的蛋白质等成分及微生物病原体的作用[3]。目前研究提示自噬相关基因与肝癌发生发展密切相关,但大多数集中在单一自噬基因上,对肝癌中总体的自噬基因表达水平及预测价值仍不明确,基于目前相关研究,本研究分析癌症基因组图谱 (the cancer genome atlas,TCGA)肝癌数据,获取肝癌组织与癌旁组织中整体的自噬相关基因表达水平,并通过分析自噬基因差异表达与临床特征的关系,探索自噬基因是否是肝癌患者预后的独立危险因素,提升自噬相关基因的预测价值,有助于临床上更加精准预测肝癌患者的生存时间。
1. 材料与方法
1.1数据收集与整理分析
从TCGA 肝癌数据库(https://www.cancer.gov/about-nci/organization/research/structural-genomics/tcga, tcga-lihc)下载424例肝癌患者原始RNA测序信息与临床信息,样本包含374例肝癌组织与50例癌旁组织。从人类自噬数据库(http://www.autophagy.lu/clustering/index.html)获取222个人类自噬相关基因,经过与mRNA表达数据比对,最终纳入210个有效的人类自噬相关基因;然后将210个ARGs与mRNA表达信息匹配,采用R语言 DEseq2、Limma、edgeR软件包分别对mRNA表达数据进行基因表达差异分析,以肿瘤组织中基因表达量为正常组织1倍以上(log2FC ≥1)且 P<0.05定义差异表达基因,获得差异表达ARGs后采用R语言软件包pheatmap包绘制热图,ggplot包绘制火山图。
1.2评估ARGs的相关生物过程和通路
为了更好的探索自噬相关差异基因在HCC发生发展中的作用,我们对19个自噬相关差异基因的主要分子功能,细胞组分,生物学功能进行了富集分析,通过R软件包enrichplot进行分析,并绘制富集分析条形图、气泡图和弦图对结果进行可视化。
1.3自噬基因差异分析与影响患者预后生存的因素
通过分析筛选纳入374例肝癌样本临床信息;为了探索基因表达水平对肝癌患者预后生存时间的影响,我们对在肝癌组织中表达具有差异性的ARGs进行单因素与多因素Cox回归分析,获取到与肝癌患者预后显著相关的ARGS后,根据回归分析的结果建立肝癌风险评分模型,对模型绘制森林图,并根据模型计算样本风险评分,按照平均值将风险评分对应的样本分为高风险组与低风险组,进一步采用COX回归分析计算风险预测模型与其他临床信息特征对肝癌患者预后生存时间的影响,并通过Kaplan-Meier绘制高风险组与低风险组五年生存曲线,评估肝癌风险评分模型对患者预后的预测效果,探索自噬基因对肝癌患者预后的关系。
1.4统计学分析方法
分析基于4.0.5版本R软件,只有当P ≤0.05 认为差异有统计学意义。
2.结果
2.1数据获取和基因差异分析
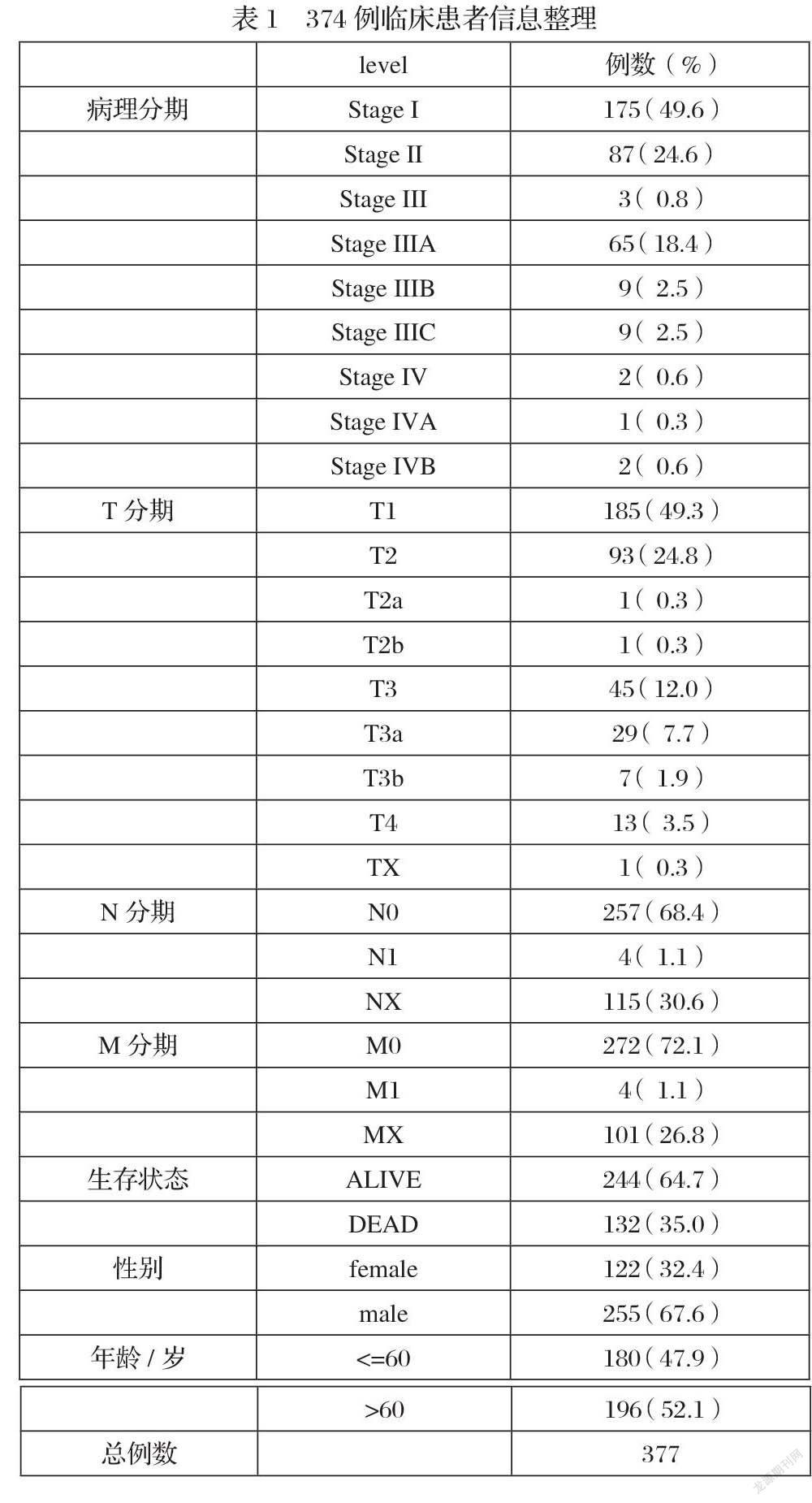
在TCGA数据库获取的374例样本临床信息如表1,本研究分析了210个ARGs在TCGA肝癌数据库中的表达差异并获取到19个差异具有显著性的基因,其中DEseq2包分析得到32个差异表达基因,上调基因数为22个,下调基因数为8个;Limma包分析得到25个差异表达基因,下调基因数为14个,上调基因数为11个;edgeR包分析得到34个差异表达基因,下调基因数为 8个,上调基因个数为26个。三个R包分析均定义为log2FC ≥1 和矫正 P<0.05。对三个R包分析结果取交集得到19个差异表达基因,分别为BIRC5,CCL2,CDKN2A,CLN3,DAPK2,DIRAS3,DLC1,FOS,FOXO1,ITGA6,MYC,NAMPT,NRG1,NRG2,PEA15,RAB24,SPHK1,TMEM74,TP73;图1显示了差异ARGs交集和在TCGA数据集中的分布热图及火山图
图1 肝癌组织与癌旁组织自噬相关基因表达情况 ①-②分别为火山图与热图,其中火山图中红色点表示上调基因,蓝色点表示下调基因。
2.2差异表达ARGs基因富集分析
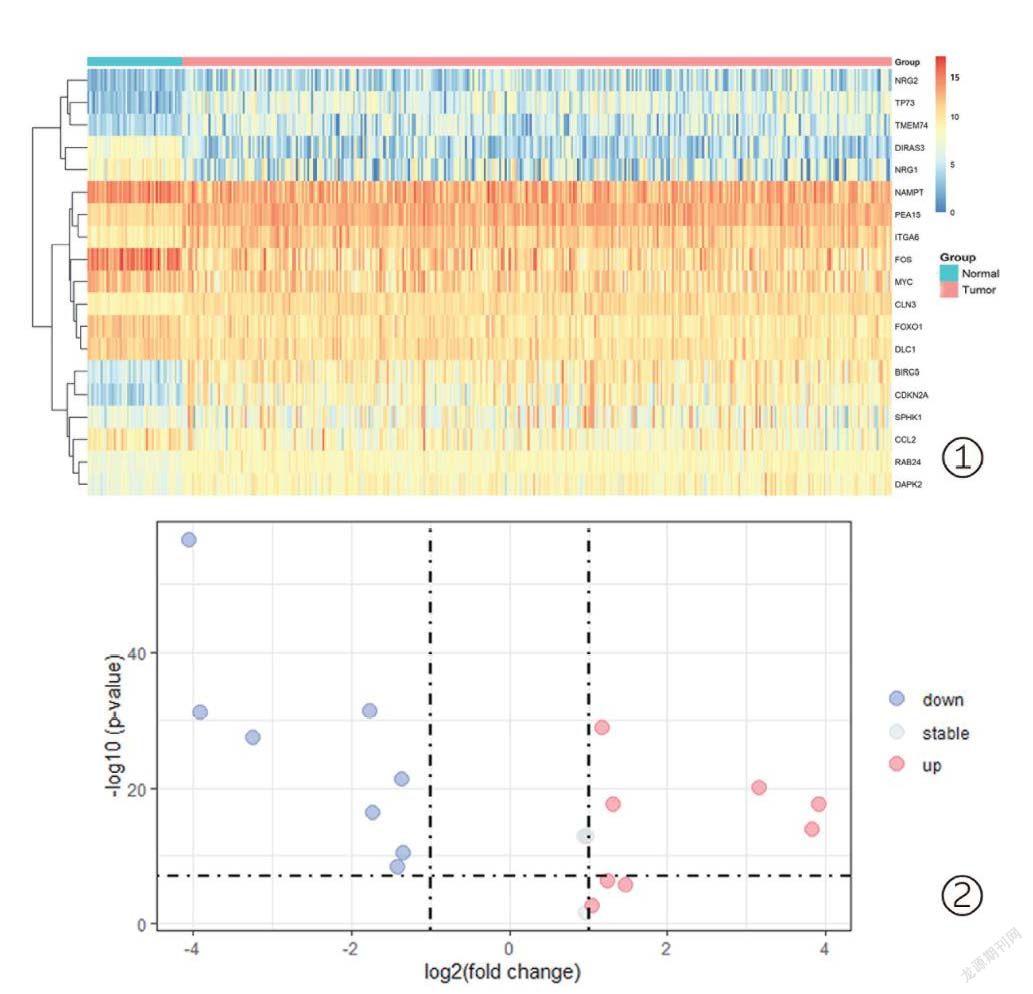
利用KEGG和GO富集分析对ARGs的分子功能、生物学过程、细胞组分进行深入分析,如图3所示,分析发现KEGG富集的通路主要包括自噬、ErbB信号通路、细胞凋亡和 p53 信号通路、IL-17信号通路、TNF信号通路。GO 富集的结果包括MAPK活性激活,ERBB2信号通路,自噬调节,转录激活等。
圖2 差异表达自噬基因的KEGG、GO通路富集功能显示ARGs主要参与自噬、ErbB信号通路、细胞凋亡和 p53 信号通路
2.3 ARGs表达与肝癌患者预后生存分析
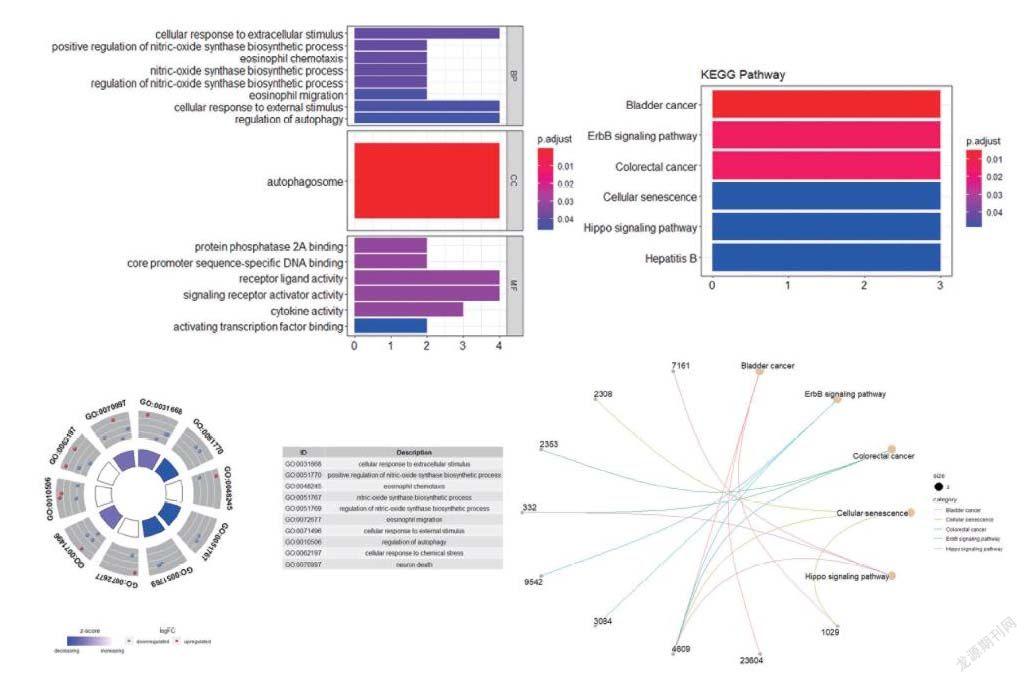
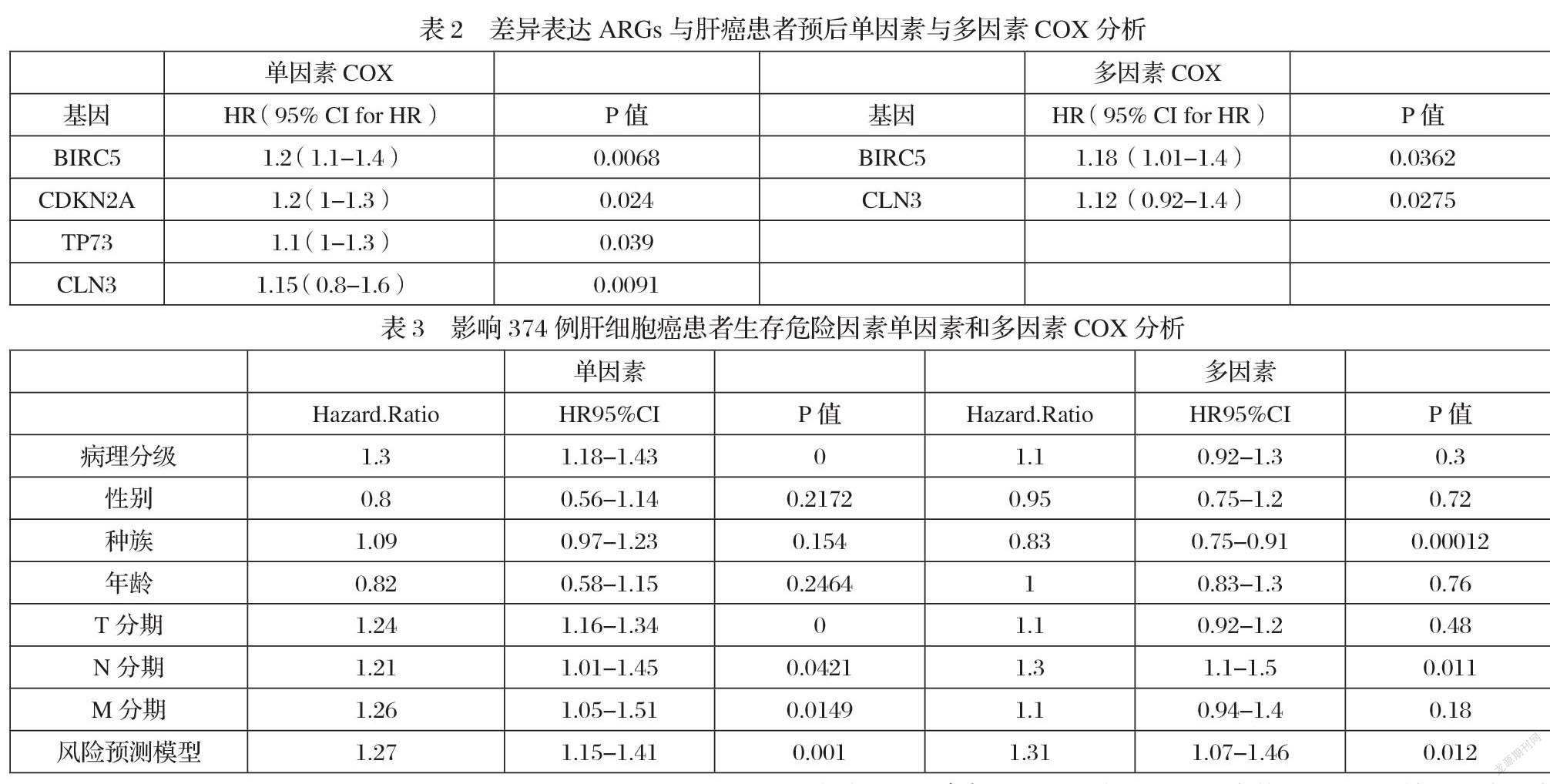
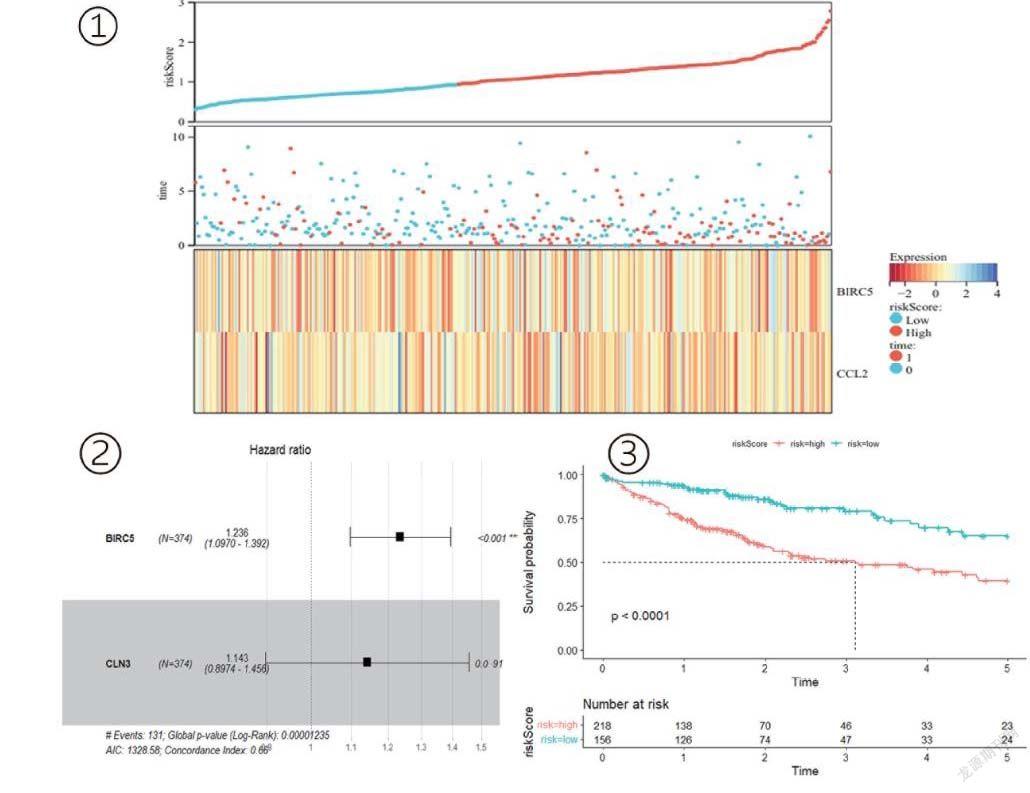
通过对差异表达的19个ARGs进行单因素COX生存分析发现BIRC5、CDKN2A、TP73、CLN3四个自噬基因与肝癌患者生存时间关系密切,(p<0.05,HR>1 为高危基因,HR<1 为低危基因);进一步对差异基因进行多因素COX生存分析发现BIRC5、CLN3对肝癌患者生存时间显著相关,见表2;为独立预测肝癌患者预后,根据COX回归分析结果构建基于加权风险评分的肝癌预后模型,风险评分计算公式:0.198*BIRC5表达量+ 0.0902*CLN3表达量;绘制K_M生存曲线图显示高风险组与低风险组的生存时间差异,以此检验该风险模型的预测能力;如表3所示,与其他临床信息相比,该风险模型是肝癌预后的独立危险因素(p<0.05);图4显示该模型高风险组的生存率明显高于低风险组(p<0.05),随着风险评分升高,HCC患者的生存率显著降低。
图3 ①在分析了不同风险分数与患者的随访时间、事件及各个基因的表达变化的关系后,可以观察到患者的生存率随着风险分数的增加明显下降。
②该模型森林图显示BIRC5是肝癌预后的独立危险因素,模型整体具有显著意义,③生存分析曲线显示高风险组生存力显著低于低风险组。
3.讨论
目前自噬与HCC的发生发展关系研究主要集中通路抑制,基因突变,耐药等多方面,但ARGs对肝癌的具体作用仍不明确,临床意义也有待研究;大部分基于生物信息学分析ARGs的研究更多的是研究单基因的表达差异与肝癌预后的影响,预测能力有限;本研究从TCGA数据库中获取肝癌数据集并采用三种R软件包分析了肝癌组织与癌旁组织中表达具有差异性的ARGs,避免了单一R软件包对ARGs的漏筛,更好的展现ARGs在肝癌中的表达情况;由此基于差异表达ARGs构建了HCC风险预测模型对肝癌患者生存时间进行预测,最终纳入2个与肝癌患者预后生存时间显著相关的ARGs,有效的提升了ARGs的临床预测价值。
本研究分析了TCGA数据库中肝癌数据集的表达谱数据,研究结果显示了ARGs在肝癌中的表达水平以及预测肝癌预后的作用。研究共得到19个差异ARGs,其中11个基因在肝癌组织的表达量上调,8个基因表达下调。通多单因素COX回归分析发现BIRC5、CDKN2A、TP73、CLN3与肝癌患者预后生存时间显著相关,为进一步缩小范围,采用多因素COX分析ARGs表达量与生存时间的关系,最终确定了BIRC5与CLN3两个差异ARGs,并利用这两个基因建立了肝癌风险预测评分模型。随后对模型的预测能力进行检验,生存分析显示通过该风险模型计算所得高风险组生存时间显著低于低风险组,表明该模型在HCC患者生存时间预测上具有一定的临床价值。
BIRC5(baculoviral inhibitor of apoptosis repeat-containing protein 5)是一种凋亡抑制蛋白,主要参与细胞凋亡抑制[4],BIRC5在多数肿瘤中高表达,通过调控细胞凋亡与细胞转化影响肿瘤迁移、复发,同时也参与血管生成,细胞有丝分裂与肿瘤耐药等,BIRC5是所有凋亡抑制的调控因子中功能最强的因子[5],基于这些特性BIRC5成为抑制凋亡靶向因子的研究热点,有研究表明BIRC5在肿瘤组织中表达量升高并对HCC患者的预后预测有着显著影响[6],与本研究得到的结果类似;CLN3是定位于核内体和溶酶体上的抗凋亡蛋白,CLN3通过抑制内源性神经酰胺作用而表达抗凋亡作用[7],Marcel Klein等[8]研究发现其与细胞内运输和调控自噬有关,在CLN3基因敲除的细胞中,溶酶体活性与溶酶体数量都出现了降低,而自噬体数量升高,提示CLN3可能通过溶酶体系统影响细胞 自噬。但BIRC5与CLN3对肝癌预后生存的影响尚未明确,有待进一步研究。
综上所述,本研究基于TCGA数据库肝癌数据集确定了ARGs的表达水平与分布状况,并对差异ARGs的生物学功能及对肝癌患者预后影响进行了深入探索,发现了19个在肝癌组织中表达具有差异性的自噬基因,并确定了2个与肝癌患者预后生存时间显著相关的自噬基因,建立了HCC风险预测模型,该模型有待在临床一线中进一步验证其准确性,帮助临床医生制定更加精准的治疗策略。
参考文献:
[1] Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424.
[2] Chen W, Zheng R, Baade P D, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-32.
[3] Mao Y, Da L, Tang H, et al. Hepatitis B virus X protein reduces starvation-induced cell death through activation of autophagy and inhibition of mitochondrial apoptotic pathway[J]. Biochem Biophys Res Commun, 2011, 415(1): 68-74.
[4] Duma N, Santana-Davila R, Molina J R. Non-Small Cell Lung Cancer: Epidemiology, Screening, Diagnosis, and Treatment[J]. Mayo Clin Proc, 2019, 94(8): 1623-1640.
[5] Shang X, Liu G, Zhang Y, et al. Downregulation of BIRC5 inhibits the migration and invasion of esophageal cancer cells by interacting with the PI3K/Akt signaling pathway[J]. Oncol Lett, 2018, 16(3): 3373-3379.
[6] Wang L, Huang J Fau - Jiang M, Jiang M Fau - Sun L, et al. Survivin (BIRC5) cell cycle computational network in human no-tumor hepatitis/cirrhosis and hepatocellular carcinoma transformation[J](1097-4644 (Electronic)).
[7] 张春树, 吕静, 毛东伟. CLN3基因在卵巢浆液性囊腺癌中的表达及耐药相关性的研究[J]. 中国优生与遗传杂志, 2015, 23(10): 13-15+140.
[8] Yasa S, Modica G, Sauvageau E, et al. CLN3 regulates endosomal function by modulating Rab7A-effector interactions. LID - jcs234047 [pii] LID - 10.1242/jcs.234047 [doi][J](1477-9137 (Electronic)).
课题名称:HBX基因突变调控PI3K/Akt/mTOR通路对肝癌細胞自噬的作用及其机制研究
课题编号:2019GXNSFDA245001
课题类型:广西自然科学基金

