食管鳞状细胞癌关键基因筛选与生物学途径分析
陆必燊,晏峻峰
(湖南中医药大学信息科学与工程学院,中国湖南 长沙 410208)
食管癌(esophageal carcinoma,EC)是一种常见的恶性肿瘤,其发病率全球排名第七,总死亡率排名第六,被认为是每年影响50万人的第六大恶性肿瘤[1~2]。食管早期癌在组织学上可分为腺癌(adenocarcinoma,AC)和鳞状细胞癌(squamous cell carcinoma,SCC)[3]。食管鳞状细胞癌(esophageal squamous cell carcinoma,ESCC)在亚洲更为常见,是一种致死性恶性肿瘤,生存率低[4~5]。在食管鳞状细胞癌治疗指南中,金标准是经胸食管次全切除加双野淋巴结切除术[6]。虽然手术切除联合辅助化疗是治疗食管鳞状细胞癌的一种有效途径,但手术与化疗带给病人的痛苦仍是巨大的。
近年来,食管鳞状细胞癌的生物标志物成为研究重点。已有研究发现,一些生长因子和相应的受体,如表皮生长因子和转化生长因子,与食管鳞状细胞癌的发生及患者预后效果相关[7]。因此,深入研究食管鳞状细胞癌细胞恶性生物学行为的潜在分子机制,将有助于鉴定可靠的分子标记物,对早期诊断、预后评估、复发监测、控制食管癌细胞增殖和新药靶标的探索非常重要。
本研究运用R语言及其相关软件包,选取GEO数据库中5个类型为阵列表达谱的食管鳞状细胞癌基因数据集为分析材料,筛选了食管鳞状细胞癌与癌旁非肿瘤组织之间的差异表达基因(differentially expressed gene,DEG),进行了GO(Gene Ontology)与KEGG(Kyoto Encyclopedia of Genes and Genomes)富集分析,并通过构建蛋白质相互作用网络(protein-protein interaction network,PPI network),筛选出食管鳞状细胞癌密切相关的关键基因与功能模块,以期能为食管鳞状细胞癌的诊断提供潜在的分子标记和治疗靶标。
1 材料与方法
1.1 材料
从GEO数据库[8](https://www.ncbi.nlm.nih.gov/geo/)下载编号为 GSE20347、GSE29001、GSE33426、GSE45168和GSE70409的食管鳞状细胞癌基因表达谱矩阵文件以及对应的平台文件。GSE20347数据集[9]基于GPL571平台,共有34例样本,包含17例食管鳞状细胞癌组织和17例正常食管组织。GSE29001数据集[10]共45例样本,包含24例正常食管组织和21例癌症组织,其平台为GPL571。GSE33426数据集[11]共有71例样本,包含12例正常组织和59例食管鳞状细胞癌组织,同样为GPL571平台。GSE45168数据集基于GPL13497平台,共10例样本,正常食管组织和癌症组织各5例。GSE70409数据集[12]基于GPL13287平台,共34例样本,正常食管组织与癌症组织各17例。
1.2 方法
1.2.1 数据处理与差异基因筛选
依次将5个食管鳞状细胞癌芯片数据集导入R Studio软件,使用R语言命令将基因表达谱矩阵文件中的基因探针ID转换为平台文件中的基因符号,以获得包含国际标准基因名称的矩阵文件。剔除没有相对应的基因名和一个探针对应多个基因的数据,对多个探针对应一个基因的数据取均值。使用limma R软件包对每个数据集进行标准化处理[13],并将所有基因表达数据进行转化。
本研究根据下载的GEO数据集中样本性状信息,将样本分为正常组织组以及癌症组织组,以校正后的 P<0.05 和|log2FC|>1(FC:fold change)为筛选标准,使用limma R软件包筛选每个数据集中的差异表达基因。使用RobustRankAggreg(RRA)R软件包整合按值排序的所有基因列表的5个数据文件[14]。
1.2.2 差异表达基因的GO以及KEGG富集分析
DAVID 6.8 数据库(https://david.ncifcrf.gov/)是用于基因富集和功能注释分析的常用数据库[15]。该数据库整合了生物学数据和分析工具,可为大规模的基因或蛋白质列表提供系统而全面的生物学功能注释。本研究采用DAVID对已识别的差异表达基因进行GO注释和KEGG通路富集分析,分析结果以P<0.05作为纳入标准。
1.2.3 PPI网络构建与hub基因及功能模块筛选
将筛选的全部差异表达基因导入STRING 11.0数据库,构建食管鳞状细胞癌差异表达基因的PPI网络图[16]。应用Cytoscape 3.7.2软件对KEGG通路分析结果和STRING数据库分析的相互作用数据进行可视化网络分析。基于Cytoscape 3.7.2软件,cytoHubba插件用于筛选食管鳞状细胞癌hub基因,MCODE(Molecular Complex Detection)插件用于筛选PPI网络中的功能模块[17]。对筛选的功能模块进行通路富集分析。利用jvenn网站(http://jvenn.toulouse.inra.fr/app/index.html)绘制hub基因的韦恩图[18]。
1.2.4 关键基因验证
利用GEPIA[19](http://gepia.cancer-pku.cn/)对筛选的关键基因在食管鳞状细胞癌组织与正常组织中的表达情况进行验证。其中,癌症组织样本选择TCGA(The Cancer Genome Atlas)数据,正常组织样本选择TCGA和GTEx(Genotype-Tissue Expression)中的数据,箱图抖动大小设置为0.4,以|log2FC|>1和P<0.05作为检验阈值。
2 结果
2.1 数据处理与差异表达基因筛选结果
对食管鳞状细胞癌芯片表达数据集GSE-20347、GSE29001、GSE33426、GSE45168 和 GSE-70409进行归一化处理。差异表达基因筛选结果表明,GSE20347数据集包含1 007个差异表达基因,其中上调基因453个,下调基因554个;GSE29001数据集筛选出1 909个差异表达基因,包括1 015个上调基因和894个下调基因;GSE-33426数据集筛选出3 811个差异表达基因,涉及1 992个表达上调的基因和1 819个表达下调的基因;GSE45168数据集筛选出1 365个差异表达基因,包括583个表达上调的基因和782个表达下调的基因;GSE70409数据集包含1 817个差异表达基因,其中上调基因829个,下调基因988个。
采用RRA软件包筛选5个数据集整合后的差异表达基因,通过等级分析鉴定了373个共同的差异表达基因,其中包括154个表达上调的基因和219个表达下调的基因。图1展示了排名前20的上调/下调基因。

图1 RRA整合后的前20个上调和下调的差异表达基因热图横坐标表示数据集ID,纵坐标表示基因名称,红色表示log2FC>0,绿色表示log2FC<0。Fig.1 Heat map of the top 20 up-and down-regulated DEGs after RRA integrationAbscissa represents dataset ID,ordinate represents gene name,red indicates log2FC>0,and green indicates log2FC<0.
2.2 差异表达基因的GO以及KEGG富集分析结果
整合的差异表达基因的GO功能分析分为以下3个部分:生物过程(biological process,BP)、分子功能(molecular function,MF)和细胞组分(cellular component,CC)。图2和图3显示了整合的差异表达基因的不同GO功能富集分布。上调与下调的差异表达基因的前15个GO富集分析结果如表1和表2所示。上调的基因主要富集在细胞外基质组织(ontology:BP)、细胞质(ontology:CC)和蛋白质结合(ontology:MF),而下调的基因主要富集在氧化还原过程(ontology:BP)、细胞外外泌体(ontology:CC)和钙离子结合(ontology:MF)。KEGG通路分析结果显示,整合的差异表达基因主要集中在以下5个途径:阿米巴病、PI3K-Akt信号转导通路、ECM-受体相互作用、黏着斑和蛋白质消化吸收(表3,图4)。使用Cytoscape软件绘制富集通路网络图,结果如图5所示。

图2 整合后上调的差异表达基因的GO富集分布Fig.2 GO enrichment and distribution of up-regulated DEGs after integration
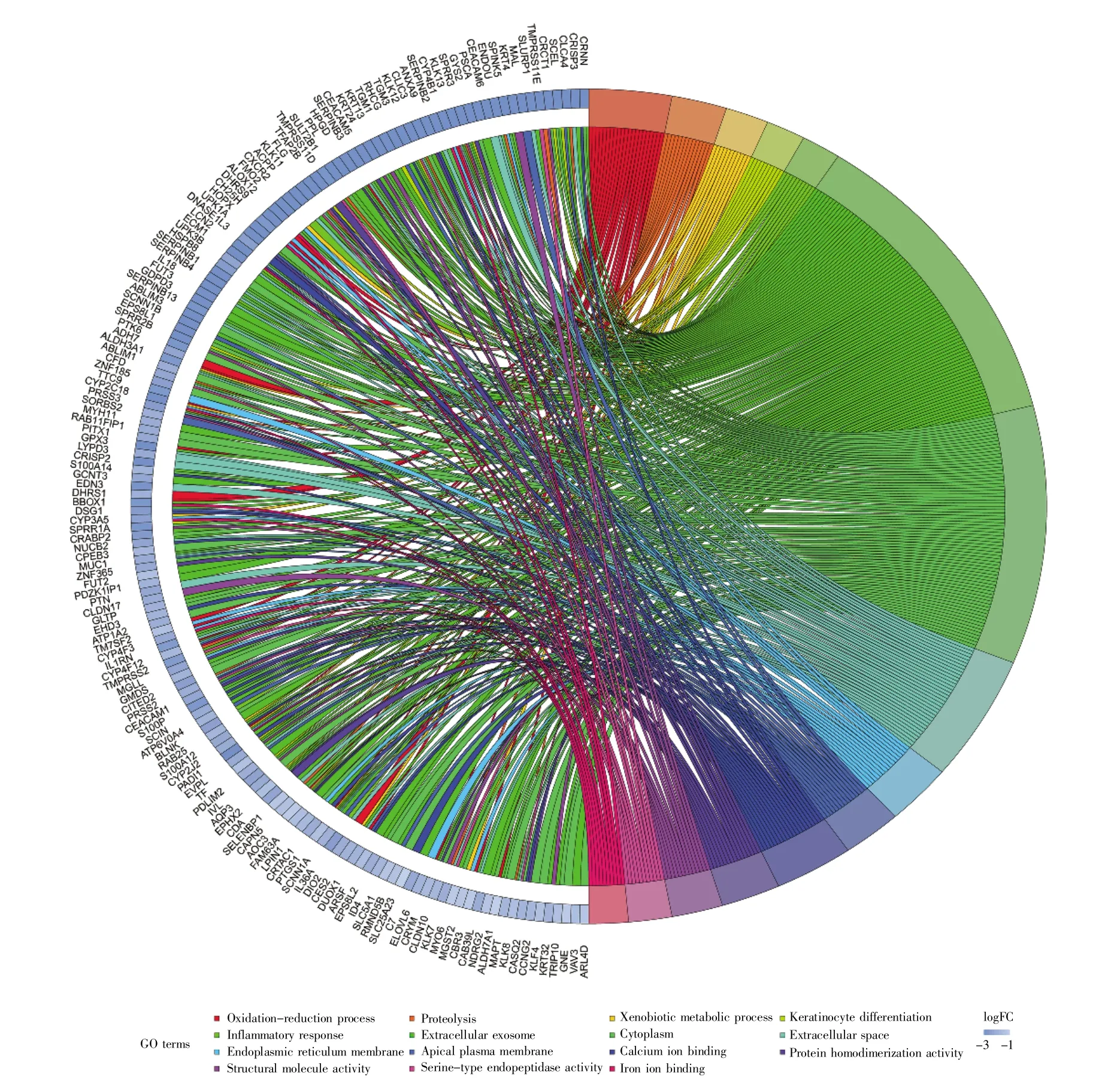
图3 整合后下调的差异表达基因的GO富集分布Fig.3 GO enrichment and distribution of down-regulated DEGs after integration

表1 与上调基因相关的前15个GO富集术语Table 1 Top 15 GO enrichment terms related to up-regulated genes

表2 与下调基因相关的前15个GO富集术语Table 2 Top 15 GO enrichment terms related to down-regulated genes

表3 整合后的差异表达基因的KEGG通路富集分析Table 3 KEGG pathway analysis of integrated DEGs
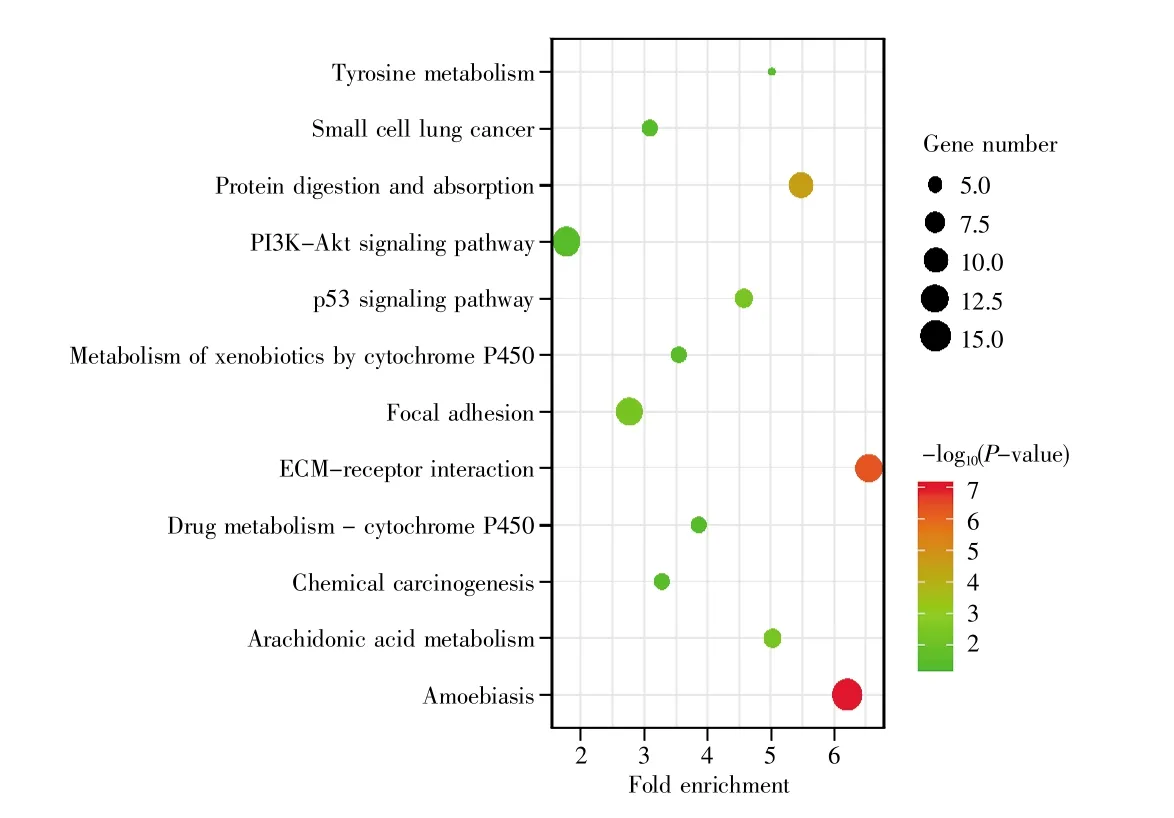
图4 整合后的差异表达基因的KEGG通路富集分析Fig.4 KEGG pathway enrichment analysis of integrated DEGs

图5 富集通路网络图蓝色代表通路,红色代表上调的基因,绿色代表下调的基因。Fig.5 Network diagram of enrichment pathwaysBlue represents pathways,red represents up-regulated genes,and green represents down-regulated genes.
2.3 Hub基因与功能模块筛选结果
将筛选得到的373个差异表达基因输入STRING数据库,构建PPI网络。下载结果并使用Cytoscape软件的cytoHubba插件筛选 Degree、MCC(maximal clique centrality)、MNC(maximum neighborhood component)、EPC(edge percolated component)4种算法得分靠前的15个hub基因,结果如表4所示。利用jvenn网站绘制4种算法筛选出的hub基因的韦恩图,得到7个共同hub基因:CDK1、KIF20A、TTK、CDC45、CCNB2、TPX2、KIF4A(图6)。

表4 Degree、MCC、MNC、EPC算法筛选的前15个hub基因Table 4 Top 15 hub genes screened by Degree,MCC,MNC,and EPC algorithms

图6 Degree、MCC、MNC、EPC算法筛选的前15个hub基因的韦恩图Fig.6 Venn diagram of the top 15 hub genes screened by Degree,MCC,MNC,and EPC algorithms
此外,使用Cytoscape软件的MCODE插件从PPI网络中筛选了11个功能模块,其中MCODE得分靠前的两个功能模块如图7所示。对这两个功能模块进行通路富集分析,结果显示:模块A的基因主要富集在细胞周期、p53信号通路和卵母细胞减数分裂;模块B的基因主要富集在ECM-受体相互作用、蛋白质消化吸收、阿米巴病、黏着斑、PI3K-Akt信号通路。

图7 两个PPI网络功能模块Fig.7 Two PPI network function modules
2.4 关键基因验证
通过GEPIA对筛选得到的关键基因进行分析,结果显示:CDK1、KIF20A、TTK、CDC45、CCNB2、TPX2、KIF4A在食管鳞状细胞癌中均表达增高(图8)。

图8 关键基因表达水平的验证结果红色代表肿瘤组织,灰色代表正常组织。Fig.8 Validation results of key gene expression levelsRed represents tumor tissue,and gray represents normal tissue.
3 讨论
在食管鳞状细胞癌的早期阶段对患者进行检测可以提高诊断的准确性,促进个性化治疗并改善预后的效果。然而,食管鳞状细胞癌的主要发生原因尚不清楚。本研究通过分析GSE20347、GSE29001、GSE33426、GSE45168和GSE70409五个基因芯片数据集,筛选得到373个食管鳞状细胞癌相关的差异表达基因,其中154个表达上调基因和219个表达下调基因。GO功能分析发现,差异表达基因主要参与细胞外基质组织、细胞质、蛋白质结合、氧化还原过程、细胞外外泌体和钙离子结合等。KEGG通路分析发现,差异表达基因主要富集在阿米巴病、PI3K-Akt信号通路、ECM-受体相互作用、黏着斑和蛋白质消化吸收等通路。PPI网络和韦恩图法共筛选得到以下7个关键基因:CDK1、KIF20A、TTK、CDC45、CCNB2、TPX2、KIF4A。
CDK1(cyclin-dependent kinase 1)的相关途径包括卵母细胞减数分裂和胞外信号调节激酶(extracellular signal-regulated kinase,ERK)信号转导[20]。其编码的蛋白质是Ser/Thr蛋白激酶家族的成员,是高度保守的蛋白激酶复合物的催化亚基,被称为M期促进因子,对于真核细胞周期的G1/S和G2/M相变至关重要[21]。Hansel等[22]发现,CDC2/CDK1在食管腺癌及其前体病变中的表达既可作为诊断癌症进展的标志物,又可以作为潜在的药物治疗靶点。有研究报道,CDK1是食管鳞状细胞癌G2/M通路的调节剂,并且CDK1与其他调节剂(例如CDC25)的组合可增强对患者预后的预测[23]。
KIF20A(kinesin family member 20A)的相关途径包括JAK/STAT3信号通路,以及高尔基体到内质网的逆行转运[24]。现有研究表明,KIF20A的上调与胃癌预后不良有关[25],其过表达通过促进细胞增殖和抑制细胞凋亡导致肺腺癌恶性转移[26],但其与食管鳞状细胞癌的相关研究报道较少。
TTK(TTK protein kinase)是一种具有磷酸化酪氨酸、丝氨酸和苏氨酸能力的双重特异性蛋白激酶,对细胞分裂的调节至关重要,与细胞增殖相关,且参与中心体复制过程[27]。已有研究发现,TTK的过表达可能参与了食管鳞状细胞癌的增殖过程[28]。
CDC45(cell division cycle 45)的相关途径包括E2F介导的DNA复制和细胞周期调控,其在真核生物DNA复制的早期步骤中起着重要作用[29~30]。Huang等[31]发现,CDC45的表达敲低在体外和体内均抑制非小细胞肺癌细胞增殖,并使细胞停滞在细胞周期的G2/M期。Ke等[32]报道,RING1和YY1结合蛋白通过下调CDC6和CDC45抑制食管鳞状细胞癌的增殖,从而抑制G1/S过渡。
CCNB2(cyclin B2)的相关途径包括卵母细胞减数分裂和调控有丝分裂细胞周期阶段过渡[33],其重要旁系同源物是CCNB1。研究表明,CCNB1的表达与食管鳞状细胞癌患者的恶性肿瘤和预后有关[34],CCNB2在食管鳞状细胞癌的发生发展中可能起着重要作用[35]。
TPX2(TPX2 microtubule nucleation factor)的相关途径包括在G2/M过渡时调控PLK1(Polo-like kinase 1)活性和基因表达[36]。TPX2在多种肿瘤类型(例如子宫颈癌和胃癌)中表达上调[37],且其表达与肝细胞癌的生长和转移有关[38]。研究表明,TPX2调节食管鳞状细胞癌细胞的增殖和侵袭性[39],且其高表达与食管鳞状细胞癌患者较差的总生存期和较短的无病生存期有关[40]。
KIF4A(kinesin family member 4A)的相关途径包括表皮生长因子受体的内吞运输和p53信号通路[41]。该基因编码的蛋白质是一种基于ATP的微管运动蛋白,参与膜细胞器的细胞内运输,与浓缩的染色体臂相关联,并且可能参与有丝分裂期间染色体完整性的维持[42]。
综上所述,本文通过对食管鳞状细胞癌及癌旁组织的差异表达基因进行分析,筛选得到7个关键基因:CDK1、KIF20A、TTK、CDC45、CCNB2、TPX2、KIF4A,其中 CDK1、TTK、TPX2、CDC45、CCNB2在食管鳞状细胞癌中的作用已有研究进行报道,而KIF20A、KIF4A在食管鳞状细胞癌中的相关研究较少。通过生物信息学方法,对食管鳞状细胞癌相关基因数据进行分析,将有助于增强人们对食管鳞状细胞癌病症发生发展机制的了解;分析得到的关键基因与生物学通路,将有望为食管鳞状细胞癌的早期诊断、预后评估及靶向治疗提供新思路。