牦牛与普通牛、水牛的X染色体基因编码区比较及密码子偏性分析
刘欣睿,王嘉博,柴志欣,武志娟,益西康珠,钟金城
(西南民族大学,青藏高原动物遗传资源保护与利用四川省教育部重点实验室,成都 610041)
牦牛(Bosgrunniens)是生活在青藏高原及其周边横断山脉地区特有畜种,为高原农牧民提供了基本的生活资源[1]。氧分压降低会导致机体组织氧气供应不足,影响动物的正常生理功能[2]。生活在高海拔地区的物种通过长期选择进化出了特殊的生理机制和形态特征,以适应高原恶劣生态环境。如硕大的肺脏和厚实的心血管壁,通过消除缺氧血管收缩驱动来适应低压缺氧[3],降低能量代谢[4]及抵抗低压低氧环境的其他生理反应等[5]。相反,普通牛(Bostaurus)及江河型水牛(Bubalusbubalis)在牦牛居住的高原地区饲养时,会遭受严重的肺动脉高压[6],使其无法适应高原生存和饲养环境。
在哺乳动物的进化历程中,性染色体较常染色体表现出明显的结构和形态差异,而性染色体在进化过程中常由常染色体转化而来,性染色体和常染色体在分子进化和种群遗传中存在差异,主要体现在性染色体在进化过程中表现出更高的遗传漂变敏感性[7],它的大部分位点更容易受到选择压力的影响,这使得性染色体上被自然选择固定下来的效应较常染色体要高[8]。其中X染色体的剂量依赖机制决定哺乳动物的性别,但相关机制尚不清楚[9]。X染色体与Y染色体不同,其受到雌性个体在减数分裂期间的X-X重组保护,使X染色体几乎保留了祖先的所有基因[10]。牛亚科动物X染色体的基因含量高度保守,雌性个体的遗传变异更多固定在X染色体上[11]。反刍动物个体在性染色体的组成方面具有差异,雌性为XX,雄性为XY,因此X染色体的重组和连锁现象仅发生在雌性个体上,这使得性染色体的重组率远低于常染色体[8],导致X染色体在进化过程中更容易保留物种特异性。
比较基因组分析是一种识别评估不同物种遗传特性和基因结构的常用工具,对深入挖掘生物学功能具有重要意义。密码子偏好性是广泛存在于生物体内的一种在编码氨基酸时偏好使用某特定同义密码子的现象[12],密码子的偏性常由突变和自然选择共同作用。目前,绝大部分关于牦牛染色体的研究都集中于常染色体[13-14]、Y染色体[15]以及线粒体DNA[16]。本研究从比较基因组学和密码子偏性水平阐述牦牛与普通牛、水牛X染色体基因结构、共线性关系及密码子偏性差异,有助于了解牦牛及其近缘物种基因的改变、缺失或获得,以及系统进化地位,为进一步解析牛亚科动物品种差异及牦牛高原适应性提供参考,同时研究密码子偏好性对优化密码子和提高目标蛋白表达也具有重要意义。
1 材料与方法
1.1 测序数据整理
本研究以牦牛、普通牛(源于Ensembl数据库,http:∥asia.ensembl.org)和水牛(源于NCBI数据库,https:∥www.ncbi.nlm.nih.gov)为材料(表1)。利用自编Perl脚本[17]进行过滤获得牦牛38 325 bp、普通牛33 376 bp、水牛57 536 bp的基因序列用于密码子偏性分析,其过滤条件为:①以ATG为起始密码子,TAG、TGA、TAA为终止密码子[18];②基因的碱基数量为3的倍数;③删除序列长度<300 bp的序列;④剔除重复基因序列。
1.2 基因组对比分析
牦牛、普通牛、水牛X染色体的CDS文件由一个自编bash脚本提取得到,并针对以下3点进行修改:①剔除重复基因,计算基因序列长度,保留最长序列;②根据位置信息和序列长度,去掉内含子坐标,修改抬头坐标信息;③修改坐标信息后的基因序列呈首尾相连的长串基因组,各基因序列间添加10 bp的NA片段消除基因间的相互影响(图1)。
以处理后的牦牛CDS文件建库,与普通牛、水牛进行BLAST比对,使用BLAST Ring Image Generator(BRIG v0.95)(http:∥brig.sourceforge.net/)[19]进行可视化,使用fasta文件的抬头信息为BRIG图添加注释信息,其注释信息在最外层标注,BLAST比对的E-value阈值设定为1e-5,其余采用默认参数。相关基因功能均从NCBI(https:∥www.ncbi.nlm.nih.gov/gene/)模块获得。

表1 物种数据来源

图1 牦牛文件的修改模式图Fig.1 Modification pattern diagram of file of Bos grunniens
1.3 基因共线性分析
提取BLAST结果中牦牛特异区段附近20 kb序列,剔除匹配长度<100 bp、匹配度<95%及抬头信息无基因名注释的比对区段。使用R包genoplotR[20]基因共线性可视化,构建的3种牛亚科动物的dna_seg基因组结构文件中以strand模块[1/-1]表示正链和负链,图中以箭头方向为5′→3′端区分基因方向;comparison结构比较对象文件以direction模块[1/-1]表示对比区段是否正向匹配,图中以交换线段表示。
1.4 密码子偏性分析
1.4.1 密码子使用偏性的测量 使用CodonW 1.4.2软件(http:∥codonw.sourceforge.net/)分析3种牛科动物X染色体的CDS序列,获得相应的有效密码子数(effective number of codon,ENC)[21]、同义密码子第3位碱基GC含量(CG3s)、同义密码子第3位碱基TCAG含量(T3s,C3s,A3s,G3s)、鸟嘌呤和胞嘧啶占比(GC%)、密码子适应指数(codon adaptation index,CAI)、密码子偏好性指数(codon bias index,CBI)、蛋白质疏水指数(Gravy)、芳香族氨基酸含量(Aromo)及各密码子在生物体内的使用频数,使用R统计计算相对同义密码子使用频率(relative synonymous codon usage,RSCU)[22]、平均值及标准差,并使用R包pheatmap、ggplot2等对数据可视化。 RSCU值能有效反映密码子的偏性情况,RSCU=1,表示该密码子无偏倚性;RSCU>1,表示该密码子的使用频率高于同义密码子(预期使用频率);RSCU<1,表示该密码子的使用频率低于同义密码子。RSCU的计算公式为:

(1)
式中,fij是第j个氨基酸的第i个密码子出现的频率;ni是同义密码子的个数。
1.4.2 ENC-plot绘图密码子偏性分析 使用R包ggplot2,以GC3s为横坐标、ENC值为纵坐标绘制ENC-plot散点图。ENC-plot能直观表示出自然选择及其他因素对密码子偏性的影响[23]。ENC的标准曲线表示在不存在自然选择的情况下密码子的偏性情况[24],若密码子的偏性更多受自然选择的影响应该坐落于标准曲线的下方;越靠近标准曲线表明选择压力对密码子的偏性影响越小。其中ENC值能反映密码子中同义密码子非均衡使用的偏好性程度,ENC的取值介于20~61之间,ENC值越大编码氨基酸时密码子使用偏性越强[25],越小偏性则越弱。ENC标准曲线计算及绘制公式:
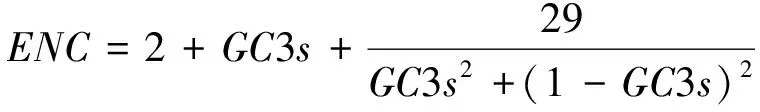
(2)
1.4.3 PR2-plot密码子偏性分析 密码子第3位碱基的组成由于突变影响具有一定的偏性,PR2-plot能很好地分析第3位碱基ATCG之间的偏性情况[26]。如果两条链的DNA均未发生突变和自然选择偏差,那么预期的绘制点将均匀地分布于线段(x=0.5,y=0.5)的左右,即碱基A=T,C=G。通常来讲密码子不会只受到突变的影响而使散点均匀的分布在偏性分析图的4个区域,当第3位碱基使用频率A/T不平衡时散落在图的上下方,C/G不平衡则散落在左右方,而平面图中心表示A=T,G=C[27]。
1.4.4 最优密码子的确定 构建高低密码组按照ENC值对CDS序列从大到小进行排序,提取前10%作为高表达密码子组,后10%作为低表达密码子组,分别计算各组RSCU值及ΔRSCU=RSCU(高表达组)-RSCU(低表达组)[28]。确定最优密码子需满足:①属于高频密码子(RSCU>1);②属于高表达密码组(ΔRSCU>0.08)。
2 结 果
2.1 基因组对比分析
为了阐述牦牛X染色体序列组成特点,以及与普通牛、水牛X染色体序列的差异,以牦牛X染色体序列为参考,分别与普通牛及水牛该序列进行比较分析,发现普通牛和水牛未匹配的20~40、400~420、570~590和950~970 kb 4个区段为牦牛特异区段。其中牦牛与普通牛的匹配度最高,水牛与牦牛之间除牦牛特异区段外,在280~300、510~530、840~860和1 090~1 100 kb区段也发现了长段未匹配区。通过基因组注释,在4个牦牛特异区段中3种牛亚科动物均能匹配到在20~40 kb区段的IL13RA1、DOCK11、WDR44、CT83、AGTR2和KLHL4基因;400~420 kb区段的HNRNPH2、GLA、RPHL36A-HNRNPH2、BTK、TIMM8A、TAF7L、DRP2、CENPI、TMEM35A、XKRX、NOX1和CSTF2基因;570~590 kb区段的CYSLTR1、TAF9B、PGK1、ATP7A、MAGT1和ATRX基因;950~970 kb区段的CXorf38、MID1IP1、OTC、SYTL5和CYBB基因,其中缺失区段被一个标记为unknown的标签注释在图中(图2)。
2.2 基因共线性分析
使用genoplotR包对提取的4个20 kb片段进行基因共线性分析。在20~40 kb区段牦牛所有基因均与普通牛、水牛具有相反排列。牦牛DOCK11基因较普通牛短,与水牛相近;牦牛KLHL13基因长度较普通牛和水牛长(图3A)。在400~420 kb区段3种牛的基因长度相近,呈现出复杂的基因线性;与普通牛和水牛相比,牦牛BTK、TIMM8A、TAF7L和XKRX基因在进化过程中呈现与前端CENPI、TIMM35A、NOX和CSTF2基因的排列交换;水牛与普通牛呈现碱基的相反排列(图3B)。570~590 kb区段牦牛与普通牛、水牛均呈现高度一致性,但水牛与牦牛、普通牛呈现碱基的相反排列(图3C),牦牛ATRX基因较普通牛、水牛长,牦牛TAF9B及ATP7A基因较另2种牛科动物短;在950~970 kb区段,牦牛与普通牛一致度较高,牦牛SYL5、CYBB基因较普通牛长,水牛仍与牦牛、普通牛具有相反的碱基排列,且水牛未匹配基因CYBB的线性区段(图3D)。
2.3 密码子偏性
Seqtk软件分析结果显示,牦牛X染色体(cDNA)长度最短为136 336 377 bp,普通牛为139 009 144 bp,水牛的最长为136 336 377 bp,长度差异可能与密码子偏性有关。
3种牛亚科动物的ENC值均不存在显著性差异,均值在50.33~50.76,ENC值均偏大(表2)。牦牛X染色体编码区的第3位碱基(ACG)含量较普通牛、水牛差异均不显著(P>0.05),第3位碱基T含量、GC3s值、GC含量、CBI值、Gravy值和Aromo值均较水牛差异显著(P<0.05),与普通牛差异不显著(P>0.05)。3种牛亚科动物X染色体基因组编码区的密码子GC含量均>0.5,表明X染色体编码区序列中GC含量>AT含量;密码子均偏向以G/C为结尾,第3位密码子以G/C结尾的偏性为牦牛<普通牛<水牛。
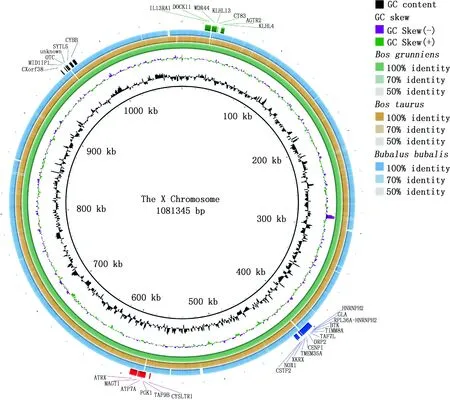
图2 牦牛、普通牛和水牛X染色体的比较基因组分析Fig.2 Comparative genome analysis of X chromosome of Bos grunniens,Bos taurus and Bubalus bubalis

A,20~40 kb片段的基因线性;B,400~420 kb片段的基因线性;C,570~590 kb片段的基因线性;D,950~970 kb片段的基因线性A,Gene linearity of that 20-40 kb fragment;B,Gene linearity of that 400-420 kb fragment;C,Gene linearity of that 570-590 kb fragment;D,Gene linearity of that 950-970 kb fragment图3 基因共线性分析Fig.3 Gene collinearity analysis

表2 CodonW分析的牛亚科基因组密码子偏性
2.4 RSCU分析
由表3可知,3种牛亚科动物X染色体基因编码区密码子RSCU值无明显差异,其中牦牛、普通牛和水牛的RSCU范围分别为0.341~2.099、0.366~2.007和0.379~2.000。牦牛有32个密码子具有偏好性(RSCU>1),普通牛有33个,水牛有31个。其中牦牛有15个密码子以CG结尾,普通牛有16个,水牛有20个。牦牛、普通牛及水牛强偏性密码子(RSCU≥1.5)均为CUG、GUG、AGA、AGG和UGA(图4A),由翻译各氨基酸的密码子占比可知(图4B),3种牛亚科动物X染色体基因编码区密码子RSCU值分布较为一致,其中强偏性密码子均分布在亮氨酸、精氨酸和缬氨酸3个氨基酸,且终止密码子中UGA具有较强偏好性。
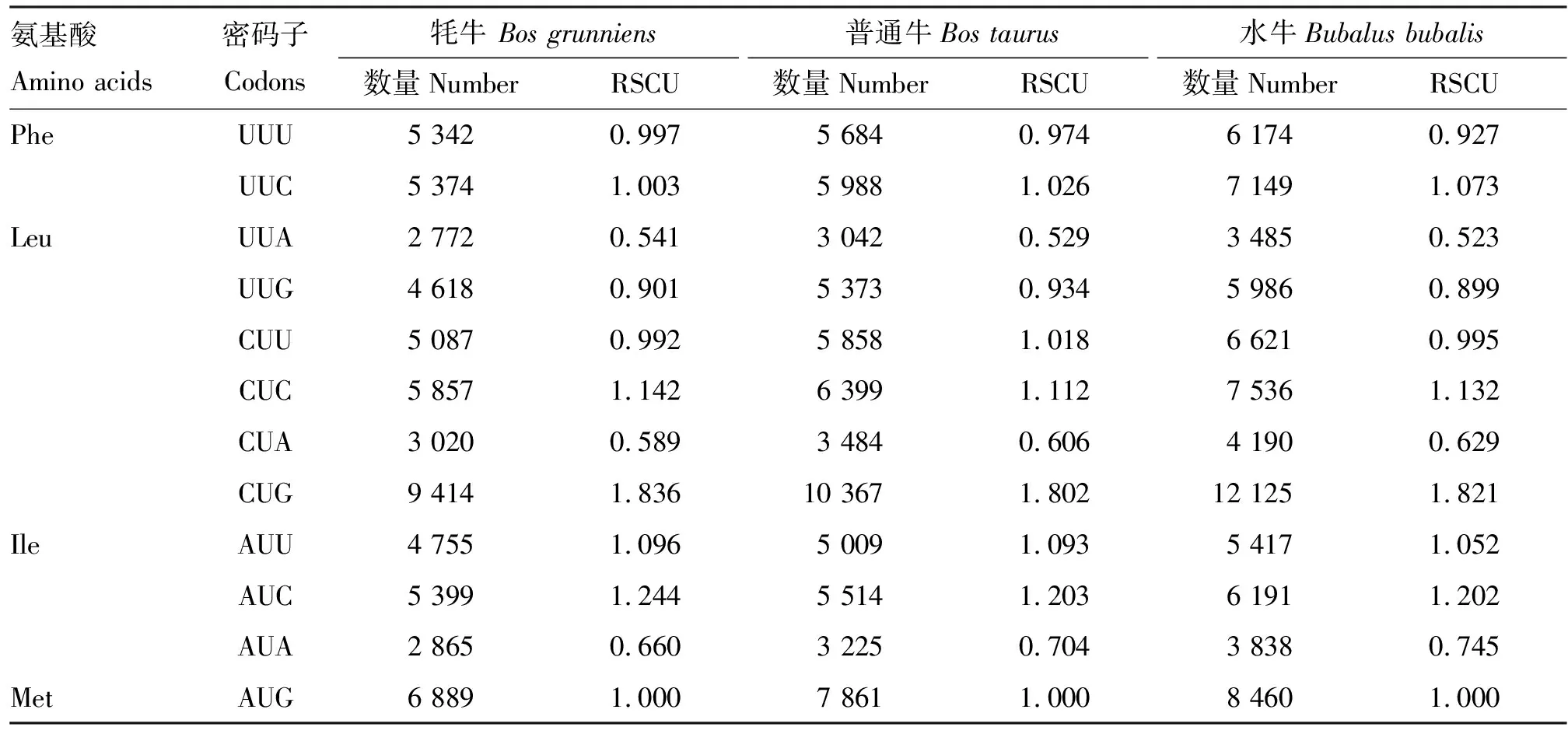
表3 牛亚科各氨基酸的 RSCU 分析
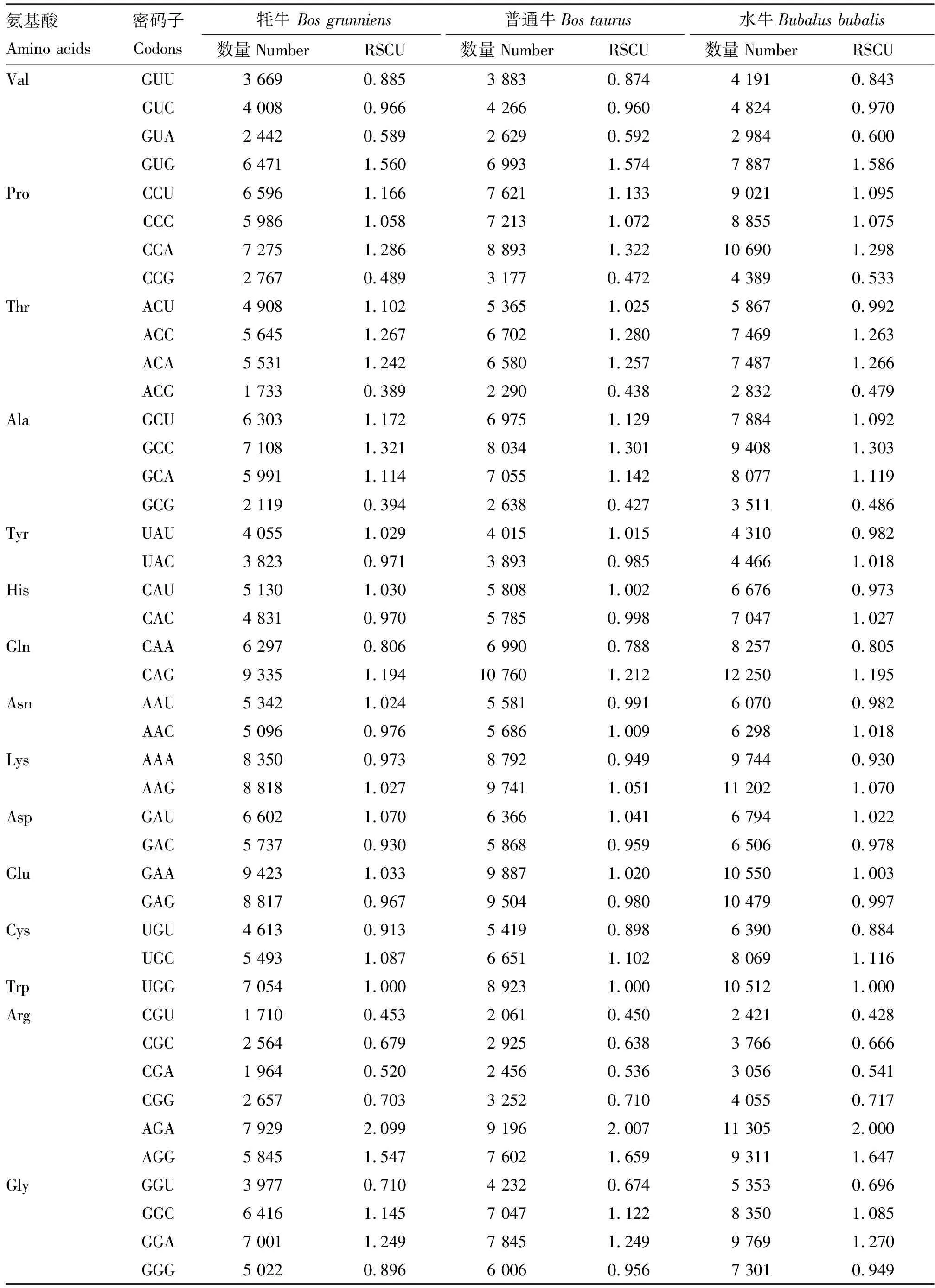
续表

续表

A,RSCU值热图分析,颜色越偏向于黄色和红色,说明该密码子的偏性越强;B,翻译各氨基酸的密码子占比,以百分比表示(左),密码子RSCU值的曲线图(右)A,Heatmap of RSCU,the color in the figure is more biased towards yellow and red,indicating that the corresponding codon bias is stronger;B,The proportion of codons translated for each amino acid,expressed as a percentage (left),and the graph of the RSCU value of codons (right)图4 RSCU分析Fig.4 RSCU analysis
2.5 ENC-plot分析
结果显示,3种牛亚科动物大部分绘制点均坐落于ENC标准曲线的下方(图5A),且牦牛绘制点相对集中,水牛相对分散。为了进一步说明3种牛亚科动物的差异,对ENC比值频数进行了统计,结果表明无论是牦牛、普通牛还是水牛的ENC比值均集中在0.05~0.15、0.15~0.25、0.25~0.35 3个组段(表4),牦牛在-0.05~0.15组段较另两者显示更为集中。
2.6 PR2-plot分析
结果显示,多数绘制点落座于X=0.5左侧,说明第3位碱基中检测到了碱基C/G的偏性,即C>G;第3位碱基A/T无明显偏性(图5B),结果与表2分析一致。3种牛亚科动物的密码子偏性不仅受突变影响,还受到一些如自然选择及其他微小因素影响。
2.7 最优密码子
牦牛中有32个偏好密码子(RSCU>1),其中有16个为最优密码子(此处仅将牦牛的结果列于表5):AUU、CCU、CCA、ACU、ACA、GCU、GCA、UAU、CAU、AAU、GAU、GAA、AGA、GGA、UCU和UCA,9个以U结尾,7个以A结尾;普通牛的33个偏好密码子中有13个最优密码子:CUU、AUU、CCU、ACU、ACA、GCU、GCA、UAU、GAU、GAA、AGA、GGA和UCU,8个以U结尾,5个以A结尾;水牛的31个偏好密码子中有9个最优密码子:AUU、CCU、GCU、GAU、GAU、GAA、GGA、UCU和UCA,6个以U结尾,3个以A结尾。牦牛、普通牛及水牛最优密码子均偏好于以A/U结尾,无C/G结尾的密码子,且牦牛较普通牛和水牛具有更多最优密码子。
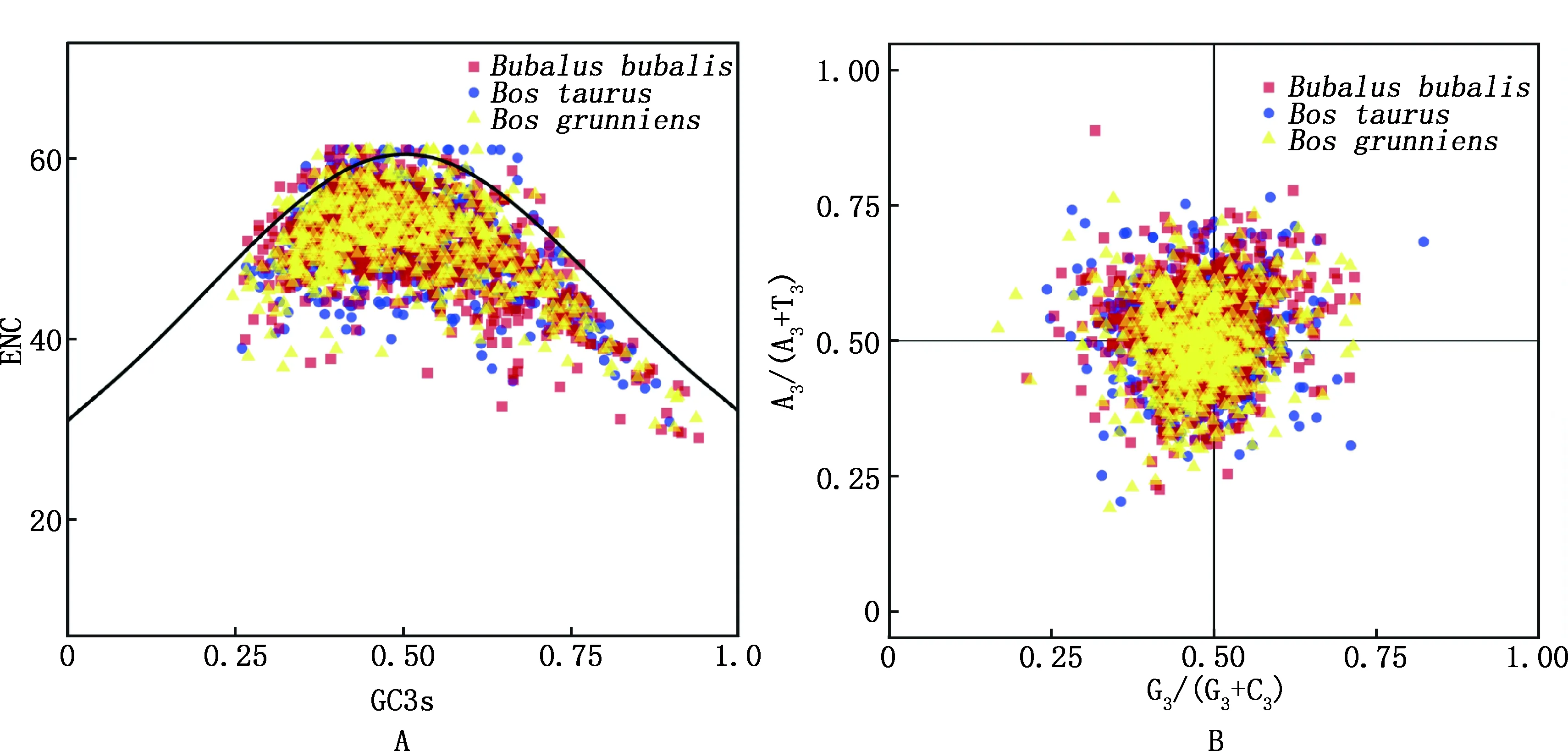
A,ENC-plot;B,PR2-plot图5 密码子偏性分析Fig.5 Codon bias analysis
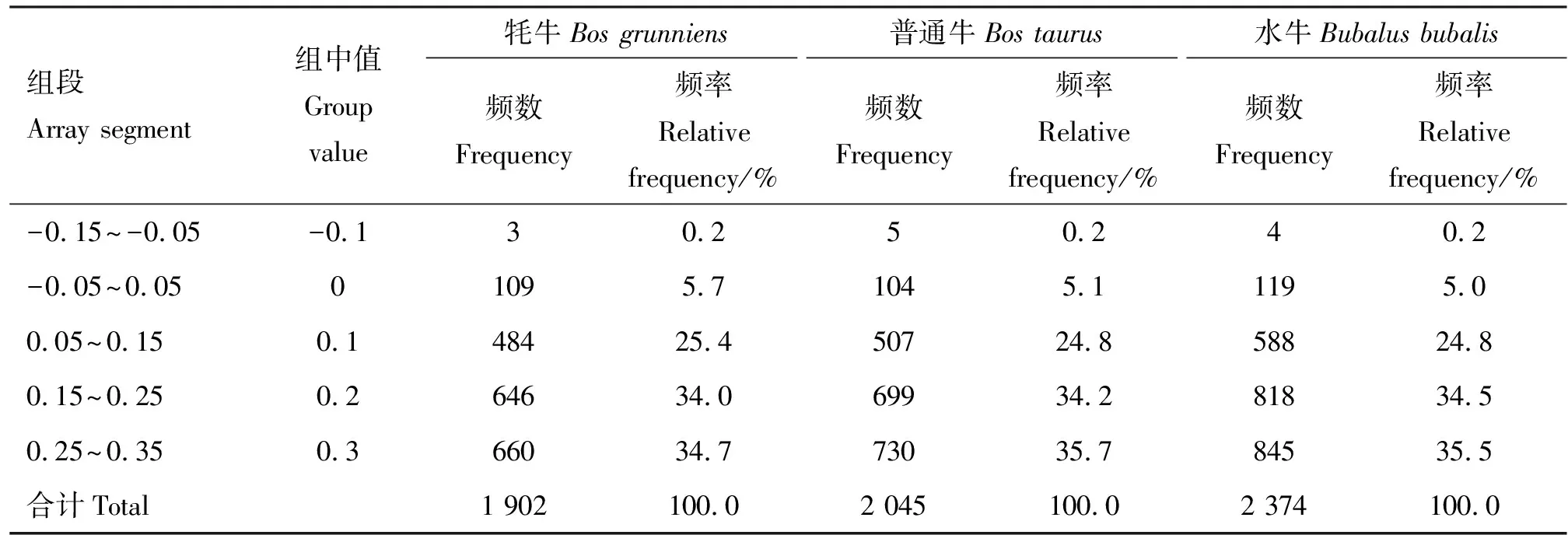
表4 ENC比值频数分布表
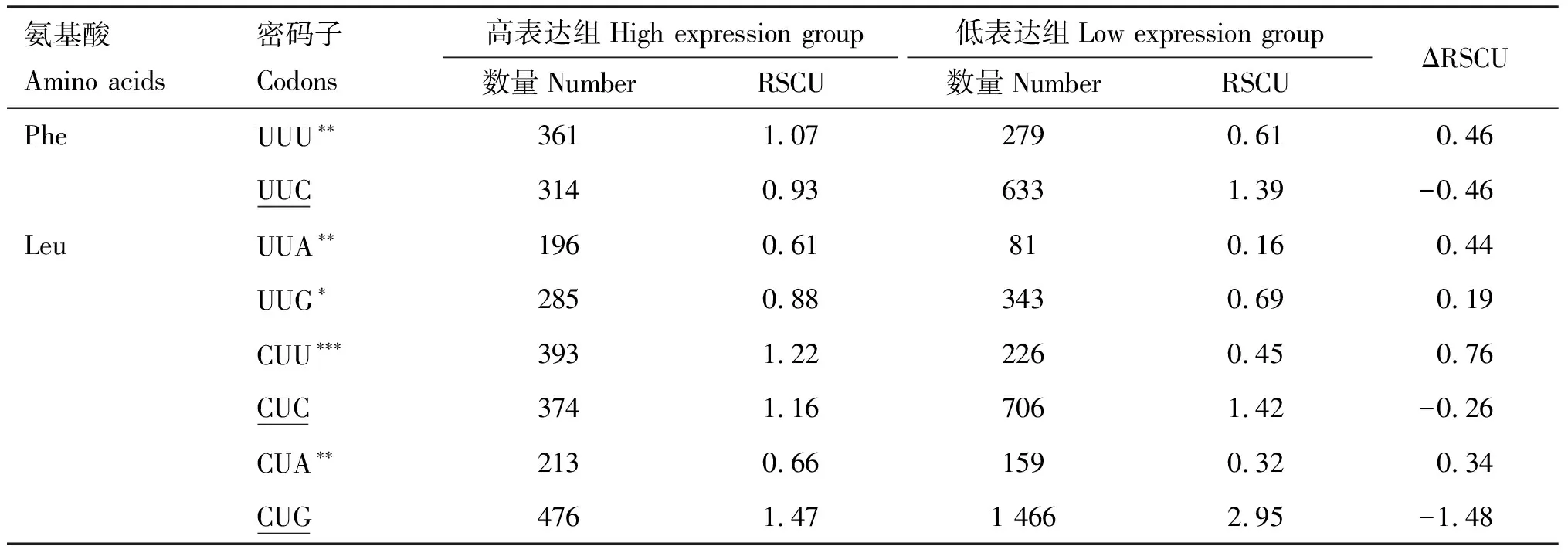
表5 牦牛X染色体基因组氨基酸相对同义密码子使用频率

续表
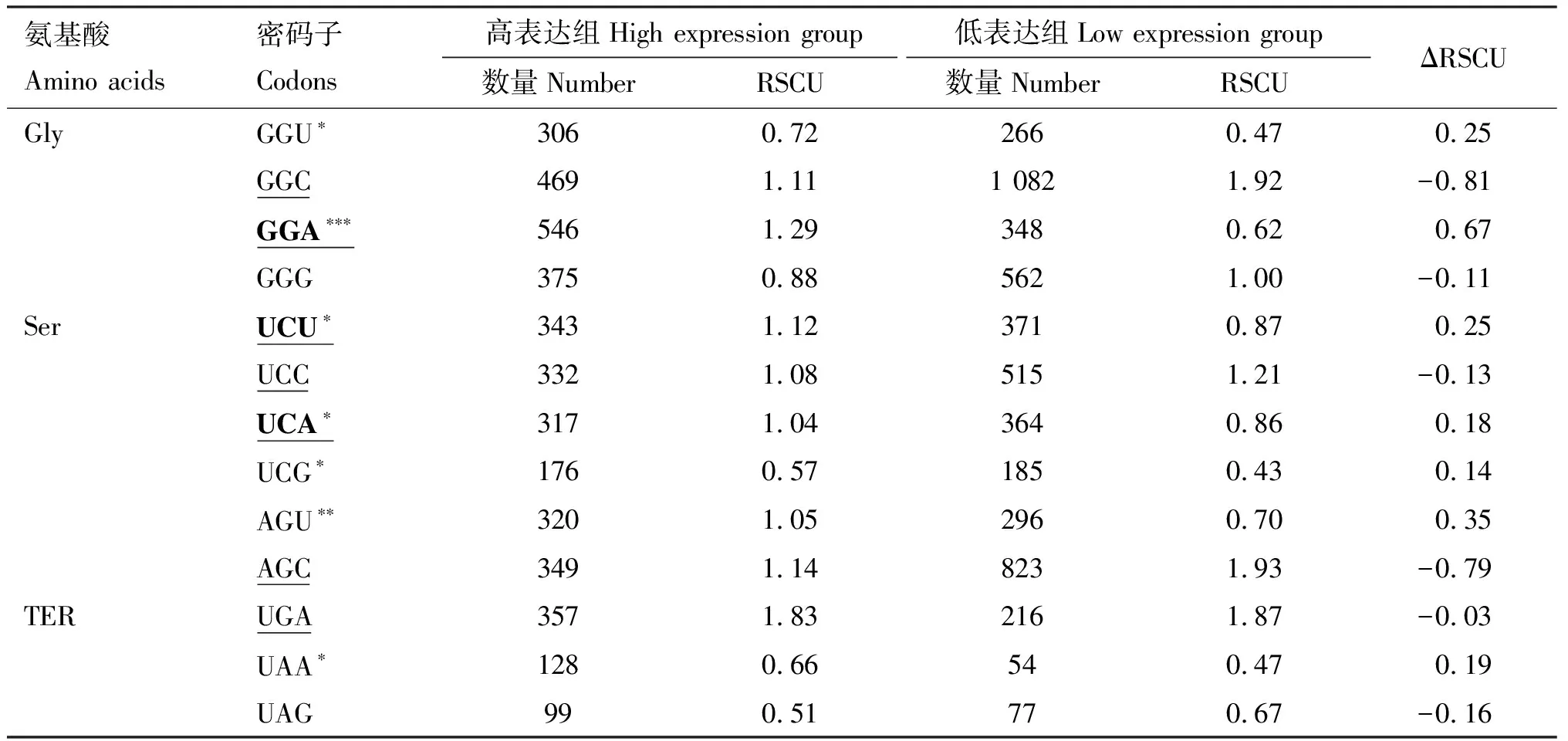
续表
3 讨 论
3.1 基因组比较
牦牛(2n=60),普通牛(2n=60)和江河型水牛(2n=50)的体细胞具有不同的染色体数。性染色体与其他染色体不仅在形态结构上存在差异,更多的体现在分子遗传方面,如更高的遗传漂变率、选择压力和突变等[5],以及X染色体的剂量依赖效应[9],均导致其固定了更多自然选择效应。对牦牛和其他牛亚科动物的X染色体序列进行比对分析将有利于探究牦牛分子遗传特性。
基因组比较结果显示,3种牛亚科动物X染色体基因组的相似性均保持在较高水平,说明X染色体较为保守。对于平原物种来说,适应高原环境需要提高血红蛋白浓度,提高心肺部功能促进气管收缩,或加快机体代谢促进血液循环等,而在牦牛特异片段附近20 kb区段注释的30个功能基因中有2个与肺部呼吸相关基因IL13RA1和AGTR2,1个与气管收缩相关基因CYSLTR1以及1个与糖酵解相关基因PGK1,其中部分基因参与机体代谢功能,但其作用机理尚不明确。糖酵解是生物体在无氧条件下代谢降解获得能量的主要途径,PGK1基因编码一种糖酵解酶,能催化1,3-二磷酸甘油酸转化为3-磷酸甘油酸,该基因也与血管形成贫血等相关[29],其是否与牦牛高原适应相关还有待进一步研究。在被标注出的4个缺失区段,牦牛与普通牛、水牛具有差异的基因KLHL13、CENPI、PGK1等均与呼吸代谢相关。共线性结果显示,3种牛亚科动物在牦牛特异的4个区段大部分基因能匹配共线性基因,且匹配区段较长,说明三者之间的进化分化时间较短,积累的变异较少,更多的保留了从祖先遗传下来的特征。但牦牛的共线性结果表现出较普通牛和水牛更多的交换线性区段,从线粒体DNA和常染色体DNA推断的进化关系表明牛亚科动物最开始分化为Bovina和Bubalina属,后Bovina分化出牦牛和普通牛,Bubalina分化出水牛,因此牦牛和普通牛相似性更高[30],可能是由于牦牛生活的特殊生态环境,使其受到更大的选择压力,在进化过程积累的变异程度也更大,以致这些交换基因在牦牛进化过程中发挥重要作用。后面可对3种牛亚科动物各基因内部的碱基交换区段进行研究,从而进一步了解物种间的基因差异。
3.2 密码子偏性
密码子第3位核苷酸位置的点突变通常导致同义替换,而不改变编码的氨基酸,因此不影响机体的稳定性。然而,非同义替换可能导致表型变化,使自然选择作用于基因。突变和自然选择是促成基因进化的2种主要因素[31]。影响密码子偏性的其他因素还包括碱基组成、基因表达、遗传漂变、无义突变、错义突变和mRNA稳定性等[32]。
本研究共检测比对了3种牛亚科动物X染色体基因组中密码子偏性程度、组成特性、高表达代表和低表达代表的密码子、核苷酸偏性的影响。对基因表达、突变或选择压力和每个氨基酸最优密码子的鉴定进行了分析。其中ENC值常被作为判断密码子偏性的重要参数,能有效反映密码子偏性情况[28]。研究发现,牦牛全基因组CDS区[13]及X染色体CDS区的密码子使用偏性情况均不明显。3种牛亚科动物X染色体密码子的ENC值不存在显著差异,均能有效地使用2个密码子来编码每个氨基酸,其ENC值均>50,高于牦牛全基因组CDS区ENC的48.58,但由于单个基因的ENC值均匀的分布于20~61之间,使得密码子偏性对X染色体的总体影响较全基因组观测的更为显著[33]。密码子的使用模式同样对碱基的组成具有显著影响[34],GC含量是判断碱基组成的重要指标,牦牛X染色体上的GC含量为51%,较普通牛无显著差异,与水牛差异显著,均无明显偏性,这符合3种牛亚科动物的进化历程[30]。研究表明,哺乳动物GC含量普遍偏高[35-36],牦牛全基因组水平GC含量同样显著高于AT含量[13]。在最优密码子分析中也发现了上述使用差异,3种牛亚科动物X染色体CDS区的最优密码子均以A/U结尾,牦牛全基因组发现的18个最优密码子[13]均以G/C结尾,这说明不同染色体可能具有不同的密码子使用模式。其中牦牛最优密码子数量最多,这有利于通过密码子优化等手段改造外源基因,提高蛋白表达水平,为牦牛的遗传育种改良提供理论参考。
本研究通过ENC-plot及PR2-plot分析发现,3种牛亚科动物X染色体的部分基因第3位碱基ATCG使用存在不均衡性,它们的密码子偏性均更大程度受自然选择的影响[28]。ENC比值频数分布提示3种牛科动物ENC期望值与ENC观测值之间差异较大,多数位于标准曲线下方,也进一步说明牦牛与其他两种牛科动物的密码子偏性受自然选择的影响较大。这与牦牛和普通牛全基因组密码子分析发现牦牛的密码子偏性更多的受突变影响而非自然选择的结果不同[13]。推测是由于X染色体的高保守性导致,突变压力较高对于哺乳动物的X染色体存在一定危害。密码子的偏性可能还与CDS基因长度相关[37],同基因组比对结果联合分析能够解释牦牛的密码子GC含量低于普通牛及水牛,较长的基因序列在进化过程中更容易受到自然选择及突变压力的影响。
4 结 论
牦牛X染色体编码区与普通牛、水牛相比,存在较多差异基因,如KLHL13、CENPI、PGK1等均与呼吸代谢相关的基因,以及由于强选择压和突变压下表现出的交换线性区段;同时牦牛、普通牛和水牛X染色体编码区密码子使用偏性相似,均偏向G/C结尾的密码子,牦牛偏性最小;牦牛筛选出最多(16个)最优密码子均以A/U结尾;且三者密码子偏性受到自然选择的影响均大于突变的影响。

