扎那纹胸鮡g型溶菌酶的cDNA克隆及组织表达分析
王运娇,吕 梦,蔡莎莎
溶菌酶最初以水解细胞壁而闻名[1],根据分子结构、催化机制及免疫特性的不同,动物溶菌酶分为3种:c型溶菌酶、g型溶菌酶和i型溶菌酶[2]。鱼类溶菌酶主要有c型溶菌酶和g型溶菌酶2种,且这2种类型的溶菌酶在组织分布、基因组结构和分子特征方面都存在很大的不同[2]。目前针对鱼类溶菌酶的研究主要集中在c型,对g型溶菌酶的报道局限于鲤形目鲤鱼、草鱼[3],鲈形目石首鱼科、鮨科[4],鲟形目鲟科[5],鲽形目牙鲆科和鳕形目鳕科[6]等。目前尚无关于鲇形目鮡科鱼溶菌酶的报道研究。扎那纹胸鮡隶属鮡科、纹胸鮡属主要分布于我国澜沧江中游及怒江上中游水网系统,是当地重要的经济渔获之一[7]。近年来,由于过度捕捞、栖息地退化或环境污染,该物种的自然种群数量急剧下降,引起人们对其长期可持续性的日益关注。人工驯养是保护扎那纹胸鮡种质的一种可行方式,但目前扎那纹胸鮡的人工养殖技术还不成熟,难以实现规模化,且常有各种感染性疾病的发生。鉴于溶菌酶在鱼类感染性疾病控制中的重要作用[8],该研究利用PCR克隆和生物信息学的方法分析鉴定了扎那纹胸鮡g型溶菌酶(G.zainaensisg-typeLysozyme,GzLysG)基因,并利用实时定量PCR技术分析了其在扎那纹胸鮡不同组织中的表达特征,为进一步阐明g型溶菌酶在扎那纹胸鮡免疫防御抵抗病原菌中的功能奠定了重要基础。
1 材料与方法
1.1 实验动物扎那纹胸鮡6条,4雄2雌,体质量100~200 g,采自四川怒江,液氮速冻,干冰运输。解剖取其肝脏、肠、肌肉、鳃、皮肤和脾脏组织,置于液氮中,保存备用。
1.2 药品与试剂TRIzol RNA分离试剂购自美国Invitrogen公司;SMARTTM cDNA Library Construction Kit(Clontech)、PrimeScriptTMRT reagent Kit with gDNA Eraser (Perfect Real Time)、TB Green®Premix DimerEraserTM(Perfect Real Time)以及PCR克隆用的Taq酶均购自宝日医生物技术(北京)有限公司;引物合成、测序由上海生工生物工程有限公司完成。
1.3 方法
1.3.1扎那纹胸鮡溶菌酶基因的克隆 利用TRIzol RNA分离试剂提取扎那纹胸鮡肝脏组织总RNA,分别利用1%琼脂糖凝胶电泳和NanoDrop GX分光光度计分析RNA的完整性和纯度。选以高质量的RNA为模板,严格按照说明书要求,利用建库试剂盒SMARTTM cDNA Library Construction Kit构建cDNA文库。参考已知鱼类g型溶菌酶的基因序列,借助Primer Premer 5.0在线软件,设计两条5′上游引物(P1:5′-ATGGCTKGCATTTTTGGAGAC-3′,P2:5′-ATGATCTGAAGARGATGGA-3′),然后利用试剂盒内的CDSⅢ/3′PCR Primer为3′随机引物进行目的基因的PCR克隆,挑选阳性克隆测序,将测序结果序列与NCBI数据库中的序列进行比对鉴定分析。
1.3.2生物信息学分析 使用ExPASy中的Translate软件(https://web.expasy.org/translate/)推导氨基酸序列。对推导的氨基酸序列,利用SignalP 5.0 Server(https://services.healthtech.dtu.dk/service.php?SignalP-5.0)进行信号肽的查找分析。利用在线软件ProtParam (https://web.expasy.org/protparam/)对溶菌酶序列的理化性质进行分析。利用CDD(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)分析溶菌酶的保守结构域。在NCBI中查找已报道的其他物种来源的溶菌酶的序列信息,利用Clustal Omega(https://www.ebi.ac.uk/Tools/msa/clustalo/)在线软件对扎那纹胸鮡溶菌酶与其他物种的氨基酸序列进行多物种序列比对。利用MEGA 6.0软件,选以邻接法构建系统发育树,参数设置选择Poisson Correction 模型,bootstraps设置为 1 000[9]。
1.3.3结构分析 利用在线预测软件SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)分析蛋白的二级结构。利用SWISS-MODEL(https://swissmodel.expasy.org/)预测扎那纹胸鮡溶菌酶的高级结构。
1.3.4组织表达谱分析 按照“1.3.1”所述方法提取扎那纹胸鮡脾脏、肝脏、肠、肌肉、鳃和皮肤6个组织的总RNA,然后按照PrimeScriptTMRT reagent Kit with gDNA Eraser (Perfect Real Time)试剂盒说明书,合成cDNA一链序列。反转录的cDNA模板用EASY Dilution稀释10倍后使用。利用Primer Premer 5.0软件设计扎那纹胸鮡溶菌酶基因和内参基因β-actin的引物。GzLysG的上下游引物分别为5′-AGTACAAGGCCACCATCGTC-3′和5′-TCACTGTTCCAGGCTCCTTT-3′,β-actin的上下游引物分别为5′-GCTGTCTTCCCTTCCATTGT-3′和5′-CTGAGCCTCATCACCAACAT-3′。按照TB Green®Premix DimerEraserTM(Perfect Real Time)试剂盒说明书在QuantStudio 7 Flex real-time PCR System(Applied Biosystems, USA)上进行qPCR反应。反应体系为:12.5 μl的TB Green Premix DimerEraser(2×),0.75 μl的PCR Forward Primer(10 μmol/L),0.75 μl的PCR Reverse Primer(10 μmol/L),2 μl的模板cDNA,9 μl的灭菌双蒸水。qPCR的反应条件为:95 ℃预变性30 s;40个循环,95 ℃变性5 s,60 ℃退火30 s,72 ℃延伸30 s。各样品的目的基因反应产物经熔解曲线确认是否为单一特异性的扩增条带。目的基因的转录水平变化采用ΔΔCt法分析处理。

2 结果
2.1 GzLysG cDNA的克隆及分子特征以肝脏总RNA为模板,利用巢氏PCR技术,成功克隆到一条编码GzLysG的全长cDNA序列(GenBank登录号:OP881998, 图1)。按照溶菌酶的类型和扎那纹胸鮡拉丁名G. zainaensis的前两个首字母将克隆得到的溶菌酶命名为GzLysG。GzLysG的cDNA序列全长558 bp,编码185个氨基酸(图1)。蛋白保守功能结构域的预测结果表明,GzLysG含有g型溶菌酶典型的鹅蛋清溶菌酶(goose egg white lysozyme, GEWL)结构域和可溶性裂解转糖苷酶(soluble lytic transglycosylases, SLT)结构域。

图1 GzLysG cDNA及其编码的氨基酸序列红色字体为半胱氨酸残基;黄色阴影为糖结合位点;框中为SLT结构域
GzLysG蛋白无信号肽序列,表明该溶菌酶为非分泌表达的蛋白质(图2)。GzLysG蛋白的氨基酸序列中含有20种常见氨基酸,其中Lys的含量最高(11.9%),其次是Gly (10.8%)和Ala(8.1%)。其分子量为20 478.20 u,理论等电点为9.16,经ExPASy-ProtParam分析判断为一种稳定的碱性亲水性蛋白质。

图2 GzLysG的信号肽分析
2.2 多物种溶菌酶基因序列比对多物种序列比对结果发现,GzLysG与日本鳗鲡(AJLysG2)和斑马鱼(DRLysG)来源的溶菌酶的同源性相对较高。且在GzLysG序列中也发现了g型溶菌酶高度保守的催化活性位点Glu(E73)、Asp(D86)和Asp(D97)(图3)。

图3 GzLysG与其它代表性g型溶菌酶的多物种序列比对绿色阴影为高度保守的氨基酸,黄色阴影和星号指示催化位点残基
2.3 GzLysG的结构特征分析GzLysG二级结构中含有6个大小不一的α螺旋结构,占比43.24%,β折叠占比11.35%,β转角占比4.86%,无规则卷曲结构占比40.54%(图4A)。Swiss model模型进一步验证了上述结果,GzLysG与鹅蛋蛋白溶菌酶(PDB:153l.1.A)结构相似,都有6个α螺旋结构(图4B)。3D结构模型显示,GzLysG分子表面有一条较深的裂缝,催化活性中心的3个残基均匀地分布在裂缝的两侧(图4B)。溶菌酶的这种特殊结构与其酶活性催化机制密切相关。

图4 GzLysG的结构预测结果
2.4 物种间溶菌酶的进化关联分析基于溶菌酶分子的蛋白序列,构建了扎那纹胸鮡与其他物种来源的溶菌酶的系统发生树。结果如图5所示,根据进化的亲缘关系,系统发生树可以被分为3个分支,c型溶菌酶,i型溶菌酶和g型溶菌酶。其中,GzLysG与斑马鱼来源的溶菌酶表现出较近的亲缘关系,且与其他物种的g型溶菌酶聚为一支。此外,通过进化发生树,还能发现不同物种的g型溶菌酶之间存在同一性高的特点,即使是同一物种的g型和c型溶菌酶也存在各自的同一性和差异性。

图5 GzLysG的进化分析
2.5 GzLysG基因的组织表达谱分析以β-actin基因为内参,利用实时定量PCR检测了GzLysG基因在扎那纹胸鮡不同组织中的表达情况。结果如图6所示,GzLysG基因在多个组织中均有表达,其在不同待检组织中的表达量差异性较大。GzLysG基因在皮肤中的表达量最高,表达量约为在肠中表达量的633倍;在肌肉和鳃中的表达量次之,分别为在肠中表达量的476和81倍;其在肝脏、脾脏和肠中的表达量较低。
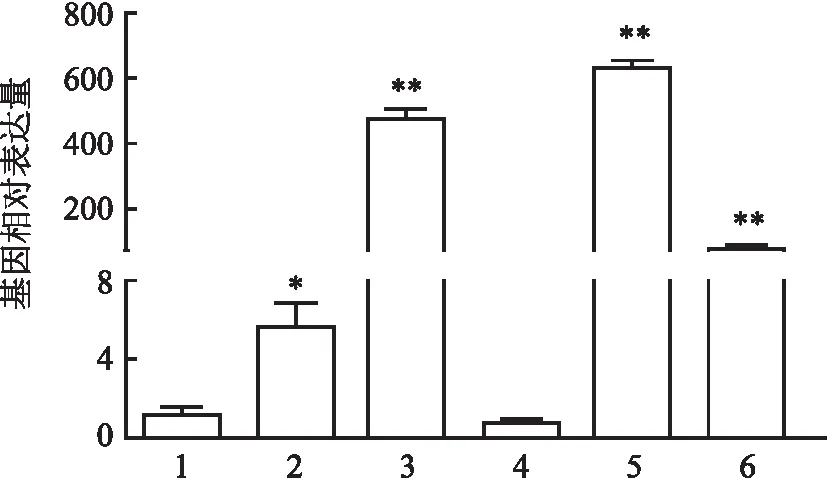
图6 GzLysG mRNA在扎那纹胸鮡不同组织中的相对表达量
3 讨论
近年来,随着水产养殖生态环境的恶化,鱼类病害发生率居高不下。水产养殖对于抗生素的过度依赖已在全球范围内造成了严重的微生物耐药性问题。因此,提高鱼类自身免疫防御能力,研发安全高效的抗生素替代品,是鱼类疾病防控的未来研究方向[8]。相较于特异性免疫,非特异性免疫系统在鱼类抵抗病原菌时发挥更为重要的作用[10]。作为宿主重要的非特异性免疫因子,鱼类溶菌酶因具有广谱、高效、生物安全的抗菌活性在防治水生动物病害方面展现出良好的应用前景[2,10]。本研究通过基因克隆和生物信息学方法获得一条扎那纹胸鮡溶菌酶GzLysG的编码序列。GzLysG具有g型溶菌酶典型的结构特征,含有3个保守的活性催化位点、GEWL结构域和SLT结构域。研究[2]发现,二硫键的存在有助于维持溶菌酶的结构稳定性,而g型溶菌酶中二硫键数目会因物种不同存在很大的变化。鸟类和哺乳动物g型溶菌酶均含有4个保守的半胱氨酸,可形成2个稳定的二硫键。鱼类中,大西洋鲑鱼中g型溶菌酶有3个半胱氨酸[11],大菱鲆和日本鳗鲡中g型溶菌酶有2个半胱氨酸,但不能形成二硫键[12],而鲤鱼、牙鲆、条石鲷等多数鱼类中g型溶菌酶均无半胱氨酸。本研究中,GzLysG序列中仅存在1个半胱氨酸残基。二硫键的缺失将会影响g型溶菌酶的结构特性,在温度变化时其活性也会改变,推测鱼类g型溶菌酶还存在其他维持结构稳定的机制。
除了抗菌活性,一些溶菌酶还被报道具有免疫调节、抗肿瘤、消化等功能[13-14]。GzLysG是一种偏碱性的亲水性蛋白,无信号肽,编码该蛋白的氨基酸中以赖氨酸含量最高,说明GzLysG主要为胞内起抗菌活性的溶菌酶。值得注意的是,GzLysG虽与斑马鱼DRLysG、日本鳗鲡AJLysG2具有较高的同源性,但与二者的组织表达模式差异明显。DRLysG和AJLysG2被报道在鳃和免疫器官如肝脏、脾脏中具有较高的表达量[12]。而GzLysG基因虽在肠、肝脏、脾脏这些免疫器官中均有被检测到,但其在皮肤和肌肉组织中表达量最高,鳃中次之。鱼类的皮肤、鳃和肌肉等是阻挡病原菌入侵的重要机械屏障,也是参与其免疫应答反应的重要组织器官。GzLysG基因在这些免疫相关组织中的高表达,提示了该溶菌酶在扎那纹胸鮡固有免疫防御中发挥着重要作用。
通过转基因技术转入溶菌酶基因,可获得抗病能力强的转基因鱼[8]。将溶菌酶作为饲料添加剂应用于水产养殖,可提高鳗鱼、草鱼等的生长性能和抗感染能力。将溶菌酶与凝结芽孢杆菌、丁酸梭菌等益生菌,与寡糖、中草药、植物精油、抗菌肽等功能性饲料添加剂共同使用,均具有良好的协同增效效果,是优秀的抗生素替代组合方案。本文克隆了GzLysG的cDNA序列,并对其分子结构特征、组织表达谱等进行了分析,初步探讨了GzLysG在扎那纹胸鮡先天免疫防御中的作用。该研究有助于扎那纹胸鮡的人工科学养殖,未来课题组将进一步围绕GzLysG的功能和作用机制开展研究。

