胃癌预后及免疫治疗反应预测基底膜相关基因标签的构建
李文倩,宫宇红,邱文生
(1 青岛大学附属医院肿瘤内科,山东 青岛 266031; 2 海军青岛特勤疗养中心中医科)
胃癌是全球发病率排第5、死亡率排第3的恶性肿瘤[1]。在过去的几年中,免疫疗法在提高胃癌病人的总生存期(OS)方面被证明是有效的[2]。然而,由于胃癌的高度异质性,免疫疗法并不适用于所有胃癌病人[3]。因此,开发能够准确预测胃癌肿瘤免疫微环境的基因标签,以识别不同的胃癌免疫亚型,确定最有可能从免疫治疗中获益的人群,是目前亟待解决的问题。在肿瘤组织中,免疫细胞浸润的程度受肿瘤周围毛细血管通透性、肿瘤刺激产生的细胞因子和趋化因子以及肿瘤微环境中细胞外基质的影响[4]。作为细胞外基质的重要组成部分,基底膜(BM)在免疫细胞浸润过程中充当重要屏障[5]。然而,BM相关分子是否能够影响免疫细胞浸润从而调节肿瘤免疫微环境仍然未知。此外,BM作为浸润性癌和原位癌区分的关键结构,在肿瘤的侵袭和转移中发挥重要作用[6]。目前尚无研究探讨BM相关分子对胃癌预后的影响。本研究基于生物信息学方法对以上问题进行了初步探讨。
1 材料和方法
1.1 BM相关基因表达数据的采集
从癌症基因组图谱(TCGA)数据库中获取32例正常样本及375例胃癌样本的RNA测序数据和临床信息作为训练集,再从基因表达综合数据库(GEO)GSE84437数据集中获取433例胃癌样本的RNA测序数据和临床信息作为验证集。从基因集富集分析(GSEA)分子特征数据库(v7.5.1版本)中获取61个BM相关基因。
1.2 与胃癌预后相关的BM相关基因标签的构建及验证
用“limma”R包分析肿瘤和正常组织间差异表达的BM相关基因(BMDEGs),将|log2FC|>1和伪发现率(FDR)<0.05作为阈值。训练集采用单变量比例风险回归(Cox回归)分析筛选胃癌预后相关的BM相关候选基因,进一步使用最小绝对值选择与收缩算子(LASSO)回归建立提示胃癌预后的BM相关基因标签。根据标签中每个BM相关基因的标准化表达水平与其相应回归系数的乘积再总和来计算每例病人的风险评分。根据中位风险评分将训练集和验证集的病人分为高、低风险组,采用生存曲线(Kaplan-Meier曲线)分析评估两组生存差异,Cox回归及受试者工作特征(ROC)曲线分析进一步验证标签的可靠性和稳定性。用“rms”R包构建列线图,预测胃癌病人1、3、5年的OS。建立校准曲线以评估使用列线图预测的OS与实际观察到的OS之间的一致性,采用ROC曲线分析比较各预测变量的预后价值,采用决策曲线分析(DCA)评估列线图模型的临床适用性。
1.3 肿瘤免疫微环境及免疫治疗反应分析
采用不同算法评估高、低风险组胃癌病人肿瘤微环境中免疫细胞的丰度,同时分析两组间免疫检查点相关基因表达、免疫功能、基质评分、免疫评分以及肿瘤微卫星不稳定性(MSI)的差异。从肿瘤免疫功能障碍和排斥(TIDE)在线网站(http://tide.dfci.harvard.edu/)下载胃癌免疫治疗反应相关数据信息,计算高、低风险组病人的TIDE评分,并利用GSE78220和PRJEB25780数据集信息分析风险评分与免疫治疗反应的关系。然后用“pRRophetic”R包分析风险评分与胃癌常用化疗药物敏感性之间的关系。
1.4 在线生信分析工具验证BMDEGs的表达
基于基因表达水平交互式分析平台(GEPIA)(http://gepia.cancer-pku.cn/detail.php)验证胃癌和正常胃组织中BMDEGs的差异表达水平。
1.5 统计学分析
应用GraphPad Prism 8.0.1软件(GraphPad Software,Inc.)和R 4.1.2软件进行数据的统计分析。连续变量比较采用Student’st检验、独立样本Wilcoxon秩和检验、Kruskal-Wallis检验和方差分析,相关性分析采用Pearson相关分析。P<0.05认为差异具有统计学意义。
2 结 果
2.1 BM相关基因标签的构建
差异分析筛选出19个BMDEGs(图1A、B)。用单变量Cox回归分析进一步筛选出8个与胃癌预后相关的BMDEGs(图1C),通过LASSO回归分析最终建立了由6个BMDEGs(CAV1、COL4A1、MMP11、PXDN、COL4A5、USH2A)组成的胃癌基因标签(图1D、E)。
2.2 BM相关基因标签预后价值的评估
病人的风险评分计算如下:风险评分=0.072×CAV1的表达+0.015×COL4A1的表达+0.077×MMP11的表达+0.152×PXDN的表达+0.054×COL4A5表达+1.188×USH2A表达。训练集(图2A)和验证集(图2B)中的病人均根据中位风险评分分为高、低风险组。训练集和验证集中风险评分较高的胃癌病人往往死亡较早,Kaplan-Meier曲线分析表明,高风险组胃癌病人的OS较短(图2C~F)。训练集单变量Cox回归分析表明,年龄和T、N、M分期以及风险评分(P<0.001,HR=3.472,95%CI=1.949~6.186)与OS之间显著相关(图2G);多变量Cox回归分析同样显示,年龄和N、M分期以及风险评分(P<0.001,HR=4.008,95%CI=2.193~7.323)是OS的独立预后因素(图2H)。验证集的单变量和多变量Cox回归分析进一步证实了训练集的结果(图2J、K)。以上结果证明我们构建的BM相关基因标签可作为胃癌病人的独立预后生物标志物。ROC曲线分析结果显示,训练集中预测胃癌病人1、3、5年生存率的风险评分曲线下面积(AUC)分别为0.627、0.638和0.717(图2I),验证集中预测1、3、5年生存率的风险评分AUC也均高于0.6(图2L)。
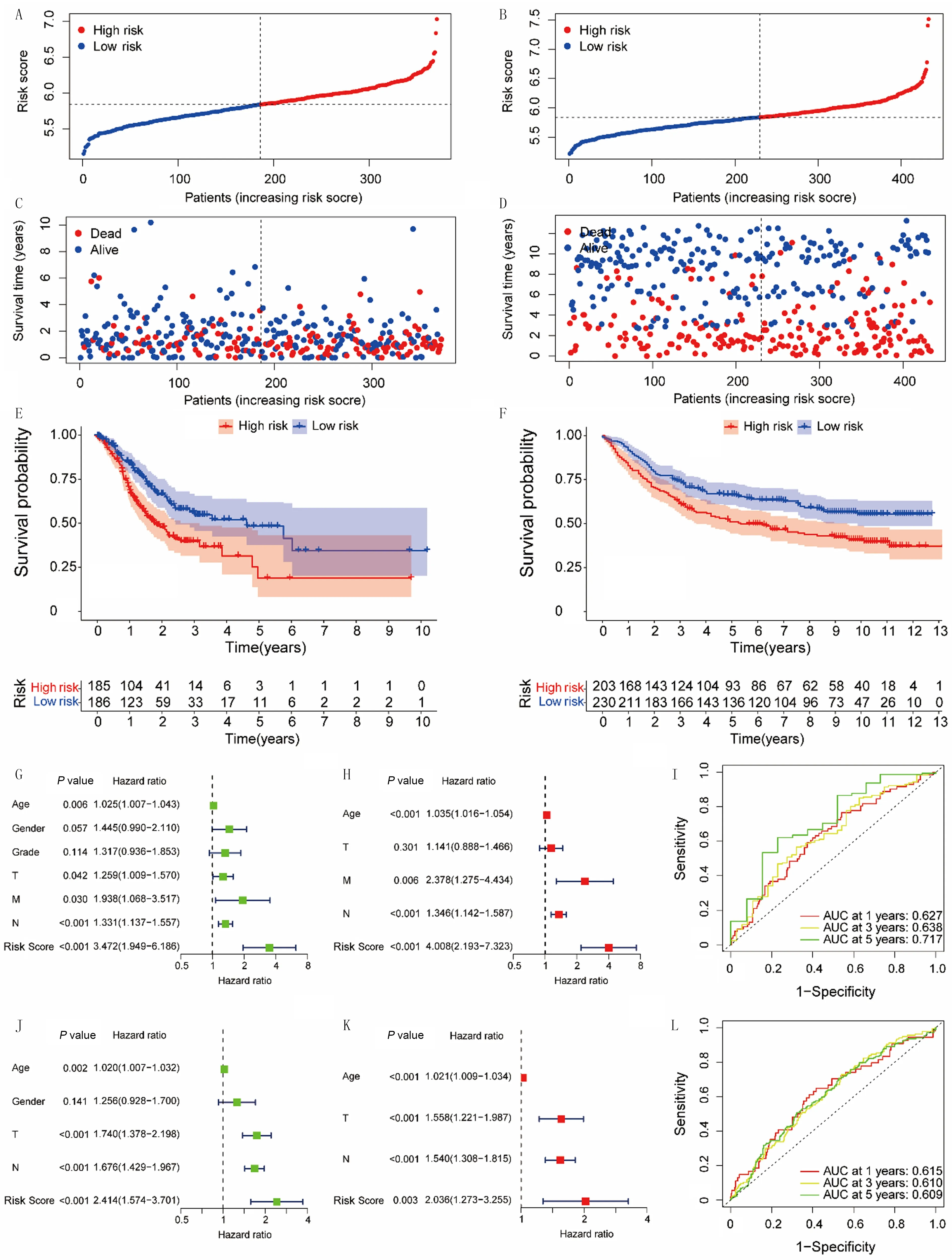
TCGA(A)和GEO(B)队列中胃癌病人的风险分数分布;TCGA(C)和GEO(D)队列中胃癌病人风险分数和生存状态关系散点图;TCGA(E)和GEO(F)队列中高、低风险组胃癌病人OS的Kaplan-Meier曲线;TCGA(G、H)和GEO(J、K)队列中临床特征和风险评分的单变量和多变量Cox回归分析森林图;TCGA(I)和GEO(L)队列中风险评分预测胃癌病人1、3、5年OS的ROC曲线。
2.3 预测胃癌OS的列线图模型的构建及评价
将多种临床风险因素与基于基因标签计算所得风险评分结合构建胃癌OS预测的列线图模型(图3A)。训练集及验证集的校准曲线显示,列线图模型预测胃癌病人OS的准确度较高(图3B、C)。两队列ROC曲线分析结果显示,列线图模型预测胃癌病人1、3、5年OS的AUC均较风险评分高(图3D、G),更显著高于其他临床风险因素(图3E、H)。DCA结果显示,列线图模型预测胃癌OS的临床适用度也较高(图3F、I)。
2.4 肿瘤免疫微环境与免疫治疗反应分析
免疫细胞浸润与风险评分间关系分析结果显示,大多数免疫细胞浸润水平与风险评分呈正相关,尤其是促进胃癌进展和导致免疫抑制的细胞(图4A)。箱线图结果亦是如此(图4B)。免疫功能差异分析结果显示,趋化因子CCR家族、抗原递呈细胞(APC)共刺激、Ⅱ型干扰素反应功能在高风险组中更强,而主要组织相容性复合体(MHC)Ⅰ类功能在低风险组中更强(图4C)。免疫检查点相关基因(ICGs)表达差异情况分析显示,高风险组中有22个基因表达水平显著增加,而低风险组中仅有4个基因(图4D)。计算两组肿瘤纯度(ESTIMATE)评分、免疫评分和基质评分结果显示,高风险组3种评分均显著高于低风险组(图4E、F、G)。上述结果表明,高风险组胃癌病人可能通过促进免疫抑制细胞浸润,ICGs表达增加,以及募集更多基质细胞介导免疫逃逸,从而导致预后不良。
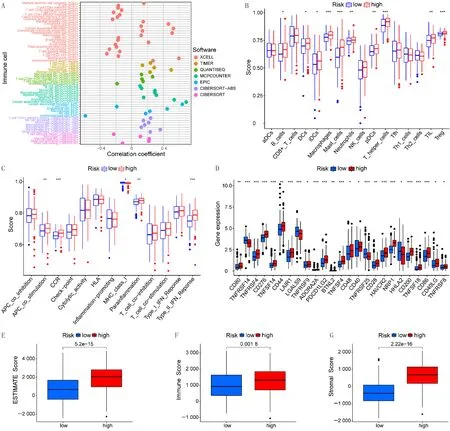
A:免疫细胞浸润和风险评分之间的相关性分析;高、低风险组间免疫细胞浸润(B)、免疫功能(C)和ICGs表达(D)的博弈图;高、低风险组ESTIMATE评分(E)、免疫评分(F)和基质评分(G)的比较。*P<0.05,**P<0.01,***P<0.001。
MSI分析结果显示,MSI-H的病人风险评分更低(图5A);TIDE分析结果显示,高风险组病人的TIDE评分更高(图5B)。利用GSE78220(图5C)及PRJEB25780(帕博利珠单抗治疗反应,图5D)免疫治疗反应数据集分析风险评分与免疫治疗反应之间的关系,结果显示,免疫治疗有反应组风险评分更低。表明高风险组胃癌病人更容易发生免疫逃逸,且对免疫治疗的反应更有限。常用化疗药物敏感性差异分析结果显示,高风险组对硼替佐米、环巴胺以及帕唑帕尼的敏感性更高(图5E~G),而低风险组则对甲氨蝶呤和丝裂霉素C更敏感(图5H、I)。以上结果表明,本研究构建的BM相关基因标签有助于临床管理和减少胃癌病人耐药的发生。
2.5 6个BMDEGs表达水平的验证
GEPIA分析结果显示,胃癌组织中COL4A1、PXDN和MMP11的表达水平显著高于正常组织,而COL4A5的表达水平则显著低于正常组织,但与先前研究不同的是,CAV1和USH2A在胃癌与正常组织中的表达差异无显著性。
3 讨 论
BM作为肿瘤进展和肿瘤微环境调节中至关重要的结构,目前还没有研究探索其相关分子在胃癌预后、肿瘤免疫微环境以及胃癌免疫治疗中的作用。本研究基于TCGA数据库,从61个BM相关基因中筛选出19个在胃癌及癌旁组织中差异表达的基因,进一步采用单变量Cox和LASSO回归进行预后分析,最后构建了一个包含6个关键BMDEGs的基因标签,该标签可对胃癌病人生存风险进行评分,评估病人预后,分析肿瘤免疫微环境,可作为评估胃癌免疫治疗效果的有效分子生物标志物。该标签所包含的6个关键基因目前已被证明会影响肿瘤的发生发展。
CAV1是洞穴的主要支架蛋白,研究发现它在胃癌组织中低表达,增加其表达可抑制胃癌发展[7]。COL4A1是Ⅳ型胶原蛋白α链(COL4A)家族的成员,可通过调节Hedgehog信号通路影响胃癌侵袭和上皮间质转化,有助于胃癌转移[8]。COL4A5同属于COL4A家族,生存分析表明,COL4A1/5表达百分比越低,胃癌病人的OS越长,且COL4A5是胃癌的独立预后分子标志物[9]。MMP11作为基质金属蛋白酶(MMP)家族的一员,在胃癌病人肿瘤组织和血浆纯化的外泌体中过表达,参与胃癌转移,影响胃癌OS[10]。PXDN催化胶原蛋白Ⅳ中的磺酰胺键,起巩固BM的作用,研究发现PXDN可抑制宫颈癌的上皮间质转化[11],但可促进黑色素瘤细胞侵袭[12]。USH2A编码Usherin蛋白,研究发现该蛋白在胃癌的早期阶段表达水平升高,可能与促进胃癌的进展有关[13]。以上文献结果增加了本研究构建的基因标签预测胃癌病人预后的可靠性。
此外,本研究还结合多种临床风险因素与基于基因标签所得的风险评分构建了一个能够更为准确预测胃癌OS的列线图模型,并从3个维度评估了列线图模型预测胃癌OS的临床价值。结果显示,列线图模型预测胃癌OS的准确度、区分度以及临床适用度均较高。
通过对BM相关分子与肿瘤免疫微环境的相关性进行分析发现,高风险组中免疫细胞浸润度较高,且浸润的免疫细胞多与肿瘤发展和抗肿瘤免疫抑制相关,如肿瘤相关巨噬细胞、肥大细胞、肿瘤相关中性粒细胞及调节性T细胞。此外,高风险组基质细胞评分以及ICGs的表达均较高,这进一步促进了高风险组免疫逃逸的发生。
过去10年中,免疫检查点阻断疗法已使一系列恶性肿瘤获得令人振奋的生存获益。目前我国已批准纳武利尤单抗联合化疗(CapeOX/FOLFOX)用于晚期胃癌一线治疗,无论PD-L1的表达如何。信迪利单抗联合CapeOX也已在我国被批准用于晚期胃癌一线治疗。此外尚有一些在其他肿瘤中获批的PD-1单抗对晚期胃癌的疗效目前也在研究中。同时,国内也有一大批PD-1抑制剂用于胃癌术前新辅助治疗的二期临床研究正在进行,目前安全性基本没有问题。免疫治疗的日益成熟应用将对早晚期胃癌病人的生存及生活质量起到明显改善作用。
TCGA根据胃癌分子及遗传特征将胃癌分为4种亚型:EB病毒阳性型、微卫星不稳定型、染色体不稳定型和基因组稳定型[14]。其中EB病毒阳性型胃癌表现出高CD8阳性T细胞浸润和PD-L1/L2表达增加,提示其对免疫检查点抑制剂具有更高的敏感性[15]。MSI-H状态是手术完全可切除胃癌病人的良好预后因素,是围手术期细胞毒性化疗(如氟尿嘧啶类)反应的阴性预测指标,也是免疫检查点抑制剂反应的阳性预测因子。美国国立综合癌症网络指南推荐对所有新确诊为胃癌的病人使用聚合酶链反应、二代测序或免疫组化方法进行MSI状态检测(https://www.nccn.org/professionals/physician_gls/pdf/gastric.pdf)。此外,肿瘤突变负荷以及PD-L1表达均是免疫检查点抑制剂治疗晚期胃癌疗效的有用预测生物标志物。本研究采用生物信息学方法构建了一个新的可预测胃癌免疫微环境及免疫治疗反应的BM相关基因标签,通过TIDE及其他免疫治疗反应数据库分析,结果表明该基因标签能够很好地预测胃癌抗PD-1治疗反应。
总之,本研究首次将BM与胃癌预后、肿瘤免疫微环境和免疫治疗反应评估联系起来,随着二代测序技术的成熟应用,本研究结果有望使胃癌的免疫治疗、分子靶向治疗及化疗更加个体化和精准化。