基于高效SNP芯片的小麦产量相关性状全基因组关联分析
刘丽华, 刘阳娜,周 悦,李宏博,张明明,屈平平,赵昌平,庞斌双
(北京市农林科学院杂交小麦研究所,杂交小麦分子遗传北京市重点实验室,农业农村部农作物DNA指纹创新利用重点实验室,北京 100097)
小麦是世界上重要的粮食作物之一,有约40%的人口以小麦为主粮(http://www.fao.org/)。随着全球人口数量的增加、气候灾害的频发、耕地资源的减少、水资源日益匮乏,全球粮食安全依然面临挑战[1],而提高小麦品种产量潜力是解决粮食安全的有效途径。株高、穗长、小穗数、穗粒数、有效分蘖数、粒长、粒宽和千粒重与小麦产量显著相关[2-5],挖掘此类性状的关联位点,有助于加快有利基因的聚合和利用,对小麦产量的遗传改良具有重要的意义。
小麦的产量相关性状是由多基因控制的数量性状,受环境的影响较大。全基因组关联分析(genome-wide association study, GWAS)和数量性状位点(quantitative trait locus, QTL)定位的连锁分析被认为是解析复杂数量性状的两种主要手段[6]。关联分析以连锁不平衡为基础,把自然群体中个体的表型同基因型的多样性结合起来分析,直接鉴定出与表型变异密切相关的具有特定功能的等位基因位点,较连锁分析的QTL定位方法具有分辨率高、构建群体耗时短、能同时检测多个等位基因、考虑更加复杂的遗传背景等优点[7-8],更容易发现重要的数量性状基因位点,挖掘出更多的功能基因,现已广泛应用于小麦[9-10]、玉米[11]、水稻[12]、大豆[13]、花生[14]等农作物的数量性状研究。
GWAS分析结果受标记密度的影响较大,标记密度越大,关联结果越精确[15]。单核苷多态性(single nucleotide polymorphism, SNP)标记,在基因组中具有分布广泛、遗传稳定、密度高、可实现高通量检测等优点,用于全基因组关联分析,解析率高。随着小麦基因组测序的发展,大量SNP标记被开发出来,各款SNP芯片和SNP高通量检测平台的问世,大力推进了基于SNP标记开展小麦产量相关性状关联分析的研究。Yerlan等[9]利用90K Illumina iSelect SNP芯片,筛选出3 245个有效SNP标记,对194份春小麦的12个农艺性状进行GWAS分析,获得12个至少在两个环境下显著关联的位点。Lozada等[16]利用90K Illumina iSelect SNP芯片,筛选出5 715个SNP标记,对239份软红冬小麦的8个产量相关性状进行GWAS分析,获得5个与产量相关性状的显著关联位点。Kumar等[17]利用35K Axiom SNP芯片,筛选出15 886个SNP标记,对205份小麦种质资源的产量相关性状进行GWAS分析,获得10个与产量显著相关的SNP位点。张红杰等[18]利用55K SNP芯片,对152份合成小麦的株高性状进行关联分析,共发现24个与株高显著关联的SNP位点。
以往报道中,用于关联分析的有效SNP标记密度相对较低,且应用高密度SNP标记基于 GWAS 挖掘小麦产量相关基因的研究报道较少。本研究以248个中国北部冬麦区小麦育成品种为材料,利用自主开发的高质量高效小麦Affymetrix BAAFS Wheat 90K SNP芯片进行基因分型,获得63 658个高质量SNP位点,开展株高、穗长、小穗数、穗粒数、有效分蘖数、粒长、粒宽和千粒重共8个产量相关性状的GWAS分析,发掘小麦产量相关性状稳定关联的SNP位点,筛选相关候选基因,为相关基因克隆和分子标记辅助选择提供理论依据。
1 材料与方法
1.1 试验材料与表型鉴定
选取248个北部冬麦区小麦育成品种分别于2017、2018和2019年在北京市海淀试验站种植。采用随机区组设计,双行区,2次重复,行长1.5 m,行距25 cm,每行30株,按当地常规方式进行田间管理。在小麦成熟收获前,随机抽取每份材料的10个单株(去掉每行两端的植株),考察其株高(plant height, PH)、穗长(spike length, SL)、小穗数(spikelet number, SN)、穗粒数(kernel number per spike, KN)、有效分蘖数(effective tiller, ET)。待收获脱粒自然晾干后,称量千粒重(thousand-kernel weight, TKW),并测量粒长(kernel length, KL)和粒宽(kernel width, KW)。
1.2 表型数据分析

1.3 DNA提取和SNP基因分型
采用CTAB法[19]提取248个品种的DNA,并将其保存在TE中。DNA质量和浓度用紫外分光光度计和琼脂糖凝胶电泳进行测定,要求DNA样本浓度在50 ng·μL-1以上,OD260/280应在1.7~2.1之间,DNA的总量应大于2 μg。
利用北京市农林科学院杂交小麦研究所自主开发的Affymetrix BAAFS Wheat 90K SNP芯片,对248个品种的自然群体进行基因分型,检测到覆盖小麦全基因组的68 519个位点。对基因型进行质量控制,质量控制标准为:去除杂合率大于10%、缺失率大于10%、MAF小于0.05且无物理位置的位点,最终获得63 658个高质量SNP位点用于本研究的关联分析。
1.4 全基因组关联分析与候选基因筛选
利用R语言(LEA)分析群体结构,PCA方法进行主成分分析;采用R语言的GAPIT软件(http://www.zzlab.net/GAPIT/)进行亲缘关系矩阵(Kinship)计算、亲缘关系聚类热力图绘制、连锁不平衡(linkage disequilibrium LD)分析;标记间的LD采用R2来衡量,计算整个基因组的LD值,绘制LD值和标记物理距离之间的散点图,然后进行LD衰减曲线的拟合,并定义LD衰减的阈值为R2=0.1[20];将PCA和Kinship纳入,利用GAPIT软件的MLM模型,对株高、穗长、小穗数、穗粒数、有效分蘖数、粒长、粒宽和千粒重共8个性状进行全基因组关联定位,当标记的P≤0.000 01 时认为标记与性状存在显著关联。将在2个及2个以上的环境中发现的位点视为稳定的位点。
针对多环境稳定显著关联SNP标记,参照中国春RefSeq V1.1基因注释信息,获取关联标记最近的基因,在Ensembl数据库(http://plants.ensembl.org/index.html)查找其基因功能。
2 结果与分析
2.1 表型数据分析
248个材料的8个性状在不同环境中均表现出较大的变异范围,说明目标性状是受多基因控制的数量性状。8个性状的方差分析表明(表1),群体各性状的基因型、环境以及基因型×环境互作中均达到显著水平(P≤0.05),除穗长在环境中为显著水平外,其他性状表现为极显著水平(P≤0.001)。各性状的平均广义遗传力差别较大,范围为0.562~0.891,所有性状均适合遗传分析。

表1 自然群体8个性状方差分析Table 1 Significant analysis of eight traits in natural population between genotype and environment
对性状间的相关性进行分析,大部分具有显著正相关性或显著负相关(表2)。株高与穗长呈极显著正相关,与有效分蘖数、粒宽呈显著正相关,与穗粒数呈显著负相关;穗部性状间,穗长分别与小穗数、穗粒数呈极显著正相关,小穗数与穗粒数呈显著正相关;但小穗数与千粒重呈极显著负相关;千粒重与粒长、粒宽呈极显著正相关,且相关系数分别为0.44和0.63。这说明产量相关性状间存在协同或抑制作用。

表2 小麦产量性状间的相关性分析Table2 Corelation analysis of yield-related traits in wheat
2.2 SNP标记分析
利用Affymetrix BAAFS Wheat 90K SNP芯片(包含84 661个SNP)分析248个样品的基因型,获得高质量多态性(PHR)标记68 519个,占比80.94%。进一步去除杂合率大于10%、缺失率大于10%、MAF值小于0.05、无物理位置的位点,剩余63 658个位点(占比75.19%)用于后续关联分析。每条染色体的标记数量变化范围为595个(4D)~5 982个(3B)(图1),A、B和D染色体组分别占比39.48%、45.03%和13.92%。全基因组标记密度为0.28 Mb/SNP,A、B和D染色体组标记密度分别为0.21、0.19和0.44 Mb/SNP。
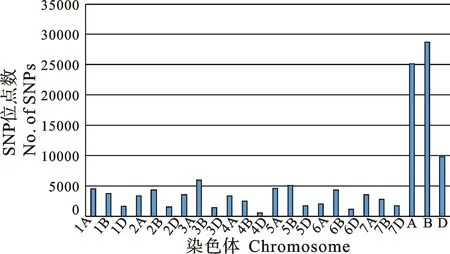
图1 SNP位点在染色体及各亚基因组上的分布
2.3 群体结构和亲缘关系分析
将63658个SNP标记均纳入群体结构、主成分以及亲缘关系分析。群体遗传结构分析表明,当K=3时,ΔK 值最大,由图2A可知,248个自然群体划分为3个亚群。有部分样品血缘比较复杂,说明自然群体间基因交流比较频繁。基于基因型的 PCA 分析(图2B)和亲缘关系分析(图2C)显示整个群体可分为3个群组, 分类结果与 Structure 结果相似。图2C结果显示除了少数品种外,大部分品种同其他品种之间的亲缘关系都并不太接近。
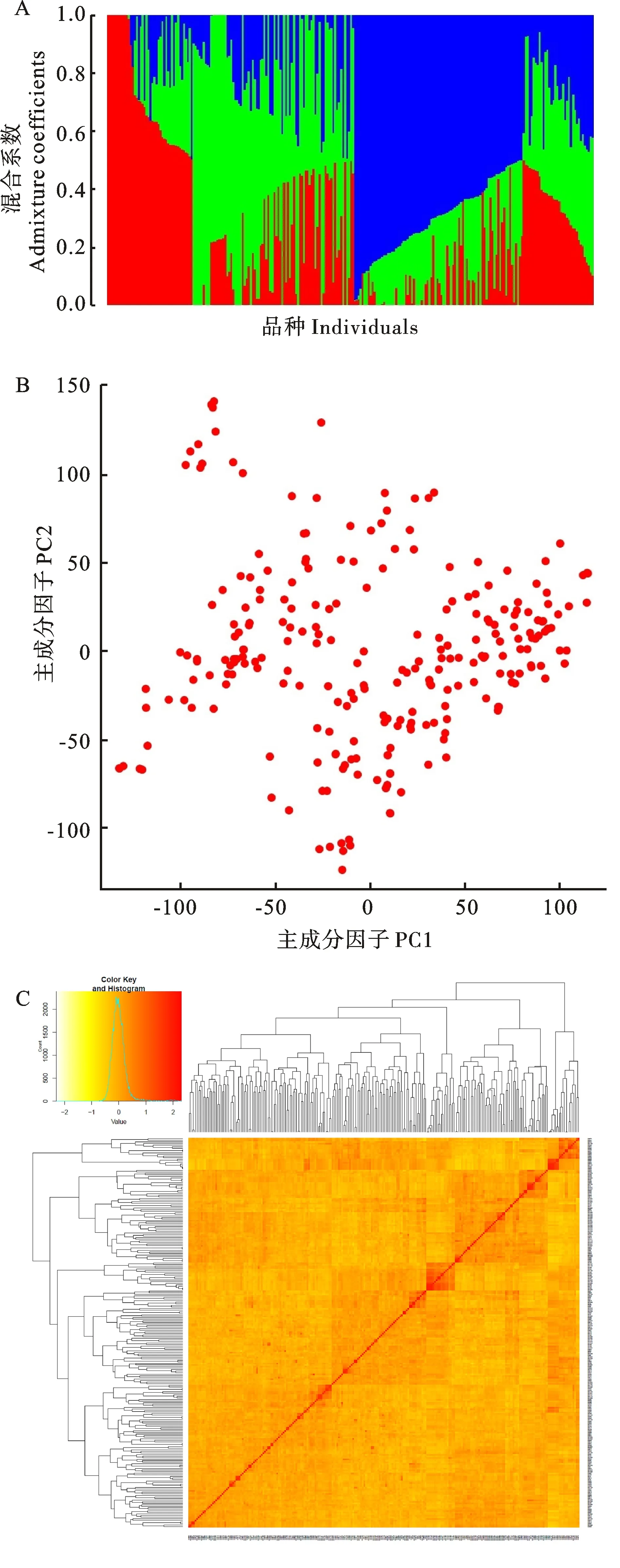
A:群体结构;B:主成分分析;C:亲缘关系。
2.4 连锁不平衡分析
将63 658个SNP标记均纳入连锁不平衡(LD)分析。从LD衰减图(图3)可以看出群体中的LD平均衰减距离为5 Mb,衰减速度较快,表明在群体进化过程中重组率较大,关联分析时所需SNP密度更大,能获得更小的候选区间,筛选时更容易。且LD衰减距离大于标记间的平均距离(0.28 Mb),说明标记对全基因组具有足够的覆盖度。

图3 连锁不平衡分析
2.5 全基因组关联分析
将63 658个SNP标记用于关联分析,共检测到158个与株高、穗长、小穗数、穗粒数、有效分蘖数、粒长、粒宽、千粒重显著关联(P≤0.000 01)的SNPs,分布在除1A、2D、3D、6B外的其它17条染色体上,在单一环境下的关联位点可解释4.05%~10.24%的表型变异(表3),有154个位点平均贡献率大于5%。其中45个SNPs在两个或两个以上环境中与性状显著关联,分布于1B、1D、2A、3A、4B、4D、5A、5B和7D染色体上,涉及株高、穗长、穗粒数、有效分蘖数、粒长、粒宽和千粒重共7个性状,解释平均表型变异的3.60%~10.51%。
有14个与株高显著关联的SNPs,分别分布在1B、4D和7D染色体上。其中位于4D染色体上的7个位点(Affx-111184993、Affx-111486973、Affx-109390358、Affx-88736027、Affx-88431037、Affx-108864084和Affx-88477950)和位于7D染色体上的3个位点(Affx-111766144、Affx-110475941和Affx-110885974)在两个以上环境中被检测到,解释3.60%~8.63%的表型变异,其中Affx-88477950对表型贡献率最高。
有23个与穗长显著关联的位点,分别分布在1B、1D、3A、5B、5D、6A、6D和7D染色体上。其中10个位点至少在两个环境中稳定表达, 如3A染色体上686.8 Mb处的6个标记(Affx-109542048、Affx-92104882、Affx-88470542、Affx-88776796、Affx-110357394和Affx-88480986)、5B(Affx-110744126)、7D(Affx-111294473)在三个以上环境同时被检测到,解释5.56%~8.42%的表型变异,其中Affx-111294473对表型贡献率最高。
有6个与小穗数显著关联的位点,分别分布在2B、3B、7A和7D染色体上,但均在单个环境中检测到,解释4.79%~8.58%的表型变异,其中Affx-109291016对表型贡献率最高。
有9个与穗粒数显著关联的位点, 分布在3A、3B、4A、4D和7D 染色体上。只有7D染色体上的Affx-111108657同时在三个以上环境中被检测到,对表型贡献率为6.06%~7.22%。
有47个与有效分蘖数显著关联的位点,分别分布在1B、5B和5D染色体上。其中位于1B染色体上的8个位点(Affx-109803920、Affx-111268269、Affx-111963063、Affx-110355319、Affx-109257091、Affx-111928763、Affx-111871302和Affx-109807825)同时在两个相同的环境中被检测到,物理位置为561.8~563.9 Mb,为同一基因座,解释6.06%~8.73%的表型变异,其中位点Affx-109807825对表型贡献率最高。
有11个与粒长显著关联的位点, 分别分布在2A、3A、4D、5A、5B和6D染色体上。其中位于2A染色体上的4个位点(Affx-88710623、Affx-110152685、Affx-109998980和Affx-110075012)和位于5B染色体上的2个位点(Affx-110374059和Affx-111349720)在两个以上环境中被检测到,解释5.45%~6.62%的表型变异,其中Affx-110374059对表型贡献率最高。2A染色体上,位于535.8~540.7 Mb的4个SNPs位点为同一个基因座;位于5B染色体上534.3 Mb处的2个SNPs位点为同一个基因座。
有24个与粒宽显著关联的位点,分别分布在2A、2B、4B、5A和7A染色体上。其中位于4B染色体上的6个位点(Affx-111258830、Affx-88421491、Affx-111007710、Affx-110710374、Affx-109181128和Affx-111875175)和位于5A染色体上的1个位点(Affx-109837870)在两个以上环境中被检测到,解释6.47%~10.51%的表型变异,其中Affx-111007710、Affx-110710374和Affx-109181128的表型贡献率最高。4B染色体上的6个位点位于36.0~37.2 Mb,为同一个基因座。
有24个与千粒重显著关联的位点,分别分布在3A、5D、7A和7B染色体上。其中位于3A染色体上的3个位点(Affx-111099384、Affx-110425849和Affx-109705103)同时在两个相同的环境中被检测到,解释7.05%~7.69%的表型变异,其中Affx-111099384和Affx-110425849的表型贡献率最高。
2.6 候选基因分析
共发现45个候选基因,其中有功能注释的基因共41个(表4),位于基因内的标记有4个。已知功能基因包括:细胞分裂素生物合成(cytokinin biosynthetic process)、水解酶(hydrolase activity)、神经酰胺代谢(ceramide metabolic process)、氧化-还原过程(oxidation-reduction process)、蛋白质结合(protein binding)、氧化还原酶(oxidoreductase activity),作用于单个供体与氧分子结合(acting on single donors with incorporation of molecular oxygen)、 两个氧原子结合(incorporation of two atoms of oxygen)、金属离子结合(metal ion binding)、ATP结合(ATP binding)、磷酸磷脂酰肌醇激酶 (phosphatidylinositol phosphate kinase activity)、转录调控(regulation of transcription)、细胞核(nucleus)、 DNA结合转录因子(DNA-binding transcription factor activity)、碳水化合物代谢(carbohydrate metabolic process)、水解酶(hydrolase activity)、RNA结合(RNA binding)、蛋白激酶(protein kinase activity)、蛋白磷酸(protein phosphorylation)等。本研究鉴定的候选基因及其功能表明植物产量涉及多种机制和复杂的代谢调控网络,然而,这些候选基因在小麦产量形成过程中的具体功能和机制还需要进一步研究。
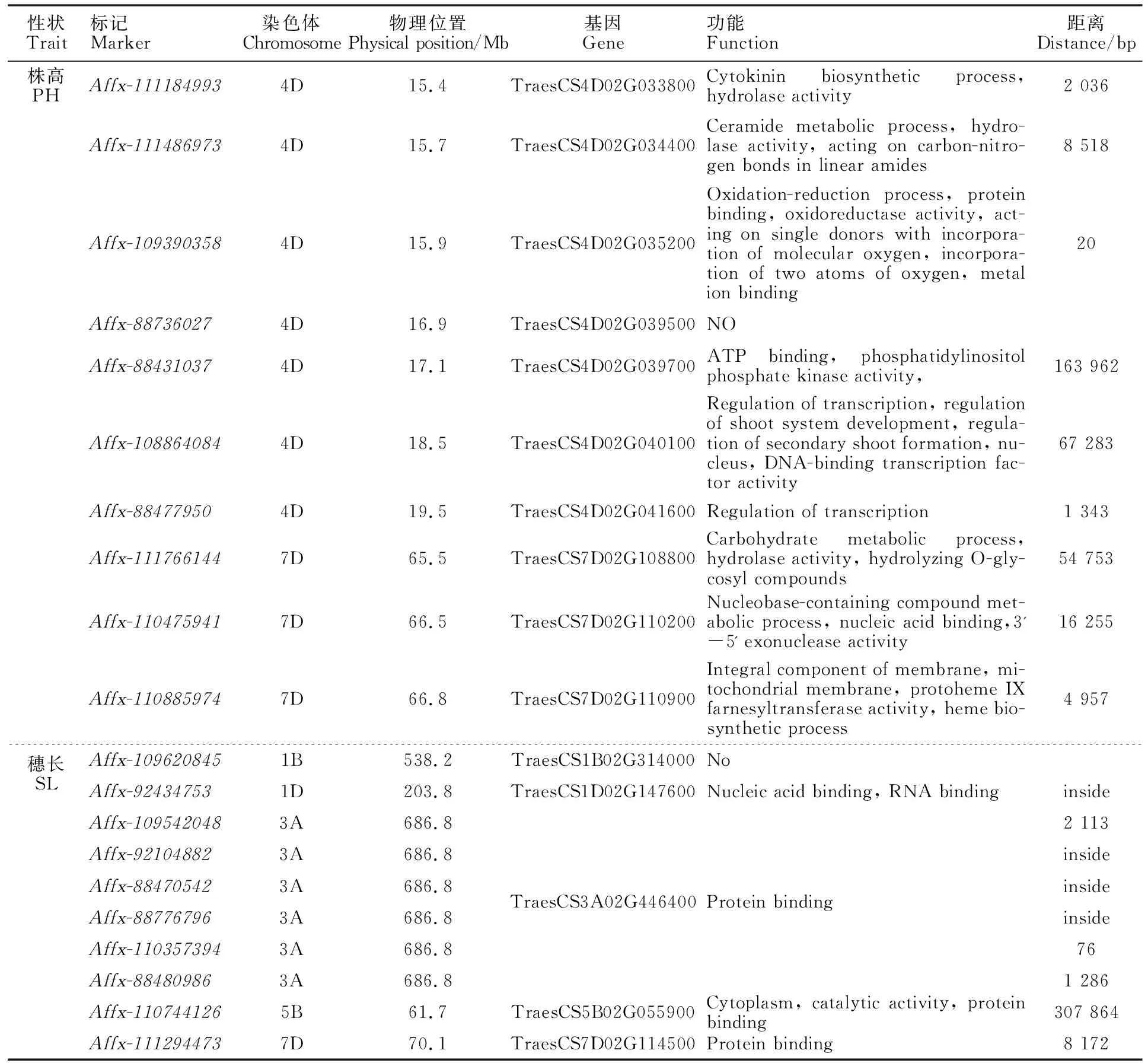
表4 稳定表达位点最近的候选基因及其功能注释Table 4 The nearest candidate genes at stable expression sites and functional annotations
3 讨论
3.1 小麦产量性状间的相关性分析
小麦产量性状包括株高、穗长、小穗数、穗粒数、有效分蘖数、粒长、粒宽、千粒重等重要指标,研究性状间的相互作用对产量的高低有重要作用。Gegas等[21]研究表明,粒长和粒宽对粒重有直接影响。Li等[22]和余曼丽等[23]研究发现千粒重与粒长、粒宽呈极显著正相关,与本研究结果一致。除此之外,本研究还发现小穗数与穗粒数呈极显著正相关,与Li等[22]和Li等[24]结果一致。说明六个相关性状之间相互关联、相互制约和相互协调的关系共同决定小麦产量高低。
3.2 小麦产量相关性状的GWAS分析
本研究利用高效90K芯片对248个育成品种组成的自然群体进行关联分析,共检测到158个与8个产量性状显著关联的SNP位点,其中45个位点至少在2个以上环境中稳定表达。有8个稳定关联位点与以往的研究结果一致(表5),37个为新发现位点。
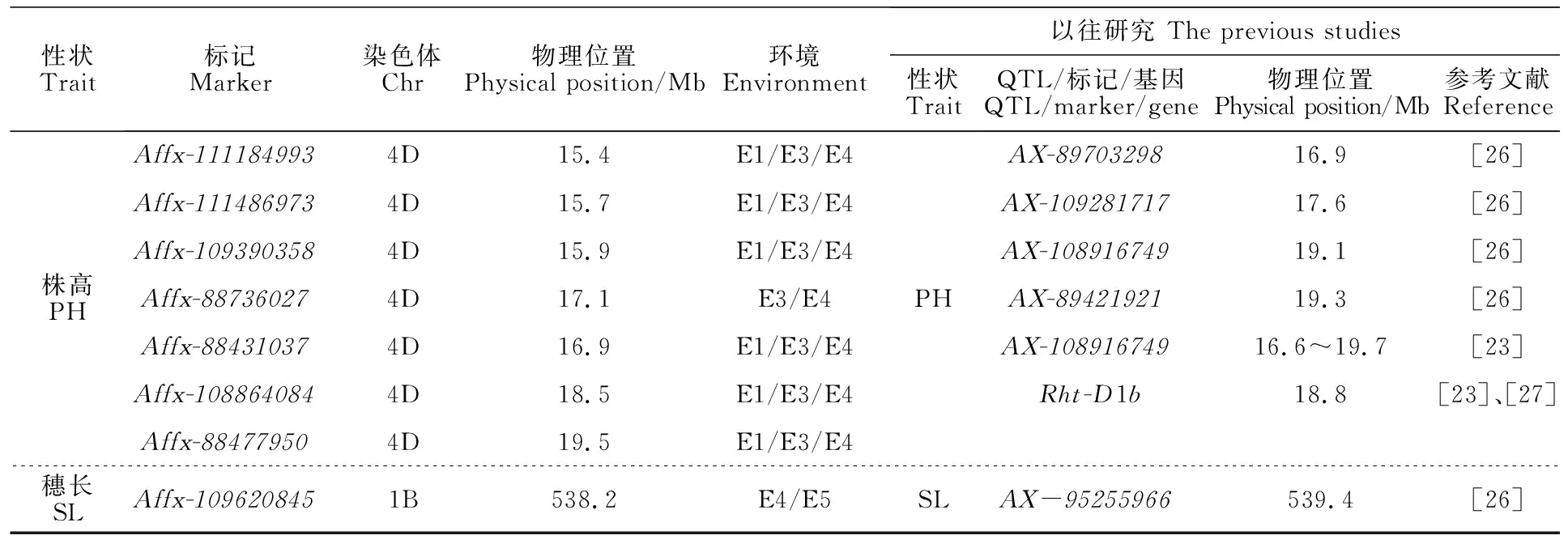
表5 小麦产量相关性状的关联位点与以往研究的比较Table 5 Comparison of the yield-related loci between the current study and previous studies
与株高稳定关联的位点有10个,分布在4D和7D染色体上。Hu等[25]在4D染色体上检测到4个株高相关位点,物理位置区间为16.9~19.3 Mb,Li等[22]发现了一个与株高相关联的位点,物理位置区间为16.6~19.7 Mb,除此之外,在其它研究中,也发现了与株高相关的等位基因Rht-D1b[26],物理位置为18.78 Mb。本研究中位于4D染色体上的7个株高相关位点的物理位置区间为15.4~19.5 Mb,其中4个位点(Affx-88736027、Affx-88431037、Affx-108864084和Affx-88477950)位于上述他人研究结果的区间内,3个位点(Affx-111184993、Affx-111486973和Affx-109390358)在附近区域,说明在此区间内可能存在控制株高的位点。位于7D染色体上的3个位点(Affx-111766144、Affx-110475941和Affx-110885974)的物理位置区间为65.5~66.8 Mb,可能为新位点。
与穗长稳定关联的位点有10个,分布在1B、1D、3A、5B和7D染色体上。在1B染色体上检测到的Affx-109620845位点与Hu等[25]定位到的位点AX-95255966相距1.2 Mb。分别在1D、3A、5B和7D染色体上检测到1、6、1和1个新位点,3A染色体上的6个位点位于同一基因座。
与穗粒数稳定关联的位点有1个(Affx-111108657),位于7D染色体上,物理位置为585.5 Mb,可能为新位点。
与有效分蘖数稳定关联的位点有8个(Affx4109803920、Affx-111268269、Affx-111963063、Affx-110355319、Affx-109257091、Affx-111928763、Affx-111871302和Affx-109807825),全部位于1B染色体上,物理位置为561.8~563.9 Mb,可判定为同一个基因座。
与粒长稳定关联的位点有6个且都为新鉴定到的位点,分别位于2A和5B染色体上。位于2A上的4个位点(Affx-88710623、Affx-110152685、Affx-109998980和Affx-110075012)为同一个基因座;位于5B染色体上的2个位点(Affx-110374059和Affx-111349720)为同一个基因座。
与粒宽稳定关联的位点有7个,分别位于4B和5A染色体上。位于4B染色体上的6个位点(Affx-111258830、Affx-88421491、Affx-111007710、Affx-110710374、Affx-109181128和Affx-111875175)为同一个基因座,为稳定新位点。位于5A染色体上的1个位点(Affx-109837870)为稳定的新位点。
与千粒重稳定关联的位点有3个(Affx-111099384、Affx-110425849和Affx-109705103),全部位于3A染色体上,为稳定的新位点。
本研究在株高和穗长中定位到的稳定位点与以往研究发现的很多位点属于同一个基因座,可对重要位点进一步开发SNP标记,为标记辅助育种提供可靠的位点。本研究同时定位到37个稳定新位点,分别是7D染色体上定位到的3个与株高相关位点;1D、3A、5B和7D染色体上定位到的9个穗长相关位点;7D染色体上定位到的1个与穗粒数相关位点;1B染色体上定位到8个与有效分蘖数相关的位点;2A和5B染色体上定位到的6个与粒长相关位点;4B和5A染色体上定位到的7个与粒宽相关位点;3A染色体上定位到的3个与千粒重相关位点。这些稳定的新位点与具有结合核酸、RNA、细胞质、蛋白质的基因以及具有催化活性和水解酶活性的基因关联(表4)。新位点的表型贡献率为3.60%~10.51%,有可能为主效位点,可在小麦产量育种中起到重要的作用,可优选这些关联位点进行功能标记开发,为分子标记辅助育种提供有效信息。

