登革病毒高度保守的HLA-A*1101限制性T细胞表位鉴定和体内免疫反应研究
陈新宇,段志良,江明华,贾庆军,徐娟娟,陈柏坤,文金生,
(1.温州医科大学附属第二医院育英儿童医院 检验科,浙江 温州 325027;2.温州医科大学 基础医学院虫媒病毒研究所,浙江 温州 325035)
登革病毒高度保守的HLA-A*1101限制性T细胞表位鉴定和体内免疫反应研究
陈新宇1,段志良1,江明华1,贾庆军2,徐娟娟2,陈柏坤2,文金生1,2
(1.温州医科大学附属第二医院育英儿童医院 检验科,浙江 温州 325027;2.温州医科大学 基础医学院虫媒病毒研究所,浙江 温州 325035)
目的:鉴定登革病毒2型(DENV-2)HLA-A*1101限制性T细胞表位并探讨其诱导的T细胞反应的特征。方法:基于DENV-2的氨基酸序列,采用T细胞表位预测软件预测HLA-A*1101限制性候选T细胞表位;基于HLA-A*1101转基因小鼠,采用ELISPOT实验、ELISA实验和CD8+T细胞(CTL)杀伤实验研究候选表位的HLAA*1101限制性和所诱导的T细胞反应的特征。结果:在合成的14条候选表位中,9条肽(NS1161-170、NS1263-272、NS36-15、NS4b44-53、NS333-42、C58-67、E30-38、NS3576-584和NS4a72-80)免疫HLA-A*1101转基因小鼠可诱导产生肽特异性分泌IFN-γ的T细胞。除了分泌IFN-γ,C58-67和NS3576-584特异性T细胞也可分泌TNF-α,而E30-38特异性T细胞更可同时分泌TNF-α和IL-6。DENV-2感染的HLA-A*1101转基因小鼠体内也可检测到针对NS4b44-53、NS333-42、C58-67、E30-38、NS3576-584和NS4a72-80的分泌IFN-γ的T细胞。采用上述6条肽的混合物免疫HLA-A*1101转基因小鼠所诱导的效应细胞可显著杀伤DENV-2感染的小鼠脾单核细胞。结论:本研究鉴定出了9条全新的DENV-2特异性HLA-A*1101限制性T细胞表位。
登革病毒;HLA-A*1101;表位/肽;转基因小鼠
登革病毒(dengue virus,DENV)感染常引起自限性登革热(dengue fever,DF),也可导致致死性登革出血热(dengue hemorrhagic fever,DHF)/登革休克综合症(dengue shock syndrome,DSS)。每年约有3.9亿人感染DENV,包括约1亿DF患者,50万DHF和(或)DSS,2万死亡病例[1-2]。大量研究表明:DENV特异性CD8+T细胞(CTL)不但可保护机体抵抗DENV的攻击、控制DENV感染和复制[3-5],而且可预防DENV二次感染时抗体依赖增强效应(antibody dependent enhancement,ADE)的发生[6]。因此,鉴定CTL表位将有助于研发新型登革候选疫苗及用于评估候选疫苗的细胞免疫效应。本研究拟预测合成DENV-2中潜在的HLA-A*1101限制性CTL表位,候选表位免疫HLA-A*1101转基因小鼠,采用酶联免疫斑点(ELISPOT)实验和ELISA实验检测肽特异性分泌IFN-γ、TNF-α和IL-6的T细胞的水平。DENV-2感染HLA-A*1101转基因小鼠,采用ELISPOT实验检测肽特异性分泌IFN-γ的T细胞的水平,最后采用CTL杀伤实验评估肽特异性T细胞对DENV-2感染的靶细胞的杀伤效应。
1 材料和方法
1.1材料
1.1.1表位预测和肽合成:基于DENV-2(NGC株;GenBank Accession No:AAC59275)氨基酸序列,采用SYFPEITHI软件(http://www.syfpeithi.de/ bin/MHCServer.dll/EpitopePrediction.htm)预测HLA-A*1101限制性CTL表位[7],并用PAProc软件(http://www.paproc.de/)分析预测的候选表位是否含有人蛋白酶体裂解位点。委托上海强耀多肽合成公司合成纯度>95%的候选表位和1条广谱性Pan-DR Th表位(PADRE:AKFVAAWTLKAAA),并溶于无菌PBS中至终浓度1 mg/mL。
1.1.2细胞株:C6/36细胞株购自美国ATCC并采用含10%胎牛血清、100 U/mL青霉素和100 μg/mL链霉素的RPMI-1640培养基培养。
1.1.3免疫小鼠分组:CB6F1-Tg(HLA-A*1101/H2-Kb)A11.01小鼠(HLA-A*1101转基因小鼠)购自美国Taconic公司并于SPF级动物房饲养和繁殖。将雌性HLA-A*1101转基因小鼠(6~8周龄)随机分成16组(每组5只)。250 μg每条肽与250 μg PADRE肽溶于500 μL PBS,然后与500 μL弗氏完全佐剂进行乳化,随后,将乳化好的肽溶液平均在5只小鼠背部皮下多点注射。1周后,同样肽与弗氏不完全佐剂的乳化液加强免疫小鼠3次(间隔1周)。对照小鼠(模拟免疫组)采用同样的免疫策略和佐剂免疫(无任何肽)。另外,6条肽的混合液(每条肽50 μg)采用上述免疫策略免疫5只小鼠。末次免疫1周后,处死小鼠,摘取小鼠脾和淋巴结(腘窝淋巴结和锁骨下淋巴结)并制备脾细胞和淋巴结细胞悬液。采用小鼠淋巴细胞分离液分离脾细胞中脾单个核细胞(splenic mononuclear cells,SMCs)。采用IFN-γ ELISPOT实验检测SMCs和淋巴结细胞中肽特异性分泌IFN-γ的T细胞的水平,采用ELISA实验检测脾细胞中分泌TNF-α和IL-6的T细胞的水平。另外,对来自6条肽混合物免疫小鼠的脾细胞,进一步采用小鼠淋巴细胞分离液分离脾细胞中SMCs,采用IFN-γ ELISPOT实验检测SMCs中肽特异性分泌IFN-γ的T细胞的水平。另外,SMCs作为效应细胞用于基于乳酸脱氢酶(LDH)释放的CTL杀伤实验。
1.2方法
1.2.1DENV-2感染HLA-A*1101转基因小鼠:将DENV-2(NGC株)加入C6/36细胞于37 ℃感染2 h,弃病毒液后于37 ℃细胞培养箱培养3~5 d。当细胞病变出现“+++”时,收集细胞上清液并采用0.22 μm过滤器过滤,滤过病毒液采用Vero细胞测定PFU后冻存于-80 ℃备用。通过小鼠尾静脉将上述滤过病毒液注入HLA-A*1101转基因小鼠体内(0.5 mL,1× 107PFU/小鼠)。4周后,处死小鼠制备脾细胞悬液并分离出SMCs。采用IFN-γ ELISPOT实验检测SMCs中单个上述候选表位特异性T细胞的水平。
1.2.2准备靶细胞:将正常的HLA-A*1101转基因小鼠的脾细胞铺于6孔细胞培养板,37 ℃培养2 h后,弃每孔上清液,采用RPMI-1640培养基轻轻洗孔3次。在感染复数(MOI)为5时,将DENV-2加入贴壁细胞(脾单核细胞)于37 ℃感染2 h,弃病毒液,加RPMI-1640维持液于37 ℃培养24 h。未感染的脾单核细胞和DENV-2感染的脾单核细胞做为靶细胞用于CTL杀伤实验。
1.2.3小鼠IFN-γ ELISPOT检测:小鼠IFN-γ ELISPOT试剂盒(预包被96孔板)购自荷兰U-CyTech公司,ELISPOT实验方法参考本课题组前期研究[8]。将上述SMCs(每孔2×105细胞)或淋巴结细胞(每孔2×105细胞)加入ELISPOT板每孔中。背景对照孔(无肽)和实验孔(每孔20 μg肽)均为双复孔。于37 ℃培养箱培养24 h后,弃细胞并洗板。依次进行:加抗小鼠IFN-γ单抗并孵育,洗板;加HRP标记的链霉亲合素并孵育,洗板;加底物并显色,洗板。最终,孔中每个斑点代表一个斑点形成细胞(spot forming cell,SFC)或肽特异性分泌IFN-γ的T细胞。采用ELISPOT读板仪计数每孔斑点数,结果表示为IFN-γ SFCs/2×105SMCs或淋巴结细胞。
1.2.4小鼠细胞因子ELISA检测:小鼠TNF-α,IL-6 ELISA试剂盒(预包被96孔板)购自达科为生物科技公司。参考说明书进行操作,将肽免疫的小鼠的脾细胞(1×106细胞)加入无菌的1.5 mL EP管(500 μL培养基)。设置背景对照(无肽)和实验孔(20 μg不同的候选表位肽)。于37 ℃培养48 h后,离心EP管后将100 μL上清转移至ELISA 96孔板中。随后按照说明书,依次加抗小鼠TNF-α或IL-6的抗体、孵育、洗板;加HRP标记的链霉亲合素、孵育、洗板;加底物并显色。采用酶标仪测定每孔液体的OD450值。根据TNF-α或IL-6标准品测定并绘制的标准曲线,计算出每孔中细胞因子的浓度(pg/mL)。
1.2.5基于LDH释放的CTL杀伤试验:细胞毒实验试剂盒购自美国Promega公司,并用于测定肽特异性T细胞杀伤DENV-2感染的小鼠脾单核细胞的能力。根据说明书,本实验在V形底96孔板中进行。6条肽混合免疫小鼠的SMCs用作效应细胞,未感染的小鼠脾单核细胞和DENV-2感染的小鼠脾单核细胞做为靶细胞。将各种细胞依次加入V形底孔中。实验孔包括恒定量的靶细胞(每孔1×104细胞)和3种不同量的效应细胞[效/靶(E:T)比分别为10:1,5:1,1:1],靶细胞自发释放孔(TS孔,每孔1×104细胞),效应细胞自发释放孔(ES孔,每孔1×105或5×104或1×104细胞),靶细胞最大释放孔(TM孔,每孔1×104细胞),培养基孔(CM孔),容积校正孔(VC孔)。37 ℃孵育8 h后,向TM孔和VC孔加入细胞裂解液,于37 ℃继续孵育1 h。离心96孔板(400 g,4 min),转移每孔50 μL上清至平底96孔板每孔中,然后每孔加入底物,于37 ℃避光反应20 min。采用酶标仪测定每孔OD490。针对每个E:T比,将各孔OD490值代入下面公式以计算靶细胞被效应细胞杀伤的百分比。
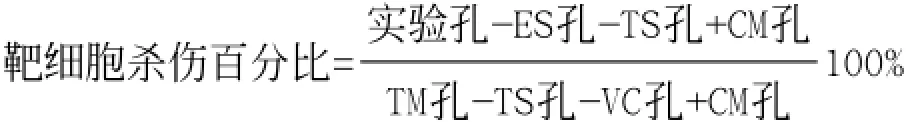
1.3统计学处理方法 采用SPSS17.0软件进行统计学分析。数据采用来表示,2组间比较采用t检验。P<0.05为差异有统计学意义。
2 结果
2.1候选表位的特征 从预测的表位中选择合成了14条预测得分高并且羧基端为人蛋白酶体裂解位点的候选表位,其中5条为9肽[NS549-57(ETDHHAVSR)、NS5571-579(LTYQNKVVR)、E30-38(CVTTMAKNK)、NS3576-584(NVEVEIWTK)和NS4a72-80(LMSGRGIGK)],9条为10肽[NS1161-170(GVFTTNIWLK)、NS1263-272(QTAGPWHLGK)、NS2a85-94(PTFAAGLLLR)、NS2a126-135(ALALGMMVLK)、NS36-15(DVPSPPPVGK)、NS4b44-53(ATTFVTPMLR)、NS333-42(YSQIGAGVYK)、NS4b77-86(GLGKGWPLSK)和C58-67(TIPPTAGILK)]。BLAST检索显示,大部分候选表位(12/14)在超过700个DENV-2分离株中保守(表1),E30-38为保守性最高的肽。另外,4条肽(NS1161-170、NS1263-272、NS5571-579和E30-38)在其他DENV血清型中也保守,见表1。
2.2候选表位诱导HLA-A*1101转基因小鼠产生肽特异性T细胞反应 14条候选CTL表位中,9条肽(NS1161-170、NS1263-272、NS36-15、NS4b44-53、NS333-42、C58-67,E30-38、NS3576-584和NS4a72-80)免疫HLA-A*1101转基因小鼠诱导产生了高水平的可分泌IFN-γ的T细胞反应,其中NS1263-272诱导的T细胞反应最强烈,见图1。而剩余5条肽(NS2a85-94、NS2a126-135、NS4b77-86、NS549-57和NS5571-579)所诱导产生的T细胞频率与背景对照差异无统计学意义。采用ELISA实验检测了肽诱导产生的T细胞在分泌IFN-γ以外是否还可分泌促炎因子(TNF-α和IL-6)。E30-38诱导的T细胞还可同时分泌TNF-α和IL-6,即使C58-67和NS3576-584特异性T细胞也可分泌TNF-α;而剩余的6条肽诱导的T细胞不分泌TNF-α和IL-6,见图2A-B。
2.3DENV-2感染HLA-A*1101转基因小鼠产生肽特异性T细胞反应 DENV-2感染小鼠的SMCs可识别6条肽(NS4b44-53、NS333-42、C58-67、E30-38、NS3576-584和NS4a72-80),表现为SMCs受到这些肽刺激时可分泌IFN-γ(图3)。另外,ELISA结果提示E30-38特异性T细胞还可分泌TNF-α和IL-6,而C58-67和NS3576-584特异性T细胞只产生TNF-α(数据本文未提供)。DENV-2感染小鼠的SMCs在受到其他8条肽刺激时既不分泌IFN-γ,也不分泌TNF-α和IL-6,见图3。
2.4候选表位免疫的HLA-A*1101转基因小鼠体内肽特异性T细胞的细胞毒效应 在混合肽免疫小鼠的SMCs中存在单个肽特异性分泌IFN-γ的T细胞;混合肽免疫小鼠的SMCs可高效杀伤DENV-2感染的小鼠脾单核细胞,靶细胞杀伤百分率与E:T比呈正比[(54.69±12.23)%,E:T=10:1;(31.21±8.79)%,E:T= 5:1;(10.85±4.38)%,E:T=1:1];而SMCs对正常的小鼠脾单核细胞无显著性杀伤效果,见图4和图5。

表1 HLA-A*1101限制性候选T细胞表位
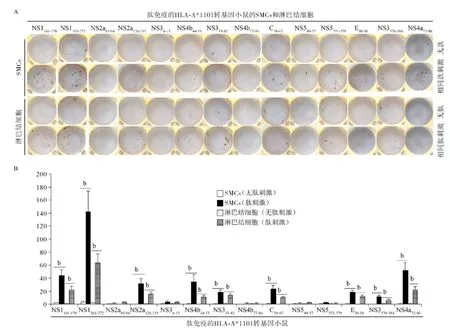
图1 肽免疫的HLA-A*1101转基因小鼠脾和淋巴结内肽特异性分泌IFN-γ的细胞的频率
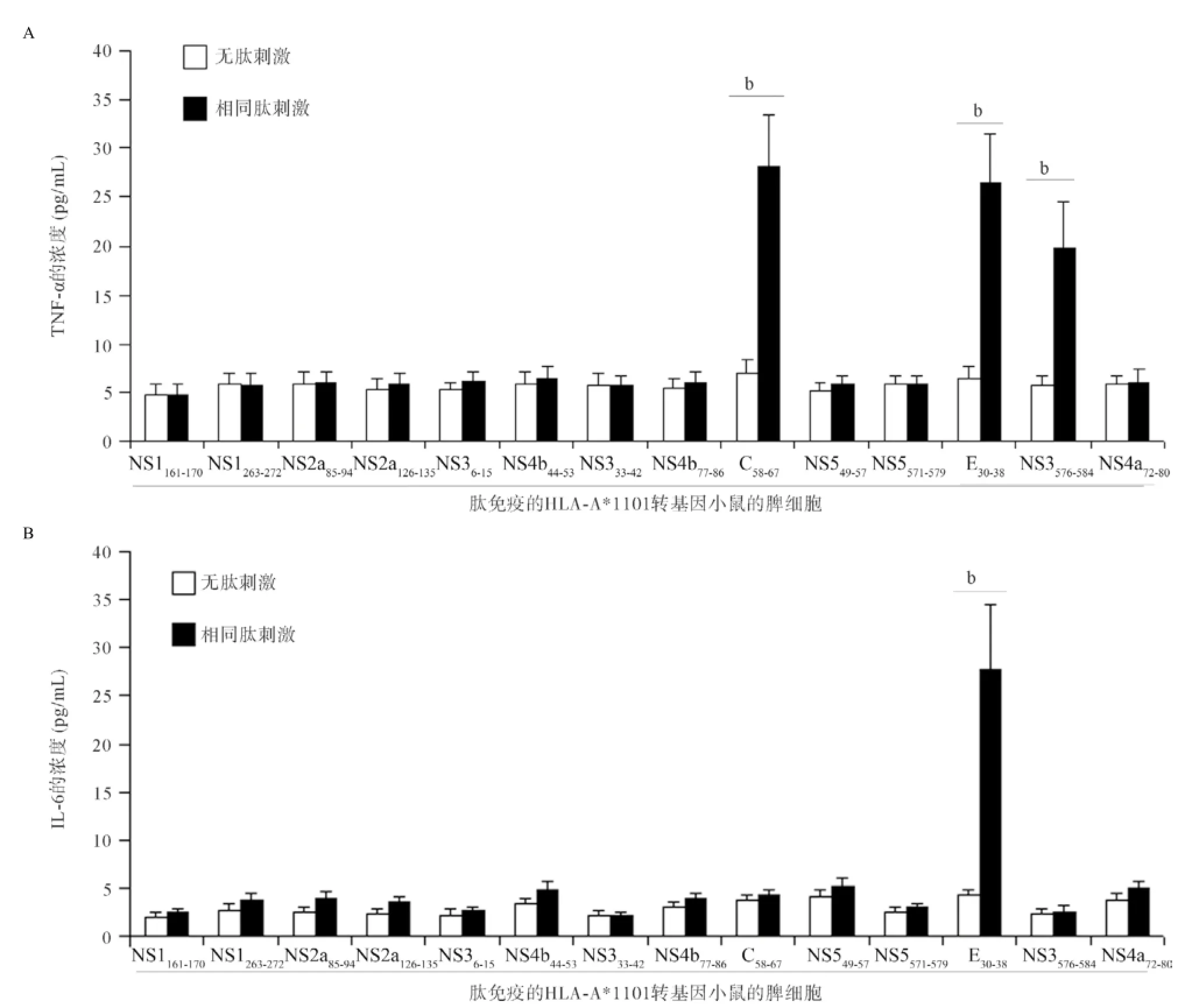
图2 肽免疫的HLA-A*1101转基因小鼠脾内肽特异性细胞分泌TNF-α和IL-6的情况
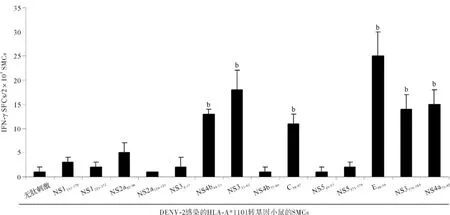
图3 DENV-2感染的HLA-A*1101转基因小鼠脾内肽特异性分泌IFN-γ的细胞

图4 6条肽混合物免疫的HLA-A*1101转基因小鼠脾内肽特异性分泌IFN-γ的细胞
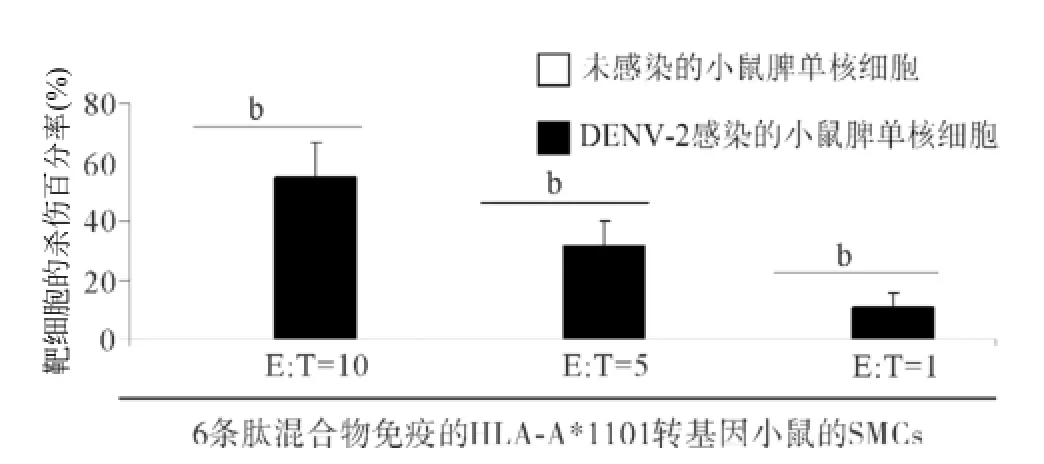
图5 6条肽混合物免疫的HLA-A*1101转基因小鼠SMCs的杀伤效应
3 讨论
考虑到HLA-A的等位基因A*1101具有广泛人群覆盖率,其在不同肤色和种族中人群覆盖率均超过20%[9]。DENV-2是中国近年来主要的流行血清型[10]。因此,本研究拟鉴定DENV-2中受HLA-A*1101限制的CTL表位并探讨其所诱导T细胞反应的特征。考虑到IFN-γ是T细胞分泌的一种可抑制DENV复制的细胞因子[11],因此我们首先评估候选CTL表位在HLAA*1101转基因小鼠内诱导的T细胞能否分泌IFN-γ。尽管14条候选表位均具有较高的预测得分,但只有9条肽可诱导显著的IFN-γ+T细胞反应。值得注意的是,所有14条候选表位免疫Balb/c和C57BL/6小鼠均未诱导具有统计学意义的IFN-γ+T细胞反应,而且模拟(无肽)免疫的HLA-A*1101转基因小鼠体内也不存在这些候选表位特异性IFN-γ+T细胞反应(数据本文未提供)。上述实验结果强烈提示上述9条肽应该受HLA-A*1101限制性,而不受小鼠MHC分子(H-2分子)限制。Imrie等[12]的研究表明:人的DENV特异性多功能性CTL能够同时分泌IFN-γ、IL-6 和TNF-α。我们前期基于HLA-A*2402转基因小鼠的研究也发现小鼠的DENV特异性多功能CTL也可分泌这些细胞因子[13]。而本研究再一次证明了这种现象的存在:E30-38、C58-67和NS3576-584可诱导多功能性T细胞反应。最新研究表明:DENV特异性多功能CTL细胞可保护机体免于感染DENV[5]。因此,这3条表位可能具有诱导保护性免疫的效应。
为了确定DENV蛋白是否可被小鼠蛋白酶体加工释放出这些候选CTL表位,进而诱导T细胞反应。将DENV-2通过血液途径感染HLA-A*1101转基因小鼠,在体外采用各个候选CTL表位刺激小鼠SMCs,采用ELISPOT实验检测SMCs在受到候选表位刺激时是否分泌IFN-γ。我们发现DENV-2感染的小鼠的SMCs可识别6条上述鉴定的表位(NS4b44-53、NS333-42、C58-67、E30-38、NS3576-584和NS4a72-80)。这些数据提示DENV的蛋白可被小鼠蛋白酶体加工产生上述6条肽,后者诱导了T细胞反应。由于DENV在小鼠细胞内复制水平低,产生的肽的浓度也偏低,因而诱导的T细胞反应的水平较弱。3条肽(C58-67、E30-38和NS3576-584)特异性T细胞分泌TNF-α和IL-6的模式同肽免疫小鼠中T细胞分泌这些细胞因子的模式一致,再次印证了这些多功能性T细胞反应。尽管NS1161-170、NS1263-272和NS36-15免疫HLA-A*1101转基因小鼠可诱导高水平的T细胞反应,但三者不被DENV-2感染小鼠的SMCs识别,提示DENV的蛋白在小鼠细胞内无法加工产生这3条肽。由于6条肽(NS4b44-53、NS333-42、C58-67、E30-38、NS3576-584和NS4a72-80)可诱导IFN-γ+ T细胞反应,并且可被DENV-2感染的HLA-A*1101转基因小鼠的脾细胞识别。因此,这些表位可做为免疫原用于评估登革候选疫苗在小鼠体内诱导T细胞免疫反应的情况。另外,由于6条表位(NS4b44-53、NS333-42、C58-67、E30-38、NS3576-584和NS4a72-80)均受HLA-A*1101分子呈递,因此我们想知道这6条肽同时免疫机体时是否存在相互干扰以及所诱导的T细胞是否可杀伤DENV-2感染的靶细胞。结果,6条肽混合免疫小鼠也可诱导产生单个肽特异性IFN-γ+T细胞(说明肽与肽之间在免疫时无明显干扰),后者可高效杀伤DENV-2感染的小鼠脾单核细胞。
研究人员仅从DENV中鉴定出了数量有限的HLAA*1101限制性表位。例如,NS3133-142(GTSGSPIIDK)和E258-266(GAMHSALAG)被鉴定为HLA-A*1101限制性表位[14]。Weiskopf等[15]报道了9条DENV-2特异性HLA-A*1101限制性表位。Appanna等[16]从DENV-2中鉴定出了HLA-A11限制性表位。将我们本研究鉴定的表位与上述研究人员报道的表位对比可知,本文鉴定的表位NS333-42(YSQIGAGVYK)比Weiskopf研究团队报道的HLA-A*1101限制性表位NS334-42(SQ IGAGVYK)多一个氨基酸残基。Vaughan等[17]报道NS4a61-80(LATVTGGIFLFLMSGRGIGK)是一个T细胞表位,但并没有确定其最短的免疫优势肽。本研究鉴定的表位NS4a72-80(LMSGRGIGK)位于上述肽序列内,因而应该是最短的免疫优势肽。Rivino等[18]从DENV-2鉴定出了一条CTL表位NS1161-175(GVFTT NIWL KLKEKQ)但未确定其HLA限制性。近期研究表明NS1162-171(VFTTNIWLKL)是一条HLA-A*2402限制性表位[12]。有趣的是,本研究证明与其高度相似的肽NS1161-170(GVFTTNIWLK)为HLA-A*1101限制性表位,二者仅有1个氨基酸残基差异。
本研究鉴定出了9条DENV-2特异性HLA-A*1101限制性表位。本研究所鉴定的表位可被用于研究CTL的功能、评估登革候选疫苗在小鼠内细胞免疫效果,甚而研发基于CTL表位的新型登革疫苗。
[1]WHITEHEAD S S,BLANEY J E,DURBIN A P,et al. Prospects for a dengue virus vaccine[J]. Nat Rev Microbiol,2007,5(7): 518-528.
[2]BHATT S,GETHING P W,BRADY O J,et al. The global distribution and burden of dengue[J]. Nature,2013,496 (7446): 504-507.
[3]TESTA J S,SHETTY V,SINNATHAMBY G,et al. Conserved MHC class I-presented dengue virus epitopes identifed by immunoproteomics analysis are targets for crossserotype reactive T-cell response[J]. J Infect Dis,2012,205(4): 647-655.
[4]ZELLWEGER R M,MILLER R,EDDY W E,et al. Role of humoral versus cellular responses induced by a protective dengue vaccine candidate [J]. PLoS Pathog,2013,9(10):e1003723.
[5]WEISKOPF D,ANGELO M A,DE AZEREDO E L,et al. Comprehensive analysis of dengue virus-specifc responses supports an HLA-linked protective role for CD8+ T cells[J]. Proc Natl Acad Sci U S A,2013,110(22): E2046-E2053.
[6] ZELLWGER R M,EDDY W E,TANG W W,et al. CD8+ T cells prevent antigen-induced antibody-dependent enhancement of dengue disease in mice[J]. J Immunol,2014,193(8):4117-4124.
[7]张跃进,柳献云,陈向敏,等. HCMV-UL138 CTL表位肽重组酵母菌的构建及其免疫效应[J]. 温州医科大学学报,2015,45(6): 406-414.
[8]钟晓芝,段志良,郭江龙,等. 丙型肝炎病毒HLA-A*1101和A*2402限制性CD8+T细胞表位的研究[J]. 温州医科大学学报,2014,44(8): 547-554.
[9]PAN Q Q,FAN S,WANG X Y,et al. The distribution of human leukocyte antigen-A,-B,and -DRB1 alleles and haplotypes based on high-resolution genotyping of 167 families from Jiangsu Province,China[J]. Hum Immunol,2011,72 (10): 872-876.
[10] JIANG L,WU X,WU Y,et al. Molecular epidemiological and virological study of dengue virus infections in Guangzhou,China,during 2001-2010[J]. Virol J,2013,10:4.
[11] DIAMOND M S,ROBERTS T G,EDGIL D,et al. Modulation of dengue virus infection in human cells by alpha,beta,and gamma interferons[J]. J Virol,2000,74(11): 4957-4966.
[12] IMRIE A,MEEKS J,GURARY A,et al. Differential functional avidity of dengue virus-specifc T-cell clones for variant peptides representing heterologous and previously encountered serotypes[J]. J Virol,2007,81(18): 10081-10091.
[13] DUAN Z L,LIU H F,HUANG X,et al. Identification of conserved and HLA-A*2402-restricted epitopes in Dengue virus serotype 2[J]. Virus Res,2015,196: 5-12.
[14] CHAKRABORTY S,CHAKRAVORTY R,AHMED M,et al. Computational approach for identifcation of epitopes in dengue virus envelope protein: a step towards designing a universal dengue vaccine targeting endemic regions[J]. In Silico Biol,2010,10(5-6): 235-246.
[15] WEISKOPF D,YAUCH L E,ANGELO M A,et al. Insights into HLA-restricted T cell responses in a novel mouse model of dengue virus infection point toward new implications for vaccine design[J]. J Immunol,2011,187(8): 4268-4279.
[16] APPANNA R,HUAT T L,SEE L L,et al. Cross-reactive T-cell responses to the nonstructural regions of dengue viruses among dengue fever and dengue hemorrhagic fever patients in Malaysia[J]. Clin Vaccine Immunol,2007,14(8): 969-977.
[17] VAUGHAN K,GREENBAUM J,BLYTHE M,et al. Metaanalysis of all immune epitope data in the Flavivirus genus:inventory of current immune epitope data status in the context of virus immunity and immunopathology[J]. Viral Immunol,2010,3(3): 259-284.
[18] RIVINO L,KUMARAN E A,JOVANOVIC V,et al. Differential targeting of viral components by CD4+ versus CD8+ T lymphocytes in dengue virus infection[J]. J Virol,2013,87(5): 2693-2706.
(本文编辑:吴彬)
Identification of dengue virus-derived highly conserved HLA-A*1101-restricted T-cell epitopes and study on their in vivo immune responses
CHEN Xinyu1,DUAN Zhiliang1,JIANG Minghua1,JIA Qingjun2,XU Juanjuan2,CHEN Bokun2,WEN Jinsheng1,2. 1.Department of Clinical Laboratory,the Second Affiliated Hospital & Yuying Children's Hospital of Wenzhou Medical University,Wenzhou,325027; 2.Institute of Arboviruses,School of Basic Medical Sciences,Wenzhou Medical University,Wenzhou,325035
Objective: To identify dengue virus serotype 2 (DENV-2)-derived HLA-A*1101-restricted T-cell epitopes and explore the characteristics of induced T cells. Methods: Based on the complete amino acid sequence of DENV-2,T-cell epitope prediction software was utilized to predict putative HLA-A*1101-restricted T-cell epitope. Based on HLA-A*1101 transgenic mice,the HLA-A*1101 restriction of epitope candidates and the characteristics of induced T cells were determined using ELISPOT assay,ELISA assay and CTL cytotoxicity assay. Results: Among 14 epitope candidated,each of 9 peptide (NS1161-170,NS1263-272,NS36-15,NS4b44-53,NS333-42,C58-67,E30-38,NS3576-584,and NS4a72-80) could induce IFN-γ-secreting T cell response in HLA-A*1101 transgenic mice. Except for IFN-γ,C58-67- and NS3576-584-specifc T cells also could secret TNF-α while E30-38-specifc T cells could secret TNF-α and IL-6 simultaneously. High levels of IFN-γ-secreting T cells were detected in DENV-2-infected HLA-A*1101 transgenic mice and directed to 6 peptides (NS4b44-53,NS333-42,C58-67,E30-38,NS3576-584,and NS4a72-80). The effector cells from HLA-A*1101 transgenic mice immunized with the mixture of 6 abovementioned peptides effciently lysed DENV-2-infected splenic monocytes. Conclusion: Nine DENV-2-derived peptides are identifed as HLA-A*1101-restricted T-cell epitopes.
dengue virus; HLA-A*1101; epitope/peptide; transgenic mice
R373.2
A DOI: 10.3969/j.issn.2095-9400.2016.08.002
2015-11-11
国家自然科学基金资助项目(81501363);浙江省自然科学基金资助项目(LQ14C010006);浙江省科技计划项目(Y2014 0653)。
陈新宇(1976-),男,浙江温州人,副主任技师。
文金生,教授,硕士生导师,Email:wjs78@wmu.edu. cn。

