基于传统培养和高通量测序方法分析羊肉加工过程中的菌群多样性
,2,*,2
(1.江苏省农业科学院农产品加工研究所,江苏南京 210014; 2.青海省青海湖肉业有限责任公司,青海海南州 813000)
肉的腐败变质主要是由于微生物的污染和酶的作用,其中微生物的入侵和大量繁殖是造成肉质腐败的主要原因。从屠宰到加工微生物的来源广泛而多样,甚至可以造成交叉污染,因此控制肉品质量保证其安全性的首要条件是防止微生物的污染繁殖,而在微生物繁殖中控制微生物中优势腐败菌的生长繁殖,是对肉品进行保鲜的有效手段。抑制腐败菌的生长首先就要控制其生物膜的形成量,这对优势腐败菌尤为重要,生物膜是细菌繁殖并不断聚集形成的一种保护膜,生物膜的存在使得细菌对杀菌剂(抗生素等)、宿主自身的免疫机制甚至是恶劣的环境都有很大的抵抗性。生物膜的这一特点,使抗生素对细菌治疗效果大打折扣[1],尤其是一些引发慢性感染疾病的致病菌更难治愈[2]。近年来对肉品中优势腐败菌的研究也越来越多,王宁[3]研究表明,冷却羊肉的腐败菌中,假单胞菌作为优势菌群占了约30%,此外肠杆菌、乳酸菌、葡萄球菌等腐败菌也常作为羊肉中的优势菌群,但所占比例在各类文献中略有不同。傅鹏等[4]发现冷却猪肉的初始菌相中也是假单胞菌属占优势。对肉的保鲜技术不断改进并延长其货架期一直是肉品研究的热点,较为传统的方法是通过低温、真空包装、气调包装[5]等延缓微生物的生长繁殖,李月明等[6]研究了生物可降解膜对肉品的保鲜作用,杨玉红[7]则利用了乳酸菌的低pH及其代谢产物(细菌素和类细菌素)来进行保鲜。
高通量测序法近来作为一种新的分子生物技术,具有通量高、准确率高、速度快的特点[8-10],能对整个样品中的微生物种类和相对数量有一定的分析,更完整地体现微生物的群落特征[11]。目前高通量测序被广泛应用于食品加工[12-13]和微生物体系如泡菜、白酒、奶酪等[14-16]的检测,甚至在检测肠道微生物[17-19]和水土中相关样品[20-21]中也有应用。
本实验擦取了屠宰至加工包装的各个过程中羊胴体表面及其能接触到的台面上的细菌,研究整个流程中羊肉表面的优势菌群及各菌株的成膜能力。旨在利用传统分离菌落方法和高通量测序法研究羊肉胴体表面的优势菌,并利用结晶紫染色研究各菌株的成膜能力,为下一步羊肉产品的抑菌保鲜提供理论基础。
1 材料与方法
1.1 材料与仪器
样品 从羊胴体表面及所接触台面擦拭所得的菌样;营养琼脂(Nutrient Agar)、营养肉汤(Nutrient Broth,NB)、假单胞菌分离琼脂(Pseudomonas Isolation Agar)、假单胞分离肉汤(Pseudomonas Isolation Broth)、胰蛋白胨大豆肉汤(Trypticase Soy Broth)、月桂酸硫酸盐胰蛋白胨肉汤(Lauryl Sulfate Tryptose Broth,LST)、MRS培养基(MRS Medium)、MRS肉汤(MRS Broth) 北京路桥技术有限责任公司;甘露醇氯化钠琼脂(Mannitol Salt Agar)、结晶紫中性红胆盐葡萄糖琼脂(Violet Red Bile Glucose Agar,VRBGA) 北京奥博星生物科技有限责任公司;细菌基因组DNA提取试剂盒 天根生化科技(北京)有限公司。
酶标仪 美国BioTek仪器公司;22331台式高速冷冻离心机 德国艾本德股份公司(Eppendorf AG);TP600梯度升降温功能PCR仪 日本Takara Bio Inc.;变性梯度凝胶电泳系统 上海培清科技有限公司。
1.2 实验方法
1.2.1 菌落总数测定 本实验选取九个取样点,其中羊胴体表面选取三处,分别为开膛前、开膛后、冲洗后,每个取样点取三只羊胴体,每只胴体三个平行面。其余的取样点为冲洗的台面、分割羊肉的台面两个、案板、分离内脏后冲洗用水、烫毛用水,每个取样点三个平行。将棉签在(5×5) cm2的取样器范围内擦拭,将沾有菌落的棉签放入装有225 mL无菌水的均质袋中振荡混合均匀,取1 mL混合均匀的菌液移入培养皿,然后倒入PCA固体培养基待其凝固之后倒置培养24~48 h至菌体长出计数。
1.2.2 菌种的分离与保存 把原始菌液稀释到一定梯度,取100 μL涂布在PCA板和选择性培养基平板上培养24~48 h,从各平板中挑取菌落密度分布比较均匀的、呈单菌落的不同菌株再次对应不同的选择性液体培养基在37 ℃恒温振荡培养,然后继续平板涂布、液体培养,至少重复纯化三次。最终将分离纯化完成的菌液与50%(v/v)的无菌甘油以1∶1的比例混合均匀置于-80 ℃冷冻保存。
1.2.3 分离菌种鉴定 将从1.2.2中分离出的43株菌的菌液过夜培养,各取1 mL离心收集细菌,用100 μL灭菌的超纯水重悬然后100 ℃水浴加热10 min使细胞膜破碎,继续在4 ℃下10000 r/min离心10 min,取上清液作为模板进行PCR扩增。PCR扩增体系为40 μL,其中2×Master Mix为20 μL、超纯水14 μL、引物27F和1492R各2 μL、模板2 μL。PCR扩增完成后,取5 μL扩增后的样品和1 μL buffer混合均匀进行琼脂糖凝胶电泳鉴定,将能显示出清晰条带的PCR扩增产物和未显示出条带的菌液送至上海生工生物技术有限公司,所用引物为27F和1492R(5′-AGAGTTTGATCMTGGCTCAG-3′,5′-GGTTACCTTG TTACGACTT-3′)。所得的测序序列结果用Blast进行对比,并选取同源性最高的菌株序列,鉴定出菌种类型并利用其序列构建系统进化树。
1.2.4 菌株成膜能力比较 将从1.2.2中分离出的43株菌株按1%的接种量接种到无菌的NB液体培养基中,混合均匀后取200 μL菌液加入96孔板,对照组为不加入菌液的液体培养基,37 ℃培养24 h。取培养好的样品,首先在570 nm处测定吸光值,此时对照组和实验组的吸光值用OD1和OD11表示。然后吸除上层培养基并用0.01 mol/L的PBS洗三次后干燥。每孔加入200 μL 1%的结晶紫染色5 min,吸除染液PBS洗三次,干燥后加入200 μL 95%的乙醇30 min,测其570 nm处的吸光值,对照组和实验组的吸光值用OD2和OD22表示。菌株成膜能力用B值表示,B值=(OD22-OD2)/(OD11-OD1)。
1.2.5 DNA的提取和PCR扩增 取未稀释的初始样品,12000 r/min低温离心15 min收集菌体沉淀。DNA的提取参照DNA提取试剂盒(Omega Bio-tek,Norcross,GA,U.S.)的说明书方法进行。以提取的DNA作为模板,以338F和806R(5′-ACTCCTACGGG AGGCAGCAG-3′,5′-GGACTACNNGGG TATCTAAT-3′)作为引物对细菌的16S rDNA V3-V4区进行序列扩增。
1.2.6 高通量测序 将初步处理的样本委托北京奥维森基因科技有限公司,利用其Illumina Miseq PE300高通量测序平台测序。通过MiSeq平台测序得到的数据,依据所得的PE reads之间的overlap关系将reads进行拼接,并根据各样品数据对序列采用质量控制和过滤[22],去除嵌合体和序列末端低于20质量值的序列,切除引物错配和数据中的短序列(一般长度小于150 bp)。按照0.97[23]以上的相似性原则对序列进行归并并生成应用于物种分类的操作分类单元(operational taxonomic units,OTU)。根据OTU的聚类分析结果与silva数据库进行对比,得出相对应的物种多样性信息[24],计算各指数并分析alpha多样性进而对菌群的多样性和丰度进行评估[25]。利用UniFrac算法比较样品间物种的群落差异并进行多样性分析。
1.3 数据处理
每个指标重复试验3次。测定结果以平均值加减标准差表示,采用Excel软件和STATISTIX 8进行数据和制图分析。显著性水平设置为P<0.05。
2 结果与分析
2.1 各取样点菌落总数
图1为各取样点测得的菌落总数,A、B、C为屠宰分割的不同时期羊胴体表面菌落数,其余为加工过程中各接触面的取样。由图可知,相对于羊胴体,各分割台面的菌落总数明显略高,经检测案板G菌落总数最高可达106CFU/cm2与冲洗过羊胴体的台面D、分割下的内脏所用的清洗用水H和分割台面F都有显著性差异(P<0.05)。而脱毛所用的烫毛水可能因为温度高其菌数略有下降,仅有104CFU/cm2,并与其他接触面有显著性差异(P<0.05)。对于羊胴体表面A、B、C三点来说,开膛后和冲洗后相对于开膛前有显著性差异(P<0.05),羊胴体经过开膛之后,其菌数显著性下降(P<0.05),可能是开膛前与开膛后之间经过了一次水洗,而经过C冲洗(水管淋洗)之后,菌数又有略微下降的趋势,但相对于冲洗之前没有显著性差异(P>0.05)。由A、B、C三点可知,在羊胴体进入加工车间之前,开膛、冲洗等操作步骤对菌落数量的影响不大。而结合所有的取样点来看,当羊胴体进入包装间后,据测得的菌落数可知在羊肉的分割过程中很有可能造成交叉污染,增加羊肉表面菌群的丰度,可能会对胴体表面的菌群结构产生影响。
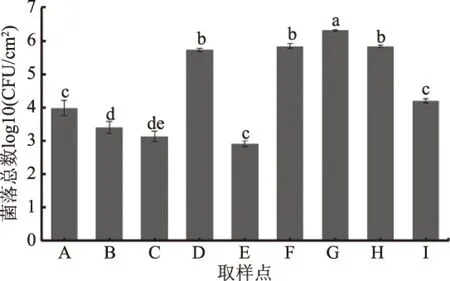
图1 各取样点菌落总数Fig.1 The number of colonies at each sampling point注:A:开膛前;B:开膛后;C:冲洗后;D:冲洗台面;E:分割台面1;F:分割台面2;G:案板;H:内脏分离水;I:烫毛水;不同小写字母代表在不同取样点菌落总数具有显著性差异(P<0.05)。
2.2 传统分离法鉴定菌株
将1.2.2中使用选择性培养基分离得到的43株菌进行测序,所得序列结果用Blast进行对比,并选取同源性最高的菌株序列,鉴定出菌株分别为嗜水气单胞菌菌株(Aeromonashydrophila)5株,气单胞菌菌株(Aeromonas)4株,肠杆菌菌株(Enterobacterstrain)7株,变形杆菌菌株(Proteus)2株,葡萄球菌菌株(staphylococcus)6株,大肠杆菌菌株(E.colistrain)4株,梭状芽胞杆菌菌株(Clostridium)2株,哈夫尼肺泡菌菌株(Hafni’salveolarbacterium)1株,泛菌属(Pantoea)2株,肌炎沙雷菌(Serratiaserrata)2株,溶酪大球菌菌株(typhimurium)1株,柠檬酸杆菌菌株(Citrobacter)2株,假单胞菌菌株(Pseudomonas)2株,克雷伯氏菌属(Klebsiellasp.)2株,不动杆菌菌株(Acinetobacter)1株。各取样点分离出的菌落情况如表1。值得一提的是,分割台面和案板上分离出的菌株种类和数量相对于其他取样点的培养基更为丰富。传统分离方法表明,气单胞菌菌株和肠杆菌菌株的数量在羊肉加工过程中的菌群结构中占据优势地位。由表1的分离结果根据所选取的同源性最高的菌株系列进行编号,构建系统进化树如下图2。

图2 基于16S rRNA基因构建的43株菌株的进化树Fig.2 The phylogenetic tree of 43 spoilagebacteria based on 16S rRNA

表1 不同培养基分离所得菌株的鉴定结果Table 1 The identification results of strainsisolated from different sampling points
2.3 各菌株成膜能力比较
将培养完成的各菌株生物膜按1.2.4所述的方法进行成膜能力计算,其结果如图3。图3的每一组柱形图的宽度表示每一类菌株的数量,柱形图越宽代表分离得到的菌株数量越多,并在菌群结构中占据优势;柱形图的高度则表明分离出的每株菌的成膜能力(B值),B值越大成膜能力越强。由图3可知,在各类菌株的成膜能力数值中,C类即肠杆菌菌株不仅成膜能力远远高于其他菌株,数量上也有一定的优势,此外嗜水气单胞菌、葡萄球菌和大肠杆菌的成膜能力和菌株数量也优于剩余的腐败菌。从这几种优势腐败菌选取的部分结晶紫染色结果来看,肠杆菌(C)和大肠杆菌(F)的染色结果相对于气单胞菌和葡萄球菌颜色更深,其整体的成膜能力数值也高于其他几类菌。

图3 从羊肉加工过程中分离的菌株Fig.3 Strains isolated from the process of mutton dispensing注:A:嗜水气单胞菌(A1-A5);B:气单胞菌(B1-B4);C:肠杆菌(C1-C7);D:变形杆菌(D1-D2);E:葡萄球菌(E1-E6);F:大肠杆菌(F1-F4);G:梭状芽胞杆菌(G1-G2);H:哈夫尼肺泡菌(H1);K:泛菌属(K1-K2);L:肌炎沙雷菌(L1-L2);M:溶酪大球菌(M1);N:柠檬酸杆菌(N1-N2);P:假单胞菌(P1-P2);Q:克雷伯菌属(Q1-Q2);R:不动杆菌(R1)。
2.4 高通量测序法测定羊肉加工过程中的菌群结构
2.4.1 测序数据统计与OTU分析 根据Illumina Miseq高通量测序得到羊肉宰杀分割过程中各取样点总菌数的序列数据,然后经Trimmomatic和FLASH软件进一步优化得到了15个样本共1347802条有效序列,主要集中在200~260 bp的区间长度内。当样品量不断增大至约40000时,OUT曲线(图4A)和Shannon曲线趋于平缓(图4B),样品中OTU数目不再显著增加,说明数据量足够,可以反映样本中大多数的微生物物种[26]。对羊胴体表面的三个样品A、B、C测得的OTU进行数据重叠得到羊胴体表面的Venn 图,然后再对所有取样点进行数据重叠得到花瓣图,结果如图5。从图5的韦恩图可知,三个胴体表面样品之间有相互重叠的部分数据,共有的微生物共有432种,也就是说三个样品之间有部分相同的菌群结构。同样地,由花瓣图可知,所有的取样点之间也具有相同的菌群结构。
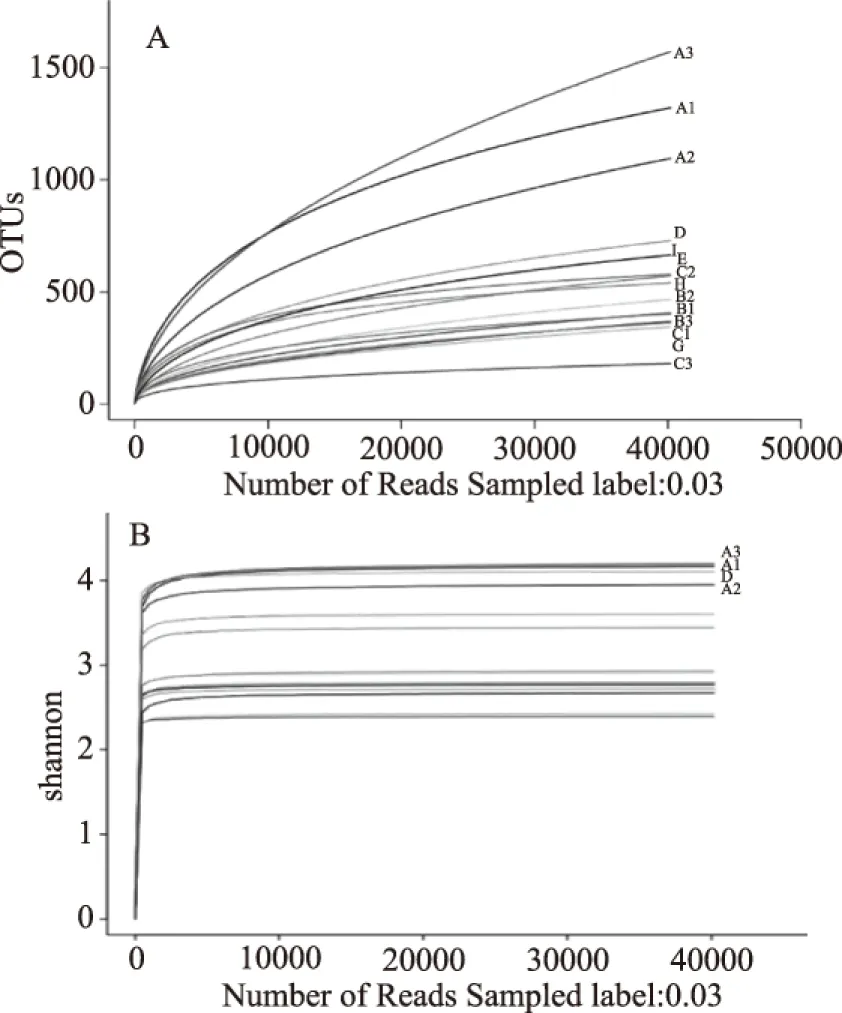
图4 稀释曲线(A)和Shannon-Wiener曲线(B)分析Fig.4 Rarefaction curves(A)and Shannon-Wiener curves(B)
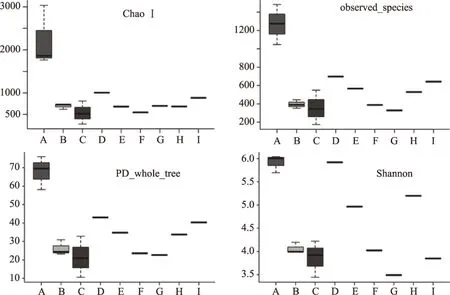
图6 Alpha多样性指数的箱图Fig.6 The box plots of Alpha diversity indexes
2.4.2 Alpha多样性指数分析 Alpha多样性主要反映待测定样品的生物多样性和物种的丰度、均匀度及其测序深度[27]。Alpha多样性包括四个指标,其中Chao I指数与菌群的丰富度呈正相关,observed_species和PD_whole_tree指数则反映了测序深度,而Shannon指数则关系到群落的多样性,其数值越大菌群的多样性越高[24],多样性结果如图6。由Chao I指数可知,开膛前的A样品Chao I指数最高,也就是其菌群丰富度最高,相对来说,经过开膛和冲洗的B、C样品丰富度大大下降,而剩余的其他样品丰富度基本跨度不大;相对来说observed_species和PD_whole_tree指数所反映的测序深度与取样数量密切相关,因此与实际样品情况存在误差;由Shannon指数可看出群落多样性的变化,由图可知,样品A的Shannon指数最高,而经过开膛和冲洗的样品B、C Shannon指数降低,说明开膛和清洗这一步骤使得羊胴体表面菌落多样性降低。同样地,对于羊胴体的后续加工过程而言,其冲洗过的台面D、分割台面E、内脏分离之后的清洗水H 的Shannon指数也较高,也就是说,羊肉分割的过程中很有可能因为其工具及操作方法而使得羊肉表面的菌落多样性增加。
2.4.3 物种组成和聚类分析 采用RDP Classifier算法对各样本序列比对分析,并在科和属的水平上表达了各取样点菌群的物种信息,其群落组成如图7。由图7可知,开膛前的羊胴体表面菌群多样性远远高于开膛后和冲洗后的羊胴体表面,由图7-a可知在科水平上A、B、C表面菌群主要有莫拉菌科(Moraxellaceae)、气单胞菌科(Aeromonadaceae)、嗜冷杆菌科(Flavobacteriaceae)、希瓦氏菌科(Shewanellaceae)、普雷沃氏菌科(Prevotellaceae)、假单胞菌科(Pseudomonadaceae)、李斯特菌科(Listeriaceae)、葡萄球菌科(Staphylococcaceae)、肠杆菌科(Enterobacteriaceae)、芽孢杆菌科(Bacillaceae)、理研菌科(Rikenellaceae)等。而由图7-b可知在属水平上A、B、C表面菌群主要有鲍曼不动杆菌属(Acinetobacter)、嗜冷杆菌属(Psychrobacter)、气单胞菌属(Aeromonas)、希瓦氏菌属(Shewanella)、金黄杆菌属(Chryseobacterium)、类香味菌属(Myroides)、黄杆菌属(Flavobacterium)、假单胞菌属(Pseudomonas)、死环丝菌属(Brochothrix)、巨型球菌属(Macrococcus)、普雷沃氏菌属(Prevotella)、水栖菌属(Enhydrobacter)等。相对于羊胴体表面,台面和案板上的菌群多样性更高,其主要菌种基本上和胴体表面一致,但比例略有不同。此外在科水平上增加了韦荣氏球菌科(Veillonellaceae)、瘤胃菌科(Ruminococcaceae)、柄杆菌科(Caulobacteraceae)、球菌科(Enterococcaceae)、芽单胞菌科(Xanthomonadaceae)、肉杆菌科(Carnobacteriaceae)等。在属水平上则增加了罗思氏菌属(Rothia)、地杆菌属(Pedobacter)、假节杆菌属(Pseudarthrobacter)、克雷伯氏菌属(Klebsiella)、短波单胞菌属(Brevundimonas)、浮游球衣菌属(Sphaerotilus)等。

图7-a 基于科水平的物种组成分析柱状图Fig.7-a Histogram of species compositionanalysis based on family level

图7-b 基于属水平物种组成分析柱状图Fig.7-b Histogram of species compositionanalysis based on genera level
根据科和属水平上的菌群物种组成及相对丰度,采用UPGMA方法对各取样点进行聚类分析,结果如图8。由图8-a、8-b可知,开膛前的三个取样点在同一分支,开膛后、冲洗后及所取水样的几个分支紧密相关,说明这几个取样点的菌群组成和相对丰度类似,而冲洗台面和案板两个取样点菌群组成和其他的取样点相比差别较大,自成一个分支。
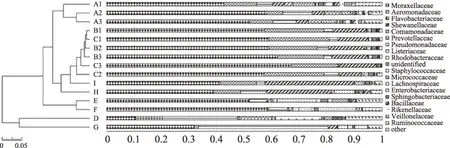
图8-a 基于科水平的样本聚类柱状图Fig.8-a Sample clustering histogram based on family level
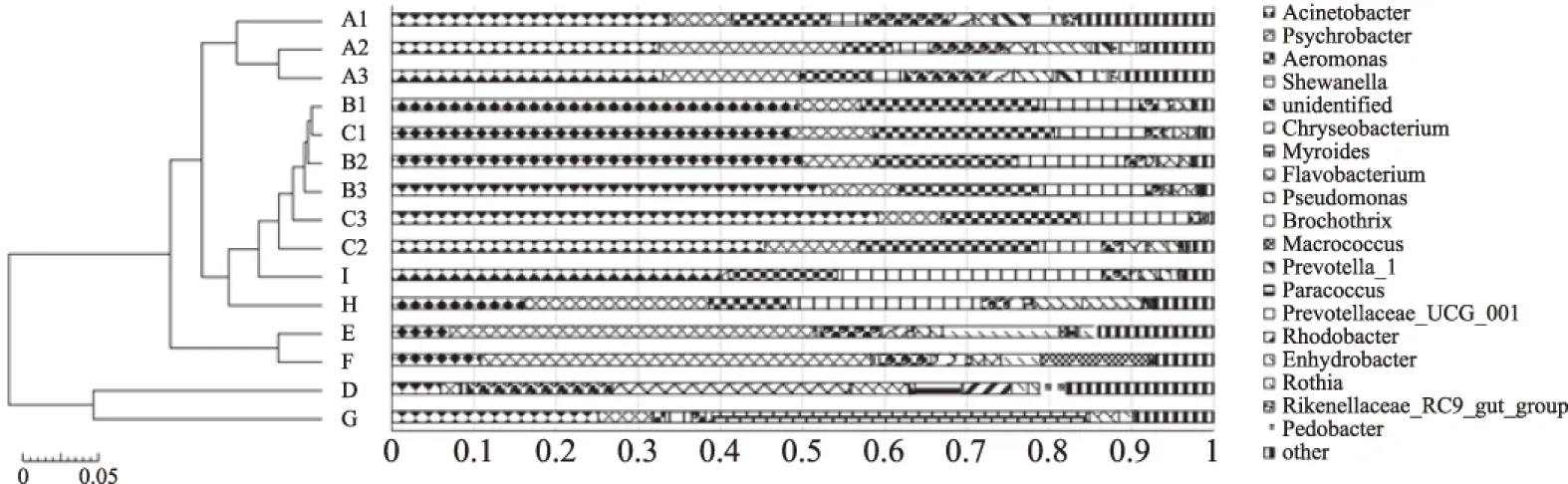
图8-b 基于属水平的样本聚类柱状图Fig.8-b Sample clustering histogram based on genera level
2.4.4 不同取样点菌群结构的主成分分析(principal component analysis,PCA) PCA是应用方差分解,对多维数据进行降解进而将差异反映在二维坐标图上,显示出更为简单明了的规律。如果样本的菌群结构组成相似,那么在PCA图上的距离也就越近[28-31]。基于OTU水平的PCA分析结果如图9。如图9所示,横纵坐标分别为PC1与PC2,其贡献率分别为38.74%和20.6%,说明这两个主成分是样品菌群结构组成差异的主要因子。其中在PC1和PC2方向上,A、B、C三个羊胴体表面的取样点中,B和C较为接近,表明在此主成分水平上开膛后和冲洗后的羊胴体表面菌群特征较为相近,但开膛后B和清洗后C样品与开膛前的样品A相比存在较大的差异,说明经开膛这一步骤后,菌群结构发生了较大的变化。冲分割台面E、F和案板G的菌群特征较接近,与洗台面D的菌群差异性较大;两种水样的菌群结构也存在差异性。
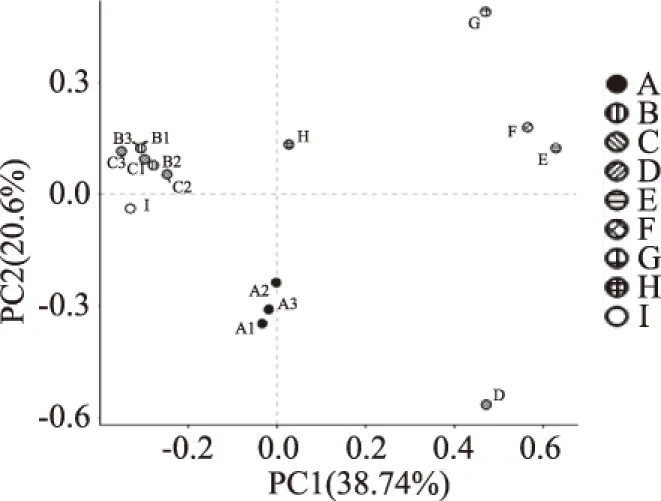
图9 不同取样点PCA Fig.9 PCA of different sampling points
3 结论
本研究采用传统分离方法和Illumina Miseq高通量测序对羊肉加工过程中的各取样点的菌群结构进行分析。Illumina Miseq高通量测序则研究了羊肉整个加工过程中取样点的菌群结构,全面地反映了羊肉加工过程的菌群结构变化。研究发现,羊胴体表面和各加工台面的菌群结构有较大的差异,相对于羊胴体,后续加工的台面等取样点菌群的多样性波动较大,但优势菌群的种类并没有多大变化,只是所占比例略有不同。总的来说,羊肉加工过程中的常见的优势菌群在科水平上主要有莫拉菌科(Moraxellaceae)、气单胞菌科(Aeromonadaceae)、嗜冷杆菌科(Flavobacteriaceae)、希瓦氏菌科(Shewanellaceae)、假单胞菌科(Pseudomonadaceae)、李斯特菌科(Listeriaceae)、葡萄球菌科(Staphylococcaceae)、肠杆菌科(Enterobacteriaceae)、芽孢杆菌科(Bacillaceae)等。在属水平上则包括鲍曼不动杆菌属(Acinetobacter)、嗜冷杆菌属(Psychrobacter)、气单胞菌属(Aeromonas)、希瓦氏菌属(Shewanella)、金黄杆菌属(Chryseobacterium)、假单胞菌属(Pseudomonas)、死环丝菌属(Brochothrix)、罗思氏菌属(Rothia)、地杆菌属(Pedobacter)、假节杆菌属(Pseudarthrobacter)、克雷伯氏菌属(Klebsiella)。
传统培养分离出了可培养菌落,并对筛选出的43株菌株成膜能力进行计算,结果表明嗜水气单胞菌菌株和肠杆菌菌株数量上有一定优势,经结晶紫染色并计算其成膜能力可知肠杆菌菌株的成膜能力最强。生物膜的存在使得细菌对环境的耐受性、抗生素的耐药性大大提高,因此若能抑制生物膜的生长,就能在很大程度上加强杀菌效果,防止细菌性感染等一系列疾病。根据本研究的结果,拟选取数量较多且成膜能力强的菌株进行抑制生物膜的分子机制的进一步研究。

