基于线粒体基因组的鲹科鱼类进化分析
王海山 ,蒋欢 ,陈治 ,杨超杰 ,叶乐
(1.海南热带海洋学院水产与生命学院,海南 三亚 572022;2.宁波大学海洋学院,浙江 宁波 315211)
鲹科(Carangidae)鱼类隶属硬骨鱼纲(Osteichthyes)、辐鳍鱼亚纲(Actinopteri)、鲈形目(Perciformes),下有4 个亚科:鲹亚科(Caranginae)、鰤亚科(Seriolinae)、鲳鲹亚科(Trachinotinae)和鰆鲹亚科(Chorineminae)。该科鱼类广泛分布于印度洋、太平洋、大西洋的热带和亚热带水域。现记录的鲹科鱼类共34 属约147 种,其中鲹亚科的种类最丰富,共20 属,占全科的50%以上[1]。中国海域有鲹科鱼类64 种,分布于东海、黄海以及南海,全部鲹科鱼类在南海均有分布,东海分布约25 种。每到春夏季,鱼群会随暖流从低纬度区洄游到高纬度出生地产卵,也有少数几种能够进入淡水流域。大部分鲹科鱼类喜欢集群游动于沿岸水域或珊瑚礁海域[2],其中体型中等的种类游速较快。根据鲹科鱼类体形特点和游动时的运动学特征,常把其游动模式运用于水下推进器的研究[3]。鲹科鱼类多是重要的海洋经济鱼类,如竹筴鱼(Trachurus trachurus)、细鳞圆鲹(Decapterus macarellus)和长体圆鲹(Decapterus macrosoma)等是南海外海渔业的重要捕捞对象[4],而五条鰤(Seriola quinqueradiata)、黄条鰤(Seriola lalandi)和卵形鲳鲹(Trachinotus ovatus)等高级食用海水鱼,也是目前我国海南、广东和广西深水抗风浪网箱养殖重要种类。为了促进我国海水养殖业的发展,不同学者对多种鲹科鱼类人工繁殖、网箱养殖技术、营养生理等展开了研究,并取得了突破性的进展[5-7]。
关于鱼类分类,目前也做了许多工作。朱元鼎和郑文莲(1958)[8]描述了在中国南海海域发现的钝鳍叶鲹(Caranx pectoralis)和海南鰆鲹(Chorinemus hainanensis)两个新种的外部形态;Suzuki(1962)[9]比较了日本海域不同鲹科鱼类的骨骼系统,探讨了鲹科鱼类的系统问题;郑文莲(1981)[10,11]采用传统形态测量和解剖学方法,从骨骼、耳石、鳞片、嗅觉器官、消化器官等系统研究了中国海域鲹科鱼类分类、分布和形态,为推进后续相关鱼类科、亚科、属、种的鉴别提供了重要参考资料;欧利国等(2020)[12,13]等基于地标点法鉴定了南海海域4 种鲹科鱼类的108 尾样品,同时依据耳石形态特征聚类分析了南海东沙群岛海域7 种鲹科鱼类,并建立了系统分类检索表,为南海鲹科鱼种识别以及分类提供了参考资料。总体而言,从18 世纪60 年代开始,关于鲹科鱼类分类是以新种描述、形态分类、生物地理分类为主。
随着生物信息统计软件以及线粒体DNA(mitochondrial DNA,mtDNA)特征被普遍应用于各种生物的系统发育分析,分子生物学在鲹科鱼类的研究中逐步开展。牛素芳等(2018)[14]分析了203 尾蓝圆鲹的线粒体DNA 细胞色素b 序列的遗传多样性和遗传结构,研究了南海北部蓝圆鲹的群体遗传变异特征;许则滩等(2015)[15]探讨了东海舟山群岛普陀海域18 尾鲹科样本的线粒体COI 序列,通过比较种间和种内差异研究了本海域鲹科鱼类的分类地位及分子系统进化关系,得出了鲹科鱼类同一属内种间遗传距离明显大于种内遗传距离的结论,并认为普陀海域的18 尾鲹分别属于7 属8 种;朱世华等(2007)[16-18]结合数据库已测定完成的3 种鲹科鱼类序列,依据线粒体DNA 控制区、细胞色素b 序列、16S rRNA 序列生成矩阵和构建的分子系统树,分析了中国海域的8 属9 种鲹科鱼类,结果支持鲹科四个亚科的分类阶元,也提出狮鼻鲳鲹(Trachinotus blochii)为卵形鲳鲹的误鉴,以及将丽叶鲹(Caranx kalla)的中文名改为“丽副叶鲹”的观点。该结论与国外鱼类分类学者David 等(2002)[19]基于形态特征分析和分子数据构建的部分鲹科鱼类的分子发育树相似。
鱼类线粒体基因组不仅是研究真核细胞核酸与蛋白质合成的模型系统,也是研究DNA 结构与复制的良好模型。线粒体基因组为母性遗传,进化主要是以碱基替换为主,进化速度较核DNA 快5~10 倍[20],被广泛应用于鱼类群体遗传结构分析和类群识别。目前,尽管16S rRNA、D-loop 区和COI序列等片段分别被运用于鲑科、鲤科、鲷科和鳀科等鱼类遗传多样性的研究中,但是,多认为线粒体上不同的区域进化速度不同,用序列更长的线粒体DNA 序列来构建统发育树可以减少信息分布的不均一性和序列长度太短而降低系统进化树的置信度带来的影响,因此其结果更精确、可靠。
本文从GenBank 数据库中下载已测定完成的33 种鲹科鱼类的线粒体基因组序列,通过Clustal X和MEGA X 软件进行DNA 序列比对和核酸多态性、遗传距离等分析,结合邻接法(Neighbor-Joining,NJ)、最小进化法(Minimum Evolution,ME)系统发育树,探讨鲹科种属间的序列差异及其亲缘关系,为鲹科的分类提供新的视角。
1 材料与方法
1.1 全基因组序列信息
通过检索NCBI 的GenBank 数据库获取33 种鲹科鱼类的线粒体基因组序列,并保存为FASTA格式。其种名、分类地位及相应序列号等相关信息见表1。
1.2 数据分析
将下载的基因组序列用Clustal X 软件进行序列比对,再辅以人工校对;运用MEGA X 软件统计序列的全长、碱基含量、变异位点数(比例)及简约位点(Parsimony informative sites)等信息(表2)。多数学者认为Kimura 双参数模型(Kimura2-Parameter,K-2-P)是评估低遗传变异序列的最佳核酸替代模型,本研究利用MEGA X 软件中Kimura 双参数法计算种属间的遗传距离;利用DnaSP 5.0 软件统计33 种鲹科鱼类线粒体基因组序列的碱基多态性;运用MEGA 软件中的邻接法及最小进化法构建33 种鲹科鱼类的系统进化树,采用Bootstrap 法检验各分支的支持率,以自展值为1 000 次做重复抽样检验,其余参数为软件默认值。
2 结果与分析
2.1 核苷酸组成
序列分析显示,鲹科鱼类线粒体基因组序列全长在16 530~16 609 bp 之间,与大多数硬骨鱼类线粒体序列长度相似[21],其中鰤属的高体鰤(Seriola dumerili)序列最短,黄条鰤的序列最长。核苷酸多样性指数为37.3%,检测到序列总的变异位点共7072个,占42.0%,简约信息位点共6 297 个,变异位点数大于简约信息位点数。转换/ 颠换比值(R)1613/1026=1.6<2.0,说明基因序列的突变达到饱和;同属的个体碱基差异为1.68%~12.89%,平均差异为6.75%,表明鲹科鱼类种间的碱基组成差异很小,呈现一定的核酸组成稳定性。线粒体全基因组碱基平均含量由高到低依次为C(29.5%)>A(27.8%)>T(26.0%)>G(16.7%),其中A+T 的含量(53.8%)大于G+C 含量(46.2%),表现出AT 含量相对丰富和GC含量相对缺乏的现象,符合硬骨鱼类线粒体碱基组成偏好于A 和T 的特点[22-24]。本文所选全部物种碱基均有不同程度的AT 偏向(表1)。
DnaSP 5.0 计算位点多态性,发现了715 个单一变异位点、5 943 个简约信息位点,其中发生种碱基突变的单一变异位点676 个、简约信息位点3 120个,发生三种碱基突变的单一变异位点36 个、简约信息位点1 363 个,发生四种碱基突变的单一变异位点3 个,简约信息位点1 460 个。DnaSP 5.0 计算表明,33 种鲹科鱼类线粒体基因组核酸多样性(Pi)为0.15549,以100 bp 为单位在总长约16 800 bp 的线粒体基因组上步移,计算获得核酸多样性曲线(图1),并结合鲹科鱼类线粒体基因结构图分析可以发现,33 种鲹科鱼类线粒体基因组在D-Loop 区域具有最高的核酸多样性(第15 861~16 072 位碱基核酸多样性指数Pi 为0.48081),其次为ND6 和ND5 区域(核酸多样性指数Pi 分别为0.26080 和0.26313),ND2 和ND1 区域也具有较高的核酸多样性指数(0.23428 和0.23282)。16S rRNA、tRNA 区域核酸多样性较低(最低为0.005)。即在33 种鲹科鱼类线粒体基因组上碱基突变频率最高的区域出现在线粒体复制D-Loop 控制区和NADH 基因的亚基位置。16S、12S rRNA 和tRNA 位置碱基突变频率较低,基因较为保守。

图1 33 种鱼线粒体基因组核酸多样性曲线Fig.1 Mitochondrial DNA diversity of 33 fish species
2.2 遗传距离矩阵
遗传距离通常反映遗传多样性的高低,物种内个体之间的遗传距离大,表明个体之间的亲缘关系远,物种间遗传多样性较高;相反的,物种内个体之间的遗传距离小,表明个体之间的亲缘关系近,遗传多样性较低。K-2-P 计算的遗传距离显示,鲹科鱼类种间遗传距离平均值为0.183;种间遗传最大值为卵形鲳鲹与高体若鲹(Carangoides equula)的0.250,最小值为黑纹小条鰤(Seriolina nigrofasciata)与白舌尾甲鲹(Uraspis helvola)的0.002。其中卵形鲳鲹与鲳鲹属外物种的遗传距离最大,平均值为0.229;北美鲳鲹(Trachinotus carolinus)与鲳鲹属外物种的平均遗传距离为0.228;布氏鲳鲹(Trachinotus blochii)与鲳鲹属外物种的平均遗传距离为0.226;泰勃圆鲹(Decapterus tabl)与其他鲹科鱼类平均遗传距离最小为0.170;细鳞圆鲹与其他鲹科鱼类平均遗传距离为0.172;丝鲹(Alectis ciliaris)与其他鲹科鱼类平均遗传距离为0.175。值得注意的是,高体若鲹与若鲹属其他种的遗传距离并不是最小的,其与甲若鲹(Carangoides armatus)、马拉巴若鲹(Carangoides malabaricus)、竹筴鱼、蓝圆鲹、日本竹筴鱼(Trachurus japonicus)和泰勃圆鲹的遗传距离分别为0.184、0.179、0.156、0.161、0.159 和0.158;与细鳞圆鲹、珍鲹(Caranx ignobilis)、乌鲹(Parastromateus niger)的遗传距离均小于与马拉巴若鲹的遗传距离(表2)。

表2 33 种鱼线粒体基因序列的种间遗传距离Tab.2 Genetic distance of 33 fish species based on mitochondrial genome
综上,鲳鲹属在进化上与其他鲹科鱼类分化较大,圆鲹属与其他鲹科鱼类遗传距离更近,在线粒体基因组水平上高体若鲹与圆鲹属鱼类亲缘关系更近,不符合传统分类地位。
2.3 系统发育树分析
早期对鲹科鱼类的分类主要以外部形态为标准,由于地区、发育阶段和环境的影响,相关研究中的特征描述或多或少存在差异,因而对鲹科鱼类各属种的分类存在争议。本文分别采用NJ 法及ME法构建了基于线粒体基因组核苷酸序列的33 种鲹科鱼类系统进化树。Bootstrap 的参数值大于70%时,其结果符合真实的系统发育史的可靠性大于95%;如果参数值小于50%,则构建的系统发育树无法反映实际情况。本实验所建的NJ、ME 系统发育树的绝大部分支的Bootstrap 都是100%,表明本研究可靠地反映了鲹科鱼类的系统发育史(图2、图3)。
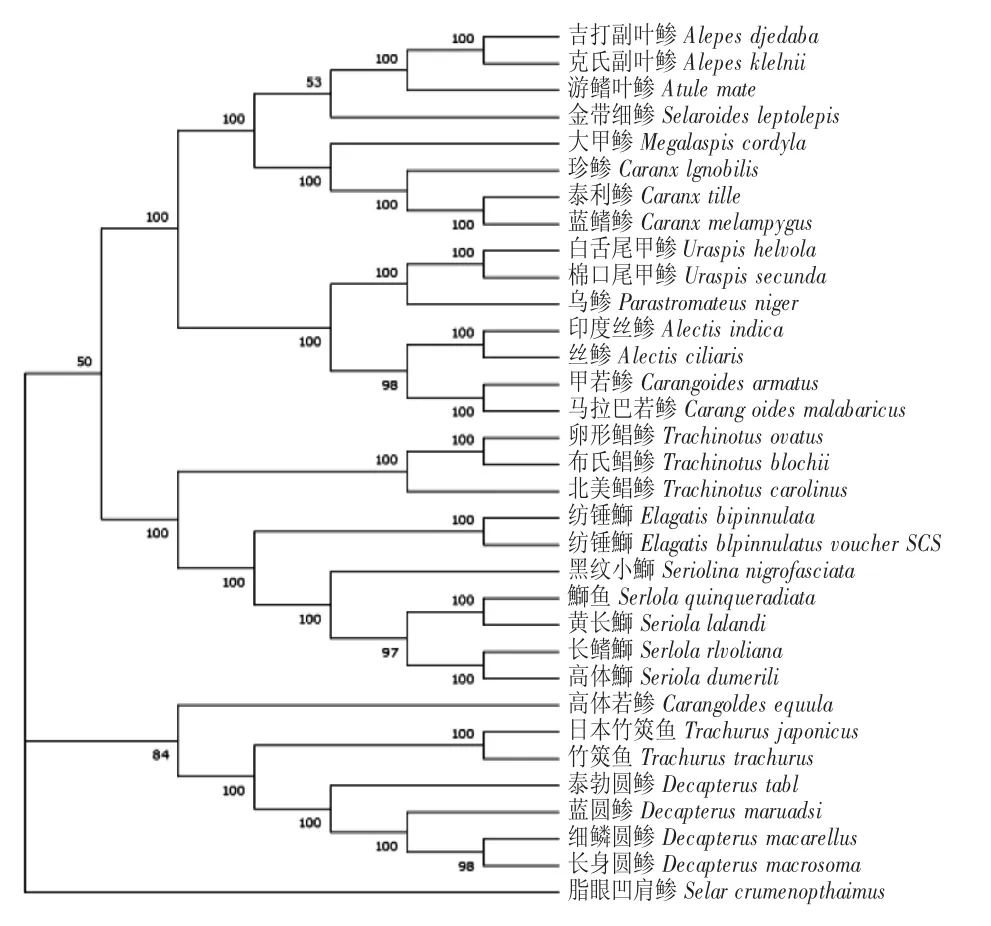
图2 33 种鱼基于线粒体序列的NJ 系统发育树Fig.2 Phylogenetic trees of 33 fish species based on the complete mitochondrial genome by NJ analysis

图3 基于线粒体序列的ME 系统发育树Fig.3 Phylogenetic trees based on the complete mitochondrial genome by ME analysis
NJ 树显示:鲹科鱼类分为3 大支系,从树的基部第1 支系可知鲹亚科的脂眼凹肩鲹(Selar crumenophthalmus)是较早单独分离出来的一支;第2支系是竹筴鱼属与圆鲹属形成姐妹群,再与高体若鲹聚为一支(置信度为50%);第3 支系由鰤亚科、鲳鲹亚科以及鲹亚科共同组成,较早分为2 个进化分支,一个分支显示鲳鲹亚科的卵形鲳鲹、布氏鲳鲹以及北美鲳鲹所组成一个进化支,另一分支包含纺锤属和鰤属,再与鲳鲹属聚集形成姐妹群。由此可知,鲳鲹亚科与鰤亚科有相当密切的亲缘关系;树的顶部为鲹亚科的鱼类,包含副叶鲹属、叶鲹属、细鲹属、大甲鲹属以及鲹属,每个属都单独成为一个支系;泰利鲹(Caranxtille)和蓝鳍鲹(Caranxmelampygus)、棉口尾甲鲹(Uraspis secunda)和白舌尾甲鲹、吉打副叶鲹(Alepes djedaba)和克氏副叶鲹(Alepes kleinii)、竹筴鱼和日本竹筴鱼、长鳍鰤(Seriola rivoliana)和高体鰤、卵形鲳鲹和布氏鲳鲹这六组物种遗传距离相同,进化速率相同;这种聚类关系与孙立元(2014)[25]的结果一致。
ME 树显示,鰤亚科进化距离最长,位于系群的基部,是鲹科的原始种群,其次是鲳鲹亚科;鲹亚科又分化出3 个分支,聚集到树枝末端——物种组成与其在NJ 树上所处的分支基本相同,只在排列顺序上有差异,表明两种方法所得到的结果一致。
3 讨论
鱼类线粒体基因组具有分子小、进化速度快、母系遗传和信息相对稳定等优势,适于从序列信息、基因组成及排列方式等角度来分析分子系统学及种群遗传学[20]。本研究中,鲹科鱼类线粒体基因组核苷酸序列的相关数据呈现AT 含量相对丰富和GC 含量相对缺乏的现象,符合硬骨鱼类线粒体碱基组成偏好于A 和T 的特点。以NJ 法和ME 法构建的鲹科3 亚科15 属33 种鱼类基因序列的系统进化树结果基本一致,均表明鲳鲹亚科与鰤亚科为姐妹群,这与朱世华等利用线粒体DNA 控制区、细胞色素b 序列、16S rRNA 序列分析结果一致。鰤亚科是3 个亚科中的最原始种群[10,11]。
早期的系统发育分析主要通过化石和现存物种的生理、形态特征来构建系统发育关系。荷兰自然学家Coenraad 和Hermann(1989)[26]首先于1844年根据从日本海域采集的高体若鲹标本进行了体长及背鳍、臀鳍、胸鳍和腹鳍数等特征的描述。1844—1907 年间,高体若鲹先后被更名为Carangoides acutus、Kaiwarinusequula、Carangoidesequula、Carangoides dasson 等。高体若鲹最开始被归为鲹属下的若鲹亚属,后因分类系统修改鲹属上升为鲹亚科,其下亚属也相应上升为属,高体若鲹归为鲹亚科的若鲹属。目前高体若鲹为鲹亚科、若鲹属的分类地位被广为接受。较多文献中竹筴鱼、日本竹筴鱼学名、中文名的使用也较为混乱。从物种的命名以及相关文献的记录都显示,鲹科鱼类中的某些种类分类地位存在争议。本研究构建的两种系统发育树均显示:若鲹属的马拉巴若鲹、甲若鲹与丝鲹属的丝鲹以及印度丝鲹(Alectis indica)形成姐妹群,而若鲹属的高体若鲹并未与甲若鲹、马拉巴若鲹聚为一支,而是与圆鲹属、竹筴鱼属的种类聚为1 支(置信度84%~99%),表明高体若鲹与圆鲹属、竹筴鱼属之间的亲缘关系较近,与若鲹属间的亲缘关系较远。本研究结果与史宝等(2019)[27]在探讨黄条鰤线粒体基因组系统进化分析中所构建的系统进化树结果基本一致,但后者并未讨论高体若鲹处于的异常分支的结果。
以往分析鲹亚科时主要基于体形、肩带下角有无深凹、上下颌有无牙齿、牙齿形状、牙列数、第二背鳍有无延长鳍条等形态学特征进行分类。如欧国利和刘必林(2020)[12]利用地标点法对鲹科鱼类耳石形态进行分类识别,将高体若鲹与短吻丝鲹、竹筴鱼以及脂眼凹肩鲹区分开来;从外部形态学分析,高体若鲹与马拉巴若鲹、甲若鲹都具有体形为卵圆形、高而侧扁的形态特征,但却在一定程度上受性别、发育阶段以及适应环境的影响。如幼鱼时,甲若鲹体侧与高体若鲹同样具六条暗色横带,但随着个体成长,甲若鲹的暗色横带逐渐消失。因此,形态学鉴定在一定程度上受性别、发育阶段以及适应环境的影响,仅依靠形态学对物种分类鉴定存在着较多问题。

