光控诱导重组系统的开发与应用
谢甜,王梅,高瑞钰,苗艳尼,张燚铭,蒋婧
光控诱导重组系统的开发与应用
谢甜,王梅,高瑞钰,苗艳尼,张燚铭,蒋婧
中国科学院分子细胞科学卓越创新中心(生物化学与细胞生物学研究所),基因组标签计划研发中心,上海 200031
位点特异性重组系统由重组酶和特异性识别位点两部分组成,是一种强大的基因操作工具,被广泛运用于生命科学研究。已开发的诱导型重组系统以时空方式精准调控细胞和动物的基因表达,被用于基因功能研究、细胞谱系示踪和疾病治疗等领域。根据诱导重组酶时空表达方式的不同,诱导型重组系统可分为化学诱导和光控诱导两种方式。光控诱导重组系统是利用光作为诱导剂,根据光控方式和对象的不同,可进一步分为光笼和光遗传学两类。光笼诱导重组系统是利用光敏基团来控制化学诱导剂或重组酶,光诱导前它们的活性被光敏基团抑制;在特定光照射后,它们的活性被恢复,进而实现光控诱导基因重组。光遗传学诱导重组系统是通过光遗传学开关介导分割型重组酶的重新激活来诱导基因重组。其中光遗传学开关由一系列基因编码的光敏蛋白组成,包括隐花色素、VIVID蛋白、光敏色素等。这些类型丰富的光控诱导重组系统为从高时空分辨率的维度解析基因的表达和功能提供了更多的工具,以满足日益复杂的生命科学研究需求。本文主要对不同类型光控诱导重组系统的开发原理及应用进行综述,比较其优缺点,最后对未来开发更多光控重组系统进行展望, 旨在为系统优化升级提供理论基础和指导。
光控诱导重组系统;光笼;光遗传学开关;位点特异性重组酶;时空调控
位点特异性重组酶(site-specific recombinases, SSRs)是一类具有催化活性,能够特异性识别DNA序列,实现位点特异性重组的基因操作工具。其中酪氨酸重组酶家族成员如Cre[1~3]、Flp[4]和Dre[5,6],由于其重组特异性和高效性,在基因工程中发挥着关键作用。这些重组酶能够在没有辅助蛋白的帮助下,实现基因组DNA的切除(excision)、倒位(inversion)和易位(translocation)[7]。因此,SSRs介导的基因重组系统是生物医学研究不可或缺的工具。
SSRs家族成员Cre重组酶因其卓越的重组效率成为小鼠遗传学中最常用的工具。Cre/重组系统来源于P1噬菌体[8],包含Cre重组酶和34 bp的位点两种组分,由Cre重组酶催化位点之间发生DNA重组[9]。Cre重组酶的表达可受启动子调控,因此操纵不同类型启动子即可调控Cre重组酶的时空表达。在过去的几十年中,已开发了一系列诱导型重组系统用于复杂的科学研究,主要包括化学诱导和光控诱导两种方式。
化学诱导重组系统(chemical-inducible recombination system)是指通过小分子化合物来介导SSRs的时空表达,已被广泛应用于在特定发育阶段和特定组织细胞中对特定基因进行遗传操作。以化学诱导Cre重组酶为例,主要包含有CreER[10]、基于Tet-on的Cre[11,12]、Di-Cre[13]和DD-Cre[14]等方式。CreER的使用最为广泛,是将Cre与雌激素受体(estrogen receptor, ER)的配体结合域(ligand binding domain, LBD)相融合。CreER通常定位于细胞质中,在他莫昔芬(tamoxifen, Tam)诱导下,融合的Cre蛋白才会进入细胞核,识别位点并发生基因重组[15,16]。基于Tet-on的Cre则是通过强力霉素(doxycycline, Dox)诱导型启动子,即TRE启动子驱动Cre的表达。Tet-on系统的另一个元件rtTA由普遍存在或组织特异性的启动子驱动表达,强力霉素可以结合并激活rtTA,使得激活的rtTA结合到TRE启动子上诱导Cre的表达[17]。另外,Di-Cre[13]和DD-Cre[14]分别是通过小分子化合物介导重组酶二聚化或稳定化从而实现Cre的时间特异性激活。其中Di-Cre是利用雷帕霉素(rapamycin, Rap)诱导FKBP12和FRB的二聚化来重新组合分割型Cre重组酶。分割型重组酶是指将重组酶拆分成两个小蛋白,当它们重新拼接组装后可恢复重组酶的功能进而催化重组反应[18,19]。然而,化学诱导重组系统存在细胞毒性、系统泄漏和潜在脱靶等缺点。此外,由于小分子化合物可自由扩散,缺乏组织特异性,多用于时间轴上的调控;并且小分子化合物扩散时间长且难以立即去除,导致时间调控不够精准[20,21]。
为了精准实现时间和空间的双调控,相比小分子化合物,光是一种理想的诱导剂,可在不同环境中自然调节许多细胞过程[22~24]。基于紫外(UV light)、蓝光(blue light)和远红光(far-red light)照射的光控诱
导重组系统(light-controlled inducible recombination system)已被开发用于时空特异地精准遗传操作。根据光控方式及对象的不同,光控诱导重组系统分为光笼和光遗传学两种类型。光笼诱导重组系统(photocaged inducible recombination system)是利用光敏基团来控制化学诱导剂或重组酶。而光遗传学诱导重组系统(optogenetic inducible recombination system)主要是利用光遗传学开关来介导分割型重组酶的重新激活。本综述将对这两大类光控诱导重组系统的开发原理及其应用进行总结比较。
1 光笼诱导重组系统
光笼(photocage)是一种可光控释放的光敏物质,其原理是通过物理或化学等方法将光敏基团安装到目标释放物的核心位置,使其处于非活性状态。在特定光照射下,目标释放物被释放出来,恢复生物天然活性[25]。光笼已被用于多种生物学研究中,包括转录研究[26]、蛋白质–蛋白质相互作用[27]、细胞迁移与增殖[28]、蛋白质磷酸化[29]以及CreER系统的基因表达调控[30]等。光笼一般包含目标释放物(target substance)和光敏基团(photosensitive group)两个基本组成部分,目标释放物可以是药物、氨基酸、多肽、蛋白质和核酸等[31~33]。根据目标释放物类型的不同,光笼诱导重组系统的应用可以分为两类:光笼化学诱导剂和光笼重组酶。
1.1 光笼化学诱导剂
光笼化学诱导剂(photocaged chemical inducer)是将化学诱导重组系统的小分子化合物进行光笼修饰。例如CreER系统的小分子化合物包括4-羟基他莫昔芬氮丙啶(4-hydroxytamoxifen aziridine)、4-羟基环芬(4-hydroxycyclofen)、他莫昔芬(Tam)和4-羟基他莫昔芬(4-hydroxytamoxifen)等,都可以设计成为光笼(图1 A)。Link等[30]合成了释放4-羟基他莫昔芬氮丙啶的光笼,在紫外光刺激下能够触发培养细胞中CreER系统激活重组,但是该系统存在背景泄露。相比之下,4-羟基环芬具有更好的光稳定性且易于合成[34]。Sinha等[35]合成了释放4-羟基环芬的光笼,在水溶液中可以渗透入斑马鱼胚胎,通过紫外照射或双光子照射后可快速恢复活性进而激活CreER重组系统,实现高时空分辨率单细胞标记以及调控基因表达。当光笼化合物作用于复杂的生物体内,其活性受药代动力学、递送障碍和光与组织复杂相互作用等多种因素的影响。释放4-羟基环芬光笼的光控功能除了在体外细胞培养系统和斑马鱼体内模型中得到证实,还在复杂生物体内被证明。Lu等[36]的实验表明,释放4-羟基环芬的光笼在类器官与小鼠体内仍具有活性,可在紫外光下有效地诱导CreER介导DNA重组,实现单细胞水平的精准基因时空调控(表1)。
光笼进行光反应释放目标物的关键是光敏基团,上述两种光笼使用的光敏基团为4,5-二甲氧基- 2-硝基苯甲醇(4,5-dimethoxy-2-nitrobenzyl alcohol)。Inlay等[37]利用另一种光敏基团——邻硝基苄基(o- nitrobenzyl)合成了释放他莫昔芬的光笼。不同的是,首先它允许直接对他莫昔芬进行光笼修饰,而无需对药物分子进行任何结构修改或衍生,其次与几乎不溶于水的他莫昔芬相比,光笼修饰后大大提高了水溶性。值得注意的是,他莫昔芬必须转化为4-羟基他莫昔芬才能有效地结合CreER[43]。因此光笼他莫昔芬的使用仅限于能将他莫昔芬转化为4-羟基他莫昔芬的部分细胞。然而体外细胞的这种转化效率低,需要使用接近毒性浓度的他莫昔芬才能实现稳健的基因重组。因此,Faal等[38]直接将4-羟基他莫昔芬共价连接邻硝基苄基基团,合成释放4-羟基他莫昔芬的光笼,其活性比释放他莫昔芬的光笼高100倍以上,可以封闭的非活性形式有效进入细胞并在细胞内光依赖性释放,实现更精准地基因重组(表1)。
Tet-on表达调控系统的化学诱导剂为四环素类似物,最常用的是强力霉素(Dox)。释放强力霉素或其类似物的光笼已被证明可以用于在不同生物系统中通过紫外线或双光子局部照射诱导基因表达,包括体外培养的细胞、离体培养的小鼠海马体、发育中的小鼠胚胎和非洲爪蟾蝌蚪[44~46]。利用这种方式可以设计基于Tet-on表达Cre的光笼重组系统,但尚未有研究者开展相关研究验证。
Di-Cre的化学诱导剂是雷帕霉素(Rap),Rap诱导的FKBP12和FRB蛋白异二聚化是常用的条件性控制生物过程的开关之一[47]。Brown等[39]开发了一种释放Rap的光笼,称为光裂解雷帕霉素二聚体(light-cleavable rapamycin dimer, dRap),可在紫外照射后裂解释放Rap,诱导形成FKBP12-Rap-FRB三元复合物。而FKBP12和FRB分别融合Cre的N端(氨基酸19~59)和C端(氨基酸60~343),随着三元复合物的形成,Cre可重构恢复活性,进而诱导基因重组(图1 B)。dRap在紫外线照射之前不会诱导FKBP12和FRB的异二聚化,很好地防止了背景泄露(表1)。

图1 基于光笼分子的光控诱导重组系统
A:光笼化学诱导剂。在光控诱导CreER/系统中,经光笼修饰的小分子化合物活性被抑制。在特定波段的光照下,光控释放的小分子化合物恢复活性,使得CreER融合蛋白与HSP90发生解离。CreER被转运进入细胞核后可识别序列,诱导两个序列之间的目标基因(gene of interest,GOI)发生重组。B:光裂解雷帕霉素二聚体dRap。紫外光照射后,dRap裂解释放天然雷帕霉素Rap,从而诱导FRB-CreC和FKBP12-CreN二聚化,使得分割型Cre重组酶重构恢复催化活性。C:光笼重组酶。紫外光照射后,光笼Cre重组酶恢复活性。
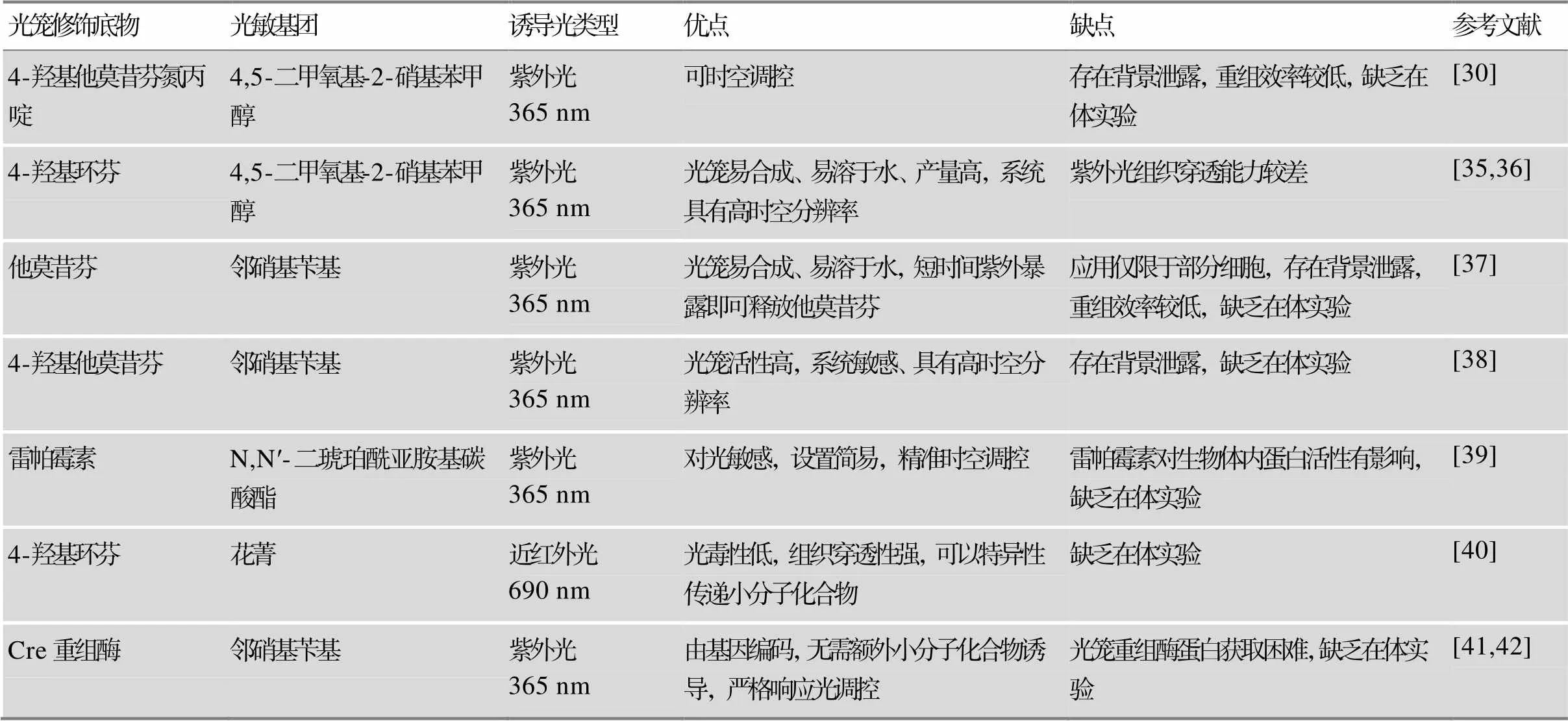
表1 光笼诱导重组系统的比较
与化学诱导重组系统相比,利用紫外光的局部照射增强了系统的时空特异性。然而,基于紫外光的光笼技术在使用中也存在一些问题,例如紫外照射会导致不同程度的DNA损伤,从而引起细胞毒性;紫外光组织穿透能力较差,需要进行多次辐照等。相比之下,近红外光的光毒性低且具有高效的组织穿透性,因此Gorka等[40]设计了一种基于花菁(cyanine)的光笼,在报告细胞系中可以依赖近红外光释放4-羟基环芬,从而结合CreER调控基因表达。这种近红外解锁策略需要进一步在体内进行验证,以便未来在复杂生理环境中应用(表1)。
1.2 光笼重组酶
光笼重组酶(photocaged recombinase)是直接对重组酶蛋白进行光笼修饰(图1 C)。Edwards等[41]设计了一种基因编码释放Cre重组酶的光笼,可以直接在Cre蛋白的Y324催化位点安装光敏基团邻硝基苄基,紫外光照射下可恢复Cre活性,实现基因重组。光笼Cre重组酶的生成方法是将携带突变的表达质粒和邻硝基苄基酪氨酸表达质粒共转化大肠杆菌[48],表达生成的光笼Cre蛋白需要分离纯化后才能使用,制备麻烦限制了其应用。Luo等[42]将突变的基因克隆入邻硝基苄基酪氨酸表达载体,无需额外的蛋白表达和纯化,转染了表达载体和报告质粒的细胞在紫外暴露下可产生功能性Cre重组酶,高效诱导报告基因重组。但这种光笼重组酶系统仅在哺乳动物细胞中得到验证,还需要更深入广泛的生物体内运用验证(表1)。
2 光遗传学诱导重组系统
光遗传学(optogenetics)是一种新兴的融合光学(optics)和遗传学(genetics)的生物技术。其中基因编码的光遗传学开关(optogenetic switch)是一类强大的光遗传学工具,由响应光刺激产生聚合反应或构象变化的光敏蛋白(photosensitive protein)组成。基于各种光敏蛋白已开发一系列光遗传学开关:隐花色素[49~52]、VIVID(VVD)[53~55]、Magnets[56]、植物光敏色素[57~59]、UVR8[60~62]、细菌光敏色素[63~65]、LOV结构域[66~68]等。隐花色素和VVD系统的光遗传学开关是最广泛使用的光遗传学工具,主要由波长为450~490 nm的蓝光控制。然而,蓝光的组织穿透力不足,需要较长的曝光时间,导致对细胞的光毒性增加,限制了蓝光光遗传学开关在动物体内的应用。使用更长波长的光源可以突破这一限制。光谱区域在700~900 nm内的远红光和近红外光谱可以穿透更深的组织或内脏器官[69,70]。由此,研究者们又开发了一系列基于响应红光或远红光的植物和细菌光敏色素的光遗传学开关,来进行胚胎组织或深部组织中的光遗传学操作。利用上述光遗传学开关和不同分割型重组酶,可以产生多样的光遗传学诱导重组系统。目前已开发的分割型重组酶包括Cre、Dre和Flp等,其中以分割型Cre重组酶的光遗传学诱导重组系统最广泛应用(表2)。
2.1 隐花色素
来源于拟南芥()中的隐花色素CRY2是一种蓝光响应光感受器,其天然结合配体为具有螺旋-环-螺旋结构的CIB1蛋白。在蓝光刺激条件下,CRY2与CIB1相互作用形成二聚体,并且在黑暗条件下二者逐渐解聚,无需外源辅助因子。CRY2蛋白含有一个保守的N端光裂合酶同源区域(photolyase homology region, PHR),是与CIB1结合的最小相互作用域。在蓝光调控下,CRY2或CRY2PHR可与全长CIB1或截短形式的CIBN发生聚合或解聚作用[49]。CRY2-CIB1二聚化系统已被广泛用于功能研究,如用于蛋白质分布和定位[49,81]、转录调节[82~85]、磷酸肌醇水平[86~88]、细胞骨架动力学[89]等。
最早将CRY2-CIB1系统和Cre重组酶联合使用的是Kennedy等[49],通过将CRY2和CIBN分别融合到分割型Cre重组酶的N端(氨基酸19~104)和C端(氨基酸106~343)。在蓝光照射下CRY2和CIBN的二聚化重构Cre恢复催化活性,介导DNA重组。这被认为是第一代光遗传学Cre重组系统(PA-Cre 1.0) (图2 A,表2)。Boulina等[90]将编码三色荧光蛋白的Brainbow基因盒与PA-Cre结合,在果蝇中诱导荧光蛋白的表达,实现了多色标记细胞的光控实时成像。Schindler等[91]利用光纤传导的蓝光照射激活PA-Cre,实现对小鼠海马体永久性基因修饰,并且使用组织穿透力较强的双光子激活可以对小鼠大脑中亚毫米精度组织进行基因表达调控。
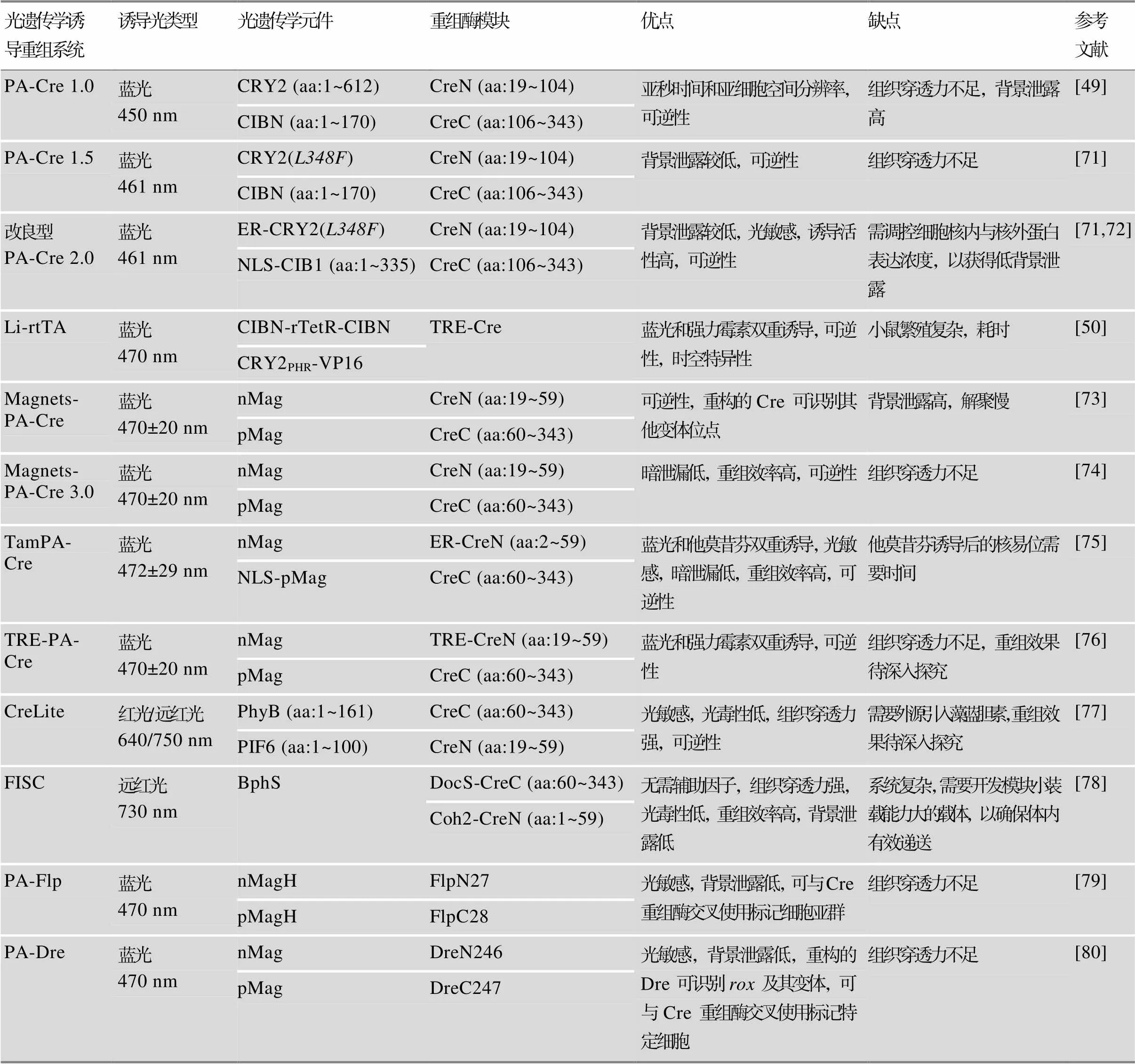
表2 光遗传学诱导重组系统的比较
尽管CRY2-CIB1系统可以在亚秒时间分辨率和亚细胞空间分辨率上实现二聚化,但是存在元件体积大、暗背景泄露、诱导效率低等局限性。通过优化CRY2-CIB1元件可以升级该系统。Taslimi等[71]发现CRY2()-CreN突变体在短暂的光照时间下可快速结合CIBN-CreC诱导DNA重组,且减缓二聚体的解聚,延长相互作用时间。该系统与PA-Cre 1.0相比,重组酶活性提高约35%,暗背景下本底泄露显著降低50%,被称为PA-Cre 1.5 (图2 A,表2)。在PA-Cre 1.5的基础上将CRY2()-CreN和CIB1-CreC构建在同一载体上,可使得重组背景极低,在哺乳动物细胞和小鼠体内实验中显示出更加灵敏的光响应性以及更高的重组效率,升级为PA-Cre 2.0[71](表2)。Meador等[72]系统地检查了PA-Cre 2.0的两个模块蛋白的表达和定位对重组效率的影响,发现PA-Cre 2.0的低暗背景是由于CRY2()- CreN的核-胞质穿梭所导致,突变削弱了CRY2的核定位,而主要表达在细胞质中。随着细胞中整体蛋白质表达的增加,细胞核中CRY2()- CreN减少。基于此,利用化学和光控双重诱导,设计产生他莫昔芬调控核转位的ER- CRY2()- CreN和核定位的NLS-CIB1-CreC构成的改良型PA-Cre 2.0,实现对DNA重组的严格控制(图2 B,表2)。
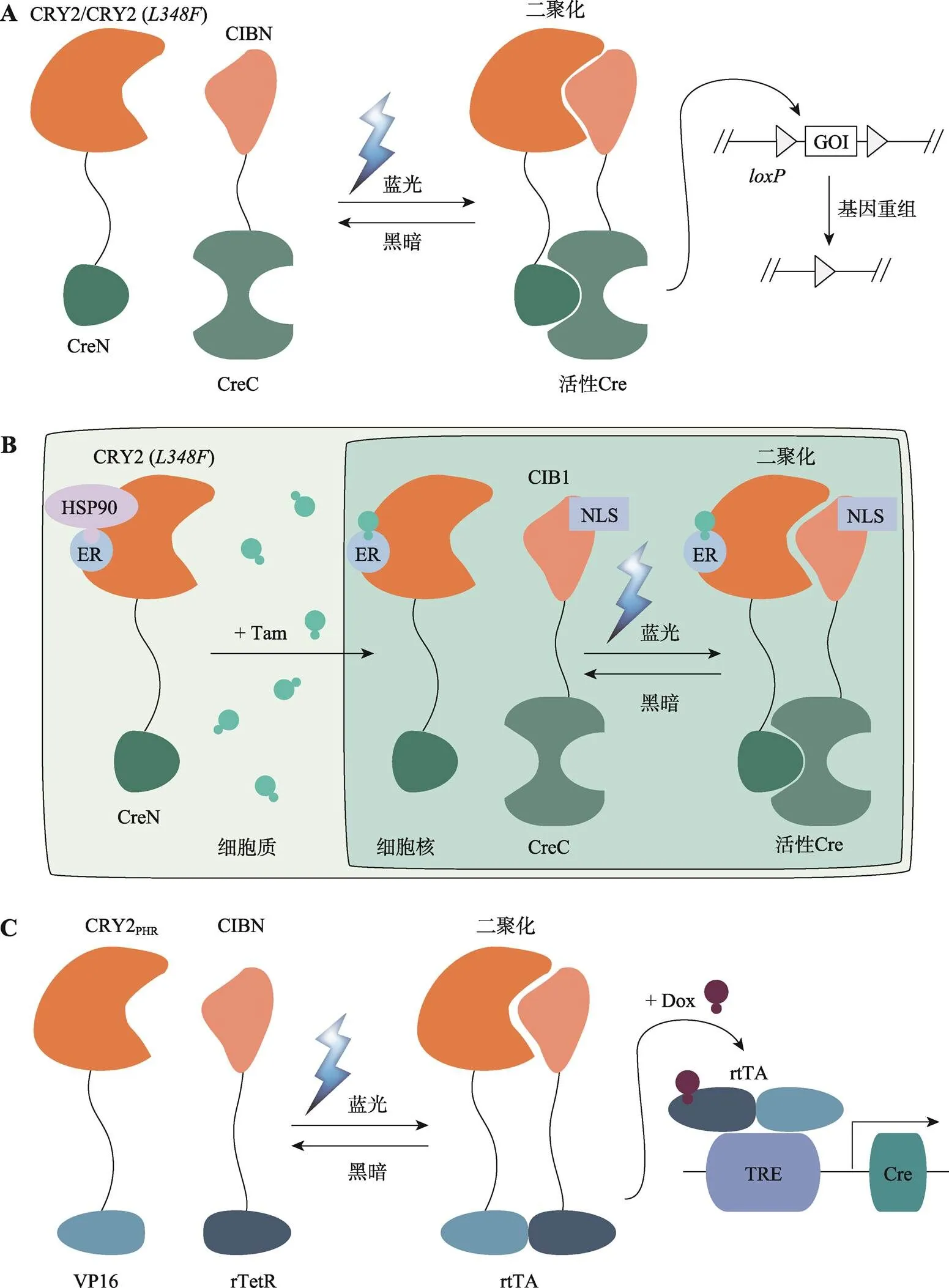
图2 基于隐花色素的光控诱导重组系统
A:CRY2或者CRY2()融合CreN,CIBN融合CreC构建的PA-Cre 1.0以及优化的PA-Cre 1.5系统。在黑暗条件下,Cre被分成两个片段对位点没有催化活性。在蓝光照射下,CRY2或者CRY2()与CIBN发生二聚化介导CreN和CreC互补重构,使得Cre迅速恢复催化活性,可识别两个位点发生DNA重组。B:光与他莫昔芬Tam双重调控的改良型PA-Cre 2.0系统,包含融合了ER的CRY2 ()-CreN和NLS-CIB1-CreC,通过Tam控制核转运和光介导组装分割片段,实现对Cre重组酶活性的双重控制,提供更为复杂的DNA重组调控。C:Li-rtTA系统。rtTA的两个功能域即DNA结合域rTetR和转录激活结构域VP16分别与CIBN和CRY2PHR相融合。蓝光刺激CRY2PHR和CIBN的二聚化,促使rTetR和VP16组合发挥完整的rtTA功能。在强力霉素Dox存在的情况下,二聚化的融合蛋白激活Tet-on系统,驱动Cre的表达。NLS:核定位信号。
利用Tet-on表达调控系统的元件也可以设计成光遗传学诱导重组系统。Li等[50]通过将Tet-on系统的rtTA元件的两个功能域VP16和rTetR分别与CRY2PHR和CIBN融合,设计了一种新型光控rtTA (Li-rtTA)系统。通过CRY2PHR和CIBN的二聚化而重新结合形成的rtTA,在强力霉素存在的情况下,能够结合TRE并驱动下游基因表达。在蓝光和强力霉素的双重诱导之后,Li-rtTA系统以时空特异的方式激活小鼠报告基因。使用rtTA作为光激活模块,能够利用许多现有的遗传工具来实现多样化的时空调控,如与基于Tet-on的Cre工具鼠交配获得的子代小鼠可以实现时空特异性基因重组(图2 C,表2)。
2.2 VIVID与Magnets
VIVID (VVD)是一种来源于丝状真菌粗糙脉孢菌()的蓝光光感受器,是目前作为光遗传学工具的最小的光感受器之一,可以响应蓝光从单体可逆地转换为同源二聚体[53~55]。但这种天然同源二聚化亲和力低,影响结合效率且解聚动力学太慢,无法精确调控蛋白质相互作用[56,92,93]。因此开发能够克服天然光感受器限制的强大且多功能的光遗传学开关至关重要。Kawano等[56]通过改造VVD开发了两种不同类型的变体,分别是带正电荷的“正磁体”(pMag)和带负电荷的“负磁体”(nMag),称为Magnets。Magnets的磁体对pMag和nMag在蓝光照射下通过静电相互作用识别吸引从而发生异源二聚化,可以防止不需要的同源二聚化。Magnets为蛋白活动和相关细胞功能的时空调控研究提供了强大的工具。该研究者将Magnets和分割型Cre重组酶联合使用,开发了一种新型Magnets-PA-Cre系统。通过Magnets的异源二聚化重新组装分割型Cre重组酶,恢复Cre重组酶的催化活性,促使在低强度或短周期脉冲的蓝光照射下能够快速介导DNA重组,该系统在体内具有高时空分辨率[73](图3 A)。与CRY2-CIB1系统不同的是,Magnets-PA-Cre系统使用的分割型Cre片段是CreN59 (氨基酸19~59)和CreC60 (氨基酸60~343),恢复活性后的Cre重组酶不仅可以识别序列还可以识别其他变体如和位点[94~96],极大地扩展了Magnets-PA- Cre的适用性。为了减少在黑暗及自然光下的系统泄露,Morikawa等[74]进一步开发了升级版本Magnets- PA-Cre 3.0,通过使用CAG启动子与2A自切割肽、优化密码子,使得该系统最大程度地减少了暗泄漏,并且具有高重组效率(表2)。
另外,利用他莫昔芬结合CreER调控核转位也可以减少黑暗背景下的系统泄露[72]。Allen等[75]将CreER系统与Magnets相结合,创建了TamPA-Cre系统(图3 B)。通过将胞质定位的ER配体结合域融合到CreN(2~59)-nMag的N端,使其与核定位的NLS-pMag-CreC(60~343)在空间上实现物理分离。这种物理分离防止了Magnets-PA-Cre系统中出现的自发、浓度依赖的nMag-pMag二聚化,从而阻止了系统泄漏。TamPA-Cre系统的光刺激模式(脉冲与连续曝光)、他莫昔芬添加时间和光控开始时间,都能调控该系统的重组效率。在他莫昔芬处理3小时后的脉冲蓝光刺激下,TamPA-Cre系统表现出对低强度、短时间蓝光暴露的高敏感性,可高效诱导DNA重组(表2)。
Tet-off是另一种基因调控系统,与Tet-on系统相反,在没有强力霉素(Dox)情况下持续性开启基因表达,而在强力霉素存在的情况下关闭基因表达[97,98]。将Tet-off系统与Magnets结合构建的tTA依赖性启动子驱动PA-Cre表达的TRE-PA-Cre小鼠,在蓝光照射下可实现时空特异性DNA重组,而且该系统可以通过强力霉素来关闭,一定程度上减少系统的背景泄露[76],但该系统在小鼠模型的基因调控效果还需要更深入的表征与研究(图3 C,表2)。
2.3 光敏色素
来自植物拟南芥的光敏色素A(phytochrome A, PhyA)是一种红光和远红光响应光感受器。在与发色团藻蓝胆素(phycocyanobilin, PCB)共价结合后,对红光和远红光均敏感,可逆地与伴侣蛋白FHY1结合(660 nm红光)或分离(730 nm远红光)[99~101]。此外,更为常用的是另一种来自拟南芥的光敏色素B (phytochrome B, PhyB)。在红光照射下,PhyB在PCB的介导下发生可逆构象变化,从而与光敏色素相互作用因子(phytochrome interaction factor, PIF)发生结合,并且这种结合在远红光照射下发生解离[102,103]。PhyB/PIF系统被用于在哺乳动物细胞中以精准的时空分辨率调控基因表达、蛋白质-蛋白质相互作用和蛋白质定位等[58,59,104]。Yen等[77]将PhyB/PIF6系统与分割型Cre重组酶相结合开发了CreLite系统,在红光照射下,PhyB-CreC和PIF6-CreN融合蛋白在PCB存在下结合在一起,从而恢复Cre重组酶活性。CreLite系统被用于培养细胞和离体器官中的时空调控基因表达(图4 A,表2)。值得注意的是,PCB对于PhyA和PhyB介导的光控开关至关重要,可它不能在哺乳动物细胞内自然合成[105],因而限制了Phy系统的应用。
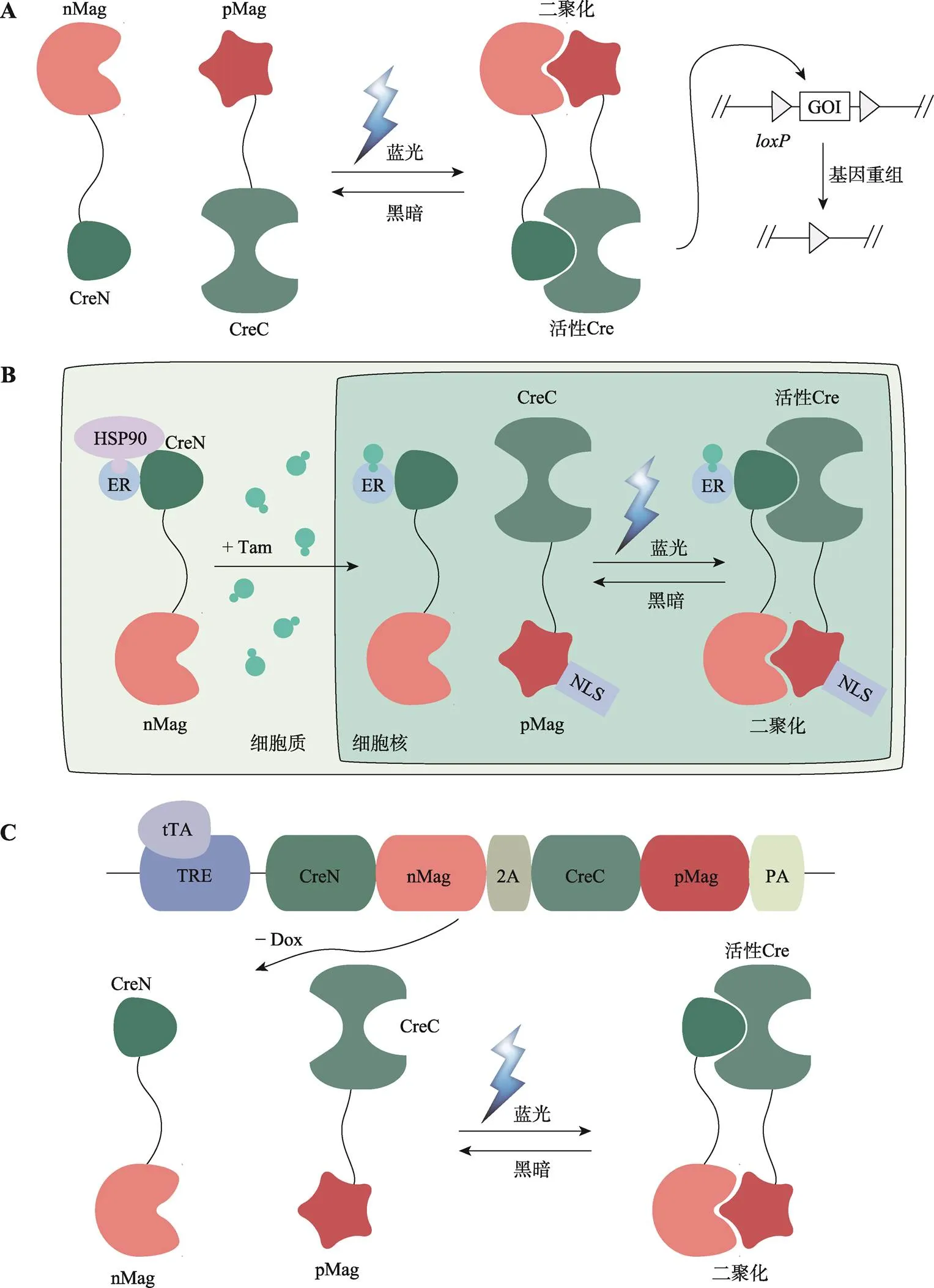
图3 基于Magnets的光控诱导重组系统
A:Magnets-PA-Cre和Magnets-PA-Cre3.0系统。蓝光照射下,nMag和pMag的二聚化重构分割型Cre重组酶活性,促使两个位点的目标基因(GOI)发生重组。B:TamPA-Cre系统。通过将胞质定位的雌激素受体(ER)融合到分割型CreN-nMag的N端,使TamPA-Cre蛋白ER-CreN-nMag与核定位的NLS-pMag-CreC在空间上分离。在他莫昔芬Tam处理和蓝光刺激下,分割型Cre重组酶随着nMag-pMag的二聚化而互补重构。C:TRE-PA-Cre系统。tTA依赖的TRE启动子驱动CreN-nMag和CreC-pMag的表达,在没有Dox情况下,蓝光照射激活nMag-pMag二聚化重构分割型Cre重组酶恢复催化活性。
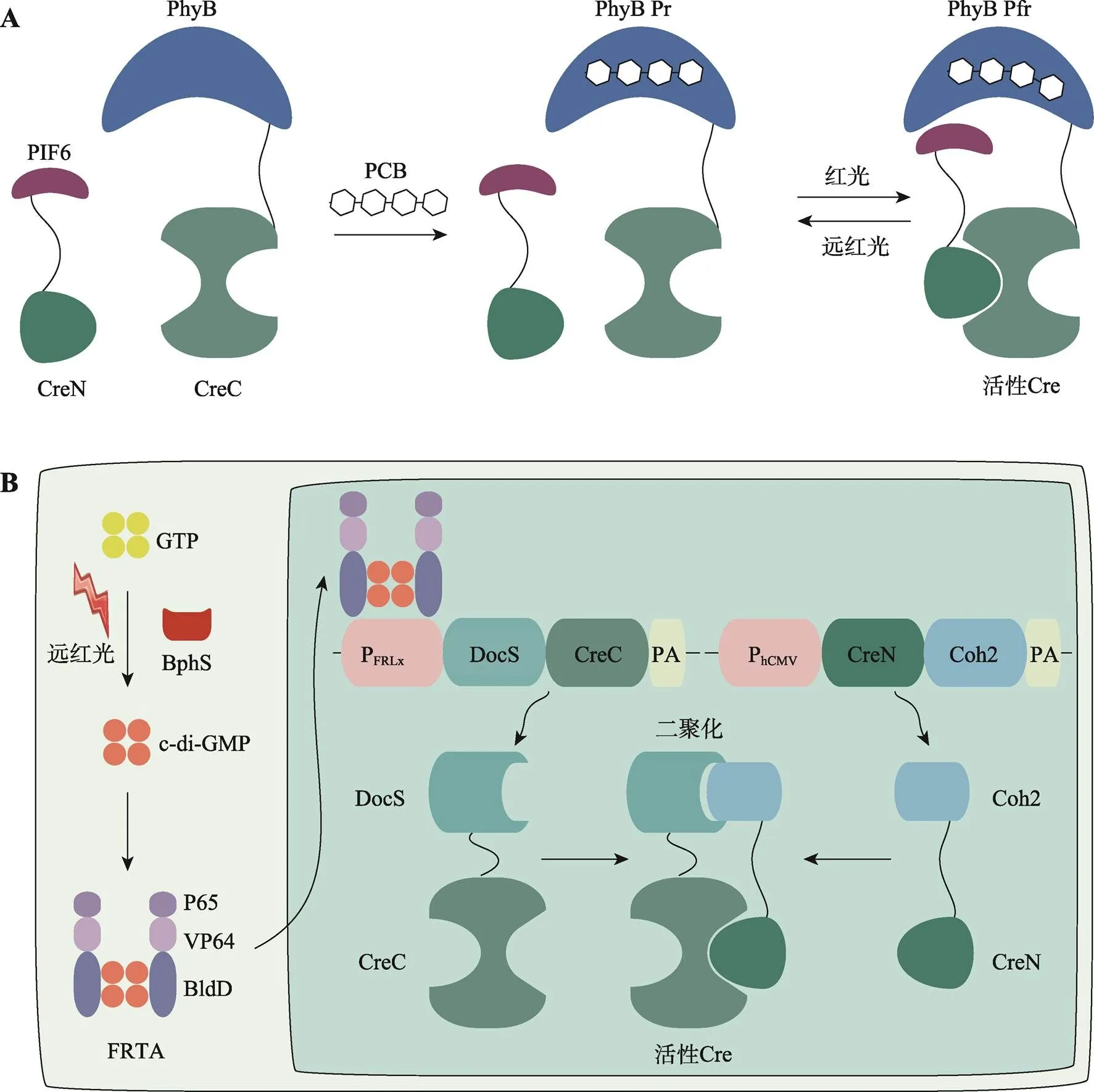
图4 基于光敏色素的光控诱导重组系统
A:基于PhyB的CreLite系统。在这个系统中,PhyB和PIF6分别与CreC和CreN融合。PhyB需要辅助因子PCB才能发挥功能。PhyB与PCB共价结合后吸收红光和红外光。当红光暴露后,PhyB发生构象变化,从失活的Pr形式(红色吸收)转变为有活性的Pfr形式(远红色吸收)。这个过程可以被远红外光逆转。Pfr状态下的PhyB和PIF6相互结合,将分割型Cre重组酶的两个片段组合重构,恢复其重组酶活性。B:基于BphS的FISC系统。在这个系统中,Cre重组酶被分为两个片段,其中CreN与Coh2融合,由组成型启动子PhCMV驱动,CreC与DocS融合,由远红光诱导启动子PFRLx驱动。远红光照射下,光感受器BphS将三磷酸鸟苷酸GTP转化为环二鸟苷酸单磷酸盐c-di-GMP,诱导远红光依赖的转录激活因子FRTA(P65-VP64-BldD)与启动子PFRLx结合,驱动DocS-CreC表达。基于Coh2和DocS结构域的强大亲和力,两个分割型Cre片段组装在一起,恢复Cre重组酶的催化活性。
细菌中也存在类似的光敏色素,如BphP1和BphS[64,106]。与Phy系统相比,细菌光敏色素使用的发色团胆绿素(biliverdin, BV)在包括哺乳动物细胞在内的真核细胞中天然存在且含量丰富,无需额外引入[107~109]。因此,细菌光敏色素已被设计成用于哺乳动物组织的多种近红外探针[110]。已经构建了基于细菌光敏色素BphP1及其天然配体PpsR2的近红外光遗传调控系统,能够介导细胞内信号传导、调控体内基因表达[63,111]。但该系统存在一些缺点如BphP1和PpsR2的蛋白质分子量相对较大,光控效率相对较低等。Wu等[78]设计了一种基于BphS的远红光诱导系统,将BphS光遗传学开关和分割型Cre重组酶联合构建了FISC (far-red light-induced split Cre/system)系统,仅通过远红光就可以实现体内基因的光遗传学调控。FISC系统表现出低背景、低细胞毒性,强大器官穿透力等优点,可高效光控介导活体小鼠内脏器官的DNA重组,性能大大优于基于蓝光诱导的光控重组系统(图4 B,表2)。
2.4 光遗传学开关与其他重组酶的联合使用
单个重组酶系统满足不了研究复杂生命过程的遗传工具需求。随着其他新型SSRs如Flp[4,79]、Dre[5]、Vika[112]和Nigri[113,114]等的发现鉴定,极大地丰富了基因操作工具。SSRs之间通常不会发生交叉重组,因而经常联合使用以实现更为精确的基因操作。已有报道表明可在小鼠体内同时使用Cre/、Flp/和Dre/这三种重组系统[115,116]。
Jung等[79]基于Magnets首次开发了一种适用于体内基因操作的高光敏和高效率的光控诱导Flp重组酶(PA-Flp),并将其与Cre重组酶联合构成Flp依赖的Cre重组系统,可光控激活Flp进而激活Cre用于小鼠的行为学研究。该系统具有高度光敏、非侵袭性、高效、易操作的优点,利用蓝光即可激活小鼠大脑深部区域的基因发生重组(表2)。Li等[80]借助分子动力学模拟,通过一系列筛选和优化,设计了一种可光控诱导的PA-Dre系统,并证明该系统在蓝光照射下不仅在细胞上,还可在小鼠的肝脏和脑部高效精准灵活地调控基因表达。此外,通过双floxed倒置开放阅读框策略,构建了Cre激活的光诱导Dre系统(Cre-activated light-inducible Dre, CALID),在小鼠体内细胞群体中实现时空特异性基因调控[80](表2)。为了针对单个神经元进行结构和功能研究,Yao等[117]研究团队基于VVD系统创建了Cre、Dre和Flp重组酶系统RecV。通过单光子或双光子照射诱导后,RecV系统能够对小鼠和斑马鱼中的单细胞或特定细胞群体进行精准基因调控。
3 结语与展望
光控诱导重组系统在近几年获得了长足的发展和应用。为了弥补化学诱导重组系统的空间局限性,可以将化学诱导剂设计成光笼,利用光控释放的方法增强化学诱导重组系统的时空调控分辨率。现有的可被光敏基团修饰的化学诱导剂主要是针对CreER和Di-Cre系统,后续可以进一步研发针对Tet-on和DD-Cre系统的光敏基团修饰化学诱导剂。另外,现有的光敏基团大多是响应紫外光诱导,会造成DNA损伤,应用范围较为局限。为此可以开发响应蓝光、红光、远红光等其他光源诱导的新型光敏基团,进而丰富光敏基团种类,拓宽应用范围,提升光笼诱导重组系统的安全性和灵活性。
由于化学诱导剂和光敏基团种类有限,合成毒性小、兼容性强的光笼仍具有挑战性。相比之下,利用光遗传学的光控二聚化开关来介导分割型重组酶重新激活的策略具有更强的灵活性和兼容性。多种类型的光遗传学开关和重组酶为精准遗传操作提供了更广泛的空间和更多的可能,以满足生物医学研究日益复杂的需求。最佳的光控诱导系统应该是安全、高效、简单、可控,并具有精准的时空调控、低泄漏、低背景、低毒性的属性。综合上述的研究,可以在两个方面进行系统升级改进。一方面,可以根据研究需要,通过不同光遗传学开关和不同重组酶组合方式来开发新的光遗传学诱导重组系统。另一方面,可以通过筛选有效的重组酶分割形式,优化光控元件的蛋白大小、组合方式、表达顺序及其在表达载体的位置,以及优化光控元件的氨基酸密码子、Linker序列、核定位信号种类与位置等途径进一步升级系统。未来,迭代更新的光控诱导重组系统将广泛用于生命科学研究领域,实现高时空分辨率遗传操作,解析尚未可知的复杂生物过程。
[1] Sternberg N, Hamilton D. Bacteriophage P1 site- specific recombination. I. Recombination between loxP sites.,1981, 150(4): 467–486.
[2] Sternberg N, Hamilton D, Hoess R. Bacteriophage P1 site-specific recombination. II. Recombination between loxP and the bacterial chromosome.,1981, 150(4): 487–507.
[3] Sternberg N. Bacteriophage P1 site-specific recombination. III. Strand exchange during recombination at lox sites.,1981, 150(4): 603–608.
[4] Golic KG, Lindquist S. The FLP recombinase of yeast catalyzes site-specific recombination in the Drosophila genome.,1989, 59(3): 499–509.
[5] Anastassiadis K, Fu J, Patsch C, Hu SB, Weidlich S, Duerschke K, Buchholz F, Edenhofer F, Stewart AF. Dre recombinase, like Cre, is a highly efficient site-specific recombinase in E. coli, mammalian cells and mice.,2009, 2(9–10): 508–515.
[6] Han XM, Zhang ZQ, He LJ, Zhu H, Li Y, Pu WJ, Han MY, Zhao H, Liu K, Li Y, Huang XZ, Zhang MJ, Jin HW, Lv Z, Tang J, Wang JJ, Sun RL, Fei J, Tian XY, Duan SZ, Wang QD, Wang LX, He B, Zhou B. A suite of new Dre recombinase drivers markedly expands the ability to perform intersectional genetic targeting.,2021, 28(6): 1160–1176.e7.
[7] Tronche F, Casanova E, Turiault M, Sahly I, Kellendonk C. When reverse genetics meets physiology: the use of site-specific recombinases in mice.,2002, 529(1): 116–121.
[8] Sauer B, Henderson N. Site-specific DNA recombination in mammalian cells by the Cre recombinase of bacteriophage P1.,1988, 85(14): 5166–5170.
[9] Brault V, Besson V, Magnol L, Duchon A, Hérault Y. Cre/loxP-mediated chromosome engineering of the mouse genome.,2007(178): 29–48.
[10] Indra AK, Warot X, Brocard J, Bornert JM, Xiao JH, Chambon P, Metzger D. Temporally-controlled site-specific mutagenesis in the basal layer of the epidermis: comparison of the recombinase activity of the tamoxifen-inducible Cre-ER(T) and Cre-ER(T2) recombinases.,1999, 27(22): 4324– 4327.
[11] Schönig K, Schwenk F, Rajewsky K, Bujard H. Stringent doxycycline dependent control of CRE recombinase in vivo.,2002, 30(23): e134.
[12] Utomo AR, Nikitin AY, Lee WH. Temporal, spatial, and cell type-specific control of Cre-mediated DNA recombination in transgenic mice.,1999, 17(11): 1091–1096.
[13] Jullien N, Sampieri F, Enjalbert A, Herman JP. Regulation of Cre recombinase by ligand-induced complementation of inactive fragments.,2003, 31(21): e131.
[14] Sando R, Baumgaertel K, Pieraut S, Torabi-Rander N, Wandless TJ, Mayford M, Maximov A. Inducible control of gene expression with destabilized Cre.,2013, 10(11): 1085–1088.
[15] Metzger D, Clifford J, Chiba H, Chambon P. Conditional site-specific recombination in mammalian cells using a ligand-dependent chimeric Cre recombinase.,1995, 92(15): 6991–6995.
[16] Feil R, Wagner J, Metzger D, Chambon P. Regulation of Cre recombinase activity by mutated estrogen receptor ligand-binding domains.,1997, 237(3): 752–757.
[17] Gossen M, Freundlieb S, Bender G, Müller G, Hillen W, Bujard H. Transcriptional activation by tetracyclines in mammalian cells.,1995, 268(5218): 1766–1769.
[18] Hirrlinger J, Scheller A, Hirrlinger PG, Kellert B, Tang WN, Wehr MC, Goebbels S, Reichenbach A, Sprengel R, Rossner MJ, Kirchhoff F. Split-cre complementation indicates coincident activity of different genes in vivo.,2009, 4(1): e4286.
[19] Tang JCY, Rudolph S, Dhande OS, Abraira VE, Choi S, Lapan SW, Drew IR, Drokhlyansky E, Huberman AD, Regehr WG, Cepko CL. Cell type-specific manipulation with GFP-dependent Cre recombinase.,2015, 18(9): 1334–1341.
[20] Gengenbacher M, Zimmerman MD, Sarathy JP, Kaya F, Wang H, Mina M, Carter C, Hossen MA, Su HW, Trujillo C, Ehrt S, Schnappinger D, Dartois V. Tissue distribution of doxycycline in animal models of tuberculosis.,2020, 64(5): e02479–e02519.
[21] Wilking N, Appelgren LE, Carlström K, Pousette A, Theve NO. The distribution and metabolism of 14C-labelled tamoxifen in spayed female mice.,1982, 50(3): 161–168.
[22] Zhang JS, Wong SHD, Wu X, Lei H, Qin M, Shi P, Wang W, Bian LM, Cao Y. Engineering photoresponsive ligand tethers for mechanical regulation of stem cells.,2021, 33(48): e2105765.
[23] Richter F, Fonfara I, Bouazza B, Schumacher CH, Bratovič M, Charpentier E, Möglich A. Engineering of temperature- and light-switchable Cas9 variants.,2016, 44(20): 10003–10014.
[24] Liu RM, Yang J, Yao J, Zhao Z, He W, Su N, Zhang ZY, Zhang CX, Zhang Z, Cai HB, Zhu LY, Zhao YZ, Quan S, Chen XJ, Yang Y. Optogenetic control of RNA function and metabolism using engineered light-switchable RNA-binding proteins.,2022, 40(5): 779–786.
[25] Mayer G, Heckel A. Biologically active molecules with a “light switch”.,2006, 45(30): 4900–4921.
[26] Pinheiro AV, Baptista P, Lima JC. Light activation of transcription: photocaging of nucleotides for control over RNA polymerization.,2008, 36(14): e90.
[27] Karginov AV, Zou Y, Shirvanyants D, Kota P, Dokholyan NV, Young DD, Hahn KM, Deiters A. Light regulation of protein dimerization and kinase activity in living cells using photocaged rapamycin and engineered FKBP.,2011, 133(3): 420–423.
[28] Miller DS, Chirayil S, Ball HL, Luebke KJ. Manipulating cell migration and proliferation with a light-activated polypeptide.,2009, 10(3): 577–584.
[29] Lemke EA, Summerer D, Geierstanger BH, Brittain SM, Schultz PG. Control of protein phosphorylation with a genetically encoded photocaged amino acid.,2007, 3(12): 769–772.
[30] Link KH, Shi YH, Koh JT. Light activated recombination.,2005, 127(38): 13088–13089.
[31] Lawrence DS. The preparation and in vivo applications of caged peptides and proteins.,2005, 9(6): 570–575.
[32] Young DD, Deiters A. Photochemical control of biological processes.,2007, 5(7): 999–1005.
[33] Tang XJ, Dmochowski IJ. Regulating gene expression with light-activated oligonucleotides.,2007, 3(2): 100–110.
[34] Sinha DK, Neveu P, Gagey N, Aujard I, Benbrahim- Bouzidi C, Le Saux T, Rampon C, Gauron C, Goetz B, Dubruille S, Baaden M, Volovitch M, Bensimon D, Vriz S, Jullien L. Photocontrol of protein activity in cultured cells and zebrafish with one- and two-photon illumination.,2010, 11(5): 653–663.
[35] Sinha DK, Neveu P, Gagey N, Aujard I, Le Saux T, Rampon C, Gauron C, Kawakami K, Leucht C, Bally- Cuif L, Volovitch M, Bensimon D, Jullien L, Vriz S. Photoactivation of the CreER T2 recombinase for conditional site-specific recombination with high spatiotemporal resolution.,2010, 7(2): 199– 204.
[36] Lu X, Agasti SS, Vinegoni C, Waterman P, DePinho RA, Weissleder R. Optochemogenetics (OCG) allows more precise control of genetic engineering in mice with CreER regulators.,2012, 23(9): 1945–1951.
[37] Inlay MA, Choe V, Bharathi S, Fernhoff NB, Baker JR Jr, Weissman IL, Choi SK. Synthesis of a photocaged tamoxifen for light-dependent activation of Cre-ER recombinase-driven gene modification.,2013, 49(43): 4971–4973.
[38] Faal T, Wong PT, Tang SZ, Coulter A, Chen Y, Tu CH, Baker JR, Choi SK, Inlay MA. 4-Hydroxytamoxifen probes for light-dependent spatiotemporal control of Cre-ER mediated reporter gene expression.,2015, 11(3): 783–790.
[39] Brown KA, Zou Y, Shirvanyants D, Zhang J, Samanta S, Mantravadi PK, Dokholyan NV, Deiters A. Light- cleavable rapamycin dimer as an optical trigger for protein dimerization.,2015, 51(26): 5702–5705.
[40] Gorka AP, Nani RR, Zhu JJ, Mackem S, Schnermann MJ. A near-IR uncaging strategy based on cyanine photochemistry.,2014, 136(40): 14153–14159.
[41] Edwards WF, Young DD, Deiters A. Light-activated Cre recombinase as a tool for the spatial and temporal control of gene function in mammalian cells.,2009, 4(6): 441–445.
[42] Luo J, Arbely E, Zhang J, Chou C, Uprety R, Chin JW, Deiters A. Genetically encoded optical activation of DNA recombination in human cells.,2016, 52(55): 8529–8532.
[43] Beverage JN, Sissung TM, Sion AM, Danesi R, Figg WD. CYP2D6 polymorphisms and the impact on tamoxifen therapy.,2007, 96(9): 2224– 2231.
[44] Cambridge SB, Geissler D, Keller S, Cürten B. A caged doxycycline analogue for photoactivated gene expression.,2006, 45(14): 2229–2231.
[45] Goegan B, Terzi F, Bolze F, Cambridge S, Specht A. Synthesis and characterization of photoactivatable doxycycline analogues bearing two-photon-sensitive photoremovable groups suitable for light-induced gene expression.,2018, 19(12): 1341–1348.
[46] Cambridge SB, Geissler D, Calegari F, Anastassiadis K, Hasan MT, Stewart AF, Huttner WB, Hagen V, Bonhoeffer T. Doxycycline-dependent photoactivated gene expression in eukaryotic systems.,2009, 6(7): 527–531.
[47] Zheng YJ, Nandakumar KS, Cheng K. Optimization of CAR-T cell-based therapies using small-molecule-based safety switches.,2021, 64(14): 9577–9591.
[48] Deiters A, Groff D, Ryu Y, Xie JM, Schultz PG. A genetically encoded photocaged tyrosine.,2006, 45(17): 2728–2731.
[49] Kennedy MJ, Hughes RM, Peteya LA, Schwartz JW, Ehlers MD, Tucker CL. Rapid blue-light-mediated induction of protein interactions in living cells.,2010, 7(12): 973–975.
[50] Li F, Lu ZW, Wu WB, Qian NN, Wang FC, Chen T. Optogenetic gene editing in regional skin.,2019, 29(10): 862–865.
[51] Bugaj LJ, Choksi AT, Mesuda CK, Kane RS, Schaffer DV. Optogenetic protein clustering and signaling activation in mammalian cells.,2013, 10(3): 249–252.
[52] Taslimi A, Vrana JD, Chen D, Borinskaya S, Mayer BJ, Kennedy MJ, Tucker CL. An optimized optogenetic clustering tool for probing protein interaction and function.,2014, 5: 4925.
[53] Zoltowski BD, Schwerdtfeger C, Widom J, Loros JJ, Bilwes AM, Dunlap JC, Crane BR. Conformational switching in the fungal light sensor Vivid.,2007, 316(5827): 1054–1057.
[54] Zoltowski BD, Crane BR. Light activation of the LOV protein vivid generates a rapidly exchanging dimer.,2008, 47(27): 7012–7019.
[55] Vaidya AT, Chen CH, Dunlap JC, Loros JJ, Crane BR. Structure of a light-activated LOV protein dimer that regulates transcription.,2011, 4(184): ra50.
[56] Kawano F, Suzuki H, Furuya A, Sato M. Engineered pairs of distinct photoswitches for optogenetic control of cellular proteins.,2015, 6: 6256.
[57] Liu HT, Yu XH, Li KW, Klejnot J, Yang HY, Lisiero D, Lin CT. Photoexcited CRY2 interacts with CIB1 to regulate transcription and floral initiation in Arabidopsis.,2008, 322(5907): 1535–1539.
[58] Shimizu-Sato S, Huq E, Tepperman JM, Quail PH. A light-switchable gene promoter system.,2002, 20(10): 1041–1044.
[59] Levskaya A, Weiner OD, Lim WA, Voigt CA. Spatiotemporal control of cell signalling using a light-switchable protein interaction.,2009, 461(7266): 997–1001.
[60] Wu D, Hu Q, Yan Z, Chen W, Yan CY, Huang X, Zhang J, Yang PY, Deng HT, Wang JW, Deng XW, Shi YG. Structural basis of ultraviolet-B perception by UVR8.,2012, 484(7393): 214–219.
[61] Cloix C, Kaiserli E, Heilmann M, Baxter KJ, Brown BA, O'Hara A, Smith BO, Christie JM, Jenkins GI. C-terminal region of the UV-B photoreceptor UVR8 initiates signaling through interaction with the COP1 protein.,2012, 109(40): 16366–16370.
[62] Crefcoeur RP, Yin RH, Ulm R, Halazonetis TD. Ultraviolet-B-mediated induction of protein-protein interactions in mammalian cells.,2013, 4: 1779.
[63] Kaberniuk AA, Shemetov AA, Verkhusha VV. A bacterial phytochrome-based optogenetic system controllable with near-infrared light.,2016, 13(7): 591–597.
[64] Ryu MH, Gomelsky M. Near-infrared light responsive synthetic c-di-GMP module for optogenetic applications.,2014, 3(11): 802–810.
[65] Shao JW, Xue S, Yu GL, Yu YH, Yang XP, Bai Y, Zhu SC, Yang LF, Yin JL, Wang YD, Liao SY, Guo SW, Xie MQ, Fussenegger M, Ye HF. Smartphone-controlled optogenetically engineered cells enable semiautomatic glucose homeostasis in diabetic mice.,2017, 9(387): eaal2298.
[66] Strickland D, Lin Y, Wagner E, Hope CM, Zayner J, Antoniou C, Sosnick TR, Weiss EL, Glotzer M. TULIPs: tunable, light-controlled interacting protein tags for cell biology.,2012, 9(4): 379–384.
[67] Woolley GA. Designing chimeric LOV photoswitches.,2012, 19(4): 441–442.
[68] Guntas G, Hallett RA, Zimmerman SP, Williams T, Yumerefendi H, Bear JE, Kuhlman B. Engineering an improved light-induced dimer (iLID) for controlling the localization and activity of signaling proteins.,2015, 112(1): 112–117.
[69] Wan S, Parrish JA, Anderson RR, Madden M. Transmittance of nonionizing radiation in human tissues.,1981, 34(6): 679–681.
[70] Gomelsky M. Photoactivated cells link diagnosis and therapy.,2017, 9(387): eaan3936.
[71] Taslimi A, Zoltowski B, Miranda JG, Pathak GP, Hughes RM, Tucker CL. Optimized second-generation CRY2-CIB dimerizers and photoactivatable Cre recombinase.,2016, 12(6): 425–430.
[72] Meador K, Wysoczynski CL, Norris AJ, Aoto J, Bruchas MR, Tucker CL. Achieving tight control of a photoactivatable Cre recombinase gene switch: new design strategies and functional characterization in mammalian cells and rodent.,2019, 47(17): e97.
[73] Kawano F, Okazaki R, Yazawa M, Sato M. A photoactivatable Cre-loxP recombination system for optogenetic genome engineering.,2016, 12(12): 1059–1064.
[74] Morikawa K, Furuhashi K, de Sena-Tomas C, Garcia-Garcia AL, Bekdash R, Klein AD, Gallerani N, Yamamoto HE, Park SHE, Collins GS, Kawano F, Sato M, Lin CS, Targoff KL, Au E, Salling MC, Yazawa M. Photoactivatable Cre recombinase 3.0 for in vivo mouse applications.,2020, 11(1): 2141.
[75] Allen ME, Zhou W, Thangaraj J, Kyriakakis P, Wu YQ, Huang ZL, Ho P, Pan YJ, Limsakul P, Xu XD, Wang YX. An AND-Gated drug and photoactivatable Cre-loxP system for spatiotemporal control in cell-based therapeutics.,2019, 8(10): 2359–2371.
[76] Takao T, Hiraoka Y, Kawabe K, Yamada D, Ming L, Tanaka K, Sato M, Takarada T. Establishment of a tTA-dependent photoactivatable Cre recombinase knock-in mouse model for optogenetic genome engineering.,2020, 526(1): 213–217.
[77] Yen ST, Trimmer KA, Aboul-Fettouh N, Mullen RD, Culver JC, Dickinson ME, Behringer RR, Eisenhoffer GT. CreLite: An optogenetically controlled Cre/loxP system using red light.,2020, 249(11): 1394–1403.
[78] Wu JL, Wang MY, Yang XP, Yi CW, Jiang J, Yu YH, Ye HF. A non-invasive far-red light-induced split-Cre recombinase system for controllable genome engineering in mice.,2020, 11(1): 3708.
[79] Jung H, Kim SW, Kim M, Hong J, Yu D, Kim JH, Lee Y, Kim S, Woo D, Shin HS, Park BO, Heo WD. Noninvasive optical activation of Flp recombinase for genetic manipulation in deep mouse brain regions.,2019, 10(1): 314.
[80] Li HY, Zhang QS, Gu YR, Wu YY, Wang YM, Wang LR, Feng SJ, Hu YQ, Zheng YS, Li YM, Ye HF, Zhou B, Lin LN, Liu MY, Yang HY, Li DL. Efficient photoactivatable Dre recombinase for cell type-specific spatiotemporal control of genome engineering in the mouse.,2020, 117(52): 33426–33435.
[81] Duan LT, Che D, Zhang K, Ong QX, Guo SL, Cui BX. Optogenetic control of molecular motors and organelle distributions in cells.,2015, 22(5): 671–682.
[82] Hughes RM, Bolger S, Tapadia H, Tucker CL. Light-mediated control of DNA transcription in yeast.,2012, 58(4): 385–391.
[83] Konermann S, Brigham MD, Trevino A, Hsu PD, Heidenreich M, Cong L, Platt RJ, Scott DA, Church GM, Zhang F. Optical control of mammalian endogenous transcription and epigenetic states.,2013, 500(7463): 472–476.
[84] Polstein LR, Gersbach CA. A light-inducible CRISPR-Cas9 system for control of endogenous gene activation.,2015, 11(3): 198–200.
[85] Nihongaki Y, Furuhata Y, Otabe T, Hasegawa S, Yoshimoto K, Sato M. CRISPR-Cas9-based photoactivatable transcription systems to induce neuronal differentiation.,2017, 14(10): 963–966.
[86] Idevall-Hagren O, Dickson EJ, Hille B, Toomre DK, De Camilli P. Optogenetic control of phosphoinositide metabolism.,2012, 109(35): E2316–E2323.
[87] Giordano F, Saheki Y, Idevall-Hagren O, Colombo SF, Pirruccello M, Milosevic I, Gracheva EO, Bagriantsev SN, Borgese N, De Camilli P. PI(4,5)P(2)-dependent and Ca(2+)-regulated ER-PM interactions mediated by the extended synaptotagmins.,2013, 153(7): 1494–1509.
[88] Kakumoto T, Nakata T. Optogenetic control of PIP3: PIP3 is sufficient to induce the actin-based active part of growth cones and is regulated via endocytosis.,2013, 8(8): e70861.
[89] Maiuri P, Rupprecht JF, Wieser S, Ruprecht V, Bénichou O, Carpi N, Coppey M, De Beco S, Gov N, Heisenberg CP, Lage Crespo C, Lautenschlaeger F, Le Berre M, Lennon-Dumenil AM, Raab M, Thiam HR, Piel M, Sixt M, Voituriez R. Actin flows mediate a universal coupling between cell speed and cell persistence.,2015, 161(2): 374–386.
[90] Boulina M, Samarajeewa H, Baker JD, Kim MD, Chiba A. Live imaging of multicolor-labeled cells in Drosophila.,2013, 140(7): 1605–1613.
[91] Schindler SE, McCall JG, Yan P, Hyrc KL, Li MJ, Tucker CL, Lee JM, Bruchas MR, Diamond MI. Photo-activatable Cre recombinase regulates gene expression in vivo.,2015, 5: 13627.
[92] Zoltowski BD, Vaccaro B, Crane BR. Mechanism-based tuning of a LOV domain photoreceptor.,2009, 5(11): 827–834.
[93] Lamb JS, Zoltowski BD, Pabit SA, Crane BR, Pollack L. Time-resolved dimerization of a PAS-LOV protein measured with photocoupled small angle X-ray scattering.,2008, 130(37): 12226–12227.
[94] Thomson JG, Rucker EB 3rd, Piedrahita JA. Mutational analysis of loxP sites for efficient Cre-mediated insertion into genomic DNA.,2003, 36(3): 162–167.
[95] Oberdoerffer P, Otipoby KL, Maruyama M, Rajewsky K. Unidirectional Cre-mediated genetic inversion in mice using the mutant loxP pair lox66/lox71.,2003, 31(22): e140.
[96] Araki K, Okada Y, Araki M, Yamamura KI. Comparative analysis of right element mutant lox sites on recombination efficiency in embryonic stem cells.,2010, 10: 29.
[97] Gossen M, Bujard H. Tight control of gene expression in mammalian cells by tetracycline-responsive promoters.,1992, 89(12): 5547–5551.
[98] Furth PA, St Onge L, Böger H, Gruss P, Gossen M, Kistner A, Bujard H, Hennighausen L. Temporal control of gene expression in transgenic mice by a tetracycline- responsive promoter.,1994, 91(20): 9302–9306.
[99] Jorissen HJMM, Quest B, Lindner I, de Marsac NT, Gärtner W. Phytochromes with noncovalently bound chromophores: the ability of apophytochromes to direct tetrapyrrole photoisomerization.,2002, 75(5): 554–559.
[100] Kami C, Mukougawa K, Muramoto T, Yokota A, Shinomura T, Lagarias JC, Kohchi T. Complementation of phytochrome chromophore-deficient Arabidopsis by expression of phycocyanobilin:ferredoxin oxidoreductase.,2004, 101(4): 1099–1104.
[101] Zhou Y, Kong DQ, Wang XY, Yu GL, Wu X, Guan NZ, Weber W, Ye HF. A small and highly sensitive red/far- red optogenetic switch for applications in mammals.,2022, 40(2): 262–272.
[102] Müller K, Engesser R, Timmer J, Nagy F, Zurbriggen MD, Weber W. Synthesis of phycocyanobilin in mammalian cells.,2013, 49(79): 8970–8972.
[103] Khanna R, Huq E, Kikis EA, Al-Sady B, Lanzatella C, Quail PH. A novel molecular recognition motif necessary for targeting photoactivated phytochrome signaling to specific basic helix-loop-helix transcription factors.,2004, 16(11): 3033–3044.
[104] Müller K, Engesser R, Metzger S, Schulz S, Kämpf MM, Busacker M, Steinberg T, Tomakidi P, Ehrbar M, Nagy F, Timmer J, Zubriggen MD, Weber W. A red/far-red light-responsive bi-stable toggle switch to control gene expression in mammalian cells.,2013, 41(7): e77.
[105] Wiltbank LB, Kehoe DM. Diverse light responses of cyanobacteria mediated by phytochrome superfamily photoreceptors.,2019, 17(1): 37–50.
[106] Kojadinovic M, Laugraud A, Vuillet L, Fardoux J, Hannibal L, Adriano JM, Bouyer P, Giraud E, Verméglio A. Dual role for a bacteriophytochrome in the bioenergetic control of Rhodopseudomonas palustris: enhancement of photosystem synthesis and limitation of respiration.,2008, 1777(2): 163–172.
[107] Ulijasz AT, Vierstra RD. Phytochrome structure and photochemistry: recent advances toward a complete molecular picture.,2011, 14(5): 498–506.
[108] Piatkevich KD, Subach FV, Verkhusha VV. Engineering of bacterial phytochromes for near-infrared imaging, sensing, and light-control in mammals.,2013, 42(8): 3441–3452.
[109] Tran MTN, Tanaka J, Hamada M, Sugiyama Y, Sakaguchi S, Nakamura M, Takahashi S, Miwa Y. In vivo image analysis using iRFP transgenic mice.,2014, 63(3): 311–319.
[110] Shcherbakova DM, Baloban M, Verkhusha VV. Near- infrared fluorescent proteins engineered from bacterial phytochromes.,2015, 27: 52–63.
[111] Redchuk TA, Kaberniuk AA, Verkhusha VV. Near- infrared light-controlled systems for gene transcription regulation, protein targeting and spectral multiplexing.,2018, 13(5): 1121–1136.
[112] Karimova M, Abi-Ghanem J, Berger N, Surendranath V, Pisabarro MT, Buchholz F. Vika/vox, a novel efficient and specific Cre/loxP-like site-specific recombination system.,2013, 41(2): e37.
[113] Karimova M, Splith V, Karpinski J, Pisabarro MT, Buchholz F. Discovery of Nigri/nox and Panto/pox site-specific recombinase systems facilitates advanced genome engineering.,2016, 6: 30130.
[114] Liu K, Yu W, Tang MX, Tang J, Liu XX, Liu QZ, Li Y, He LJ, Zhang LB, Evans SM, Tian XY, Lui KO, Zhou B. A dual genetic tracing system identifies diverse and dynamic origins of cardiac valve mesenchyme.,2018, 145(18): dev167775.
[115] Plummer NW, Evsyukova IY, Robertson SD, de Marchena J, Tucker CJ, Jensen P. Expanding the power of recombinase-based labeling to uncover cellular diversity.,2015, 142(24): 4385–4393.
[116] Karimova M, Baker O, Camgoz A, Naumann R, Buchholz F, Anastassiadis K. A single reporter mouse line for Vika, Flp, Dre, and Cre-recombination.,2018, 8(1): 14453.
[117] Yao SQ, Yuan P, Ouellette B, Zhou T, Mortrud M, Balaram P, Chatterjee S, Wang Y, Daigle TL, Tasic B, Kuang XL, Gong H, Luo QM, Zeng SQ, Curtright A, Dhaka A, Kahan A, Gradinaru V, Chrapkiewicz R, Schnitzer M, Zeng HK, Cetin A. RecV recombinase system for in vivo targeted optogenomic modifications of single cells or cell populations.,2020, 17(4): 422–429.
Development and application of light-controlled inducible recombination systems
Tian Xie, Mei Wang, Ruiyu Gao, Yanni Miao, Yiming Zhang, Jing Jiang
The site-specific recombination systems are composed of recombinases and specific recognition sites, which are powerful tools for gene manipulation and have been extensively used in life sciences research. Inducible recombination systems have been developed to precisely regulate gene expression in a spatiotemporal manner in cells and animals for applications such as gene function research, cell lineage tracing and disease treatment. Based on different spatiotemporal expression methods of recombinases, inducible recombination systems can be divided into two categories: chemical- controlled and light-controlled inductions. Light-controlled inducible recombination systems that utilize light as inducer consist of photocage and optogenetics in accordance with optical control patterns and objects. Photocaged inducible recombination systems are using photosensitive groups to control chemical inducers or recombinases. Their activities are inhibited by photosensitive groups before light induction and recovered after specific light irradiation, leading to light-controlled inducible gene recombination. While optogenetic inducible recombination systems rely on reactivations of split recombinases that mediated by optogenetic switches. Optogenetic switches are composed of a series of gene-encoded photosensitive proteins, including cryptochromes, VIVID, phytochromes, etc. These types of light-controlled inducible recombination systems provide more possibilities for analyzing gene expression and function from the dimension of high spatiotemporal resolution to meet the increasingly complex demands of life science research. In this review, we summarize the developing principles and applications of different types of light-controlled inducible recombination systems, compare their advantages and disadvantages, and prospect the development of more light-controlled recombination systems in the future, with the aims to provide theoretical basis and guidance for system optimization and upgrade.
light-controlled inducible recombination system; photocage;optogenetic switch;site-specific recombinase;spatiotemporal control
2022-05-15;
2022-06-28;
2022-07-13
国家重点研发计划专项(编号:2020YFA0509001),国家自然科学基金项目(编号:31801057)和上海市科学技术委员会科技计划项目(编号:21140905100,22140903500)资助[Supported by the National Key Research and Development Program of China (No. 2020YFA0509001), the National Natural Science Foundation of China (No. 31801057), and Shanghai Municipal Commission for Science and Technology Grants (Nos. 21140905100, 22140903500)]
谢甜,硕士,工程师,研究方向:基因组标签计划与基因编辑。E-mail: tian.xie@sibcb.ac.cn
蒋婧,博士,副研究员,研究方向:基因组标签计划与基因编辑。E-mail: jiangjing@sibcb.ac.cn
10.16288/j.yczz.22-158
(责任编委: 郭伟翔)