重症新型冠状病毒肺炎患者遗传易感性研究进展
刘文兵,刘丹,闫进,刘欣,王前飞
重症新型冠状病毒肺炎患者遗传易感性研究进展
刘文兵1,2,刘丹1,闫进1,刘欣1,2,王前飞1,2
1. 中国科学院北京基因组研究所(国家生物信息中心),中国科学院精准基因组医学重点实验室,北京 100101 2. 中国科学院大学,北京 100049
新型冠状病毒肺炎是由新型冠状病毒感染引起的全球大流行疾病。患者呈现出无症状感染、轻症到(危)重症不同严重程度的临床表现。部分重症患者因发生细胞因子风暴而出现多器官功能衰竭并最终导致死亡。除性别、年龄、基础疾病(如高血压、糖尿病)等增加重型感染风险外,宿主先天遗传缺陷也被认为与疾病严重程度(包括细胞因子风暴的发生)密切相关。在重症患者中,相继发现与病毒识别、杀伤等相关的关键基因(如、等)先天遗传变异。本文主要总结了宿主抗病毒免疫应答机制及与新型冠状病毒感染严重程度相关的先天变异基因,以期为新型冠状病毒肺炎的早期干预和分层治疗提供遗传学依据。
重症新型冠状病毒肺炎患者;先天遗传变异;免疫缺陷
新型冠状病毒(severe acute respiratory syndrome coronavirus 2, SARS-CoV-2)是具有外套膜包裹的正链单链RNA病毒,序列长度约30 kb,与SARS病毒基因组序列相似度为80%[1]。SARS-CoV-2的结构蛋白包括刺突蛋白、包膜蛋白、膜蛋白和核衣壳蛋白[2]。刺突蛋白包含S1和S2两个亚基,S1包含受体结合区负责识别宿主细胞膜受体,S2负责病毒与细胞膜融合[3]。SARS-CoV-2通过刺突蛋白感染宿主,引发呼吸系统疾病新型冠状病毒肺炎(coronavirus disease 2019, COVID-19)[4]。截止到2022年4月23日,世界卫生组织报道全球共有505,817,953人被感染,确认死亡人数高达6,213,876人。COVID-19患者临床表现具有明显的异质性。轻症患者表现出发烧、咳嗽、乏力等症状,多数患者预后良好。重症患者可出现呼吸困难及低血氧等临床表现。少数患者可进展为危重症,出现细胞因子风暴导致的急性呼吸窘迫综合征及多器官功能衰竭,其中重症监护室治疗的危重症患者死亡率高达50%[5]。在临床上需要加强对SARS-CoV-2感染人群的监测,提供重症相关的预警指标,预防患者(危)重症化。与重症相关的风险因素除年龄、吸烟、高血压、糖尿病等之外,还有病毒感染者的先天遗传因素。以往严重的流感病毒(如甲型流感病毒)感染者中,发现其存在细胞毒杀伤通路相关基因先天突变,提示先天遗传因素在呼吸道病毒感染中发挥重要作用[6]。COVID-9患者的遗传易感性包括:基础疾病相关基因变异、病毒感染(入侵、复制、释放、传播)及宿主抗病毒免疫应答(固有免疫、适应性免疫)相关基因变异等方面。本文将重点关注病毒感染、宿主抗病毒免疫应答过程中的先天遗传因素,包括病毒入侵相关的受体基因、抗原抗体系统相关的ABO血型、人类白细胞抗原(human leukocyte antigen, HLA)、I型干扰素信号通路相关基因和细胞因子风暴产生相关基因的关键多态性位点和先天罕见变异位点,讨论了病毒入侵和宿主抗病毒免疫应答过程相关的遗传学研究,梳理了重症患者可能存在的潜在先天遗传因素。加强对SARS-CoV-2感染人群的遗传学研究,发现宿主先天遗传因素在该疾病发生、发展中作用,将有助于阐明重症化的分子遗传机制,进而寻找潜在的遗传标志物,为实现对重症高风险人群及早鉴别干预提供遗传学依据。
1 病毒入侵和宿主抗病毒免疫应答过程
SARS-CoV-2的刺突蛋白与宿主细胞的血管紧张素转换酶2 (angiotensin-converting enzyme 2, ACE2)受体结合[7],位于宿主细胞膜表面的跨膜丝氨酸蛋白酶2 (transmembrane serine protease 2, TMPRSS2)会对其进行水解活化[8]。然后病毒通过膜融合进入细胞质,脱壳后释放RNA,完成感染。在细胞质中,病毒利用宿主细胞的核糖体进行翻译。SARS-CoV-2感染细胞后,宿主的固有免疫系统迅速反应,参与抗病毒免疫应答。主要为中性粒细胞通过诱导多种死亡模式杀伤病毒;自然杀伤细胞(natural killer cell, NK)通过释放颗粒酶与穿孔素对病毒感染细胞进行杀伤;巨噬细胞对病毒及其感染细胞进行吞噬[9]。位于细胞膜表面Toll样受体(toll-like receptors, TLRs)或者细胞质RIG-I样受体(retinoic acid-inducible gene I-like receptors, RLRs)等模式识别受体与SARS- CoV-2的RNA结合,通过相应的配体β干扰素TIR结构域衔接蛋白(TIR-domain-containing adaptor inducing interferon-β, TRIF)、髓样分化因子88 (myeloid differentiation primary response protein 88, MyD88)和线粒体抗病毒信号蛋白(mitochondrial antiviral- signaling protein, MAVS)激活下游信号通路,启动NF-kB信号通路调控炎症因子应答,以及活化干扰素调节因子3/7 (interferon regulatory factor 3/7, IRF3/7)促进I型干扰素表达[10]。I型干扰素抑制病毒复制,且能够结合相邻细胞表面的干扰素α/β受体1/2 (interferon alpha/beta receptor 1/2, IFNAR1/2),激活下游JAK-STAT信号通路,促进干扰素刺激基因表达[11]。干扰素刺激基因可抑制病毒复制。
宿主的适应性免疫应答在抗病毒感染中起重要作用。B细胞摄取病毒感染细胞的抗原后,可分化为能够合成和分泌抗体杀伤病毒感染的浆细胞,也可以将抗原呈递给T细胞。T细胞识别抗原呈递细胞加工处理的抗原之后,进行增殖和分化,可分化为细胞毒性T细胞(cytotoxic T lymphocyte, CTL)。CTL释放颗粒酶和穿孔素杀伤病毒感染的细胞,以此清除病毒感染细胞[12]。在宿主抗病毒免疫应答过程中,如果SARS-CoV-2无法及时被清除,会持续刺激免疫细胞(巨噬细胞、NK细胞、T细胞等)释放大量炎性细胞因子(白介素、干扰素、趋化因子等),进而吸引更多的免疫细胞形成反馈循环,产生细胞因子风暴[13]。大量炎性细胞因子聚集于肺泡,并且破坏血管使得体液和血细胞进入肺泡,导致水肿和急性呼吸窘迫综合征,加重患者病情[14]。细胞因子风暴常发生在危重症患者中,例如中国武汉一项COVID-19患者临床特征研究发现危重症患者血浆中存在高表达水平的多种炎性细胞因子[15]。图1汇总了上述病毒的入侵和宿主的抗病毒免疫应答过程,以及涉及到的重要基因。
研究表明COVID-19的易感性和严重程度会受到宿主先天遗传因素的影响。解析重症COVID-19患者的遗传基础可对其发病遗传机制和精准治疗提供新思路。筛选潜在的重症感染遗传标志物,这将有助于疾病的及早发现和进行风险分层。研究者发现COVID-19与多个基因上的突变/多态性位点相关联,例如病毒入侵相关的基因、ABO血型、HLA、I型干扰素信号通路和细胞因子风暴产生相关基因(表1)。
2 病毒入侵相关基因ACE2和TMPRSS2的遗传变异研究
SARS-CoV-2感染宿主需要经过两个步骤,首先病毒的刺突蛋白受体结合区与ACE2氨基末端催化结构域结合,随后TMPRSS2水解活化病毒刺突蛋白诱导其与宿主细胞膜融合[8]。ACE2是细胞表面的功能性受体,在心脏、肾脏和肺等组织中高表达,这些靶器官都可能成为病毒感染和疾病进展的场所[16]。已有研究表明基因的多态性和高血压的发生相关,重症COVID-19患者常伴有高血压等基础疾病,人群中基因多态性可能与COVID-19易感性和严重程度有一定的关联性[17]。Horowitz等[18]通过整合4个欧美人群的基因组数据,利用2,630个COVID-19患者和704,016健康人数据进行全基因组关联分析,发现COVID-19人群携带基因的rs190509934多态性位点比例较低(=4.5×10−13)。在COVID-19患者中,携带rs190509934的患者重症风险较低(风险比为0.69)[18]。对2035例样本的肝组织转录组数据分析发现基因的rs190509934多态性位点显著降低的表达,可提供对SARS- CoV-2感染的保护,并且与疾病严重程度相关[18]。Benetti等[19]整合5个中心的6,930名对照意大利人的外显子测序数据,发现基因的rs41303171多态性位点出现在欧洲人群(频率为0.011),几乎不出现在东亚人群。通过模拟分析预测rs41303171多态性位点影响ACE2蛋白的结构和稳定性,造成TMPRSS2无法水解活化病毒刺突蛋白和ACE2结合体,影响病毒进入细胞的过程[19]。上述基因的多态性位点影响表达或蛋白结构,可能影响病毒入侵过程,对病毒感染风险和疾病的严重程度有一定贡献。基因突变在COVID-19的发生过程中同样起到重要作用。Andolfo等[20]对欧洲6,406个COVID-19患者和902,088健康人数据进行全基因组关联分析,发现COVID-19人群携带基因的rs12329760多态性位点比例较低(=3.13×10−6)。基因的rs12329760多态性位点位于编码区。该位点可能降低TMPRSS2蛋白质稳定性,影响与ACE2的结合,进而抑制病毒的入侵,是保护性位点[20]。Wang等[21]对332例无症状和重症COVID- 19患者进行全外显子测序,发现基因的rs12329760多态性位点在重症患者中频率较低,提示该位点可能影响COVID-19患者疾病的严重程度。基因的rs12329760多态性位点在东亚人(频率为0.384)和欧洲人(频率为0.232)频率的差异,可能与意大利患者发病率和死亡率较高相关[22]。由此可见,和基因的多态性位点在不同种族发生频率具有差异,这提示不同种族人群感染SARS-CoV-2的遗传多态性位点存在区别。
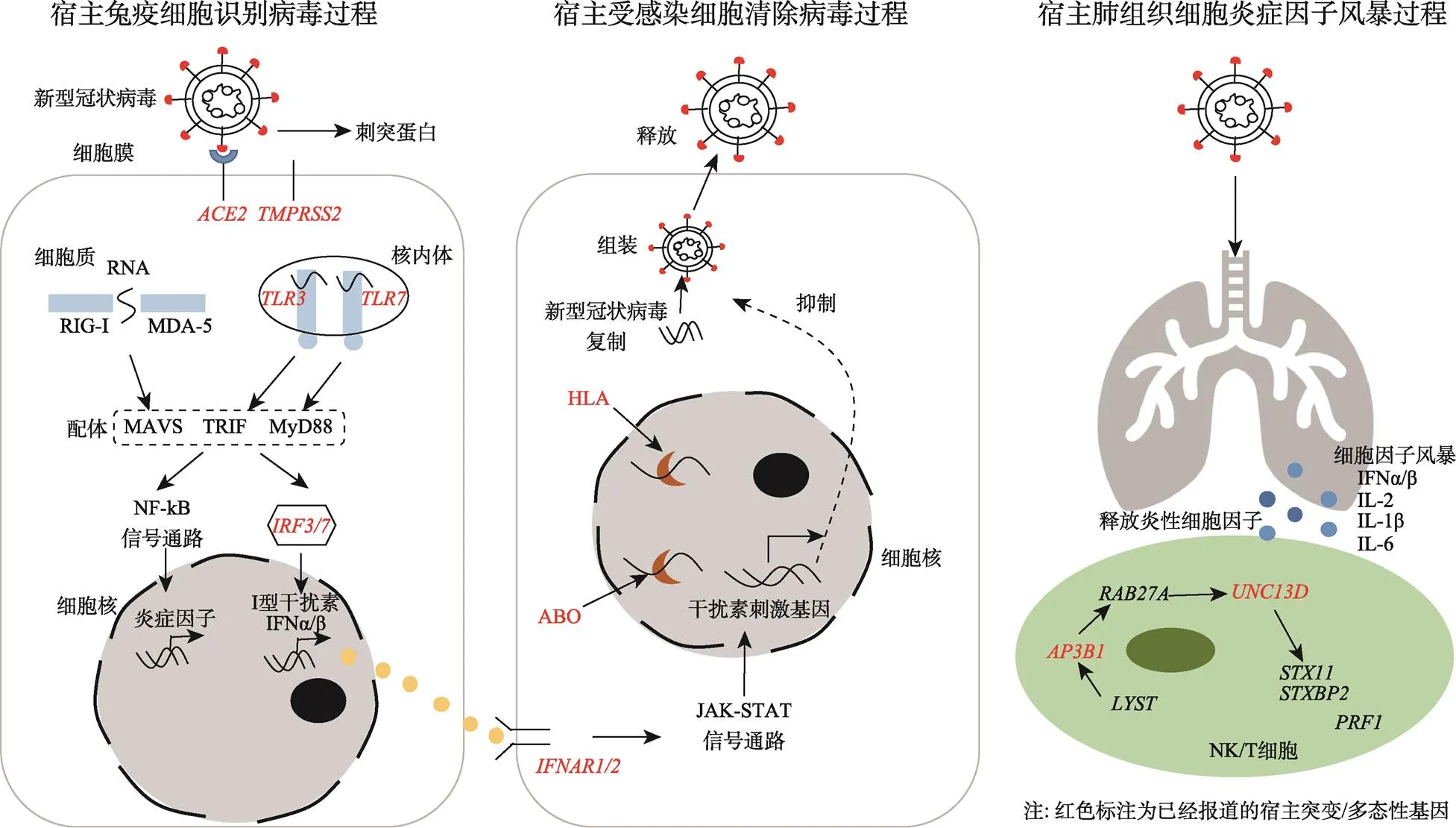
图1 宿主抗新型冠状病毒免疫应答机制
SARS-CoV-2通过刺突蛋白与宿主的ACE2受体结合,经宿主的TMPRSS2水解活化后,进入宿主细胞释放RNA。宿主通过模式识别受体识别病毒RNA,通过相应的配体激活下游信号通路,促进炎症因子和干扰素的表达,触发抗病毒免疫应答。I型干扰素可作用于具有相应受体的相邻细胞,并且激活下游的JAK-STAT信号通路,促进干扰素刺激基因表达。干扰素刺激基因可抑制病毒复制、组装等过程。在宿主抗病毒免疫过程中,NK或T细胞可通过细胞毒杀伤过程消灭病毒感染的细胞。如果上述过程出现缺陷,病毒无法及时被清除,会持续刺激免疫细胞。聚集的免疫细胞释放大量炎性细胞因子,产生细胞因子风暴破坏肺组织。根据参考文献[49]修改绘制。
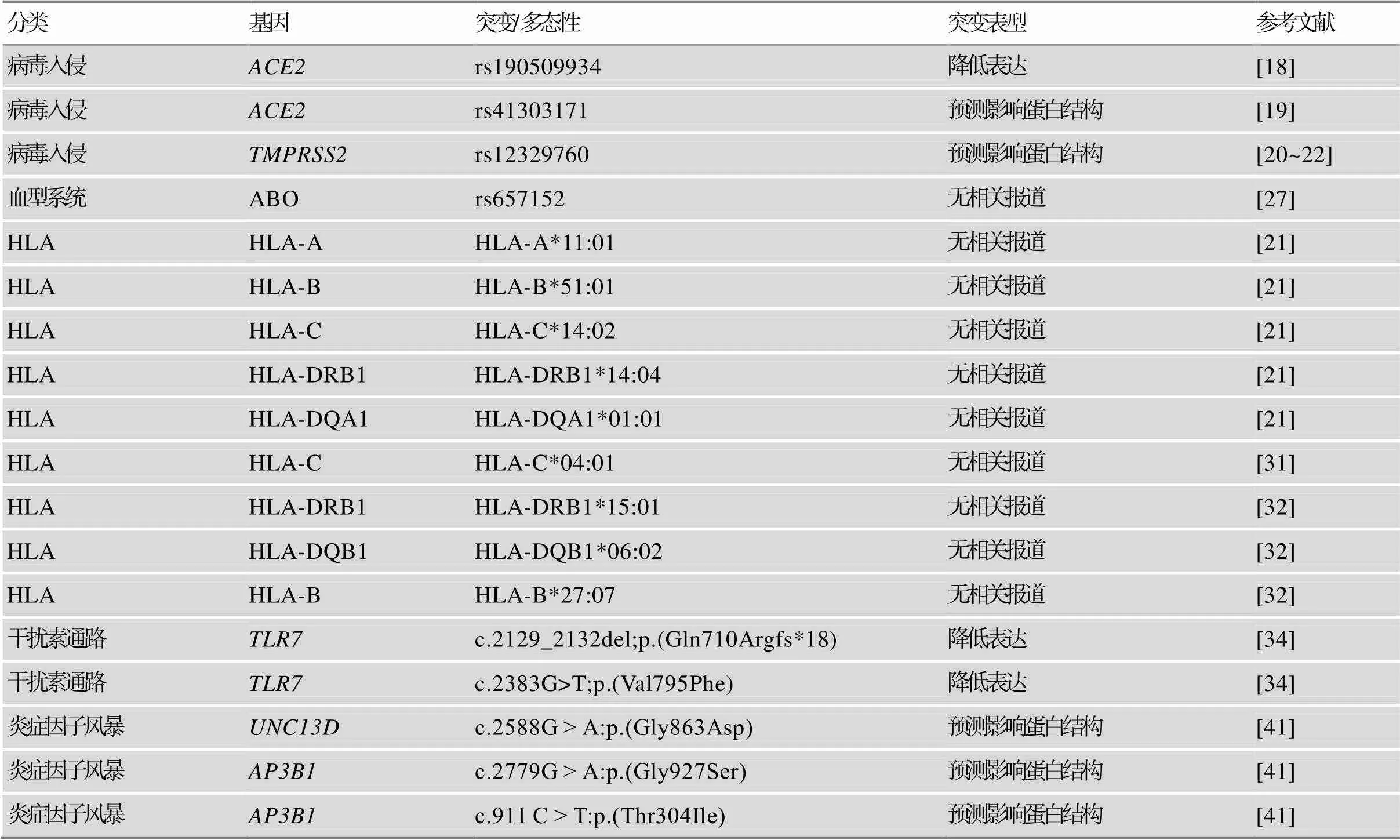
表1 重症COVID-19相关基因汇总
3 ABO血型相关的遗传变异研究
A、B、O、AB四种遗传表型是根据红细胞表面抗原的低聚糖结构决定,A型血的寡糖序列末端残基是N-乙酰半乳糖胺,B型血是半乳糖,O型血缺少这两种残基,AB型血两种残基都存在[23]。在血液中天然存在ABO抗原的对应抗体:A血型血清中存在抗B抗体;B血型血清中存在抗A抗体;O血型血清中存在抗A和抗B抗体;AB血型不存在相应的抗体。血型天然抗体主要是IgM (Immunoglobulin M),IgM也是抗病毒免疫应答中最早出现的抗体,当前研究显示感染性疾病与血型之间存在关联[24]。COVID-19发生也可能与ABO血型有一定的关联性[25]。Zhao等[26]统计中国武汉市3694健康对照和1775名COVID-19患者的ABO血型人群占比,发现COVID-19患者中A型血占比显著高于正常人,O型血占比显著低于对照人群,可能与血型天然抗体有关,提示不同ABO血型人群感染SARS-CoV-2的风险存在差异。一项研究对意大利和西班牙1980例重症COVID-19患者和2381名对照个人进行全基因组关联分析,发现9q34.2位点rs657152与重症感染显著相关(<5×10−8)[27]。基因座9q34.2处共有38个显著相关的变异位点,与ABO血型相关的位点重合。矫正性别和年龄发现A型血的人群重症风险高(风险比为1.45),O型血的人群重症风险低(风险比为0.65)[27]。抗A抗体可以干扰病毒与宿主细胞的粘附,从而阻止病毒刺突蛋白与细胞表面ACE2之间的相互作用,提示A血型人群可能更易感染病毒[28]。A抗原可引起更多的P-选择素和粘附分子附着于内皮细胞增加心血管疾病的风险[29]。上述研究表明A型血人群SARS-CoV-2感染及重症风险较高,O型血相对风险较低,ABO血型可能作为COVID-19患者易感性和严重程度的标志物。
4 白细胞抗原HLA相关的遗传变异研究
HLA在抗病毒免疫的过程中起着重要的作用,主要分为内源性抗原的递呈分子HLA-I (HLA-A、HLA-B和HLA-C)和外源性抗原的递呈分子HLA-II (HLA-DP、HLA-DQ和HLA-DR)[30]。HLA相关基因的多态性与SARS-CoV-2感染和严重程度之间的关联在多项研究中被报道。Wang等[21]对比了69例重症和215例轻症COVID-19中国患者的HLA等位基因频率,发现I类分子HLA-A*11:01(=0.008512)、HLA-B*51:01 (=0.007017)和HLA-C*14:02 (= 0.003028)显著富集于重症患者,也发现II类分子HLA-DRB1*14:04 (=0.01027)和HLA-DQA1*01:01 (=0.03947)显著富集于重症患者,提示这些位点是重症COVID-19患者的风险位点。其中HLA-B*51:01与自身免疫性疾病的发生相关,这提示该位点相关的宿主免疫应答异常可能是导致患者不良临床结局的原因[21]。Weiner等[31]整合了4个欧美国家的435个患者的基因组数据进行荟萃分析,发现HLA-C*04:01 (=0.0074)为重症COVID-19患者的风险位点,并且进行了独立验证。携带HLA-C*04:01的患者HLA结合亲和力较低,导致患者对病毒免疫应答能力下降,这可能是携带HLA-C*04:01的患者严重临床结局的潜在生物学解释[31]。Novelli等[32]通过对比99例意大利患者和1017健康人群HLA的多态性发现HLA-DRB1*15:01 (=0.0015)、HLA-DQB1*06:02 (=0.0001)和HLA-B*27:07 (=0.00001)显著性富集于COVID-19患者中,提示HLA的多态性位点和SARS-CoV-2感染相关。HLA等位基因的频率可能是COVID-19严重程度的潜在标志物。值得注意的是,不同人群观察到HLA等位基因频率不同,仍需要大规模的人群验证才能针对不同的人群进行精准治疗,相应的人群设计相应的SARS-CoV-2疫苗,有利于增强人群抵抗力。
5 I型干扰素抗病毒应答相关基因遗传变异研究
I型干扰素通路在宿主抗SARS-CoV-2免疫应答中起重要作用。SARS-CoV-2感染后,宿主免疫细胞通过模式识别受体识别SARS-CoV-2响应病毒感染,促进I型干扰素的表达,进而启动JAK-STAT信号通路诱导干扰素刺激基因的表达,实现抗病毒的功能[33]。参与病毒识别功能的基因包括、、等,调节干扰素生成相关基因包括和等,参与抗病毒功能的基因包括、和JAK-STAT信号通路相关基因,这些基因的先天突变可能影响重症COVID-19患者抗病毒免疫应答。一项COVID-19患者家系研究发现两个重症COVID-19年轻男性患者中分别携带基因的先天移码突变(c.2129_2132del;p.[Gln710Argfs*18])和错义突变(c.2383G>T;p.[Val795Phe])[34]。携带上述基因突变的重症患者,其外周血单核细胞中基因表达低。对突变的重症患者和非SARS-CoV-2感染者的外周血单核细胞进行对比分析,发现在TLR7激活剂处理下,重症患者的I型干扰素相关基因、未发生显著上调[34]。这提示发生失活突变后,未能启动I型干扰素反应。失活突变与干扰素应答功能受损相关,这为理解重症新冠病毒患者的致病机制提供新视角。Zhang等[35]通过对比659例重症COVID-19患者和534名轻症或无症状患者基因组先天突变,发现3.5%重症COVID-19患者存在I型干扰素途径相关基因(等)先天突变,同时在等I型干扰素途径相关基因突变的细胞系中发现等基因表达较低,且产生干扰素水平较低,更容易感染病毒。与轻症或无症状患者相比,重症COVID-19患者存在多种I型干扰素途径相关基因缺陷,这提示I型干扰素异常与重症疾病发生相关。当前基于SARS-CoV-2重症感染家系研究以及疾病–对照研究中发现I型干扰素抗病毒应答的罕见突变,这些基因先天突变与疾病的严重程度具有密切关系,将为临床风险分层提供遗传学依据。
6 炎症因子风暴相关基因遗传变异研究
重症COVID-19患者促炎性细胞因子IL-6、TNF-α等显著升高,呈现出细胞因子风暴的特征[36]。目前细胞因子风暴被认为是急性呼吸窘迫综合征和多器官衰竭的主要原因之一[37],有效抑制细胞因子风暴是防止COVID-19患者病情恶化和挽救患者生命的重要途径。COVID-19患者和噬血细胞综合征(hemophagocytic lymphohistiocytosis, HLH)患者的临床特征、免疫学和组织病理学数据有相似之处,存在共同的炎症分子机制[38]。已有研究表明EB病毒感染是HLH发生的关键因素,原发性免疫相关基因缺陷可能会引发EB病毒相关的噬血细胞综合征[39]。携带HLH易感基因缺陷的患者在SARS-CoV-2感染下也有可能触发炎症因子风暴,包括、、、、、等基因的先天突变[40]。Luo等[41]通过对233例不同临床表现的COVID-19患者进行全外显子测序,发现高细胞因子水平患者显著富集HLH相关基因和上的先天突变。携带和基因突变的患者重症率和死亡率较高[41]。上述突变均位于基因的保守结构域,被预测可能损害蛋白的结构,进而影响基因功能。这两个基因功能异常导致患者细胞毒杀伤功能缺陷,无法有效清除病毒,引起宿主免疫系统过度激活。诸如HLH相关基因类似的免疫缺陷基因的先天突变,可能是重症COVID-19患者产生炎症因子风暴的关键遗传因素。这提示尽早检测感染者是否存在免疫缺陷对重症COVID-19早期诊断和有效治疗具有重要的指导意义。
7 结语与展望
先天遗传因素在疾病发生发展中的作用值得被关注。原发性免疫缺陷人群对病毒抵抗力差并更易引发严重的病毒感染相关疾病。在重症COVID-19人群中同样发现干扰素信号通路、NK/T细胞毒杀伤功能相关基因发生先天变异,并且细胞毒杀伤功能基因异常显著出现于细胞因子表达水平高的重症感染者中。上述研究均提示抗病毒免疫功能先天缺陷/失调增加个体成为重症/危重症感染者的风险。关注重症感染者先天遗传基础的研究日益增加,这将对未来防治危重症新冠感染提供遗传学依据。目前,已筛选出的候选基因/位点尚未开展深入的分子机制探索和功能性验证;如对候选基因的精细作用机理进一步探索,将有助于理解免疫系统在抗冠状病毒感染的关键节点并提供潜在的分子治疗靶点。同时,仍需要基于独立人群的大样本验证已知的候选基因,进一步明确关键的候选变异基因/位点[42]。各项研究所鉴定的与疾病关联性关键基因涉及到SARS-CoV-2入侵相关基因和、ABO血型、白细胞抗原HLA、干扰素应答相关基因TLRs、细胞毒杀伤通路基因等。如何整合上述突变基因信息、并结合已有其他SARS-CoV-2感染人群单细胞图谱[43],逐步构建系统性和综合性的SARS-CoV-2感染相关基因知识库,仍是值得研究者思考的科学问题。综合利用上述信息,将有助于应对SARS-CoV-2及日后可能出现的其他冠状病毒流行性疾病。
基于现有研究,病毒入侵相关基因多态性位点发生频率呈现出种族差异性[22]。这提示遗传学研究以及鉴定关键候选基因,需考虑人种差异。出现SARS-CoV-2无症状感染的免疫机理、无症状感染人群是否存在特异性的免疫基因先天变异同样是值得进一步探讨和研究的方向。
SARS-CoV-2的变异毒株Delta和Omicron相继出现,病毒的传播效率越来越高,这种演化路径在流感病毒中很常见;在日后,SARS-CoV-2有可能成为一种类似于流感病毒的地方性流行病毒[44,45]。与Delta相比,Omicron变异毒株感染后,无症状或轻症感染者比例增高[46,47],但是仍有21%的Omicron变异毒株感染导致的住院患者存在严重的临床结局[48]。这提示早发现SARS-CoV-2重症感染者仍是公共卫生领域值得重视的问题之一。探索与COVID-19严重程度相关的先天遗传因素十分重要,鉴别高风险人群有助于降低住院率和重症死亡率,为国家医疗事业减轻负担。
综上所述,遗传学是SARS-CoV-2以及其他冠状病毒感染中不容忽视的研究方向。个体免疫系统先天遗传差异将影响病毒感染及疾病发展程度。综合现有临床指标并结合遗传学指征,将为未来实现早期甄别高危感染人群、及早干预、精准分层治疗提供新的思路。
[1] Lu RJ, Zhao X, Li J, Niu PH, Yang B, Wu HL, Wang WL, Song H, Huang BY, Zhu N, Bi YH, Ma XJ, Zhan FX, Wang L, Hu T, Zhou H, Hu ZH, Zhou WM, Zhao L, Chen J, Meng Y, Wang J, Lin Y, Yuan JY, Xie ZH, Ma JM, Liu WJ, Wang DY, Xu WB, Holmes EC, Gao GF, Wu GZ, Chen WJ, Shi WF, Tan WJ. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding., 2020, 395(10224): 565–574.
[2] Shamsi A, Mohammad T, Anwar S, Amani S, Khan MS, Husain FM, Rehman MT, Islam A, Hassan MI. Potential drug targets of SARS-CoV-2: from genomics to therapeutics., 2021, 177: 1–9.
[3] Xia XH. Domains and functions of spike protein in SRAS-COV-2 in the context of vaccine design., 2021, 13(1): 109.
[4] Guan WJ, Ni ZY, Hu Y, Liang WH, Ou CQ, He JX, Liu L, Shan H, Lei CL, Hui DSC, Du B, Li LJ, Zeng G, Yuen KY, Chen RC, Tang CL, Wang T, Chen PY, Xiang J, Li SY, Wang JL, Liang ZJ, Peng YX, Wei L, Liu Y, Hu YH, Peng P, Wang JM, Liu JY, Chen Z, Li G, Zheng ZJ, Qiu SQ, Luo J, Ye CJ, Zhu SY, Zhong NS, China Medical Treatment Expert Group for COVID-19. Clinical characteristics of coronavirus disease 2019 in China., 2020, 382(18): 1708–1720.
[5] Wang DW, Hu B, Hu C, Zhu FF, Liu X, Zhang J, Wang BB, Xiang H, Cheng ZS, Xiong Y, Zhao Y, Li YR, Wang XH, Peng ZY. Clinical characteristics of 138 hospitalized patients with 2019 novel coronavirus-infected pneumonia in Wuhan, China., 2020, 323(11): 1061–1069.
[6] Jouanguy E, Béziat V, Mogensen TH, Casanova JL, Tangye SG, Zhang SY. Human inborn errors of immunity to herpes viruses., 2020, 62: 106–122.
[7] Samavati L, Uhal BD. ACE2, much more than just a receptor for SARS-COV-2., 2020, 10: 317.
[8] Hoffmann M, Kleine-Weber H, Schroeder S, Krüger N, Herrler T, Erichsen S, Schiergens TS, Herrler G, Wu NH, Nitsche A, Müller MA, Drosten C, Pöhlmann S. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor., 2020, 181(2): 271–280.e8.
[9] Cavalcante-Silva LHA, Carvalho DCM, Lima ÉA, Galvão JGFM, da Silva JSF, Sales-Neto JM, Rodrigues-Mascarenhas S. Neutrophils and COVID-19: the road so far., 2021, 90: 107233.
[10] Prompetchara E, Ketloy C, Palaga T. Immune responses in COVID-19 and potential vaccines: lessons learned from SARS and MERS epidemic., 2020, 38(1): 1–9.
[11] Solimani F, Meier K, Ghoreschi K. Janus kinase signaling as risk factor and therapeutic target for severe SARS- CoV-2 infection., 2021, 51(5): 1071–1075.
[12] Cox RJ, Brokstad KA. Not just antibodies: B cells and T cells mediate immunity to COVID-19., 2020, 20(10): 581–582.
[13] Ragab D, Salah Eldin H, Taeimah M, Khattab R, Salem R. The COVID-19 cytokine storm; what we know so far., 2020, 11: 1446.
[14] Channappanavar R, Perlman S. Pathogenic human coronavirus infections: causes and consequences of cytokine storm and immunopathology., 2017, 39(5): 529–539.
[15] Huang CL, Wang YM, Li XW, Ren LL, Zhao JP, Hu Y, Zhang L, Fan GH, Xu JY, Gu XY, Cheng ZS, Yu T, Xia JA, Wei Y, Wu WJ, Xie XL, Yin W, Li H, Liu M, Xiao Y, Gao H, Guo L, Xie JG, Wang GF, Jiang RM, Gao ZC, Jin Q, Wang JW, Cao B. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China., 2020, 395(10223): 497–506.
[16] Beyerstedt S, Casaro EB, Rangel ÉB. COVID-19: angiotensin-converting enzyme 2 (ACE2) expression and tissue susceptibility to SARS-CoV-2 infection., 2021, 40(5): 905–919.
[17] Devaux CA, Rolain JM, Raoult D. ACE2 receptor polymorphism: Susceptibility to SARS-CoV-2, hypertension, multi-organ failure, and COVID-19 disease outcome., 2020, 53(3): 425–435.
[18] Horowitz JE, Kosmicki JA, Damask A, Sharma D, Roberts GHL, Justice AE, Banerjee N, Coignet MV, Yadav A, Leader JB, Marcketta A, Park DS, Lanche R, Maxwell E, Knight SC, Bai X, Guturu H, Sun D, Baltzell A, Kury FSP, Backman JD, Girshick AR, O'Dushlaine C, McCurdy SR, Partha R, Mansfield AJ, Turissini DA, Li AH, Zhang M, Mbatchou J, Watanabe K, Gurski L, McCarthy SE, Kang HM, Dobbyn L, Stahl E, Verma A, Sirugo G, Regeneron Genetics C, Ritchie MD, Jones M, Balasubramanian S, Siminovitch K, Salerno WJ, Shuldiner AR, Rader DJ, Mirshahi T, Locke AE, Marchini J, Overton JD, Carey DJ, Habegger L, Cantor MN, Rand KA, Hong EL, Reid JG, Ball CA, Baras A, Abecasis GR, Ferreira MA. Genome- wide analysis in 756,646 individuals provides first genetic evidence that ACE2 expression influences COVID-19 risk and yields genetic risk scores predictive of severe disease., 2021.
[19] Benetti E, Tita R, Spiga O, Ciolfi A, Birolo G, Bruselles A, Doddato G, Giliberti A, Marconi C, Musacchia F, Pippucci T, Torella A, Trezza A, Valentino F, Baldassarri M, Brusco A, Asselta R, Bruttini M, Furini S, Seri M, Nigro V, Matullo G, Tartaglia M, Mari F, Study G-CM, Renieri A, Pinto AM. ACE2 gene variants may underlie interindividual variability and susceptibility to COVID-19 in the Italian population., 2020, 28(11): 1602–1614.
[20] Andolfo I, Russo R, Lasorsa VA, Cantalupo S, Rosato BE, Bonfiglio F, Frisso G, Abete P, Cassese GM, Servillo G, Esposito G, Gentile I, Piscopo C, Villani R, Fiorentino G, Cerino P, Buonerba C, Pierri B, Zollo M, Iolascon A, Capasso M. Common variants at 21q22.3 locus influence MX1 and TMPRSS2 gene expression and susceptibility to severe COVID-19., 2021, 24(4): 102322.
[21] Wang F, Huang SJ, Gao RS, Zhou YW, Lai CX, Li ZC, Xian WJ, Qian XB, Li ZY, Huang YS, Tang QY, Liu PH, Chen RK, Liu R, Li X, Tong X, Zhou X, Bai Y, Duan G, Zhang T, Xu X, Wang J, Yang HM, Liu SY, He Q, Jin X, Liu L. Initial whole-genome sequencing and analysis of the host genetic contribution to COVID-19 severity and susceptibility., 2020, 6(1): 83.
[22] Asselta R, Paraboschi EM, Mantovani A, Duga S. ACE2 and TMPRSS2 variants and expression as candidates to sex and country differences in COVID-19 severity in Italy., 2020, 12(11): 10087–10098.
[23] Dean L. ABO blood group. In: Pratt VM, Scott SA, Pirmohamed M, Esquivel B, Kane MS, Kattman BL, Malheiro AJ, editors. Medical Genetics Summaries. Bethesda (MD): National Center for Biotechnology Information (US), 2012.
[24] Ewald DR, Sumner SC. Blood type biochemistry and human disease., 2016, 8(6): 517–535.
[25] Liu NY, Zhang TT, Ma LN, Zhang HQ, Wang HC, Wei W, Pei H, Li H. The impact of ABO blood group on COVID-19 infection risk and mortality: a systematic review and meta-analysis., 2021, 48: 100785.
[26] Zhao J, Yang Y, Huang H, Li D, Gu DF, Lu XF, Zhang Z, Liu L, Liu T, Liu YK, He YJ, Sun B, Wei ML, Yang GY, Wang XH, Zhang L, Zhou XY, Xing MZ, Wang PG. Relationship between the ABO blood group and the coronavirus disease 2019 (COVID-19) susceptibility., 2021, 73(2): 328–331.
[27] Severe Covid GG, Ellinghaus D, Degenhardt F, Bujanda L, Buti M, Albillos A, Invernizzi P, Fernández J, Prati D, Baselli G, Asselta R, Grimsrud MM, Milani C, Aziz F, Kässens J, May S, Wendorff M, Wienbrandt L, Uellendahl- Werth F, Zheng TH, Yi XL, de Pablo R, Chercoles AG, Palom A, Garcia-Fernandez AE, Rodriguez-Frias F, Zanella A, Bandera A, Protti A, Aghemo A, Lleo A, Biondi A, Caballero-Garralda A, Gori A, Tanck A, Carreras Nolla A, Latiano A, Fracanzani AL, Peschuck A, Julià A, Pesenti A, Voza A, Jiménez D, Mateos B, Nafria Jimenez B, Quereda C, Paccapelo C, Gassner C, Angelini C, Cea C, Solier A, Pestaña D, Muñiz-Diaz E, Sandoval E, Paraboschi EM, Navas E, García Sánchez F, Ceriotti F, Martinelli- Boneschi F, Peyvandi F, Blasi F, Téllez L, Blanco-Grau A, Hemmrich-Stanisak G, Grasselli G, Costantino G, Cardamone G, Foti G, Aneli S, Kurihara H, ElAbd H, My I, Galván-Femenia I, Martín J, Erdmann J, Ferrusquía- Acosta J, Garcia-Etxebarria K, Izquierdo-Sanchez L, Bettini LR, Sumoy L, Terranova L, Moreira L, Santoro L, Scudeller L, Mesonero F, Roade L, Rühlemann MC, Schaefer M, Carrabba M, Riveiro-Barciela M, Figuera Basso ME, Valsecchi MG, Hernandez-Tejero M, Acosta- Herrera M, D'Angiò M, Baldini M, Cazzaniga M, Schulzky M, Cecconi M, Wittig M, Ciccarelli M, Rodríguez-Gandía M, Bocciolone M, Miozzo M, Montano N, Braun N, Sacchi N, Martínez N, Özer O, Palmieri O, Faverio P, Preatoni P, Bonfanti P, Omodei P, Tentorio P, Castro P, Rodrigues PM, Blandino Ortiz A, de Cid R, Ferrer R, Gualtierotti R, Nieto R, Goerg S, Badalamenti S, Marsal S, Matullo G, Pelusi S, Juzenas S, Aliberti S, Monzani V, Moreno V, Wesse T, Lenz TL, Pumarola T, Rimoldi V, Bosari S, Albrecht W, Peter W, Romero-Gómez M, D'Amato M, Duga S, Banales JM, Hov JR, Folseraas T, Valenti L, Franke A, Karlsen TH. Genomewide association study of severe covid-19 with respiratory failure., 2020, 383(16): 1522–1534.
[28] Shibeeb S, Khan A. ABO blood group association and COVID-19. COVID-19 susceptibility and severity: a review., 2022, 44(1): 70–75.
[29] Dai XF. ABO blood group predisposes to COVID-19 severity and cardiovascular diseases., 2020, 27(13): 1436–1437.
[30] Dendrou CA, Petersen J, Rossjohn J, Fugger L. HLA variation and disease., 2018, 18(5): 325–339.
[31] Weiner J, Suwalski P, Holtgrewe M, Rakitko A, Thibeault C, Müller M, Patriki D, Quedenau C, Krüger U, Ilinsky V, Popov I, Balnis J, Jaitovich A, Helbig ET, Lippert LJ, Stubbemann P, Real LM, Macías J, Pineda JA, Fernandez- Fuertes M, Wang XM, Karadeniz Z, Saccomanno J, Doehn JM, Hübner RH, Hinzmann B, Salvo M, Blueher A, Siemann S, Jurisic S, Beer JH, Rutishauser J, Wiggli B, Schmid H, Danninger K, Binder R, Corman VM, Mühlemann B, Arjun Arkal R, Fragiadakis GK, Mick E, Comet C, Calfee CS, Erle DJ, Hendrickson CM, Kangelaris KN, Krummel MF, Woodruff PG, Langelier CR, Venkataramani U, García F, Zyla J, Drosten C, Alice B, Jones TC, Suttorp N, Witzenrath M, Hippenstiel S, Zemojtel T, Skurk C, Poller W, Borodina T, Pa-Covid SG, Ripke S, Sander LE, Beule D, Landmesser U, Guettouche T, Kurth F, Heidecker B. Increased risk of severe clinical course of COVID-19 in carriers of HLA-C*04:01., 2021, 40: 101099.
[32] Novelli A, Andreani M, Biancolella M, Liberatoscioli L, Passarelli C, Colona VL, Rogliani P, Leonardis F, Campana A, Carsetti R, Andreoni M, Bernardini S, Novelli G, Locatelli F. HLA allele frequencies and susceptibility to COVID-19 in a group of 99 Italian patients., 2020, 96(5): 610–614.
[33] Sa Ribero M, Jouvenet N, Dreux M, Nisole S. Interplay between SARS-CoV-2 and the type I interferon response., 2020, 16(7): e1008737.
[34] van der Made CI, Simons A, Schuurs-Hoeijmakers J, van den Heuvel G, Mantere T, Kersten S, van Deuren RC, Steehouwer M, van Reijmersdal SV, Jaeger M, Hofste T, Astuti G, Corominas Galbany J, van der Schoot V, van der Hoeven H, Hagmolen Of Ten Have W, Klijn E, van den Meer C, Fiddelaers J, de Mast Q, Bleeker-Rovers CP, Joosten LAB, Yntema HG, Gilissen C, Nelen M, van der Meer JWM, Brunner HG, Netea MG, van de Veerdonk FL, Hoischen A. Presence of genetic variants among young men with severe COVID-19., 2020, 324(7): 663– 673.
[35] Zhang Q, Bastard P, Liu ZY, Le Pen J, Moncada-Velez M, Chen J, Ogishi M, Sabli IKD, Hodeib S, Korol C, Rosain J, Bilguvar K, Ye JQ, Bolze A, Bigio B, Yang R, Arias AA, Zhou QH, Zhang Y, Onodi F, Korniotis S, Karpf L, Philippot Q, Chbihi M, Bonnet-Madin L, Dorgham K, Smith N, Schneider WM, Razooky BS, Hoffmann HH, Michailidis E, Moens L, Han JE, Lorenzo L, Bizien L, Meade P, Neehus AL, Ugurbil AC, Corneau A, Kerner G, Zhang P, Rapaport F, Seeleuthner Y, Manry J, Masson C, Schmitt Y, Schlüter A, Le Voyer T, Khan T, Li J, Fellay J, Roussel L, Shahrooei M, Alosaimi MF, Mansouri D, Al-Saud H, Al-Mulla F, Almourfi F, Al-Muhsen SZ, Alsohime F, Al Turki S, Hasanato R, van de Beek D, Biondi A, Bettini LR, D'Angio M, Bonfanti P, Imberti L, Sottini A, Paghera S, Quiros-Roldan E, Rossi C, Oler AJ, Tompkins MF, Alba C, Vandernoot I, Goffard JC, Smits G, Migeotte I, Haerynck F, Soler-Palacin P, Martin-Nalda A, Colobran R, Morange PE, Keles S, Çölkesen F, Ozcelik T, Yasar KK, Senoglu S, Karabela ŞN, Rodríguez-Gallego C, Novelli G, Hraiech S, Tandjaoui-Lambiotte Y, Duval X, Laouénan C, COVID-STORM Clinicians, COVID Clinicians, Imagine COVID Group, French COVID Cohort Study Group, CoV-Contact Cohort, Amsterdam UMC Covid-19 Biobank, COVID Human Genetic Effort, NIAID-USUHS/ TAGC COVID Immunity Group, Snow AL, Dalgard CL, Milner JD, Vinh DC, Mogensen TH, Marr N, Spaan AN, Boisson B, Boisson-Dupuis S, Bustamante J, Puel A, Ciancanelli MJ, Meyts I, Maniatis T, Soumelis V, Amara A, Nussenzweig M, García-Sastre A, Krammer F, Pujol A, Duffy D, Lifton RP, Zhang SY, Gorochov G, Béziat V, Jouanguy E, Sancho-Shimizu V, Rice CM, Abel L, Notarangelo LD, Cobat A, Su HC, Casanova JL. Inborn errors of type I IFN immunity in patients with life-threatening COVID-19., 2020, 370(6515): eabd4570.
[36] Hu BY, Huang SY, Yin LH. The cytokine storm and COVID-19., 2021, 93(1): 250–256.
[37] Chousterman BG, Swirski FK, Weber GF. Cytokine storm and sepsis disease pathogenesis., 2017, 39(5): 517–528.
[38] Ye Q, Wang BL, Mao JH. The pathogenesis and treatment of the `cytokine storm' in COVID-19., 2020, 80(6): 607–613.
[39] Bousfiha A, Jeddane L, Picard C, Ailal F, Bobby Gaspar H, Al-Herz W, Chatila T, Crow YJ, Cunningham-Rundles C, Etzioni A, Franco JL, Holland SM, Klein C, Morio T, Ochs HD, Oksenhendler E, Puck J, Tang MLK, Tangye SG, Torgerson TR, Casanova JL, Sullivan KE. The 2017 IUIS phenotypic classification for primary immunodeficiencies., 2018, 38(1): 129–143.
[40] Canna SW, Marsh RA. Pediatric hemophagocytic lymphohistiocytosis., 2020, 135(16): 1332–1343.
[41] Luo H, Liu D, Liu WB, Wang GX, Chen LT, Cao Y, Wei J, Xiao M, Liu X, Huang G, Wang W, Zhou JF, Wang QF. Germline variants in UNC13D and AP3B1 are enriched in COVID-19 patients experiencing severe cytokine storms., 2021, 29(8): 1312–1315.
[42] Cotsapas C, Saarela J, Farmer JR, Scaria V, Abraham RS. Do monogenic inborn errors of immunity cause susceptibility to severe COVID-19?, 2021, 131(14): e149459.
[43] Ren XW, Wen W, Fan XY, Hou WH, Su B, Cai PF, Li JS, Liu Y, Tang F, Zhang F, Yang Y, He JP, Ma WJ, He JJ, Wang PP, Cao QQ, Chen FJ, Chen YQ, Cheng XL, Deng GH, Deng XL, Ding WY, Feng YM, Gan R, Guo C, Guo WQ, He S, Jiang C, Liang JR, Li YM, Lin J, Ling Y, Liu HF, Liu JW, Liu NP, Liu SQ, Luo M, Ma Q, Song QB, Sun WJN, Wang GX, Wang F, Wang Y, Wen XF, Wu Q, Xu G, Xie XW, Xiong XX, Xing XD, Xu H, Yin CH, Yu DD, Yu KZ, Yuan J, Zhang B, Zhang PP, Zhang T, Zhao JC, Zhao PD, Zhou JF, Zhou W, Zhong SJ, Zhong XS, Zhang SY, Zhu L, Zhu P, Zou B, Zou JH, Zuo ZT, Bai F, Huang X, Zhou PH, Jiang QH, Huang ZW, Bei JX, Wei L, Bian XW, Liu XD, Cheng T, Li XP, Zhao PS, Wang FS, Wang HY, Su B, Zhang Z, Qu K, Wang XQ, Chen JK, Jin RH, Zhang ZM. COVID-19 immune features revealed by a large-scale single-cell transcriptome atlas., 2021, 184(7): 1895– 913.e19.
[44] Callaway E. Beyond Omicron: what’s next for COVID’s viral evolution., 2021, 600(7888): 204–207.
[45] Phillips N. The coronavirus is here to stay — here’s what that means., 2021, 590(7846): 382–384.
[46] Maslo C, Friedland R, Toubkin M, Laubscher A, Akaloo T, Kama B. Characteristics and outcomes of hospitalized patients in South Africa during the COVID-19 Omicron wave compared with previous waves., 2022, 327(6): 583–584.
[47] Murray CJL. COVID-19 will continue but the end of the pandemic is near., 2022, 399(10323): 417–419.
[48] Wolter N, Jassat W, Walaza S, Welch R, Moultrie H, Groome M, Amoako DG, Everatt J, Bhiman JN, Scheepers C, Tebeila N, Chiwandire N, du Plessis M, Govender N, Ismail A, Glass A, Mlisana K, Stevens W, Treurnicht FK, Makatini Z, Hsiao NY, Parboosing R, Wadula J, Hussey H, Davies MA, Boulle A, von Gottberg A, Cohen C. Early assessment of the clinical severity of the SARS-CoV-2 omicron variant in South Africa: a data linkage study., 2022, 399(10323): 437–446.
[49] Carter-Timofte ME, Jørgensen SE, Freytag MR, Thomsen MM, Brinck Andersen NS, Al-Mousawi A, Hait AS, Mogensen TH. Deciphering the role of host genetics in susceptibility to severe COVID-19., 2020, 11: 1606.
Genetic predisposition in patients with severe COVID-19
Wenbing Liu1,2, Dan Liu1, Jin Yan1, Xin Liu1,2, Qianfei Wang1,2
The coronavirus disease 2019 (COVID-19) is a global pandemic caused by severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) infection. COVID-19 has a variety of clinical manifestations, ranging from asymptomatic infection or mild symptoms to severe symptoms. Severe COVID-19 patients experience cytokine storm, resulting in multi-organ failure and even death. Male gender, old age, and pre-existing comorbidities (such as hypertension and diabetes ) are risk factors for COVID-19 severity. Recently, a series of studies suggested that genetic defects might also be related to disease severity and the cytokine storm occurence. Genetic variants in key viral immune genes, such asandhave been identified in severe COVID-19 patients from previous reports. In this review, we summarize the mechanisms underlying immune responses against SARS-CoV-2 and genetic variants that associated with the severity of COVID-19. The study of genetic basis of COVID-19 will be of great benefit for early disease detection and intervention.
severe COVID-19 patients; germline variants; immunodeficiency
2022-03-06;
2022-05-25;
2022-06-10
北京市自然科学基金资助项目(编号:M21022)[Supported by Beijing Natural Science Foundation (No. M21022)]
刘文兵,在读博士研究生,研究方向:遗传学。E-mail: liuwenbing@big.ac.cn
王前飞,博士,研究员,研究方向:白血病基因组学。E-mail: wangqf@big.ac.cn
10.16288/j.yczz.22-058
(责任编委: 岑山)