植物孤基因研究进展
姜明亮,郎红,李晓楠,祖野,赵靖,彭沈凌,刘振,战宗祥,朴钟云
植物孤基因研究进展
姜明亮1,郎红1,李晓楠2,祖野2,赵靖1,彭沈凌1,刘振1,战宗祥2,朴钟云2
1. 吉林农业科技学院农学院,吉林 132101 2. 沈阳农业大学园艺学院,沈阳 110866
孤基因(orphan genes)处在一个特殊的进化分支上,和其他任何已鉴定的基因没有显著的序列相似性。孤基因普遍存在于每个物种中,比较基因组学分析发现所有已测序的物种中均包含一部分孤基因,不同的筛选条件所获得的数量不等。孤基因经常与各种胁迫响应、物种特异性进化和物质代谢调节相关联。但多数孤基因没有被很好的注释,甚至没有可以被识别的功能结构域,为孤基因的功能表征带来了一定困难。相对于保守基因而言,孤基因的研究较少,这就导致了孤基因的重要性可能被“埋没”。本文从孤基因起源与进化、植物孤基因筛选及功能等方面进行了综述,并分析了目前存在的挑战和未来的研究重点与解决方案,以期为研究孤基因功能及其作用机制提供理论基础。
植物孤基因;起源;进化;筛选;基因功能
随着下一代测序技术的发展,越来越多的物种基因组被测序,进而科研人员对孤基因(orphan genes)的关注与研究也日益增加[1]。孤基因,也叫谱系特异性基因(lineage-specific genes)、分类学上限制基因(taxonomically restricted genes, TRGs),或称为orphans[2~4]。这些基因与之前已经鉴定出来的基因没有序列相似性,孤基因的概念最先在酵母()基因组测序研究中被提出,人们发现酵母基因组中有将近30%的基因属于孤基因范畴[5~9]。这类基因也可被细分为科特异、属特异或种特异孤基因等。研究表明,孤基因可能与各种生物胁迫和非生物胁迫响应、重要的物种特异性适应过程或物种特异性性状相关,也可能参与某些重要的信号途径或对于某些新的防御机制起到重要作用[10~12]。孤基因的起源有多种方式,例如从非编码序列衍生,或起源于基因复制、驯化的转座子、早期移码突变和重叠/反义读框等[11,13]。此外,孤基因进化速度非常快,并伴随着更低的转录水平或组织特异性表达[14]。许多研究已经证实,鉴定孤基因的策略主要是通过比较分析已经测序的各个物种基因组来实现的[1]。
1 孤基因的起源与进化
自下一代测序技术(next generation technologies)问世以来,对众多基因组分析表明,孤基因广泛存在于各种生物体中[15,16]。孤基因代表着分类学中最特殊的一个方面,位于基因组学、遗传学、比较和结构生物学、系统发育生物学和进化论的交叉点,这就使得利用传统方法研究孤基因变得尤为困难[17]。有机体中蛋白数量相对恒定,促使物种进化过程中基因的产生和灭绝要处于一个动态平衡的状态,同时假设众多孤基因组成了一个巨大的、动态的“新基因库”,比较合理的解释就是部分新进化基因的产生源于孤基因的进化成熟[18]。随着孤基因的出现,并在基因组中固定下来,能够参与生命活动的调控网络,是其进化成熟的标志[11]。孤基因来源于新产生的蛋白编码基因,是新兴的从非基因序列中产生的,被认为是古老基因的后裔,但其编码序列已经变得难以被识别[6,19]。
分类单元中的保守基因能为研究物种核心进程提供依据,而分类单元中的孤基因则能为物种进化进程和生物功能的研究奠定基础[20]。研究表明,通过对孤基因的基因组背景和序列进行分析,可以追溯其起源,有些可以追溯到基因复制(gene duplication)的高度歧化产物[21,22],或起源于重叠/反义阅读框(overlapping or anti-sense open reading frames),或从非编码序列区域(non-coding sequence regions)衍生,但是因基因复制而产生新功能的旁系同源基因(paralogues)并不属于孤基因的范畴[11,20,23]。一些孤基因含有转座因子,促进了孤基因的形成,这已经成为孤基因进化的主要模式,也就是常说的孤基因衍生于驯化的转座子(domesticated transposons)[6]。也有许多研究表明,早期移码突变(early frame-shift mutations)产生的新基因中也包含一部分孤基因,由于测序的物种越来越多,基因组密度也越来越大,这一起源方式会变得更容易被识别[24]。研究普遍认为非基因区域是新基因起源之地,但是现在对长链非编码RNA (long non-coding RNA, lncRNA)的调控模式、序列和功能结构还知之甚少,因此这类基因是如何形成的还很难被预测[17]。
孤基因可能是从非编码序列重新进化而来,或者是从旧的编码序列中衍生出来[11]。研究表明,拟南芥()中超过50%的孤基因和琴叶拟南芥()的非编码区域具有相似性,推断这些孤基因的出现似乎是从头形成()[25]。研究证实,一个物种基因库中从头产生的新基因可能比零星的基因复制更普遍,也是孤基因产生的主要方式之一[26~30]。新产生的孤基因在适应环境或人工驯化过程中随着时间变成熟[31]。对拟南芥、酵母等物种孤基因分析发现,新进化孤基因的蛋白质长度通常更短,这主要是由于孤基因在进化过程中拥有更少的外显子,而在某些物种中则是外显子长度显著变短[32,33]。近来研究发现,大豆()中21.7%的基因为最近起源的孤基因,且主要以单拷贝形式存在[34]。孤基因的成熟是随机的、且随着时间的推移而变得特异化[35]。孤基因的进化显得更加快速或“激进”。水稻()孤基因中的微卫星(microsatellites)和低复杂度区域(low-complexity regions)显得更为普遍,这些区域一度被认为是基因进化的强大推动力[25]。Plissonneau等[36]分析了小麦()真菌病原菌菌株()的完整基因组的进化孤区域,表明病原体具有广泛的染色体结构多态性,可能推动毒力的进化。此外,孤基因是富含编码假定效应子的基因,其中包括一个在小麦感染病菌期间上调最大的假定效应子基因[36]。年轻基因的前景如何主要取决于其能否安全快速的融入到重要的生命进程或代谢网络中,对酵母中孤基因的研究表明,调控过程出现很快,但是在蛋白与蛋白互作方面发展的较为缓慢[37]。虽然孤基因的起源和进化仍然是一个谜,但是孤基因的出现有助于人们理解物种进化是如何发生的,因此需要使用基因组和转录组等数据来加速对孤基因的筛选和研究。
2 植物孤基因的筛选
2.1 影响植物孤基因筛选的因素
随着许多物种基因组和转录组数据的陆续公布,使得孤基因和保守基因的筛选和鉴定变为可能[38]。目前,孤基因的筛选和鉴定主要通过与表达序列标签(expressed sequences tags, ESTs)、非冗余数据库(non-redundant database, nrdb)或已经完成测序的各种基因组进行比较分析来完成。影响孤基因筛选的因素有很多,主要包括:
(1)筛选程序。比较分析的程序主要围绕BLAST (Basic Local Alignment Search Tool)展开,包括BLASTP、TBLASTN、BLASTN、TBLASTX、PSI- BLAST和BLAT (BLAST-Like Alignment Tool)等。在相似条件下,不同程序的组合筛选所获得的孤基因数量不同。
(2)-value。利用不同-value筛选得到的孤基因数量大不相同,例如对杨树()基因组中孤基因的筛选,不同-value使得筛选结果存在较大差异,-value越大,所获得的孤基因数量越少,反之越多[39,40]。目前,普遍采用的-value为1E-03甚至更低,而最高的1E-01相对较为宽松,但较少采用。
(3)所比对的近源物种基因组数量以及数据库更新时间。比对的近源物种基因组数量越多可能得到的孤基因越少,但质量越高。数据库随着时间的推移而更新,注释的基因数量逐步增加,保守基因更容易被识别,从而在孤基因中被剔除掉,数据的准确性也越来越高。
(4)基因组组装和注释质量。不同基因组版本影响孤基因的筛选,尤其是基因组的注释质量对孤基因的筛选影响更大。例如水稻TIGRv5基因组中的孤基因比TIGRv2中少,但准确性却得到大幅度提升[39,41]。此外,还会有更多的条件影响孤基因的筛选,如所属不同谱系中的孤基因、特定胁迫条件中的孤基因等。
2.2 植物孤基因的普遍存在性
随着越来越多的物种基因组测序的完成,人们发现孤基因普遍存在于每个物种中,占比从小于1%~32%不等,且利用不同条件所筛选得到的孤基因数量不同[14,42]。植物孤基因的筛选工作始于拟南芥,Lin等[20]将拟南芥基因组序列与8个已测序的植物基因组序列、植物基因组数据库组装的特异转录本和UniProt数据库中的序列进行比较分析,鉴定了1324个拟南芥特异基因(lineage-specific genes,) (表1),通过一系列生物信息学分析证实了孤基因在蛋白水平上进化速度较快,同时也表明在花器官中具有更高的甲基化程度。而利用更高的-value (1E-01)进行筛选,在拟南芥中只鉴定到165个孤基因,并发现一些孤基因在胁迫条件下优先在花、根中表达,或上调表达[39]。同样,Donghue等[25]也对拟南芥孤基因进行了筛选,共鉴定了958个拟南芥特异基因,并证明孤基因的表达具有组织特异性,在多个组织和多个发育阶段中呈现低表达水平,同时孤基因在响应一系列非生物胁迫过程中显著富集。
Jiang等[43,44]利用更严格的筛选方法,将白菜()基因组和89个已测序植物基因组、植物基因组数据库中的267个物种的特异转录本和nrdb等进行比对,最终在白菜基因组中鉴定了529个芸薹种孤基因(orphan genes,)。通过对水稻孤基因的筛选和分析,利用不同的筛选程序和-value等,分别筛选获得37个[45]和1926个[41]孤基因。Guo等[41]对水稻中筛选得到的1926个孤基因进行分析,结果表明微卫星在孤基因进化和表达过程中起到重要作用。Yang等[39]借助于BLASTP和TBLASTN程序分别从水稻和杨树中鉴定到638和109个孤基因,证实孤基因在少外显子的基因上进行富集,同时也表明草本和木本植物、单子叶植物和双子叶植物之间的功能存在差异。此外,杨树孤基因可作为研究碳固定和生物燃料的候选基因[39]。Lin等[40]利用杨树基因组和30个已经测序的基因组进行比对分析,筛选得到40个孤基因,和杨树基因组以外的任何序列没有相似性,同时也没有功能注释,并通过一系列实验证实了这些孤基因可能在木本植物中参与谱系特异性生物学进程。
Li等[46]利用BLAST和基于微阵列的基因组杂交方法,鉴定了578个豇豆()孤基因。Graham等[47]利用BLAST比较分析鉴定了2525个豆科(Leguminosae)特异EST contigs,这些基因中只有不到3%和已经表征的豆科基因存在明显的同源性,分析表明孤基因中的一类CCPs (Cys cluster proteins)中的大多数基因的进化是通过本地复制和歧化选择而来。此外,多个植物基因组中的孤基因被鉴定,包括蜡烛果()[48]、甜橙()[49~51]、木豆()[52]、高粱()[53,54]、玉米()[54]、二穗短柄草()[54]、烟草()[55]、马铃薯()[55,56]、番茄()[55]、辣椒()[55]、矮牵牛()[55]、小麦[53]、苹果(Borkh.)[57]和葡萄()[58,59]等。对孤基因的序列特征分析表明,与进化保守基因相比,孤基因具有更低的多外显子基因占比、更高的GC含量和更短的序列长度,在进化上显得更加“激进”和快速。
随着比较基因组学的不断发展,多个筛选和鉴定孤基因的数据库、软件、程序或网页被建立,如ORFanFinder在线服务器(http://cys.bios.niu.edu/ orfanfinder)[60]、ATTED-II共表达数据库(http:// atted.jp)[61]、GreenPhylDB数据库(http://greenphyl. cirad.fr)[62,63]、小麦孤基因数据库TOGD[64]、禾本科孤基因数据库(http://bioinfo.ahau.edu.cn/pogd)[54]和SMOTE-ENN-XGBoost等[65]。多个物种的孤基因已经被筛选和鉴定,针对孤基因表达的模式分析证实了其在进化过程中可能起到的重要作用。
综上所述,孤基因的筛选工作证实了孤基因的普遍存在性,代表性植物孤基因的筛选结果见表1。但是,绝大部分孤基因的功能还是未解之谜,今后需要进一步的生物学实验来验证及解析其功能。
3 植物孤基因功能研究
孤基因表达具有高度特异性和低水平表达特征,参与多种生物和非生物胁迫响应,具有调节物质代谢和影响物种特异性进化等功能[66]。虽然孤基因功能研究较为缓慢且成果较少,但随着植物中第一个拟南芥孤基因(, AT3G30720)功能被表征[11,67~69],关于植物孤基因功能研究的报道不断涌现,正逐步揭开其神秘的面纱。
3.1 特异调控表达模式
拟南芥中发现的孤基因与保守基因相比,在花组织中的甲基化程度更高,并且具有更高的组织特异性和更低的表达水平[20,25]。研究表明,39个、47个和39个水稻特异基因分别在花组织、雌蕊组织和根组织中表现出较高水平的特异表达[39]。7个、12个、15个和5个杨树特异基因分别在雌花、木质部、形成层和叶组织中表现出相对较高的特异表达水平[39]。Lin等[40]研究表明,孤基因在幼叶或成熟叶、茎皮和根中的表达与对应的组织发育有较强关联。大多数在白菜的不同器官、组织和发育阶段均有表达,少数表现出组织特异性、器官特异性或发育阶段特异性表达模式[43]。豇豆孤基因也被认为是在人工选择条件下,维持驯化作物农艺性状和适应性性状平衡的一个主要因素[46]。此外,蜡烛果特异基因(-specific genes,)呈现高度组织特异性表达[48];甜橙孤基因优先在愈伤组织中表达[49]。因此,植物孤基因表现出了特异调控表达模式。
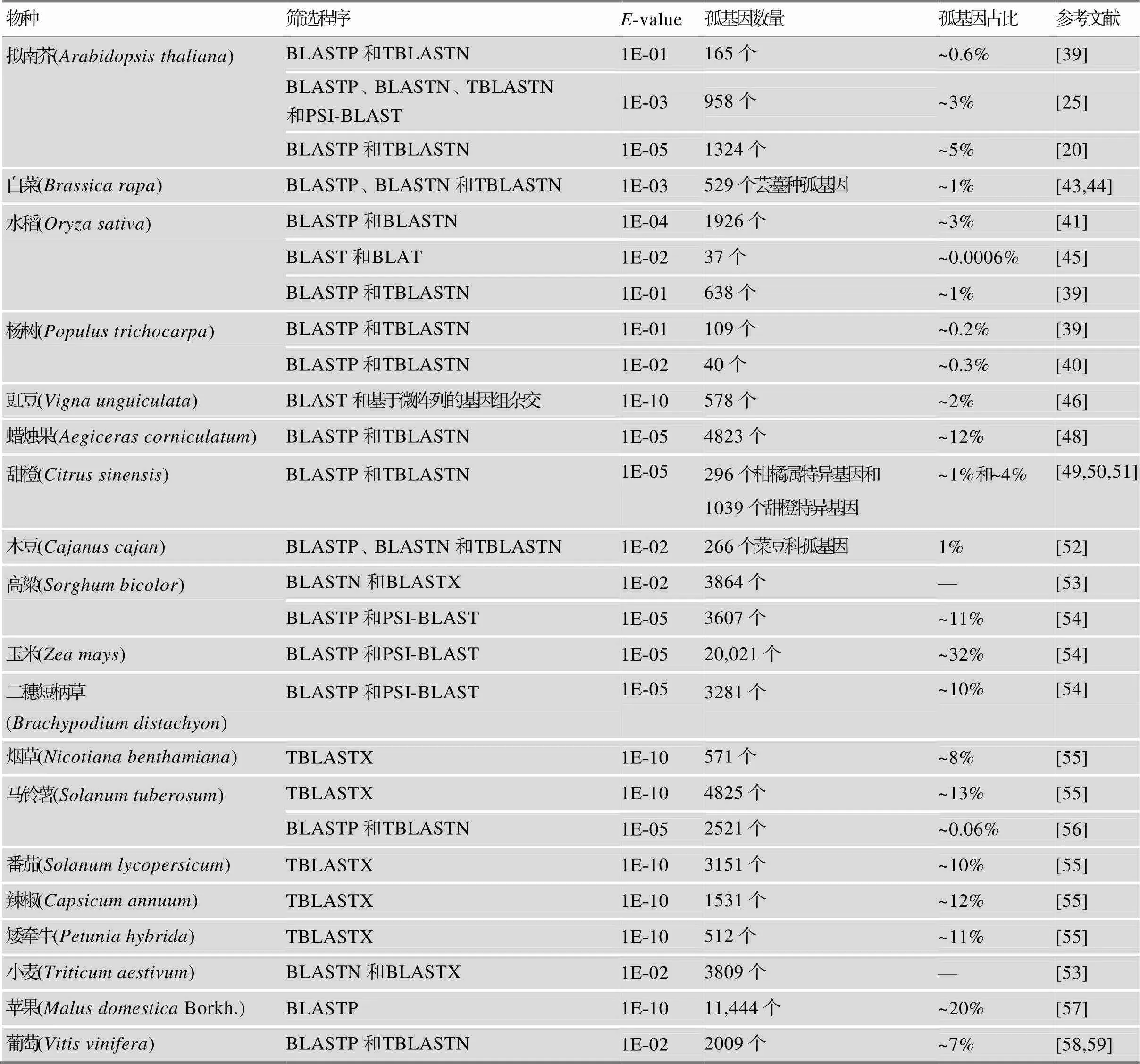
表1 代表性植物孤基因筛选结果
—:表示参考文献中未计算孤基因的比例;BLAST:Basic Local Alignment Search Tool;BLAT:BLAST-Like Alignment Tool。
3.2 胁迫响应
高度胁迫特异响应是孤基因的重要特征之一[70]。在拟南芥中,十字花科特异基因()及其在甘蓝()中的同源基因()的超量表达能提高植株对维管束枯萎病的抗性,同时超量表达增强了拟南芥的耐旱特性[71]。在烟草中,超量表达和基因也能提高转基因植株对病菌的抗性,这说明孤基因在不同物种中具有相似功能。同样,在拟南芥和大豆中超量表达拟南芥孤基因增强了植株对蚜虫、真菌和病毒的抗性,同时表明基因通过调节初级代谢和环境变化之间的复杂关系,来参与对生物/非生物干扰的优化和耐受性调节[11,72,73]。2个拟南芥特异性POFs (proteins with obscure features)也能提高氧化应激的耐受性[74]。小麦中的早熟禾亚科特异基因()能增强宿主对产毒性禾谷镰刀菌()的抗性[31]。利用酵母双杂交文库筛选和双分子荧光互补等方法发现TaFROG能与TaSnRK1α蛋白(sucrose non-fermenting-related kinase 1α)相互作用,这种互作增强了小麦对病菌的抗性,为小麦育种提供了新的抗性基因源[31]。TaFROG还能在植物体内与NAC-like转录因子TaNACL-D1和TaSnRK1α形成不同的蛋白质复合物,构成了小麦族内响应病害调控网络的一部分[75]。此外,Osp24 (orphan secreted protein 24)通过与TaFROG竞争和TaSnRK1α的结合区域,进而发挥细胞质效应因子的作用,证实了宿主和真菌病原体的孤蛋白在相互作用过程中的抵消作用[76]。Osp24和TaFROG分别是病原体和宿主中的孤蛋白,在真菌与植物的相互作用中,二者都可能受到共同进化的影响[76]。另一项关于小麦孤基因的研究发现,孤基因和能与小麦赤霉病菌分泌的小蛋白相互作用,这种互作增强了小麦对赤霉病的抗性,表明孤基因是小麦和赤霉病菌共同进化的重要组成部分,是调节抗性的潜在候选基因[77]。根肿菌()作为一种土传病害,能特异性侵染多种十字花科植物,导致根肿病的发生[78,79]。白菜在遭受根肿菌侵染条件下,52个中的41个能响应根肿菌胁迫,其中39个上调表达,结果表明可能在芸薹属植物应对生物胁迫中起重要作用[43]。然而,在白菜与根肿菌相互作用中的功能还需要深入研究。水稻AA基因组谱系特有的羟基肉桂酰酪胺(hydroxycinnamoyl tyramine, HT)基因簇(、、和),被证实有助于增强水稻抗病性,该基因簇通常在特定组织或胁迫下表现出共表达模式[80]。此外,水稻中新发现的抗病孤基因,能在单子叶植物和双子叶植物中诱导高度敏感反应,表明新抗性基因可扩展到野生水稻之外,包括野生禾本科植物[81]。对单子叶植物特有的脂肪族酚胺–羟基肉桂酰腐胺(hydroxycinnamoyl putrescine, HP)基因簇的遗传和生化分析表明,和对水稻免疫和细胞死亡具有积极的调节作用[82]。
孤基因是可用于识别参与某些环境适应的新基因,被视为一种有价值的基因资源。在拟南芥中超量表达唇形科特异孤基因()能对根冠性状产生重大影响,并调节植株对干旱胁迫敏感性[83]。深入的机理研究发现,的5′-UTR表达变化是通过与负调节因子MYB基因SIN_ 1023179 (SiMYB181)的相互作用进行调节[83]。研究证实千穗谷()中存在的一组独特的孤基因,被认为可能是维持植物对不同胁迫条件下耐受能力的一个重要因素[84]。86个蜡烛果特异基因被证实主要参与多个逆境胁迫响应途径,包括植物激素信号转导、苯丙烷生物合成、光合作用、过氧化物酶体和磷酸戊糖途径[48]。超量表达豇豆孤基因能增强转基因植株的耐旱性,证实是提高豇豆抗旱性的新基因[46]。低温胁迫能显著诱导麻疯树()/家族孤基因在叶片与根中的表达,表明基因参与麻疯树低温响应过程,同时为选育麻疯树耐低温新品种提供了候选基因[85]。相反,水稻孤基因对水稻低温耐受能力具有负调控作用[86]。通过转基因验证,表明基因也与水稻种子淹水条件下的快速生长相关[87]。
小麦孤基因在非生物和/或生物胁迫下具有应激反应性与可诱导性,表明其潜在的重要性[88]。研究人员选择了1007个功能未知的拟南芥基因,其中包含一部分孤基因,评估了其对应的纯合子T-DNA插入突变体对缺氧、低温、高温、盐分、氧化和渗透胁迫的反应[12]。结果表明,其中832个突变体对多种非生物胁迫表现出耐受性或敏感性,这意味着功能未知的基因可能在非生物胁迫反应信号或一般适应机制中发挥重要作用[12]。以上这些功能未知的基因构成了一个具体的基因库,可以在植物应对各种环境胁迫的防御策略中扮演着重要角色。
3.3 物质代谢调节
遗传和生化证据表明,在控制淀粉含量中发挥重要作用[67]。研究表明,能调节拟南芥叶片的碳氮分配,其RNAi株系的淀粉含量显著增加,蛋白含量显著下降[67];将基因转化到大豆后,显著提高了T1到T3代大豆种子中蛋白质含量,表型和野生型几乎无差异,这为大豆蛋白的生产提供了新思路。另有研究表明,随着花粉的发育表达量逐渐增加,并且在成熟花粉粒中达到最高值[11,68,69]。拟南芥过表达株系的淀粉含量减少、蛋白含量增加,而RNAi株系在一天周期内光照结束时表现出叶片淀粉含量增加、蛋白质含量减少[67,69]。基因在物质代谢调节中起着重要作用,在不同物种中也具有相似的功能[89]。例如,大豆中过表达显著增加了叶片和种子中的蛋白质含量,而种子中的油和纤维含量、叶片中淀粉含量显著减少,且转基因植株和野生型植株的表型无差异[68]。水稻过表达植株的叶片和种子中淀粉含量显著减少、蛋白质含量显著增加;玉米过表达植株的种仁中蛋白质含量显著增加、淀粉含量显著减少[69]。深入研究发现,AtQQS和转录调控子AtNF-YC4 (nuclear factor Y, subunit C4)互作,且拟南芥过表达植株的表型与过表达的表型相似,增加了蛋白质含量、降低了淀粉含量。同样,过表达和均减少了烟草叶片中淀粉含量、提高了叶片和种子中的总蛋白质含量,证明了二者在调节碳氮分配中的作用[73]。以上结果表明,一个物种的孤基因通过与保守蛋白互作,参与到另一个物种的调控网络中,因此可以作为碳氮分配的调节剂。另一项研究证实,过表达拟南芥十字花科特异基因(,)显著降低了植株中淀粉含量,而敲除植株中淀粉含量显著增加,推测可能在的下游调控网络中发挥作用,尤其在初级代谢、适应内部和外部环境变化、影响衰老过程的整合中发挥作用[90]。
以大白菜(L. ssp.)基因为目标序列构建的拟南芥过表达转基因库中,17个株系的果糖含量相对于野生型显著增加(如OE)、4个显著减少;29个株系葡萄糖含量显著增加(如OE)、1个株系显著减少;20个株系蔗糖含量显著增加、10个株系显著减少(如OE);34个株系总可溶性糖含量增加(如OE)、1个株系显著减少、8个株系无显著变化,这表明可广泛影响可溶性糖代谢[44]。以OE为例进行深入研究发现,大白菜基因敲除植株互补了OE株系表型,即敲除植株中的蔗糖含量相对于野生型显著增加,果糖、葡萄糖和总可溶性糖含量显著减少,机理研究表明可能通过依赖于蔗糖合成酶的方式影响可溶性糖含量[44]。这些结果表明,在可溶性糖代谢中起着重要的作用,可能是改善白菜营养品质的重要基因。
对水稻AA基因组谱系特有基因簇的研究发现,该基因簇区域内的转座子元件显著富集,该研究不仅发现了一个参与苯丙烷代谢的基因簇,且阐明了基因簇形成的关键原因,研究结果为植物抵御病原体提供了新的代谢池[80]。Fang等[82]研究表明,超量表达控制HP代谢物合成的基因簇(、或)提高了水稻对真菌病原体稻瘟菌的抗性,同时超量表达和会促进HP的积累,为抗病分子育种提供了重要理论依据。
3.4 影响物种特异性进化
孤基因的生物功能对于阐明物种进化进程和生物适应性具有重要意义[91]。Hanada等[92]报道了植物串联重复世系特异性扩张在植物适应环境刺激过程中起到重要作用。此外,水稻和拟南芥中的种特异性孤基因在花粉和精细胞中呈现高表达,并与新基因的起源相关联[93]。近来的研究还揭示了转移到核基因组的线粒体DNA数量与陆生植物中孤基因数量之间的高度相关性,表明线粒体基因组可能在陆生植物的核孤基因进化中发挥作用[94]。普通小麦中的显性雄性不育基因被证实属于小麦亚族特异基因,赋予小麦、大麦()和二穗短柄草的雄性不育性,其在花药中的表达与逆转录酶元件插入启动子有关[95]。另一项关于小麦孤基因的研究证实,大多数孤基因主要在生殖相关组织中优先表达,尤其是在初生雄蕊中[88]。水稻孤基因对水稻的快速生长具有重要作用,该研究为更好地在育种中利用孤基因提供了理论依据[87]。孤基因影响物种特异性进化的实例相对较少,开展相关研究的难度相对较大,未来还需要深入探索。
4 结语与展望
高通量测序技术促进了全基因组测序的发展,这使得每一个新测序物种中的孤基因被陆续发现,该部分独特的基因也不断带来惊喜。孤基因可能作为物种进化过程中重要的持续不断的基因源,孤基因的产生可能使物种形成了一种新的适应机制。孤基因的产生、变成熟亦或消亡,可能是受到了进化压力的影响,阐明孤基因进化进程,可能会有助于人们理解物种进化趋势。到目前为止,人们对孤基因的了解主要基于比较基因组研究和表达分析。然而,植物孤基因的功能分析仍然面临诸多挑战。幸运的是,科研人员在试图揭开孤基因功能的奥秘时,许多有代表性的研究成果不断涌现。例如针对[31]、[44]和[67]等基因的功能表征,为孤基因的分子基因组学奠定了坚实的基础。未来,还有许多需要深入研究的方向,例如:孤基因是如何参与到调控网络中发挥作用的?哪些因素影响孤基因的进化?孤基因在进化过程中是如何在一个基因组中固定下来并发挥重要作用的?
为了更好、更全面的研究孤基因的功能及其作用机制,除生物信息学分析方法外,需要借助于最新的技术方法来进行探索。例如,通过CRISPR (clustered regularly interspaced short palindromic repeats)技术[96~98],构建基因敲除突变体库,加速孤基因功能表征;通过特定条件下的转录组学或蛋白组学提前确定具有表达证据的孤基因,从而为其功能表征提供有力的依据;也可以通过酵母单杂交(yeast one-hybrid, Y1H)文库筛选[99]、酵母双杂交(yeast two-hybrid, Y2H)文库筛选[100]、免疫沉淀–质谱联用(immunoprecipitation-mass spectrometry, IP-MS)筛选[101]等技术鉴定孤基因的上下游调控网络,解析孤基因作用机制,这些都将为在育种中应用孤基因作为重要基因源提供理论基础和实践指导。
[1] Campbell MA, Zhu W, Jiang N, Lin HN, Ouyang S, Childs KL, Haas BJ, Hamilton JP, Buell CR. Identification and characterization of lineage-specific genes within the Poaceae., 2007, 145(4): 1311–1322.
[2] Weisman CM, Murray AW, Eddy SR. Many, but not all, lineage-specific genes can be explained by homology detection failure., 2020, 18(11): e3000862.
[3] Wilson GA, Bertrand N, Patel Y, Hughes JB, Feil EJ, Field D. Orphans as taxonomically restricted and ecologically important genes., 2005, 151(Pt 8): 2499–2501.
[4] Fischer D, Eisenberg D. Finding families for genomic ORFans., 1999, 15(9): 759–762.
[5] Dujon B. The yeast genome project: what did we learn?, 1996, 12(7): 263–270.
[6] Domazet-Lošo T, Tautz D. An evolutionary analysis of orphan genes in., 2003, 13(10): 2213–2219.
[7] Tautz D, Neme R, Domazet-Lošo T. Evolutionary origin of orphan genes., 2013, 1–8.
[8] Yin YB, Fischer D. Identification and investigation of ORFans in the viral world., 2008, 9: 24.
[9] Bianchi MM, Sartori G, Vandenbol M, Kaniak A, Uccelletti D, Mazzoni C, Rago JPD, Carignani G, Slonimski PP, Frontali L. How to bring orphan genes into functional families., 1999, 15(6): 513–526.
[10] Toll-Riera M, Bosch N, Bellora N, Castelo R, Armengol L, Estivill X, Albà MM. Origin of primate orphan genes: a comparative genomics approach., 2009, 26(3): 603–612.
[11] Arendsee ZW, Li L, Wurtele ES. Coming of age: orphan genes in plants., 2014, 19(11): 698–708.
[12] Luhua S, Hegie A, Suzuki N, Shulaev E, Luo XZ, Cenariu D, Ma V, Kao S, Lim J, Gunay MB, Oosumi T, Lee SC, Harper J, Cushman J, Gollery M, Girke T, Bailey-Serres J, Stevenson RA, Zhu JK, Mittler R. Linking genes of unknown function with abiotic stress responses by high-throughput phenotype screening., 2013, 148(3): 322–333.
[13] Striepen B, Pruijssers AJP, Huang JL, Li C, Gubbels MJ, Umejiego NN, Hedstrom L, Kissinger JC. Gene transfer in the evolution of parasite nucleotide biosynthesis., 2004, 101(9): 3154–3159.
[14] Palmieri N, Kosiol C, Schlötterer C. The life cycle oforphan genes., 2014, 3: e01311.
[15] Khalturin K, Hemmrich G, Fraune S, Augustin R, Bosch TCG. More than just orphans: are taxonomically- restricted genes important in evolution?, 2009, 25(9): 404–413.
[16] Johnson BR, Tsutsui ND. Taxonomically restricted genes are associated with the evolution of sociality in the honey bee., 2011, 12: 164.
[17] Nelson PA, Buggs RJA eds. Next generation apomorphy: the ubiquity of taxonomically restricted genes. Cambridge: Cambridge University Press, 2016, 237–263.
[18] Tautz D, Domazet-Lošo T. The evolutionary origin of orphan genes., 2011, 12(10): 692–702.
[19] Doerks T, Copley RR, Schultz J, Ponting CP, Bork P. Systematic identification of novel protein domain families associated with nuclear functions., 2002, 12(1): 47–56.
[20] Lin HN, Moghe G, Ouyang S, Iezzoni A, Shiu SH, Gu X, Buell CR. Comparative analyses reveal distinct sets of lineage-specific genes within., 2010, 10: 41.
[21] Cai J, Zhao RP, Jiang HF, Wang W.origination of a new protein-coding gene in., 2008, 179(1): 487–496.
[22] Lynch M, Conery JS. The evolutionary fate and consequences of duplicate genes., 2000, 290(5494): 1151–1155.
[23] Silveira AB, Trontin C, Cortijo S, Barau J, Del Bem LEV, Loudet O, Colot V, Vincentz M. Extensive natural epigenetic variation at a de novo originated gene., 2013, 9(4): e1003437.
[24] Wissler L, Gadau J, Simola DF, Helmkampf M, Bornberg-Bauer E. Mechanisms and dynamics of orphan gene emergence in insect genomes., 2013, 5(2): 439–455.
[25] Donoghue MT, Keshavaiah C, Swamidatta SH, Spillane C. Evolutionary origins of Brassicaceae specific genes in., 2011, 11: 47.
[26] Carvunis AR, Rolland T, Wapinski I, Calderwood MA, Yildirim MA, Simonis N, Charloteaux B, Hidalgo CA, Barbette J, Santhanam B, Brar GA, Weissman JS, Regev A, Thierry-Mieg N, Cusick ME, Vidal M. Proto-genes andgene birth., 2012, 487(7407): 370–374.
[27] Prabh N, Rödelsperger C., divergence, and mixed origin contribute to the emergence of orphan genes in., 2019, 9(7): 2277–2286.
[28] Schlötterer C. Genes from scratch-the evolutionary fate ofgenes., 2015, 31(4): 215–219.
[29] Zhang WY, Gao YX, Long MY, Shen BR. Origination and evolution of orphan genes andgenes in the genome of., 2019, 62(4): 579–593.
[30] Singh U, Wurtele ES. How new genes are born., 2020, 9: e55136.
[31] Perochon A, Jianguang J, Kahla A, Arunachalam C, Scofield SR, Bowden S, Wallington E, Doohan FM.encodes aorphan protein that interacts with SnRK1 and enhances resistance to the mycotoxigenic fungus., 2015, 169(4): 2895–2906.
[32] Albà MM, Castresana J. On homology searches by protein blast and the characterization of the age of genes., 2007, 7: 53.
[33] Domazet-Lošo T, Brajković J, Tautz D. A phylostratigraphy approach to uncover the genomic history of major adaptations in metazoan lineages., 2007, 23(11): 533–539.
[34] Tang K, Yang RL. Origin and evolution of soybean protein-coding genes., 2019, 54(3): 316– 327.
唐康, 杨若林. 大豆蛋白编码基因起源与进化. 植物学报, 2019, 54(3): 316–327.
[35] Hamari Z, Amillis S, Drevet C, Apostolaki A, Vágvölgyi C, Diallinas G, Scazzocchio C. Convergent evolution and orphan genes in the Fur4p-like family and characterization of a general nucleoside transporter in., 2009, 73(1): 43–57.
[36] Plissonneau C, Stürchler A, Croll D. The evolution of orphan regions in genomes of a fungal pathogen of wheat., 2016, 7(5): e01231–e01316.
[37] Seoighe C, Wolfe KH. Yeast genome evolution in the post-genome era., 1999, 2(5): 548–554.
[38] Beike AK, Lang D, Zimmer AD, Wüst F, Trautmann D, Wiedemann G, Beyer P, Decker EL, Reski R. Insights from the cold transcriptome of: global specialization pattern of conserved transcriptional regulators and identification of orphan genes involved in cold acclimation., 2015, 205(2): 869–881.
[39] Yang XH, Jawdy S, Tschaplinski TJ, Tuskan GA. Genome- wide identification of lineage-specific genes in,and., 2009, 93(5): 473–480.
[40] Lin WL, Cai B, Cheng ZM. Identification and characterization of lineage-specific genes in., 2014, 116(2): 217–225.
[41] Guo WJ, Li P, Ling J, Ye SP. Significant comparative characteristics between orphan and nonorphan genes in the rice (L.) genome., 2007, 2007: 21676.
[42] Murali TM, Wu CJ, Kasif S. The art of gene function prediction., 2006, 24(12): 1474–1476.
[43] Jiang ML, Dong XS, Lang H, Pang WX, Zhan ZX, Li XN, Piao ZY. Mining of-specific genes () and their induction in different developmental stages and understress in., 2018, 19(7): 2064.
[44] Jiang ML, Zhan ZX, Li HY, Dong XS, Cheng F, Piao ZY.orphan genes largely affect soluble sugar metabolism., 2020, 7(1): 181.
[45] Jin GH, Zhou YL, Yang H, Hu YT, Shi Y, Li L, Siddique AN, Liu CN, Zhu AD, Zhang CJ, Li DZ. Genetic innovations: transposable element recruitment andformation lead to the birth of orphan genes in the rice genome., 2021, 59(2): 341–351.
[46] Li GJ, Wu XY, Hu YW, Muñoz-Amatriaín M, Luo J, Zhou W, Wang BG, Wang Y, Wu XH, Huang LJ, Lu ZF, Xu P. Orphan genes are involved in drought adaptations and ecoclimatic-oriented selections in domesticated cowpea., 2019, 70(12): 3101–3110.
[47] Graham MA, Silverstein KAT, Cannon SB, VandenBosch KA. Computational identification and characterization of novel genes from legumes., 2004, 135(3): 1179–1197.
[48] Ma DN, Ding QS, Guo ZJ, Zhao ZZ, Wei LF, Li YY, Song SW, Zheng HL. Identification, characterization and expression analysis of lineage-specific genes within mangrove species., 2021, 296(6): 1235–1247.
[49] Xu YT, Wu GZ, Hao BH, Chen LL, Deng XX, Xu Q. Identification, characterization and expression analysis of lineage-specific genes within sweet orange ()., 2015, 16: 995.
[50] Xu Q, Chen LL, Ruan XA, Chen DJ, Zhu AD, Chen CL, Bertrand D, Jiao WB, Hao BH, Lyon MP, Chen JJ, Gao S, Xing F, Lan H, Chang JW, Ge XH, Lei Y, Hu Q, Miao Y, Wang L, Xiao SX, Biswas MK, Zeng WF, Guo F, Cao HB, Yang XM, Xu XW, Cheng YJ, Xu J, Liu JH, Luo OJ, Tang ZH, Guo WW, Kuang HH, Zhang HY, Roose ML, Nagarajan N, Deng XX, Ruan YJ. The draft genome of sweet orange ()., 2013, 45(1): 59–66.
[51] Wu GZ. The identification of thespecific genes and their origin pattern analysis[Dissertation]. Huazhong Agricultural University, 2016.
吴桂枝. 柑橘特异基因的鉴定及其起源方式分析[学位论文]. 华中农业大学, 2016.
[52] Varshney RK, Chen WB, Li YP, Bharti AK, Saxena RK, Schlueter JA, Donoghue MTA, Azam S, Fan GY, Whaley AM, Farmer AD, Sheridan J, Iwata A, Tuteja R, Penmetsa RV, Wu W, Upadhyaya HD, Yang SP, Shah T, Saxena KB, Michael T, McCombie WR, Yang BC, Zhang GY, Yang HM, Wang J, Spillane C, Cook DR, May GD, Xu X, Jackson SA. Draft genome sequence of pigeonpea (), an orphan legume crop of resource-poor farmers., 2011, 30(1): 83–89.
[53] Tao Y. Construction of an EST-based digital northern platform for common wheat genes and identification of wheat specific genes relative to rice[Dissertation]. Nanjing Agricultural University, 2010.
陶晔. 小麦基因电子表达分析平台的构建及相对于水稻的小麦特异基因的鉴定[学位论文]. 南京农业大学, 2010.
[54] Yao CS, Yan HW, Zhang XD, Wang RF. A database for orphan genes in Poaceae., 2017, 14(4): 2917–2924.
[55] Rensink WA, Lee YD, Liu J, Iobst S, Ouyang S, Buell CR. Comparative analyses of six solanaceous transcriptomes reveal a high degree of sequence conservation and species-specific transcripts., 2005, 6: 124.
[56] Cheng ZN. Identification, characterization, expression and function analysis of lineage-specific genes in potato [Dissertation]. Huazhong Agricultural University, 2019.
程正楠. 马铃薯种系特异性基因的鉴定、特征描述、表达和功能分析[学位论文]. 华中农业大学, 2019.
[57] Velasco R, Zharkikh A, Affourtit J, Dhingra A, Cestaro A, Kalyanaraman A, Fontana P, Bhatnagar SK, Troggio M, Pruss D, Salvi S, Pindo M, Baldi P, Castelletti S, Cavaiuolo M, Coppola G, Costa F, Cova V, Dal Ri A, Goremykin V, Komjanc M, Longhi S, Magnago P, Malacarne G, Malnoy M, Micheletti D, Moretto M, Perazzolli M, Si-Ammour A, Vezzulli S, Zini E, Eldredge G, Fitzgerald LM, Gutin N, Lanchbury J, Macalma T, Mitchell JT, Reid J, Wardell B, Kodira C, Chen ZT, Desany B, Niazi F, Palmer M, Koepke T, Jiwan D, Schaeffer S, Krishnan V, Wu CQ, Chu VT, King ST, Vick J, Tao QZ, Mraz A, Stormo A, Stormo K, Bogden R, Ederle D, Stella A, Vecchietti A, Kater MM, Masiero S, Lasserre P, Lespinasse Y, Allan AC, Bus V, Chagné D, Crowhurst RN, Gleave AP, Lavezzo E, Fawcett JA, Proost S, Rouzé P, Sterck L, Toppo S, Lazzari B, Hellens RP, Durel CE, Gutin A, Bumgarner RE, Gardiner SE, Skolnick M, Egholm M, Van de Peer Y, Salamini F, Viola R. The genome of the domesticated apple (Borkh.)., 2010, 42(10): 833–839.
[58] Liang YH, Chen F, Wang G, Wang M, Feng GQ, Cheng ZM. Bioinformatics identification, analysis of gene structure and expression characters on lineage-specific genes of grapevine., 2013, 36(6): 19–24.
梁英海, 陈飞, 王刚, 王敏, 冯冠乔, 程宗明. 葡萄谱系特有基因生物信息学识别、基因结构与表达特性分析. 南京农业大学学报, 2013, 36(6): 19–24.
[59] Li CH, Cai B, Liang YH, Jia SL. The expression analysis and function prediction of orphan genes in grape., 2013, 32(2): 240–245.
李成慧, 蔡斌, 梁英海, 贾士禄. 葡萄孤儿基因的表达分析和功能推测. 基因组学与应用生物学, 2013, 32(2): 240–245.
[60] Ekstrom A, Yin YB. ORFanFinder: automated identification of taxonomically restricted orphan genes., 2016, 32(13): 2053–2055.
[61] Aoki Y, Okamura Y, Tadaka S, Kinoshita K, Obayashi T. ATTED-II in 2016: A plant coexpression database towards lineage-specific coexpression., 2016, 57(1): e5.
[62] Conte MG, Gaillard S, Lanau N, Rouard M, Périn C. GreenPhylDB: a database for plant comparative genomics., 2008, 36: D991–D998.
[63] Valentin G, Abdel T, Gaëtan D, Jean-François D, Matthieu C, Mathieu R. GreenPhylDB v5: a comparative pangenomic database for plant genomes., 2021, 49(D1): D1464–D1471.
[64] Gao QJ, Yan HW, Xia EH, Zhang SH, Li SW. TOGD: a database of orphan genes in., 2019, 22(5): 961–966.
[65] Gao QJ, Jin X, Xia EH, Wu XW, Gu LC, Yan HW, Xia YC, Li SW. Identification of orphan genes in unbalanced datasets based on ensemble learning., 2020, 11: 820.
[66] Moxon ER, Higgins CF. Functions for orphan genes., 1997, 389(6647): 120.
[67] Li L, Foster CM, Gan QL, Nettleton D, James MG, Myers AM, Wurtele ES. Identification of the novel protein QQS as a component of the starch metabolic network inleaves., 2009, 58(3): 485–498.
[68] Li L, Wurtele ES. Theorphan gene ofmodulates carbon and nitrogen allocation in soybean., 2015, 13(2): 177–187.
[69] Li L, Zheng WG, Zhu YB, Ye HX, Tang BY, Arendsee ZW, Jones D, Li R, Ortiz D, Zhao XF, Du CL, Nettleton D, Scott MP, Salas-Fernandez MG, Yin Y, Wurtele ES.orphan gene regulates carbon and nitrogen partitioning across species via NF-YC interactions., 2015, 112(47): 14734–14739.
[70] Jiang ML. Identification oforphan genes and construction and evaluation of its transgenic library [Dissertation]. Shenyang Agricultural University, 2020.
姜明亮. 芸薹种孤基因的鉴定及其转基因库的构建和评价[学位论文]. 沈阳农业大学, 2020.
[71] Yadeta KA, Valkenburg DJ, Hanemian M, Marco Y, Thomma BPHJ. The Brassicaceae-specificgene provides resistance to vascular wilt pathogens., 2014, 9(2): e88230.
[72] Qi MS, Zheng WG, Zhao XF, Hohenstein JD, Kandel Y, O’Conner S, Wang YF, Du CL, Nettleton D, MacIntosh GC, Tylka GL, Wurtele ES, Whitham SA, Li L.orphan gene and its interactor NF-YC4 reduce susceptibility to pathogens and pests., 2019, 17(1): 252–263.
[73] Tanvir R, Ping WL, Sun JP, Cain M, Li XJ, Li L.orphan gene andboost protein accumulation and pest resistance in tobacco ()., 2022, 317: 111198.
[74] Luhua S, Ciftci-Yilmaz S, Harper J, Cushman J, Mittler R. Enhanced tolerance to oxidative stress in transgenicplants expressing proteins of unknown function., 2008, 148(1): 280–292.
[75] Perochon A, Kahla A, Vranić M, Jia JG, Malla KB, Craze M, Wallington E, Doohan FM. A wheat NAC interacts with an orphan protein and enhances resistance to Fusarium head blight disease., 2019, 17(10): 1892–1904.
[76] Jiang C, Hei RN, Yang Y, Zhang SJ, Wang QH, Wang W, Zhang Q, Yan M, Zhu GR, Huang PP, Liu HQ, Xu JR. An orphan protein ofmodulates host immunity by mediating proteasomal degradation of TaSnRK1α., 2020, 11(1): 4382.
[77] Brennan CJ, Zhou BB, Benbow HR, Ajaz S, Karki SJ, Hehir JG, O’Driscoll A, Feechan A, Mullins E, Doohan FM. Taxonomically restricted wheat genes interact with small secreted fungal proteins and enhance resistance toblotch disease., 2020, 11: 433.
[78] Jiang YF, Zhan ZX, Piao ZY, Zhang CY. Progresses and prospects of germplasms innovation for clubroot resistance and genetic improvement in., 2018, 44(11): 1592–1599.
江莹芬, 战宗祥, 朴钟云, 张椿雨. 油菜抗根肿病资源创新与利用的研究进展与展望. 作物学报, 2018, 44(11): 1592–1599.
[79] Yang ZQ, Jiang YF, Gong JF, Li Q, Dun BC, Liu DX, Yin FF, Lei Y, Zhou XQ, Wang HY, Wang J, Zhan ZX, Shah N, Nwafor CC, Zhou YW, Chen P, Li Z, Li SS, Wang BR, Xiang J, Zhou YM, Li ZY, Piao ZY, Yang QY, Zhang CY. R gene triplication confers European fodder turnip with improved clubroot resistance., 2022, 20(8): 1502–1517.
[80] Shen SQ, Peng M, Fang H, Wang ZX, Zhou S, Jing X, Zhang M, Yang CK, Guo H, Li YF, Lei L, Shi YH, Sun YY, Liu XQ, Xu CP, Tohge T, Yuan M, Fernie AR, Ning Y, Wang GL, Luo J. An-specific hydroxycinnamoyl tyramine gene cluster contributes to enhanced disease resistance., 2021, 66(23): 2369–2380.
[81] Wang CY, Chen S, Feng AQ, Su J, Wang WJ, Feng JQ, Chen B, Zhang MY, Yang JY, Zeng LX, Zhu XY., a small orphan gene harboring promoter trap for, leads to the durable resistance topv.., 2021, 14(1): 48.
[82] Fang H, Shen SQ, Wang D, Zhang F, Zhang CY, Wang ZX, Zhou QQ, Wang RY, Tao H, He F, Yang CK, Peng M, Jing XY, Hao ZY, Liu XL, Luo J, Wang GL, Ning YS. A monocot-specific hydroxycinnamoylputrescine gene cluster contributes to immunity and cell death in rice., 2021, 66(23): 2381–2393.
[83] Dossa K, Zhou R, Li DH, Liu AL, Qin L, Mmadi MA, Su RQ, Zhang YJ, Wang JQ, Gao Y, Zhang XR, You J. A novel motif in the 5’-UTR of an orphan gene ‘’ modulates root biomass in sesame., 2021, 19(5): 1065–1079.
[84] Cabrales-Orona G, Délano-Frier JP eds. Searching for an identity: functional characterization of taxonomically restricted genes in grain amaranth. Switzerland: Springer International Publishing, 2021, 97–124.
[85] Wang HB, Gong M, Guo JY, Xin H, Tang LZ. Molecular cloning and prokaryotic expression of orphan geneofgene family in., 2018, 54(9): 60–69.
王海波, 龚明, 郭俊云, 辛胡, 唐利洲. 麻疯树基因家族孤儿的克隆与原核表达分析. 林业科学, 2018, 54(9): 60–69.
[86] Liu CA. The role of orphan genein chilling tolerance of rice[Dissertation]. Nanchang University, 2018.
刘长爱. 孤儿基因在水稻耐低温中的作用[学位论文]. 南昌大学, 2018.
[87] Shuai YL. The function of orphan genein rice growth promotion[Dissertation]. Nanchang University, 2020.
帅艳玲. 孤儿基因在水稻促生过程中的功能[学位论文]. 南昌大学, 2020.
[88] Ma SW, Yuan Y, Tao Y, Jia HY, Ma ZQ. Identification, characterization and expression analysis of lineage- specific genes within., 2020, 112(2): 1343– 1350.
[89] O’Conner S, Neudorf A, Zheng W, Qi M, Zhao X, Du C, Nettleton D, Li L eds. Fromto crops: theorphan gene modulates nitrogen allocation across species. Switzerland: Springer International Publishing, 2018, 95–117.
[90] Jones DC, Zheng WG, Huang S, Du LC, Zhao XF, Yennamalli RM, Sen TZ, Nettleton D, Wurtele ES, Li L. A clade-specificgene connects primary metabolism and senescence., 2016, 7: 983.
[91] Zhang HP, Yin TM. Advances in lineage-specific genes., 2015, 37(6): 544–553.
张焕萍, 尹佟明. 谱系特有基因研究进展. 遗传, 2015, 37(6): 544–553.
[92] Hanada K, Zou C, Lehti-Shiu MD, Shinozaki K, Shiu SH. Importance of lineage-specific expansion of plant tandem duplicates in the adaptive response to environmental stimuli., 2008, 148(2): 993–1003.
[93] Cui X, Lv Y, Chen ML, Nikoloski Z, Twell D, Zhang DB. Young genes out of the male: an insight from evolutionary age analysis of the pollen transcriptome., 2015, 8(6): 935–945.
[94] O’Conner S, Li L. Mitochondrial fostering: the mitochondrial genome may play a role in plant orphan gene evolution., 2020, 11: 600117.
[95] Ni F, Qi J, Hao QQ, Lyu B, Luo MC, Wang Y, Chen FJ, Wang SY, Zhang CZ, Epstein L, Zhao XY, Wang HG, Zhang XS, Chen CX, Sun L, Fu DL. Wheatencodes for an orphan protein that confers male sterility in grass species., 2017, 8: 15121.
[96] Shan QW, Gao CX. Research progress of genome editing and derivative technologies in plants., 2015, 37(10): 953–973.
单奇伟, 高彩霞. 植物基因组编辑及衍生技术最新研究进展. 遗传, 2015, 37(10): 953–973.
[97] Li X, Shi W, Geng LZ, Xu JP. Genome editing in plants directed by CRISPR/Cas ribonucleoprotein complexes., 2020, 42(6): 556–564.
李霞, 施皖, 耿立召, 许建平. CRISPR/Cas核糖核蛋白介导的植物基因组编辑. 遗传, 2020, 42(6): 556– 564.
[98] Ma XL, Liu YG. CRISPR/Cas9-based genome editing systems and the analysis of targeted genome mutations in plants., 2016, 38(2): 118–125.
马兴亮, 刘耀光. 植物CRISPR/Cas9基因组编辑系统与突变分析. 遗传, 2016, 38(2): 118–125.
[99] Liu ZN, Yuan L, Sundaresan V, Yu XL. Screening and identification ofupstream transcription regulators in., 2019, 41(5): 430– 438.
刘振宁, 袁黎, Venkatesan Sundaresan, 余小林. 拟南芥基因上游转录调控因子筛选及鉴定. 遗传, 2019, 41(5): 430–438.
[100] Fields S, Song O. A novel genetic system to detect protein-protein interactions., 1989, 340(6230): 245–246.
[101] Marcon E, Jain H, Bhattacharya A, Guo HB, Phanse S, Pu SY, Byram G, Collins BC, Dowdell E, Fenner M, Guo XH, Hutchinson A, Kennedy JJ, Krastins B, Larsen B, Lin ZY, Lopez MF, Loppnau P, Miersch S, Nguyen T, Olsen JB, Paduch M, Ravichandran M, Seitova A, Vadali G, Vogelsang MS, Whiteaker JR, Zhong GQ, Zhong N, Zhao L, Aebersold R, Arrowsmith CH, Emili A, Frappier L, Gingras AC, Gstaiger M, Paulovich AG, Koide S, Kossiakoff AA, Sidhu SS, Wodak SJ, Gräslund S, Greenblatt JF, Edwards AM. Assessment of a method to characterize antibody selectivity and specificity for use in immunoprecipitation., 2015, 12(8): 725–731.
Progress on plant orphan genes
Mingliang Jiang1, Hong Lang1, Xiaonan Li2, Ye Zu2, Jing Zhao1, Shenling Peng1, Zhen Liu1, Zongxiang Zhan2, Zhongyun Piao2
Orphan genes are located in a special evolutionary branch and have no significant sequence similarity with any other identified genes. Orphan genes are prevalent in every species, comparative genomics analyses found that all sequenced species contained a portion of orphan genes, and the number of orphan genes obtained by distinct screening conditions is different. Orphan genes are often associated with various stress responses, species-specific evolution and substance metabolism regulation. However, most of the orphan genes have not been well annotated or even have no recognizable functional domains, which brings some difficulties to the functional characterization of orphan genes. Compared with conserved genes, there is less research on orphan genes, which leads to the possibility that the importance of orphan genes may be “unrewarded”. In this review, we summarize the origin and evolution of orphan genes, plant orphan gene screening and functions, and analyse the existing challenges and future research priorities and solutions, which provide theoretical basis for the study of orphan gene function and action mechanisms.
plant orphan genes; origin; evolution; screening; gene functions
2022-05-10;
2022-07-23;
2022-08-08
第五批吉林省青年科技人才托举工程项目(编号:QT202123)资助[Supported by the Fifth Batch of Jilin Province Youth Science and Technology Talent Promotion Project (No. QT202123)]
姜明亮,博士,讲师,研究方向:蔬菜分子生物学。E-mail: jiangmingliang@jlnku.edu.cn
郎红,博士,讲师,研究方向:植物分子生物学。E-mail: langhong@jlnku.edu.cn
姜明亮和郎红并列第一作者。
战宗祥,博士,讲师,研究方向:芸薹属蔬菜作物的资源创新与重要性状的形成和调控机制。E-mail: zhanzxiang@syau.edu.cn
朴钟云,博士,教授,研究方向:蔬菜分子生物学。E-mail: zypiao@syau.edu.cn
10.16288/j.yczz.22-154
(责任编委: 孔令让)