基于高密度SNP标记重构猪多品种群体系谱
杨雨婷,张 兴,牛安然,闫之春,龚华忠,丁偌楠,4,马 黎,4
(1.新希望六和养猪研究院数据与算法实验室,成都 610095;2.新希望六和养猪研究院,青岛 266100;3.新希望六和育种事业部,成都 610095; 4.德州市现代生猪养殖技术创新中心,德州 263000)
系谱是猪育种的重要信息来源之一,建立完整、准确的系谱对确定种猪真实身份、提高育种值估计的准确性、实现科学引种选育和选配有重要价值。种猪错误的系谱会通过“金字塔”式的猪只繁育体系在群体中大量积累、快速放大,导致后代近交快速上升、选育进展缓慢,对育种带来巨大的负面影响[1-3]。真实、完整、准确的系谱是实现种猪自繁育、自更新,摆脱外国种猪公司对国内生猪发展的制约、培育出中国自有种猪的基础之一[4-5]。
生产中误配精液、仔猪未记录调栏、耳牌脱落、耳号识读录入错误等都会造成猪只个体信息及父母信息记录错误,从而导致记录的系谱错误或缺失[6],目前我国畜群中系谱错误率一般在5%~30%之间[7-9]。新投产猪场引种建群时需引入大批同代次猪只,种猪培育及场间联合育种也需要从外部群体引种以避免因闭群繁育、猪群高度近交带来的后代性能退化和畸形频发,这些引种都需要真实准确的系谱来保证育种效果[10]。引种猪只通常由供种场提供系谱,引种猪只常见的上溯三代的系谱无法完全体现引种猪群的潜在近交。近年来,随场间联合育种和外引种猪逐渐增多,加之猪场现场生产管理难度大,猪群系谱缺失或错漏的情况频发,对育种工作造成一定影响,对核心猪群进行系谱校正已成为选育自有纯种猪的基础。
目前,用于亲子鉴定的方法根据原理可分为排除法、似然法和基因重构法3类[7],家畜中多使用PCR法对待鉴定个体的数个位点进行基因分型,再使用排除法或似然法对成对的亲子关系进行判断[8,11-12]。这种方法的成本较低,但需提前筛选数个高多态性位点[13],且仅能对亲子关系的个体对进行分析,实际生产中通常需要对父母未知的同代次群体重构系谱,少数位点进行亲子鉴定的方法难以满足这种需求。现有研究多是使用模拟数据或是对数十至数百个样本的小群体进行鉴定[7,13],对大规模真实生产群体的研究较少。
本研究使用高密度SNP芯片获得的大量标记推测共祖片段,对缺乏父母基因型的同代次长白猪和杜洛克猪群体重构系谱,并与记录的真实系谱相比较,填补使用基因组信息重构多品种、大规模真实生产猪群的空白。
1 材料与方法
1.1 试验材料
本研究选取四川某猪场2017—2021年出生的1 471头曾祖代纯种猪,其中986头为杜洛克猪,485头为长白猪,所有个体均为从国外某种猪公司单一来源引种后本场纯繁后代。两品种记录系谱共包含2 342头纯种猪,其中包括1 493头杜洛克猪和849头长白猪,系谱记录时间为2015—2021年,系谱记录最大世代数为8代,芯片分型个体平均记录系谱代数约为5代。
1.2 基因型数据及质控
采集1 471头纯种猪的耳组织样本,乙醇固定后寄送至纽勤生物科技(上海)有限公司,使用Illumina GeneSeek GGP Porcine 50K 芯片进行分型,每一个体基因组数据包括50 697个SNPs位点。该芯片基于Sscrofa 10.2全基因组信息进行设计,根据Sscrofa11.1全基因组信息将其SNPs位点所对应的MAP文件进行更新,更新后的MAP文件用于后续分析。使用PLINK v1.90b6.21对SNPs分型数据文件进行质量控制,剔除SNPs位点检出率<0.9、次等位基因频率<0.05、位于性染色体上或染色体位置未知的SNPs位点,剔除SNPs检出率<0.9的个体。
1.3 家系划分
种猪生产中一般采用“一公多母”的生产方式,通常种公猪的后代数远远多于种母猪,因此以公猪作为划分家系的主要依据。使用系谱计算亲缘关系时,将有同一曾祖父的个体视为一个家系。使用基因组信息计算亲缘关系时,将可推断为同一曾祖父的或有四级以内亲属关系且有可追溯共同祖先的个体划分为一个家系;当父母来自不同家系或近交等原因导致某一个体可被划入多个家系时,将个体归为其父系所在家系。
1.4 亲缘相关系数估计
1.4.1 同源片段推断 两个体组成的一组个体对上的每个SNP位点对应的等位基因有两种同源类型,一种为状态同源,另一种为血缘同源。状态同源(identical by state, IBS)片段,即状态相同片段,指两个体间基因片段的核苷酸序列相同。状态同源片段仅考虑等位基因本身的相似性,这种相似性是遗传过程中累积起来的突变导致的,可能是源于某个共同的祖先,也有可能仅来自于随机突变。血缘同源(identity by descent, IBD)片段,即血缘相同片段,又称共祖片段(common ancestor fragment),指两个体间核苷酸序列相同的基因片段来自某一个共同祖先。在不发生重组的遗传过程中,子代通过配子从父本和母本处分别继承一组同源染色体,实际遗传过程中由于同源染色体非姐妹染色单体重组等原因,子代的单条染色体与父母不完全一致,根据连锁不平衡将多个连续相同且来自同一祖先的等位基因序列连接为一个共祖片段。根据个体对间染色体片段共享等位基因情况将染色体划分为IBD0、IBD1和IBD2片段,分别代表该段染色体上两个体间每个SNP位点的2个等位基因中有0个、1个或2个血缘相同。使用PLINK v1.90b6.21构建IBS遗传距离矩阵,用于衡量个体间相似性、评价亲缘关系。使用KING 2.2.7 估计群体内每一组个体对间IBD0、IBD1和IBD2片段位置及比例,用于推断个体间亲缘关系。
1.4.2 SNPs亲缘相关系数计算 根据Chen等[14]提出的公式,在剔除检出率低、缺失及无法确定染色体位置的SNPs位点后,对剩余常染色体上的SNPs位点,根据位点间相似性估计个体间基因组亲缘相关系数,计算公式如下所示:

(1)

(2)

1.4.3 系谱亲缘相关系数计算 将个体间的亲缘相关系数定义为两个个体因具有共同祖先而造成的加性基因效应间的相关,个体x从其父亲s和母亲d处分别获得的配子中随机抽取一对等位基因,这对等位基因在两配子中是同源的概率即为个体x自身近交系数,用F表示,即:
式中,axsxd代表个体x父母s、d间亲缘相关系数,由公式可知后代的近交系数为其父母间亲缘相关系数的一半,个体与自身的加性遗传相关为1加个体本身的近交系数。因此可以通过假设后代的方式计算任意两个体间的亲缘相关系数。使用加性遗传效应计算个体间亲缘相关系数的公式如下所示:
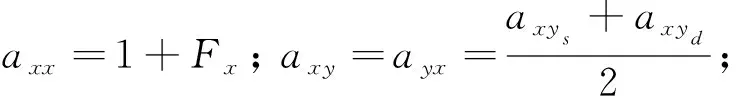
式中,a代表个体间亲缘相关系数,F代表个体自身近交,x、y分别代表待计算亲缘相关系数的两个个体,xs、xd分别代表个体x的父亲和母亲,ys、yd分别代表个体y的父亲和母亲,rsd代表个体s、d间亲缘相关系数。
根据配种及分娩记录确定系谱,系谱中包括个体号、父亲个体号及母亲个体号三列,使用python 3.8编写脚本计算系谱中所有个体的加性遗传相关矩阵及个体系谱代次。矩阵每一行或每一列均代表一个个体,矩阵中每一元素代表对应行和列的两个个体间加性遗传相关,即两个个体间的亲缘相关系数;个体系谱代次定义为从该个体向祖先可追溯到的最远通径上的个体数量,亦即该个体可追溯系谱的深度。
2 结 果
2.1 描述性统计
2.1.1 系谱描述性统计 全部1 471头个体均有记录系谱,以芯片分型个体为起点向上追溯系谱获得群体系谱记录,系谱记录中共包含2 342头猪,出生年份在2015—2021年,平均系谱深度为5.47代。其中杜洛克猪群体共986头,平均系谱深度5.53代;长白猪群体共485头,平均系谱深度5.36代。两品种群体性别、出生年份、性别及相应系谱代次见表1。

表1 两品种群体系谱描述性统计
2.1.2 基因组信息描述性统计 对SNP芯片获得基因组分型数据进行质控后,全部1 471头芯片分型个体均通过质控,45 784个SNPs通过质控,通过质控的位点在各常染色体上的分布情况见图1,图中横坐标代表染色体编号,纵坐标代表染色体位置,灰度表示SNPs在每条染色体上分布疏密程度。由图1可见,通过质控的SNPs位点覆盖全部常染色体,但在常染色体两端分布相对密集,这一结果与染色体重组热点区域分布及芯片设计位点分布相符。
2.2 系谱亲缘关系分析
利用记录系谱计算加性遗传相关矩阵得到群体内个体间亲缘相关系数,根据个体间亲缘相关系数进行聚类,构建系谱亲缘关系聚类图。由图2可知,长白群体共存在39个家系,大部分家系留种数量较为均衡,少数家系个体较多,个别家系在选育过程中留种较少,因此消失。由图3可知,杜洛克群体共存在41个家系,相较长白群体家系间留种更均衡,但家系间亲缘关系更近。
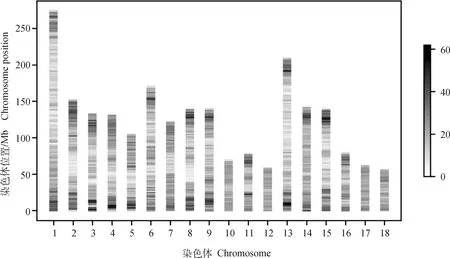
图1 质控后常染色体SNPs分布Fig.1 Distribution of SNPs on autosomes after quality control
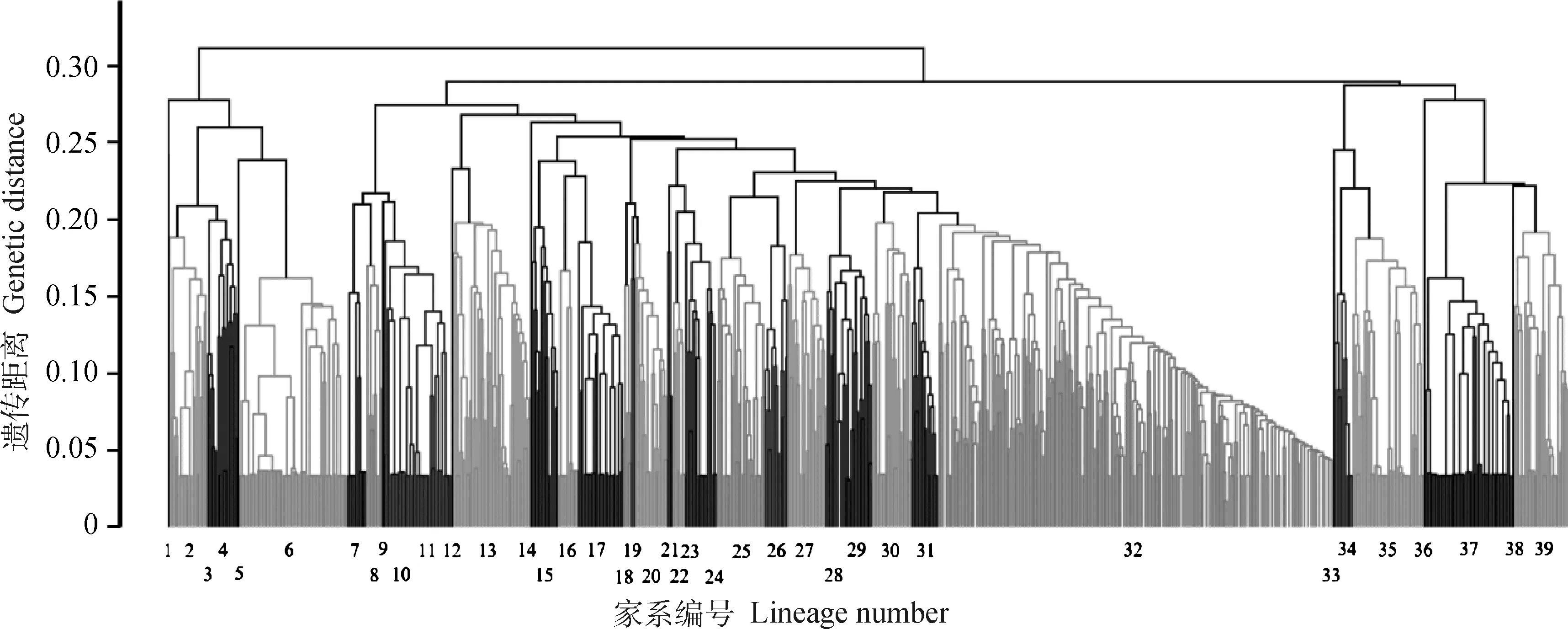
进化树中竖线代表个体,以灰度区分相邻家系,下图同The vertical lines in the evolutionary tree represent individuals, and neighboring lineages are distinguished by grayscale, the same as below图2 长白系谱亲缘系数聚类图Fig.2 Landrace genealogical kinship coefficient clustering map
2.3 基因组亲缘关系
根据群体中每组个体对间IBD1片段和IBD2片段占比做散点图,根据Chen等[14]提出的方法,用IBD1片段和IBD2片段比例判断个体对间亲缘关系,以不同颜色标记不同亲缘关系的个体对,如图4所示。
1 471头芯片分型种猪共推断出29 476对亲缘关系,亲缘关系按个体间联系紧密程度不同,从近到远依次划分为重复分型个体或同卵双胞胎、亲子关系、全同胞关系、半同胞关系、三级亲属关系、四级亲属关系和无关个体,图4中仅展示了三级及以上亲缘关系个体对。亲子及三级亲属关系个体对间几乎不存在IBD2片段,即几乎不存在同一对等位基因的两个位点均为同源的情况;重复分型个体或同卵双胞胎几乎全部位点均为同源,即几乎全部位点均相同,因此IBD2接近1而IBD1接近0;全同胞或父母间存在亲缘关系的半同胞个体对介于两者之间,既存在IBD2片段也存在IBD1片段,这一结果与预期和IBD片段估计原理一致,证明推断IBD片段在该群体中可有效划分个体对间亲缘关系。
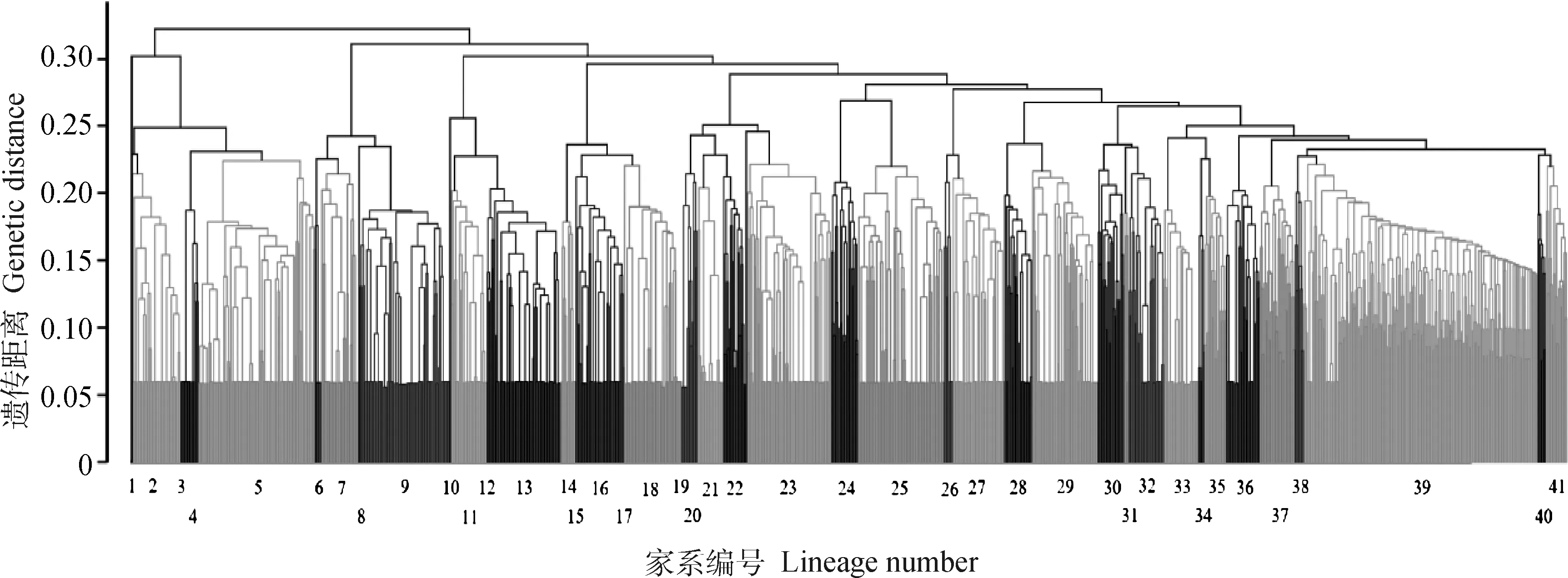
图3 杜洛克系谱亲缘系数聚类图Fig.3 Duroc genealogical kinship coefficient clustering map

图4 IBD1-IBD2比例分布图Fig.4 IBD1-IBD2 scale distribution
图5为某个体A与其全同胞个体间IBD片段在常染色体上的分布情况,图6为同一个体A与其父亲间IBD片段在常染色体上的分布情况。可见IBD1和IBD2片段在常染色体上分布并不均匀,在各常染色体两端分布较密集,靠近中段着丝粒处分布则较为稀疏,这一现象在亲子关系对间更为明显。IBD片段的分布情况除受群体遗传结构影响外,主要与染色体重组区域有关,Dimitromanolakis等[15]认为这种不均匀分布与染色体上存在重组冷热区有关,某一区域遗传结构相对稳定则较少发生重组,因此IBD片段在该段染色体上分布也会较为稀疏。其次,IBD片段的分布也与芯片设计位点有关,SNP芯片结果在质控后分布不均匀,位点分布较密集区域更易出现IBD片段。
2.4 重构系谱
根据推断的亲属关系重构系谱,按个体间亲属关系进行聚类,由于本研究所用群体内杜洛克猪和长白猪均为纯种且不存在杂交,因此混合群体在计算个体间亲缘关系及构建家系图时自然分成两组,对比个体毛色可轻易推断每组家系所属品种,故对两品种分别构建系谱。
使用个体间亲子关系及二级亲属关系绘制群体内亲缘关系图,使用基因组信息构建的杜洛克猪和长白猪系谱图如图7和图8所示,可见杜洛克群体间关系更为紧密,长白群体存在较多小家系。基因组信息共将杜洛克猪分为32个家系,平均代次为4.1代,将长白猪分为38个家系,平均代次为3.6代,与系谱记录基本一致。

图5 全同胞个体对间IBD片段分布Fig.5 Distribution of IBD sharing between full-sibling pairs

图6 亲子对间IBD片段分布Fig.6 Distribution of IBD sharing between parent-offspring pairs

图中圆点代表个体,黑线相连两个体为亲子关系,灰线相连两个体为二级亲属关系,下图同The dots in the figure represent individuals, the two dots connected by black line is parent-offspring, and the two dots connected by gray line is 2nd-degree, the same as below图7 基因组构建杜洛克猪家系图Fig.7 Genomically constructed Duroc pig lineage map
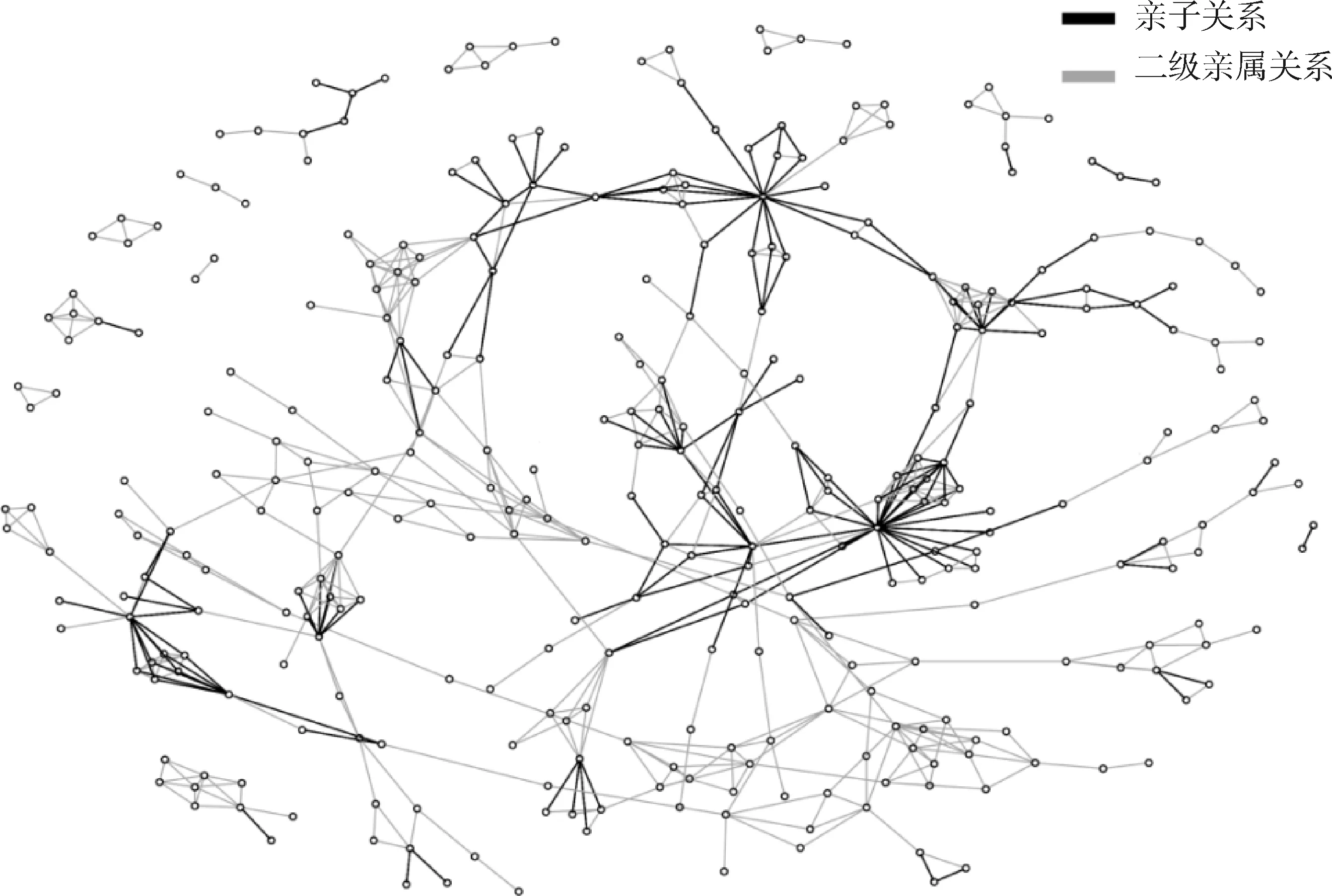
图8 基因组构建长白猪家系图Fig.8 Genomically constructed Landrace pig lineage map
为确定基因组信息重构系谱准确性,通过严格控制选取群体的配种、分娩、调栏及采样过程,保证所记录的系谱及采样分型结果准确无误,获得115头有芯片分型信息且有准确记录系谱的个体作为验证群体,以芯片分型猪只为起点向上追溯三代系谱,得到包含360头猪的系谱记录。进行芯片分型的115头猪个体间IBS遗传距离在0.132~0.475之间。根据个体间共享IBS和IBD片段比例矩阵绘制的热力图如图9所示,可见IBD矩阵较IBS矩阵颜色区分度更大,即对有亲缘关系的个体和无关个体的区分度更好。同时,从IBD片段矩阵构建的热力图中可以显著观察到由同父异母的半同胞关系形成的橙红色区域。
使用IBD片段法推断所选群体内个体间基因组亲缘关系,并与记录系谱相比较,不同亲缘关系的推断数量及推断比例见表2。由表2可见,基于共祖片段法利用基因组信息共推断出选取群体702对亲缘关系,全部亲子关系和全同胞关系均可被准确推断,同时共祖法能额外推断出个体间系谱未记录的三级和四级亲缘关系。

对角线:个体自身IBD与IBS片段相似程度;对角线上方:个体间共享IBD片段比例;对角线下方:个体间共享IBS片段比例。两个体间亲缘关系越近则共享比例越大,越接近红色,反之则接近蓝色。图中数字代表个体号Diagonal: The degree of similarity between individual IBD and IBS; Above the diagonal: Proportion of IBD shared between individuals; Below the diagonal: Proportion of IBS shared between individuals. The closer the kinship between two individuals the greater the shared ratio, the closer to red, and vice versa, the closer to blue. The numbers in the figure represent individual numbers图9 共享IBD-IBS片段比例热力图Fig.9 Sharing IBD-IBS segment scale heat map
以其中一长白家系为例,该家系共包括三代21头纯种长白猪,其中19头有芯片分型信息,各世代间无交叉且均有系谱记录。使用IBD片段法推断该家系个体间基因组亲缘关系,各亲缘关系IBD1、IBD2及基因组共享IBD比例见表3。基于基因组亲缘关系重构系谱图和基于配种及分娩等生产记录构建系谱图如图10所示。
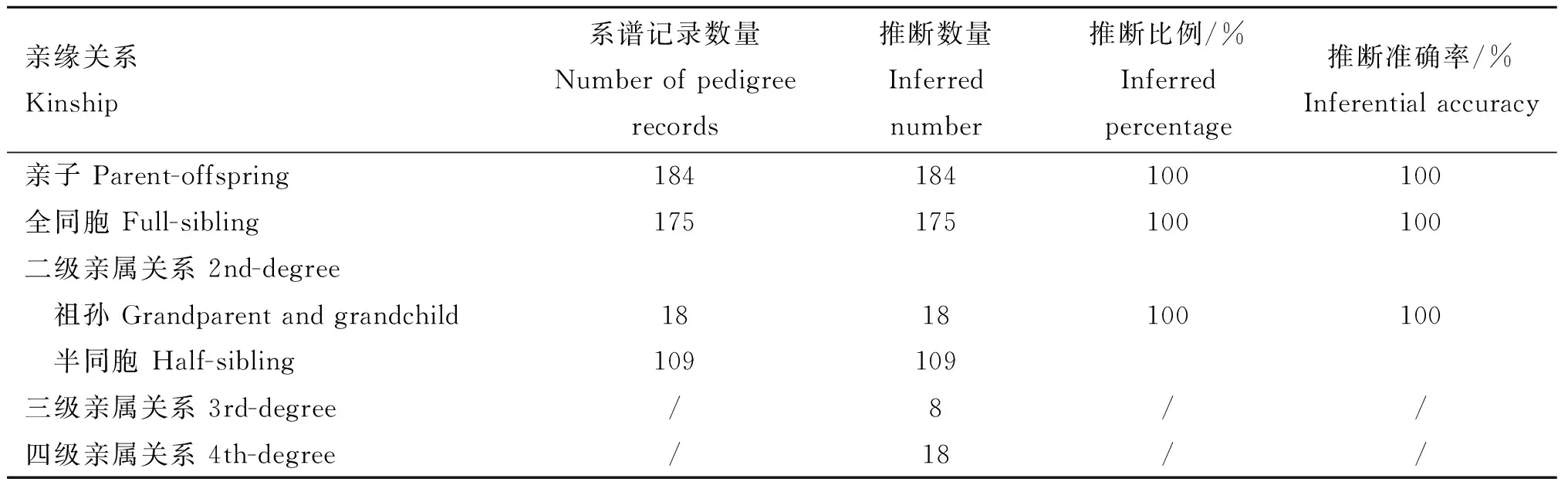
表2 基因组推断亲缘关系
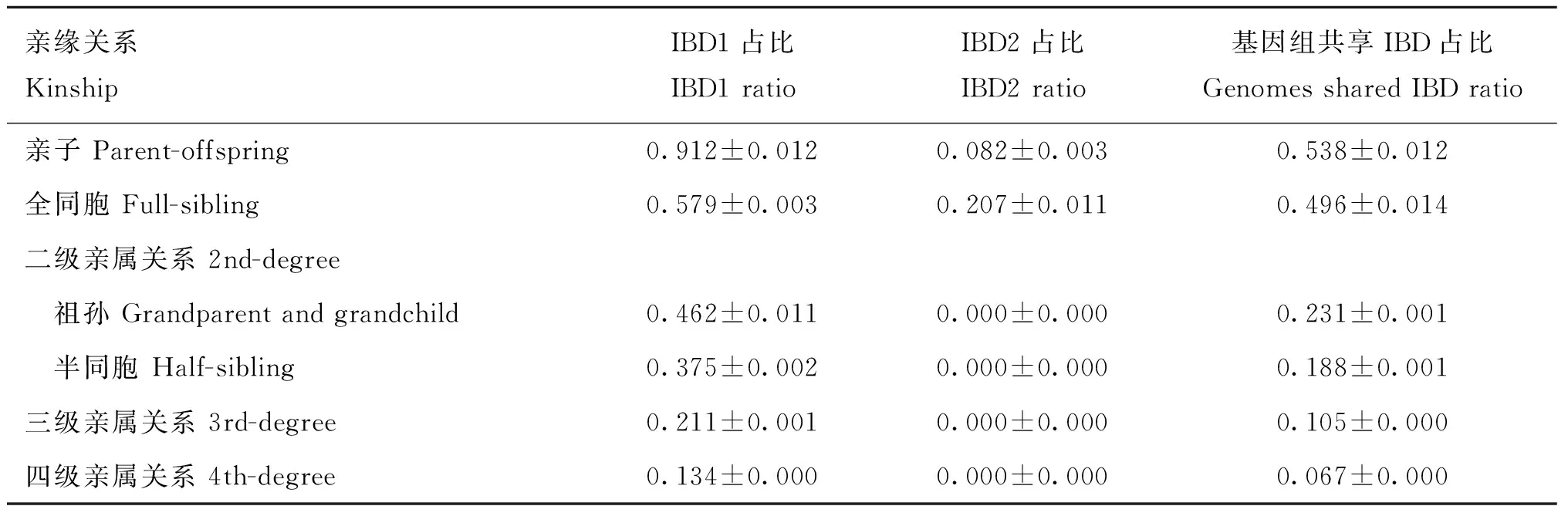
表3 长白家系内不同亲缘关系IBD占比
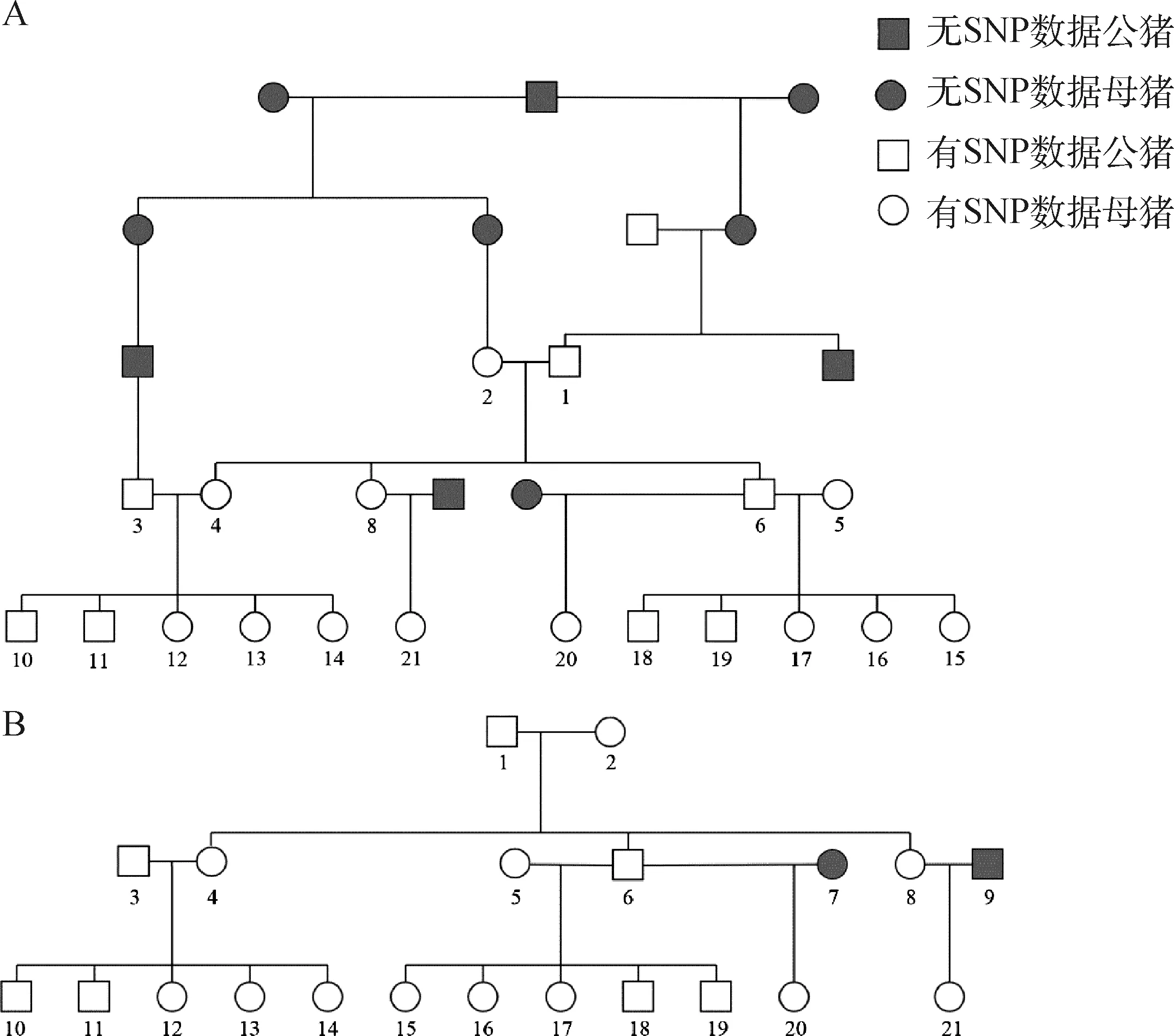
A.长白家系基因组重构系谱;B.长白家系记录系谱。图中数字代表个体号A. Genomic information-based pedigree of the Landrace lineage; B. Recorded pedigree of the Landrace lineage. The numbers in the figure represent individual numbers图10 长白家系系谱Fig.10 Pedigree of the Landrace lineage
可见基因组信息可推断出全部亲子、全同胞及二级亲属关系,且推断结果与记录系谱一致,证明其准确性。除此之外,基因组信息还可额外推断出常见三代系谱未记录的亲缘关系,如基因组推测个体3与个体4有三级亲属关系,但其在以个体10~21为起点向上追溯三代的系谱内没有可见亲缘关系,继续向上追溯系谱后发现个体3与个体4有共同祖父,亦可体现基因组重构系谱的准确性。
2.5 系谱与基因组亲缘关系比较
根据个体间基因组亲缘关系矩阵和系谱亲缘关系矩阵绘制上述115头个体组成的验证群体热力图如图11所示,可见基因组亲缘关系矩阵较系谱亲缘关系矩阵颜色区分度更大,即对有亲缘关系的个体和无关个体的区分度更好。从亲缘关系矩阵热力图中可以显著观察到没有系谱记录关联的个体间也存在基因组关联,即图11对角线下方中个体1~14与个体15~66间出现的浅蓝色方格。同时,基因组亲缘关系矩阵也可以体现出全同胞个体间真实的遗传相关,而不是系谱亲缘关系矩阵中平均的遗传相关,如图11中个体20~24对角线下方矩阵深浅不一的方格。

对角线:个体与自身亲缘关系;对角线上方:个体间系谱亲缘关系矩阵;对角线下方:个体间基因组亲缘关系矩阵。两个体间亲缘关系越近越接近红色,反之则接近蓝色。图中数字代表个体号Diagonal: The kinship of an individual to itself; Above the diagonal: Pedigree kinship matrix between individuals; Below the diagonal: Genomic kinship matrix between individuals. The closer the kinship between two individuals the closer to red, and vice versa, the closer to blue. The numbers in the figure represent individual numbers图11 亲缘关系矩阵热力图Fig.11 Relationships matrix heat map
3 讨 论
系谱是种猪育种的重要信息来源之一,建立完整、准确的系谱对确定种猪真实身份、提高育种值估计准确性、实现科学引种选育和选配有重要价值,可以进一步加快猪的遗传进展。然而系谱错误在遗传研究和育种生产中普遍存在,畜群中系谱错误率一般在5%~30%之间[7-9],对育种工作造成巨大影响。
畜禽中通常根据数个至数百个SNPs位点或微卫星标记使用似然法确定亲子关系,如余国春[13]利用120个SNPs位点确定24头藏猪及二元猪群体间亲子关系,Van Ba等[11]利用19个STR标记确定9个品种639头猪亲子关系。但基于亲子间相似性的似然法仅能确定亲本与后代间的亲缘关系[16],因生产群体常是多代次混杂的复杂群体遗传结构,且似然法判断亲子间关系时计算方法较为复杂[17],因此限制了其在大规模真实生产群体中的使用。
使用高密度SNP标记重构系谱的方法已经广泛应用于法医[18-19]和人类群体遗传结构[20-22]分析,但其在畜禽中的应用尚在探索阶段,通常使用模拟数据探究方法可行性,如周子文等[23]利用模拟的高密度SNP标记建立个体亲缘关系矩阵用于分析个体间遗传关联;使用真实数据的研究主要集中在地方品种保种群体[24-25]和奶牛[26-27]中,这些群体育种历史较长,记录详细、群体小、世代间隔大,遗传背景相对清晰,而对大规模多代次的商业化生产群体进行系谱重构的相关研究较少,特别是对外三元猪的研究少之又少。
本研究通过推断共祖(IBD)片段的方法分析真实多品种生产群体间亲缘关系,IBD片段分布情况与前人研究相近[28],但从图5和图6的结果中可以看到,使用KING推断出的IBD片段存在片段破裂的情况,与前人研究结果类似[29]。这种IBD片段特别是IBD1片段破裂的情况可能与个别位点发生突变、缺失或分型错误有关[15],同时也与芯片密度和芯片上位点分布情况有关[30],Bjelland等[28]认为连接相邻IBD片段有助于提高亲缘关系推断准确性,Zhou等[31]认为使用摩尔根距离或碱基个数确定IBD片段也会影响推断效果。
同时,群体平均近交水平也对IBD片段推断效果有一定影响,近交会增加群体内IBD片段比例,如个体67~115所组成的群体由于多代闭锁繁育,较个体15~66组成的群体近交水平更高,因此个体间推测亲缘关系系统性偏高,在图9的IBS-IBD热力图中可见高近交群体颜色相较于低近交群体更红,即平均亲缘关系更近,这一现象与前人研究结果一致[32-34]。
基因组亲缘关系矩阵计算需要使用个体SNP信息,需要对参与计算的个体进行SNP分型,相较于仅使用系谱计算的系谱亲缘关系矩阵成本较高;且由于SNP信息远远多于系谱信息,基因组亲缘关系矩阵计算时算力消耗大、计算时间长。但基因组亲缘关系矩阵除可体现系谱记录无关联个体间遗传相关、全同胞及半同胞个体间遗传相关差异外,还能在一定程度上反映群体整体近交情况。对生产核心群及外引种猪亲本进行SNP分型并构建基因组亲缘关系矩阵,与后代仔猪重构亲本系谱后的系谱亲缘关系矩阵进行合并,有利于以较低成本在普遍存在近交及系谱记录不全的生产猪群中计算育种值并开展选育选配工作。
本研究使用基因组推断亲缘关系重构的基因组系谱与记录系谱结果基本相符,但存在少量差异。系谱重构的准确率受多种因素影响,配种及分娩等生产记录、生产操作和管理水平、重构系谱所用位点类型和数量、使用的系谱重构方法等都对系谱重构准确性有影响。
重构系谱需要根据生产记录的配种、分娩记录和性别栋舍等信息辅助判断亲子对是否准确,通过每对亲子对的验证确定整个重构系谱的准确性。没有出生记录的猪由于难以确定可能的父母信息,在父母双方都没有基因分型数据的情况下只能通过全同胞、半同胞等亲缘关系推断可能的父母,且由于缺少出生时间、栋舍等信息,仅依据远缘亲属关系很难匹配出正确的父母关系。例如某一个体仅有其本身及其父亲基因分型数据,由于生产上同一公猪会同时与配多头母猪,因此这种情况下也很难确定待构建系谱的仔猪母亲,继而难以构建完整系谱。
计算亲子间关系的方法也是影响系谱校正的准确性的重要因素。例如似然法通过计算非父排除概率判断亲子关系,将非父排除概率低于某一阈值的亲子对判定为真实亲子对[35-36]。由于不同群体平均的次等位基因频率不同,计算得到的平均错误率也不同[6],因而这一阈值通常根据错误率的群体经验分布确定,真实亲子对与随机个体对间错误率的分布是连续的[26],极少数个体对会被错误判定关系,因此这一阈值的确定会影响系谱校正的准确性。以严格规范的生产操作为主,辅以对少部分丢失记录或记录不清晰的猪重构系谱将是保证系谱真实准确的有效途径。
4 结 论
本试验使用Illumina Porcine 50K 芯片质控后的45 784个SNPs位点,应用IBD片段法分析四川地区1 471头杜洛克猪和长白猪群体混合群体的群体内个体对间亲缘关系并重构系谱,推测的亲缘关系及重构系谱与生产记录相符。应用IBD片段法分析中高密度SNP标记数据推测亲缘关系并重构系谱的方法可应用于杜洛克猪和长白猪实际生产育种中,为其选种选配、计算育种值及GWAS挖掘等工作提供基础,进一步加快种猪改良进程。

