基于GASF与MSVM的放射性核素识别方法
周思益 张江梅 刘灏霖,3 冯兴华 张草林
(1.西南科技大学信息工程学院 四川绵阳 621010;2.核废物与环境安全国防重点学科实验室 四川绵阳 621010;
3.中国科学技术大学自动化系 合肥 230026)
核素识别技术是保障核安全的一项基本技术,对于核安全发展具有重要的现实意义[1]。核素识别是指在真实探测环境中利用探测器接收放射性同位素发出的γ射线并生成能谱数据,对能谱进行分析实现对核素种类的识别。传统核素识别的核心思想是寻找能谱中的全能峰,在能量刻度基础上与核素库中核素能量进行对比,得到核素判别信息[2]。但是基于传统寻峰核素识别方法在真实能谱衰减(环境噪声和探测距离加大造成能谱被淹没)环境中误差较大,识别率较低,不能得到很好的识别效果。近年来,随着机器学习的发展[2],基于机器学习的新兴核素识别方法逐渐出现,主要有基于神经网络[3-5]、AdaBoost集成学习理论[6]、模糊理论[7]和基于信息理论空间投影[8]等方法,这些方法虽然在一定程度上提高了核素识别的准确率,但不能有效处理重叠峰,且难以准确消除“假峰”。此外,大多数的研究者都是直接将一维的能谱数据作为模型输入去寻找核素能谱的有效特征,有效特征不易于提取,而采用全能峰面积、位置等作为输入特征,不能有效克服噪声影响。
针对以上问题,笔者从全谱分析的角度进行核素识别,将原始能谱视作一维序列,提出基于分段聚合近似的格拉姆角和场(Gram angle sum field,GASF)算法将一维能谱数据二维化,从新的角度、新的维度去认识和探究核素能谱信息,利用双向二维主成分分析(Bidirectional two dimensional principal component analysis,2D-2D-PCA)对二维能谱进行空间投影,获取不同核素能谱的特征空间,实现对核素的识别,提高核素识别准确率。
1 算法分析
1.1 基于GASF的一维数据二维化方法
格拉姆角场(Gramian matrix field,GAF)是基于格拉姆矩阵提出的一种序列转化方法[9]。GAF的基本思路是将经典的笛卡尔坐标系下的序列转移到极坐标系中,利用三角函数可将一维的序列转化为二维的GAF矩阵,实现一维序列二维化。对于一个待转化的核素能谱序列Y={y1,y2,…yN},将能谱序列中的每一个值通过式(1)进行处理,使处理后的能谱序列中的每一个值处于[-1,1]。
式中:φi表示反三角化后的角度且φ∈[0,π];ri表示第i个道址的半径;N表示道址总数。因此,能谱中的计数值会随着道址数的不断增加而在极坐标上发生弯曲。
通过利用每个点之间的角度和格拉姆矩阵来进行GAF定义,格拉姆矩阵如式(3)所示:
式中I=[1,1,…1],是一个单位行向量。
通过将两个数的内积公式定义为如式(4)所示的形式:
GAF根据采用的三角函数是余弦函数还是正弦函数会分别得到两种不同的格拉姆场,分别是格拉姆角和场(GASF)和格拉姆角差场,由于GASF可实现逆变换,故本文采用余弦函数构造GASF。
通过GASF矩阵就可以将一维的能谱序列转化为二维形式,同时保留了能谱序列中所有的关键信息。以60Co的能谱序列为例,采用GASF对其实现二维化,将结果以GASF热力图形式进行展示,整个过程如图1所示。
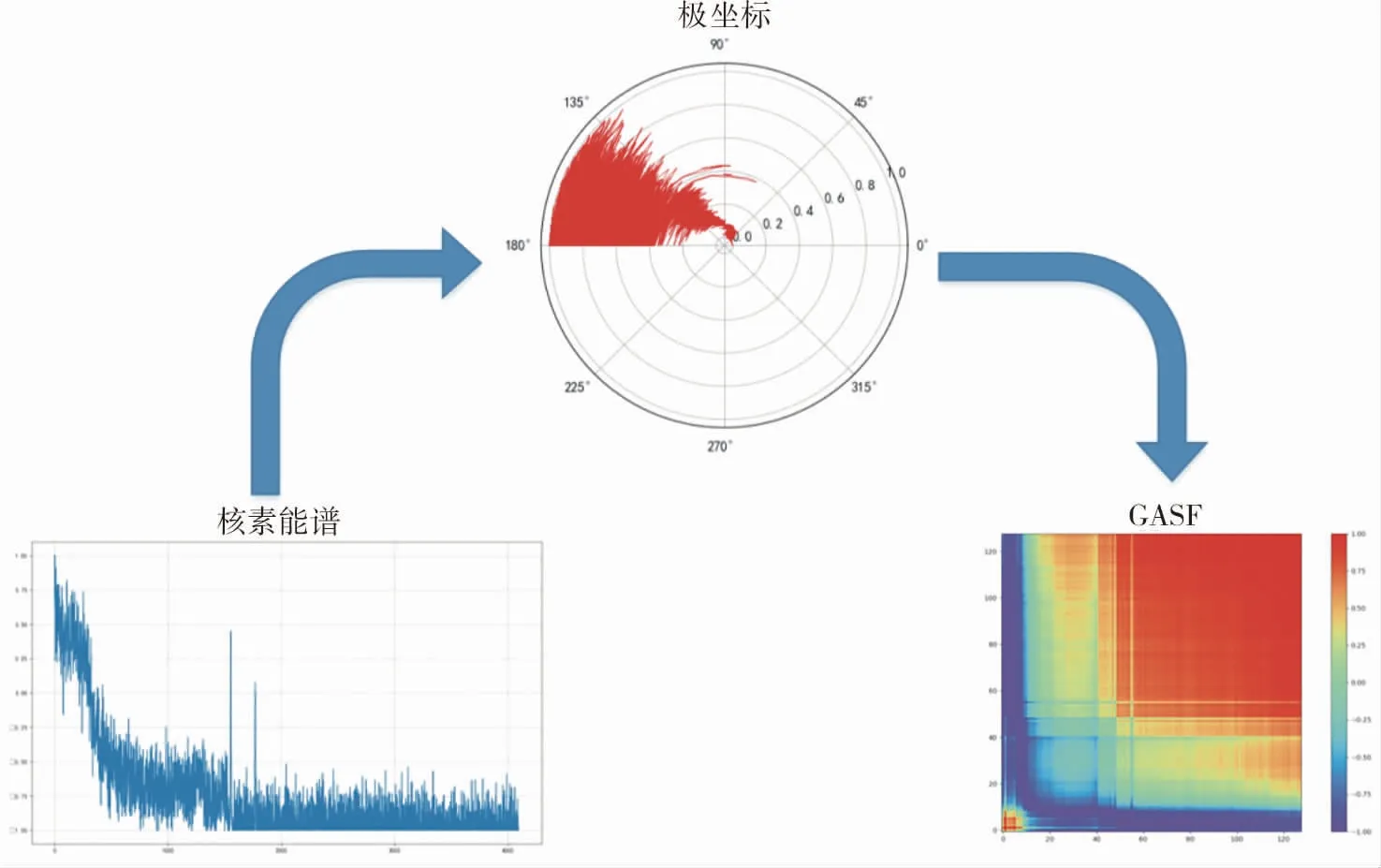
图1 基于GASF的核素能谱二维化过程图Fig.1 Two-dimensional process diagram of nuclide energy spectrum based on GASF
1.2 2D-PCA理论原理
1.2.1 1D-PCA理论原理
一维主成分分析(One-dimensional principal component analysis,1D-PCA)即传统的主成分分析方法,是最重要和最常用的一种降维方式,它通过一个线性变换将原始空间中的样本投影到新的维度空间中[10]。1D-PCA只适合用于一维数据的处理,其思想是通过线性变换找一组最优的单位正交基(即主成分),用它们的线性组合来重建原样本,并使重建后的样本和原样本的误差最小。
1D-PCA基本原理如下:假设存在N组1×M维数据样本Xi(i=1,2,…N),对其进行1D-PCA等价于寻找样本集的协方差矩阵C=XXT的前n(1≤n≤M)个特征值对应的特征向量矩阵W,W即为样本数据Xi进行空间变换的最佳投影矩阵。然后,对每一个样本Xi进行如下变换:
其中:Zi为Xi经过1D-PCA变换在新空间中超平面上的投影;Fi为Xi经过1D-PCA变换的特征向量。
1.2.2 2D-PCA理论原理
2D-PCA是1D-PCA的继承和发展。对于二维矩阵,1D-PCA在构造协方差矩阵时需要把样本矩阵变成一维向量,之后将所有样本向量构成一个大规模的协方差矩阵,然后求解此协方差矩阵的特征值及特征向量来构建映射矩阵。2D-PCA分析二维图像矩阵时,协方差矩阵是直接在图像矩阵上计算的,因此可以保留图像的空间结构信息,并且可以避免维数灾难和小样本问题,这也使得其在图像处理方面具有广泛的应用[11-12]。
2D-PCA的基本原理如下:假设X为n维列向量,将m×n维的矩阵A通过矩阵X映射到另一个子空间,即有:
式中Y为投影后的特征向量矩阵。
2D-PCA通过最大化投影数据的总离散度来寻找最优投影矩阵,其投影样本的总离散度可以由投影特征向量Y的协方差矩阵的轨迹来表征,即:
式中G=E[(A-EA)T(A-EA)],称为图像协方差矩阵。因此,式(9)可改写为:
图像协方差矩阵G通过简单的方式计算:
2D-PCA的投影准则是寻找d个投影轴X=[X1,X2,…Xd]使得准则J(X)最大,即有:
对于每个给定的样本Ai矩阵进行如下变换:
由此可得经过2D-PCA变换的图像特征向量矩阵Y。
1.2.3 2D-2D-PCA理论原理
2D-2D-PCA是对2D-PCA的进一步改进与发展[13]。2D-PCA可直接由二维数据构造协方差矩阵,避免了二维矩阵转化成一维向量时的计算负担。但2D-PCA仅仅只在水平方向上对二维数据进行了压缩降维,而忽略了竖直方向上的数据冗余性。为了解决这一问题,提出改进的2D-PCA方法,即2D-2D-PCA,其能够同时对原始的二维数据在水平和竖直方向进行2D-PCA降维,拥有良好的数据压缩和降维效果。2D-2D-PCA方法的实现过程如下:
(1)选取M个二维样本数据,每个样本数据的大小为m×n,同时计算M个样本数据的平均样本矩阵,分别从水平和竖直方向上构造2D-PCA的协方差矩阵G和C,其计算公式如下:
式中:矩阵G的维度为n×n;矩阵C的维度为m×m。
(2)分别求取矩阵G和矩阵C的特征值与特征向量。选取矩阵G的前p个最大特征值对应的特征向量构成行方向上的最佳投影矩阵X,矩阵大小为n×p,同时选取矩阵C的前q个最大特征值对应的特征向量构成列方向上的最佳投影矩阵Z,矩阵大小为q×m。
(3)对于每个给定的样本矩阵Ai在X和Z上进行压缩投影,获得经过2D-2D-PCA方法压缩降维的特征矩阵为:
从式(17)可知,使用2D-2D-PCA方法可将大小为m×n的样本数据投影为q×p维特征矩阵。2D-2D-PCA方法很好地压缩了原始二维数据中的维数据,同时将原始数据的关键信息也很好保留下来。
将2D-2D-PCA方法运用到放射性核素识别中,将一维能谱数据利用GASF方法进行二维化。将一个一维能谱序列视为宽平稳随机信号,将其表达为一个长度为n的序列:
对一维能谱序列y进行GASF后,得到对应GASF矩阵,其矩阵大小为n×n,通常n的大小为1 024,2 048或4 096,导致获得的GASF矩阵非常大,故采用2D-2D-PCA对其进行数据降维并提取特征。整个过程如图2所示。

图2 基于S变换与2D-2D-PCA的二维能谱特征提取Fig.2 Two-dimensional energy spectrum feature extraction based on S transform and 2D-2D-PCA
1.3 基于Mahalanobis距离的SVM分类器设计
支持向量机(SVM)是在统计学习理论的结构风险最小化理论基础上发展而来的一种机器学习方法[14-15],其具有较强的学习能力与泛化能力,能够有效解决小样本、非线性以及局部极值等问题,在处理二分类和多分类的问题上具有优异的准确性。但传统的SVM在进行分类时存在只抽取样本局部信息的缺点,忽略了不同样本间包含的属性信息,影响了SVM泛化能力的提高。而Mahalanobis距离判别是样本类间可分性度量分析的一种可靠方法[16],它能够将不同样本间包含的隐藏信息考虑进来,利用样本的全局信息寻找各种特征之间的联系,因此能够有效度量不同样本总体的相似程度。结合SVM和Mahalanobis提出基于Mahalanobis距离的SVM(MSVM)分类器,设计一种用于确定二维化核素能谱的类间的分类算法。该算法基本思想如下:
假设存在多个训练样本,其类间和类内协方差矩阵分别表示为Sb和Sw,同时设St=Sb+Sw为样本的总体协方差矩阵。在SVM方法中通过分类决策函数f(x)=wTx+b确定分类超平面,结合间隔最小原则,可以将其转化为二次优化问题,即:
基于考虑样本的全局信息,本文在优化问题中采用wTStw去代替wTw,将其代入式(19)获得的支持向量机则表示为:
在式(20)中,样本的总体协方差矩阵计算为St=E{[φ(x)-E(φ(x))][φ(x)T-E(φ(x)T)]},C为惩罚参数,ζi代表每个样本的松弛变量。
对式(20)进行拉格朗日变换[17],有:
式中ai为拉格朗日乘数。同时对马氏距离定义有:
将上式代入式(21)则基于Mahalanobis距离的支持向量机为:
从而得到基于Mahalanobis距离的支持向量机的决策函数:
式中s为支持向量的数目。
1.4 本文算法概述
根据前文所述,本文针对在真实能谱衰减环境中许多传统核素识别方法只利用部分能谱曲线、易受噪声影响及识别准确率较低等问题,设计基于GASF和2D-2D-PCA的γ能谱核素特征提取算法,同时设计基于MSVM的核素识别分类器,实现对核素能谱的高性能识别,整个算法过程如下:
(1)将原始γ能谱核素数据通过GASF转化为二维图像矩阵,并划分为训练集和测试集;
(2)对变换后的二维图像矩阵进行2D-2D-PCA变换,进行数据压缩和特征提取;
(3)设计基于Mahalanobis距离的SVM分类器,对训练数据进行训练并利用测试集进行验证,获得分类结果。
本文方法的整体系统框图如图3所示。
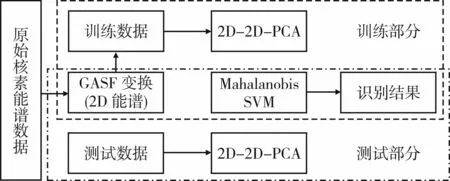
图3 算法整体系统框图Fig.3 Overall system block diagram of the algorithm
2 核素能谱识别
2.1 核素能谱数据获取及预处理
较多的核素样本数据是核素准确识别的前提。本文使用Geant 4软件,基于蒙特卡罗方法生成核素能谱,同时结合NaI探测器进行实验测试获取真实环境下的核素能谱作为核素能谱样本集。核素能谱样本主要包括60Co,137Cs,152Eu,60Co+137Cs,60Co+152Eu和137Cs+152Eu等6种单一类别核素以及混合核素。部分核素能谱样本如图4所示,可以看出所有的能谱数据都含有部分环境噪声干扰。笔者采用GASF算法和2D-2D-PCA算法对所获得的核素能谱进行特征提取,不同的核素能谱经过GASF变换后,获得的二维化图像如图5所示。可以看出,不同核素二维化后存在明显差异。由于经过S变换后的二维矩阵的维数过大,不仅增加计算负担,而且含有较多的冗余信息,故采用2D-2D-PCA对其进行数据压缩和降维,便于后续核素识别。
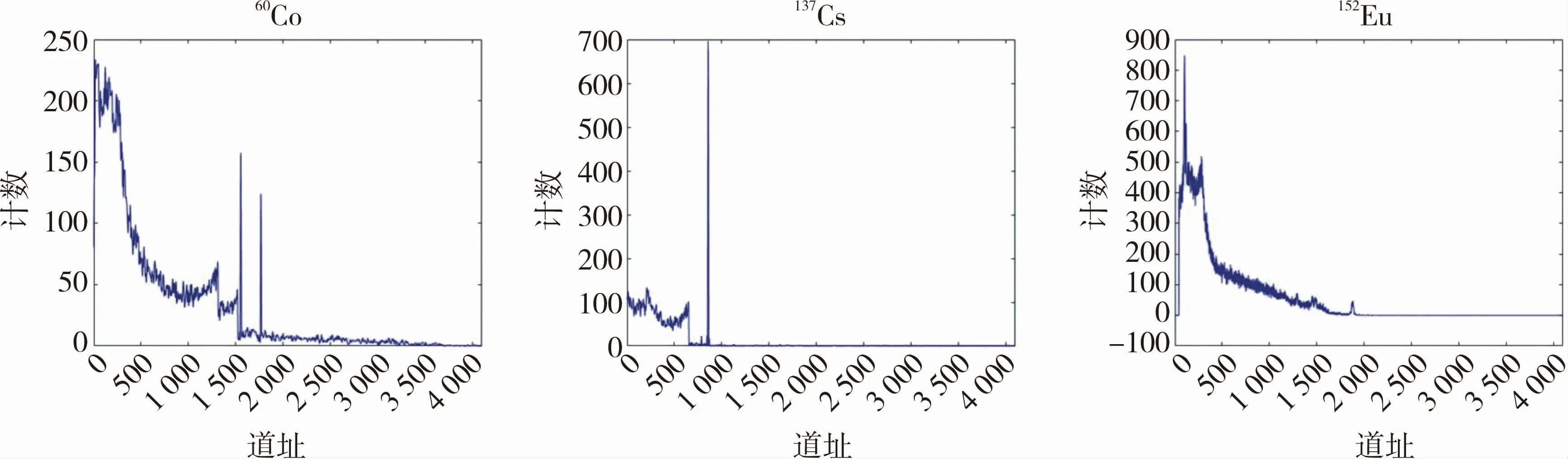
图4 不同的核素能谱样本Fig.4 Energy spectrum samples of different nuclides

图5 不同核素的GASF变换图Fig.5 GASF transform diagrams of different nuclides
2.2 核素识别模型训练
原始核素能谱数据经过预处理后,将得到的二维核素能谱数据按照7∶3的比例分成训练样本和测试样本,利用本文所设计的MSVM分类器对核素进行分类识别。此外,为获得MSVM 的最优参数设定,本文引入遗传算法(Genetic algorithm,GA)来寻找最佳参数。
将训练样本作为输入,通过GA来进行最优参数的设定,如图6所示,得到惩罚因子c和关联参数g的最优参数值,并且平均分类准确率大于95%,最佳分类准确率为100%。
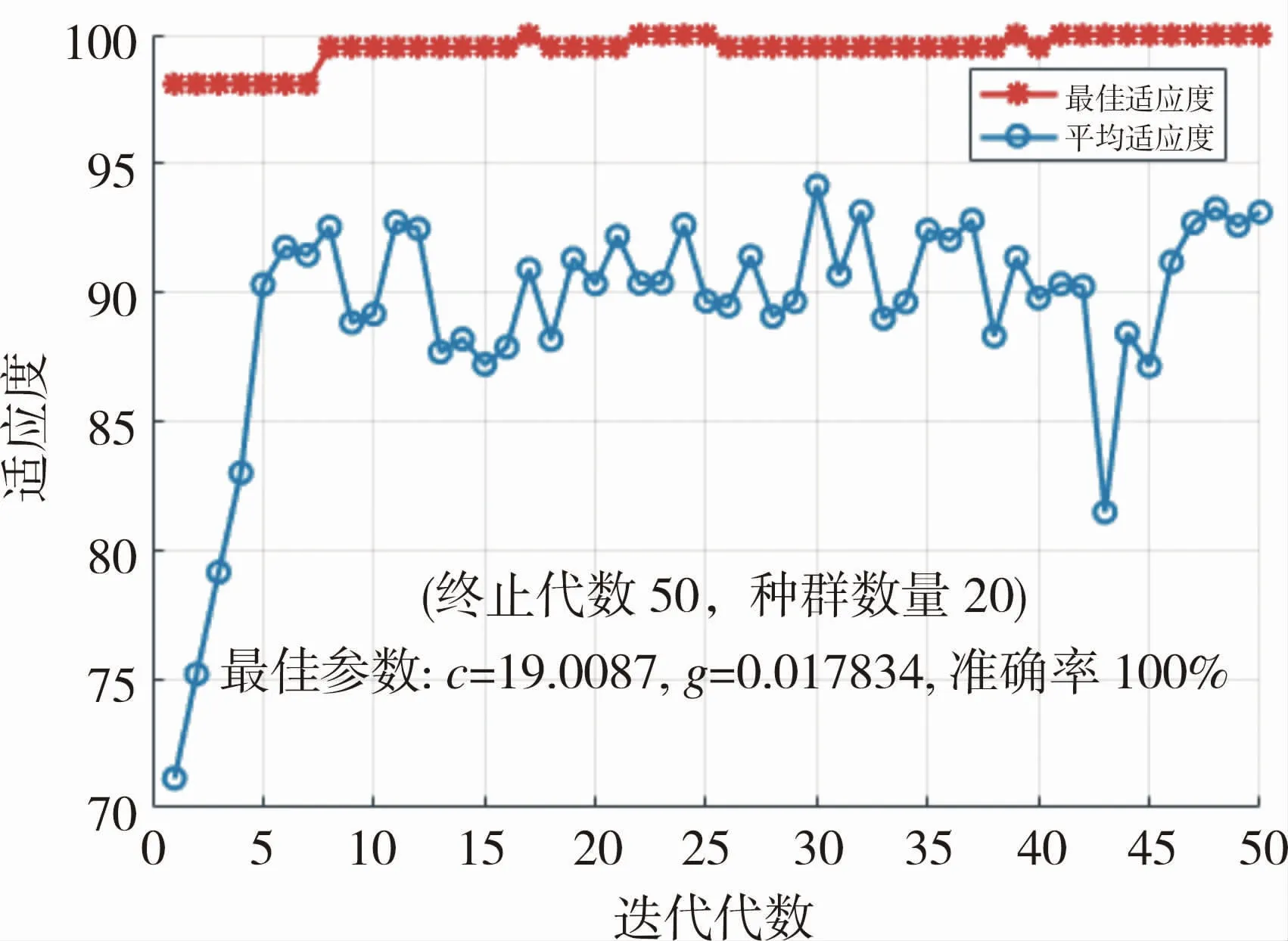
图6 GA参数寻优Fig.6 Parameter optimization by GA
2.3 仿真核素数据识别结果及分析
在该实验中,所使用的核素能谱数据都是基于Gent 4仿真软件获取的核素能谱仿真数据,仿真核素能谱识别包括单一核素的识别和混合核素的识别,将未参与模型训练的测试样本作为输入,利用训练好的MSVM对6种不同核素能谱数据进行识别,同时与传统的寻峰方法和基于奇异值分解的支持向量机方法[18]进行对比,核素识别结果如表1所示。
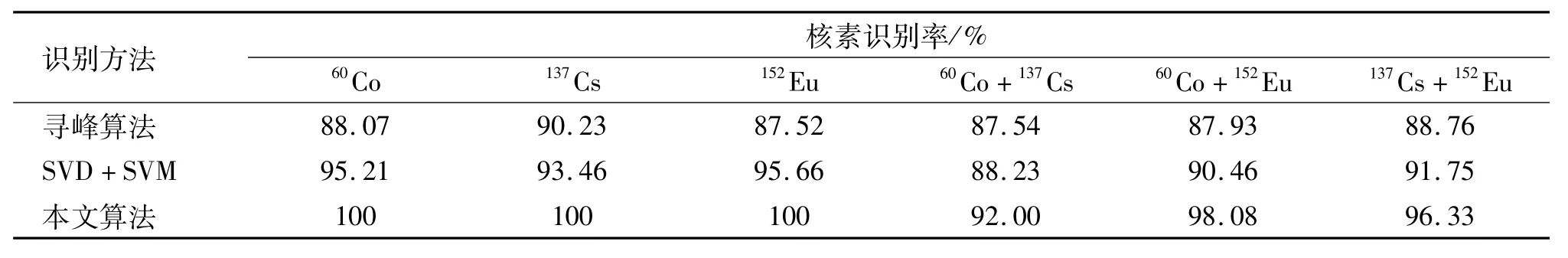
表1 仿真核素能谱识别结果Tab le 1 Recognition results of simulated nuclide energy spectrum
表1结果表明,本文算法对仿真的γ核素能谱具有良好的识别效果,能够准确识别单一核素和混合核素,同时模型训练速度快,能够自动进行参数寻优,在本文的数据集下,其平均识别速度在38 s内。寻峰算法和基于奇异值分解的支持向量机方法虽然对单一核素的识别效果较好,但对于混合核素的识别性能都有一定程度的下降。此外,传统的寻峰算法为获得较好的识别率需要调节各种参数,基于奇异值分解的支持向量机方法在进行奇异值分解时也需要选择合适的特征值才能获得较好的核素识别准确率。
2.4 真实环境下核素识别结果
在本节实验中,利用NaI(Tl)探测器(能量探测范围为50 keV~3 MeV,能量分辨率低于8%(137Cs,662 keV))获取不同探测距离的核素能谱数据,根据放射性源的放射强度、NaI(Tl)探测器的探测效率和成谱时间,将每次探测的测量时间定为每组60 s,同时为了减少NaI(Tl)探测器本身统计涨落的影响,在实验过程中会就同一个探测点进行10次重复实验,即在同一探测点测量10组核素能谱。此外,对同一类放射性源设定不同的探测距离,每次探测距离间隔为10 cm,探测距离范围为0~20 cm。通过实验验证本文算法对真实环境下采集的核素能谱的识别性能。真实环境中不同距离下的核素识别结果如表2所示。

表2 真实环境中不同距离下的核素识别结果Table 2 Nuclide identification results at different distances in real environment
表2结果表明,在探测距离较小时,本文算法对不同核素的识别正确率相对较高,同时可以看出,在探测距离为20 cm内的条件下,本文提出的核素识别算法对真实探测环境中得到的不同探测距离的核素能谱均有较高的识别正确率,平均识别率均高于96%,表现出良好的识别性能。
3 结论
本文通过格拉姆角和场算法将一维能谱数据二维化,从全谱分析和二维角度去探究核素能谱的信息,设计并训练了一种基于Mahalanobis距离的SVM分类器,并结合遗传算法对模型参数进行设置和优化,与其他核素识别算法相比能够有效提高核素识别的准确性。本文算法在真实环境下对单一核素和混合核素具有良好的识别正确率。

