基于科教融合培养大学生拔尖创新能力的表观遗传学综合实验课程
欧秀芳,吴莹,李宁,姜丽丽,刘宝,宫磊
遗传学教学
基于科教融合培养大学生拔尖创新能力的表观遗传学综合实验课程
欧秀芳,吴莹,李宁,姜丽丽,刘宝,宫磊
东北师范大学,分子表观遗传学教育部重点实验室,长春 130024
科教融合是高校培养拔尖创新人才的有效途径。表观遗传学是对经典遗传学的拓展,针对本校拔尖人才培养体系中表观遗传学实验内容的缺失,我们首次进行了表观遗传学科教融合、协同育人的尝试,将本实验室前期在水稻中开展的基因的研究成果进行实验教学转化,建成了一个集前沿性、研究型和创新性为一体,以学生独立实验为中心,考核方式多元化的表观遗传学综合实验课程。本课程以基因突变体和野生型为实验材料,将表观遗传上游酶促基因突变与基因组内特定位点的DNA甲基化和基因组稳定性联系起来。通过本课程的实践,学生对表观遗传修饰的重要作用有了更深刻理解,科研能力得到极大提升,强力支撑了本专业拔尖创新人才的培养。
科教融合;表观遗传学综合实验;实验教学
创新型人才的培养是国家长远发展的大计。党的二十大明确提出要深入实施科教兴国战略,建立和完善我国自主培养拔尖人才的培养体系。2018年,教育部发布了《关于加快建设高水平本科教育全面提高人才培养能力的意见》,强调高校要以高水平科学研究支撑本科人才培养,要将最新的科研成果及时转化为教育教学内容(http://www.moe.gov.cn/ srcsite/A08/s7056/201810/t20181017_351887.html)。因此科教融合是高校培养拔尖创新人才的有效途径。
表观遗传学是对经典遗传学的拓展,解析了遗传学上提到的环境如何导致可遗传的基因表达变化,并最终产生可遗传新表型的问题。本实验室前期研究发现,水稻()中主要负责维持CG甲基化的酶基因的纯合突变可引起全基因组范围内不同位点发生DNA甲基化变异和转座子活性的改变,并且在水稻的不同器官(叶片和愈伤)中表现出变异模式和程度的差异[1,2],该成果完善了植物中基因功能的理论,证明了DNA甲基化在调控植物正常生长发育中的重要作用。
东北师范大学生物科学专业是教育部“拔尖计划2.0”人才培养基地,《表观遗传学》理论课程是“拔尖计划2.0”学生培养必修课程。针对本校人才培养中表观遗传学实验内容的缺失,依托“分子表观遗传学教育部重点实验室”,2018年我们将上述本实验室的科研成果转化为实验课程资源,构建了一门综合表观遗传学最新研究领域和前沿技术的独立的“表观遗传学综合实验课程”。该课程是“拔尖计划2.0”学生必修课程,学生在预修了遗传学、分子生物学、表观遗传学、遗传学基础实验以及分子生物学基础实验的基础上,在第5学期进行修读,课程共计36学时,每年学生人数40人左右,分两个班级进行平行授课。课程采用理论和实践相结合的方式进行,主要探究基因突变对水稻特定序列DNA甲基化和转座子活性的影响。通过本课程的实践,培养学生独立思考、自主分析和解决科研中存在问题的创新能力;培养学生通过阅读、研究、观察和实践手段获取知识的自主学习能力以及通过团队协作学习知识和技能的交流合作能力。目前本课程已经过6轮教学实践检验和持续改进,显著提高了学生的科研能力,全面提升了人才培养质量。
1 表观遗传学实验引入本科生课堂的重要意义
表观遗传学是指不需要DNA序列变异而发生的可遗传的基因表达改变[3]。表观遗传机制包括DNA甲基化、组蛋白修饰、染色质重塑以及非编码RNA丰度变化等[4~6]。研究表明,表观遗传变异在生物进化、作物改良和人类健康中都具有重要作用[7~9]。
目前,全国各高校无论是基础实验还是综合实验大都针对经典遗传学设计而开展,缺少表观遗传学实验内容。本校的遗传学综合实验,也存在上述问题,并且综合实验内容与基础实验内容重叠较多,研究性内容不足,实验内容多为“验证实验现象、培养动手能力”的课程,教学内容上缺少设计性和创新性,与本学科的科研成果融合明显不足,缺少对学生整体科研思维的培养。教学模式上也与基础实验差别不大,仍然主要以“教师教为中心”,学生根据教师的讲解或示范进行操作,缺少对学生主动思考和探索能力的培养。考核方式仍然较单一,过程性评价不足,多采取实验报告或总结论文的考核方式,对创新能力的综合考评不够,导致现有遗传学综合实验的教学内容和模式对学生科研能力培养的支撑度显著不足,不能契合拔尖创新人才的培养目标。《表观遗传学》是本校生物科学专业“拔尖计划2.0”学生的必修课程,多年理论教学发现学生缺少理论知识的实践转化,表观遗传学实验内容在教学体系中完全缺失。因此在本科生培养方案中引入表观遗传学综合实验对于完善生物科学专业学生的实验体系,培养具有原始创新能力的拔尖人才具有重要意义。
2 从科研成果中挖掘适用于实验教学的题材
DNA甲基化是一种极其重要且相对稳定并可遗传的表观遗传修饰,其变化会影响基因表达和基因组稳定性[10,11]。生物固有的DNA甲基化水平和模式改变会导致生物表型异常甚至死亡,但一定范围内的甲基化变异又为适应和物种进化提供了物质基础[12]。在植物中,DNA甲基化可发生在CG、CHG和CHH位点(H代表A/T/C三种碱基中的任意一种)[13~16],其中CG位点的甲基化是最普遍发生的形式[13]。植物中CG甲基化主要由MET1 (methyltransferase 1)负责维持。在拟南芥()中,纯合突变显著降低了全基因组范围内CG甲基化的水平(从24.6%下降到0.42%),植株发育明显异常[17~19]。拟南芥是双子叶模式植物的代表,然而许多重要作物如小麦()、玉米()和水稻都属于单子叶植物,因此本实验室前期研究提出一个重要科学问题:基因在单、双子叶之间的功能是否保守?
水稻是单子叶模式植物的代表。水稻中是维持CG甲基化的主要功能基因[20~22]。本实验室前期对基因的纯合和杂合突变体的幼苗、长期培养的愈伤组织进行了系统研究,发现纯合突变显著降低基因组范围内CG甲基化水平(从39.88%下降到9.69%),说明基因在单、双子叶之间具有功能保守性。但是水稻纯合突变体幼苗仅能在培养基条件下存活14天,长至3 cm左右后即死亡[1],而拟南芥纯合突变体能完成整个发育过程[19,23,24]。此外,在正常培养条件下,拟南芥杂合突变体后代表现出明显的表型分歧[23],而水稻杂合突变体的表型和野生型没有明显差别[1],这表明基因在单、双子叶之间具有明显的功能差异。
在植物中,DNA甲基化与转座子稳定性密切相关。本实验室对连续3年继代培养的纯合突变体、杂合突变体和野生型愈伤组织进行了研究,发现在纯合突变体的愈伤组织中发生了大规模的转座子的转座爆发[2],说明CG甲基化在维持转座子的稳定性和基因组稳定性方面具有重要作用[1,2]。上述研究成果完善了植物中基因的功能,证明了DNA甲基化在调控植物正常生长发育中的重要作用。
本课程以上述研究为基石,教师以理论课上提出的科学问题“基因在单、双子叶之间的功能是否相同?”为核心,对其进行表观遗传学综合实验的转化,该研究成果聚焦表观遗传学的理论和应用前沿,将理论和实践相结合,科研和教学相融汇,契合本校生物学一级学科拔尖创新人才的培养目标。
3 科研成果向创新实验转化的具体实践
科研成果在具体转化时不能将整个研究成果作为实验内容,在可行性上,要考虑本科生的实验周期和实施成本;在内容设计上,要具有系统性、创新性和高阶性,能够真正体现培养学生的创新意识和科研探索精神;在实施过程中,要注意培养学生独立、完整的科研思维;在转化过程中,教师的每一步设计都要体现对学生科研能力的培养,引导学生主动思考、主动探索。课程设计的整体思路突出学生的主导地位,学生自己负责分析和解决问题,教师发挥引导和答疑解惑的作用。课程内容及具体学时安排见表1。
3.1 科学问题的提出
教师课前讲解实验课程相关理论知识,即植物中DNA甲基化的建立、维持及其生物学功能,重点关注DNA甲基化在维持基因组稳定性方面的作用。以双子叶模式植物拟南芥为例,介绍基因的作用及其突变后对拟南芥基因组DNA甲基化和表型的影响,接下来引导学生思考,提出科学问题:单、双子叶植物中的功能是否相同?根据拟南芥相关研究,提出以下问题:(1)水稻中拷贝有几个?(2)基因突变对水稻基因组稳定性、DNA甲基化以及表型有何影响?本实验室前期研究是基于组学分析,这种大尺度的分析并不适用于本科实验,因此在实验设计时,教师引导学生将科学问题具体化,即“基因突变对水稻特定序列DNA甲基化和转座子活性的影响”。
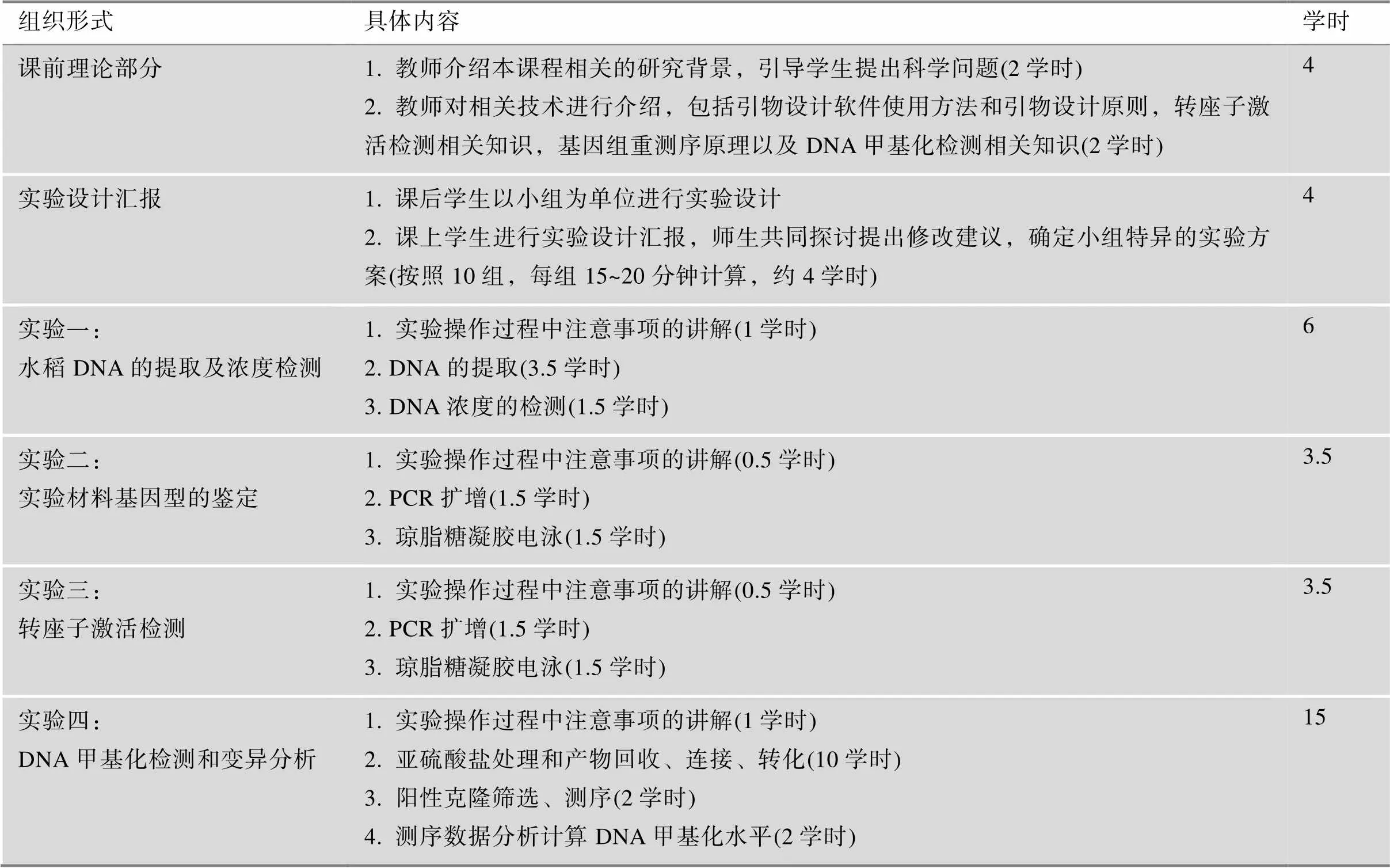
表1 课程安排
3.2 实验材料
在组织培养条件下,水稻粳稻日本晴(L. ssp., cv. Nipponbare)基因组中反转座子被激活,插入到基因,再生获得基因杂合突变体,本实验室所用杂合突变体水稻材料最初来源于日本生物科学研究所,杂合突变体自交后可获得纯合突变体()、杂合突变体()和野生型()。本实验以杂合突变体自交5~9代(2018年~2022年)获得的纯合突变体()、杂合突变体()和野生型()幼苗,以及由以上3种基因型种子诱导继代培养3~7年(2018年~2022年)的愈伤组织为实验材料。学生根据所要解决的科学问题选择实验材料,如科学问题为“纯合突变对水稻愈伤组织中特定转座子的转座活性和DNA甲基化的影响”,学生可以选择该基因的野生型以及纯合突变体的愈伤组织作为实验材料进行研究。
3.3 实验方法
3.3.1 水稻基因组DNA提取及浓度检测
学生每4人为一组,组内再分为两个小组,进行实验中的生物学重复和技术重复。教师为学生提供实验材料,学生可采用改良的CTAB法[25]或植物基因组DNA提取试剂盒(天根生化科技北京有限公司,DP305)进行水稻幼苗和/或愈伤组织基因组DNA的提取,并利用Nanodrop微量分光光度计和/或琼脂糖凝胶电泳对DNA的质量和浓度进行检测。该部分从技能层面使学生掌握植物基因组DNA提取方法和DNA质量评估方法;从思维角度培养学生应用多种实验方法对结果进行相互验证的科研思维。
3.3.2 实验材料基因型鉴定
学生利用水稻插入突变体数据库(RiceInsertion Mutant Database)查找插入基因的具体位点、插入方向以及插入位点上下游的序列信息,利用primer5软件设计引物,以3.3.1所提取的基因组DNA为模板,通过PCR扩增和琼脂糖凝胶电泳,确定材料基因型。该部分从技能层面让学生学会应用水稻突变体数据库和引物设计软件,明确PCR引物设计的基本原则以及PCR反应的原理;从思维角度使学生明确科研中实验材料准确的重要性。
3.3.3 转座子激活检测
学生根据教师讲解的转座子激活相关知识,查阅文献,利用公开数据库或实验室已有的基因组重测序数据,在水稻全基因组范围内选择感兴趣的转座子,通过设计引物、位点特异的PCR扩增以及琼脂糖凝胶电泳,检测基因突变对水稻特定转座子转座活性的影响。重测序数据和分析方法参考实验室前期科研成果[2]。该部分从技能层面使学生掌握基因组重测序的原理和基因组内转座子多态性分析的原理;从思维角度培养学生根据不同实验目的选择适合实验方案的科研思维。
3.3.4 DNA甲基化检测和变异分析
学生利用特定位点亚硫酸盐测序法检测特定序列DNA甲基化情况。具体实验过程包括:亚硫酸盐测序引物的设计;亚硫酸盐处理基因组DNA;PCR扩增目的产物的回收、与T载体连接、转化;阳性克隆筛选、测序以及测序数据分析。实验方法参照用于本课程转化的科研成果[1]。该部分从技能角度培养学生团队协作设计DNA甲基化检测实验方案的能力;从思维角度培养学生将大尺度(基因组范围)和小尺度(特点位点)结合起来综合分析问题的科研思维。
3.4 代表性实验结果
3.4.1 水稻幼苗和愈伤组织基因组DNA的提取及浓度检测
图1A展示了某小组利用不同方法提取的野生型(+/+)和纯合突变体(–/–)愈伤组织和幼苗的基因组DNA,并用Nanodrop微量分光光度计检测DNA的质量和浓度,发现从愈伤组织中所提取的DNA浓度明显低于幼苗中,并且试剂盒法提取的DNA的浓度要高于改良的CTAB法,但两种方法所提取的DNA纯度(260/280)无差异(图1A)。将图1A中的DNA稀释10倍后利用琼脂糖凝胶电泳进行检测,以λDNA(浓度为10 ng/µL)作为检测DNA完整性和估测浓度的标准,通过亮度估测浓度发现与Nanodrop所测浓度基本一致,可进一步确定所提取的基因组DNA的浓度(图1B)。

图1 DNA质量和浓度的检测
A:Nanodrop微量分光光度计检测水稻基因组DNA;B:琼脂糖凝胶电泳检测水稻基因组DNA(稀释10倍)。λDNA浓度为10 ng/µL,+/+代表野生型,–/–代表纯合突变体。
3.4.2 PCR扩增鉴定实验材料基因型。
根据水稻插入突变体数据库提供的信息,反向插入到基因的第6个外显子(图2A)。PCR引物设计时需要两对引物组合使用(图2A),引物对A位于插入位点两端(primer1和primer2),引物对B的一侧位于插入位点上游(primer1),另一侧位于的3′端(primer3),该设计巧妙的共用一个引物primer1,与primer2组成了引物对A,与primer3组成了引物对B,由于本身长度为4.3 kb,通过控制PCR反应程序中的延伸时间为1分钟(1分钟大约可扩增1 kb的序列长度),即可根据两对引物扩增条带的有无来判断材料的基因型。利用图2B中的引物序列进行扩增,如仅在引物对A扩增出目的条带(500 bp),则基因型为野生型(+/+);仅在引物对B中扩增出目的条带(500 bp),则基因型为纯合突变体(–/–);如在引物对A和B中均能扩增出目的条带(500 bp),则该基因型为杂合突变体(+/–)(图2C)。
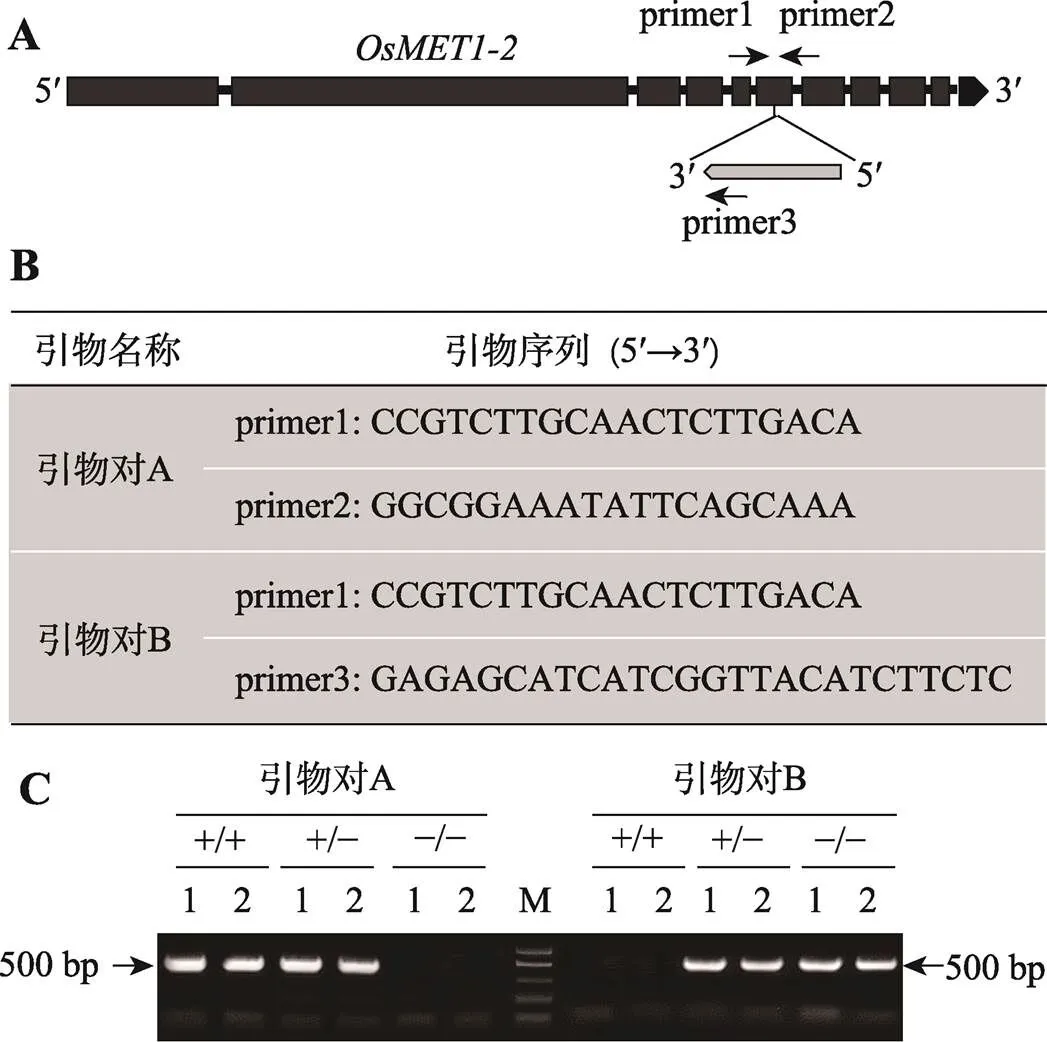
图2 实验材料基因型鉴定
A:反向插入到的第六个外显子及引物设计的位置;B:用于鉴定基因型所用的引物信息;C:引物对A和B在不同基因型中扩增出的目的条带。+/+代表野生型,+/–代表杂合突变体,–/–代表纯合突变体。M:100 bp DNA Ladder。
3.4.3 转座子激活及其上游序列DNA甲基化变异检测
本部分重点突出学生自主探究,学生可在水稻全基因组范围内选择感兴趣的转座子进行研究,通过PCR扩增和琼脂糖凝胶电泳检测转座子的转座激活情况。如某小组选择进行研究,属于MITEs类转座子,全长430 bp,的序列具有高度保守性,在粳稻中约有50个拷贝,与水稻基因组进化相关,研究发现能在特定环境条件诱导下激活[26~28]。通过与野生型(+/+)愈伤组织相比,在纯合突变(–/–)的2个单独继代培养的愈伤组织中均检测到的纯合跳出(图3A),在–/–的2号愈伤组织中检测到的纯合插入(图3B)。
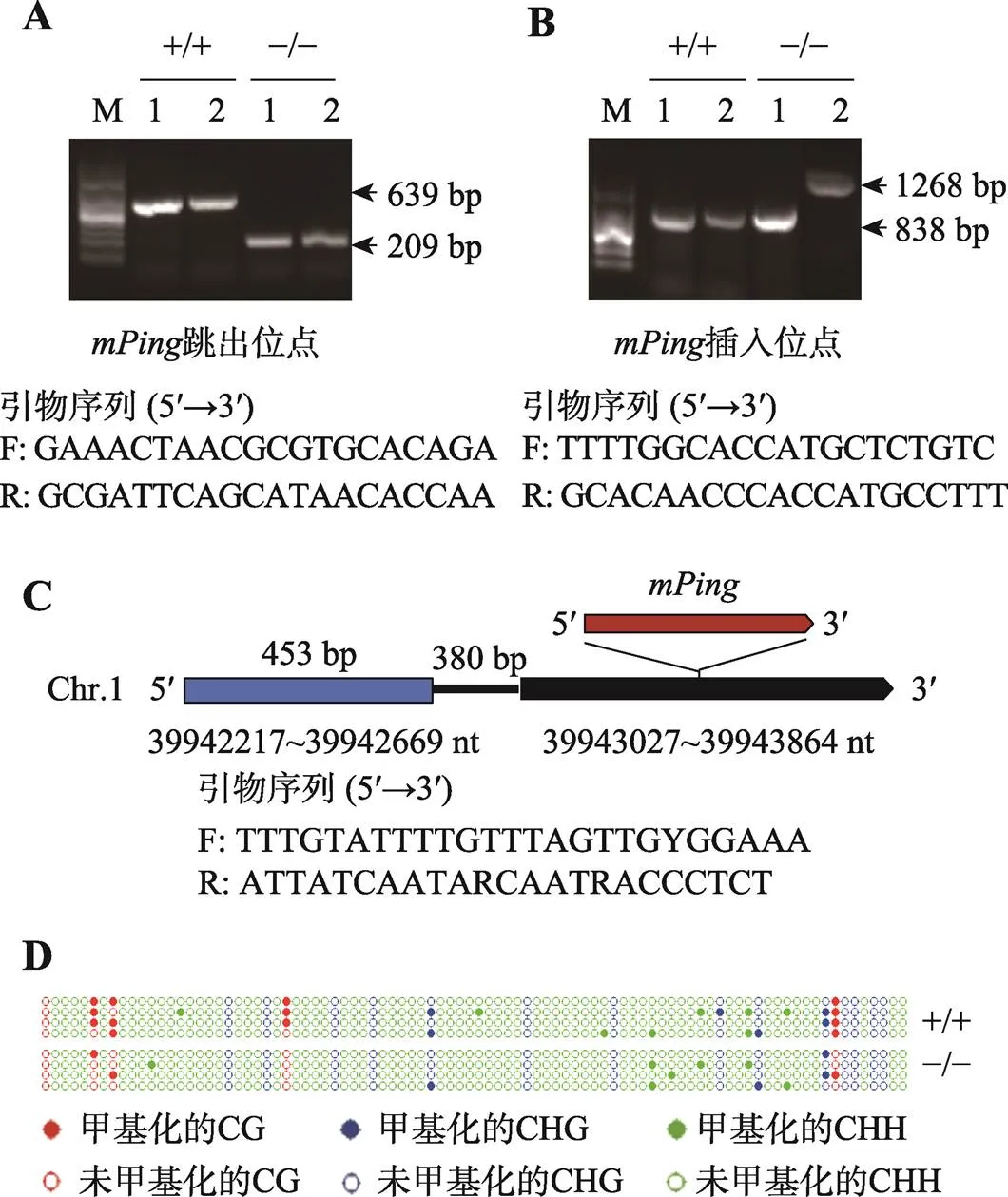
图3 转座子转座活性检测和DNA甲基化分析
A:在愈伤组织中与野生型(+/+)相比,纯合突变体(–/–)发生了的跳出;B:在愈伤组织中与野生型(+/+)相比,纯合突变体(–/–)发生了的插入;C:插入位点及上游序列信息;D:亚硫酸盐测序分析野生型(+/+)与纯合突变体(–/–)中DNA甲基化情况。M:100 bp DNA Ladder。
在距离插入位点上游833~380 bp (目的序列长度453 bp)处设计引物(图3C),检测插入与其上游序列DNA甲基化的相关性,在453 bp的序列中,包括5个CG位点,将–/–和+/+进行对比,发现在–/–中CG甲基化显著降低(图3D),而CHG和CHH甲基化无明显变化。说明的插入可能与其上游序列CG甲基化降低相关。
3.5 多元的成绩评价方式
本课程采取过程性的多元化评价体系,包括撰写与本课程相关的研究综述、以小组为单位的研究设计及汇报、学生个人实验操作的规范性评价、实验记录和研究论文。每项评定方式满分均为100,各项评价方式对学生的具体要求、考核目的、评价标准以及所占比例见表2。
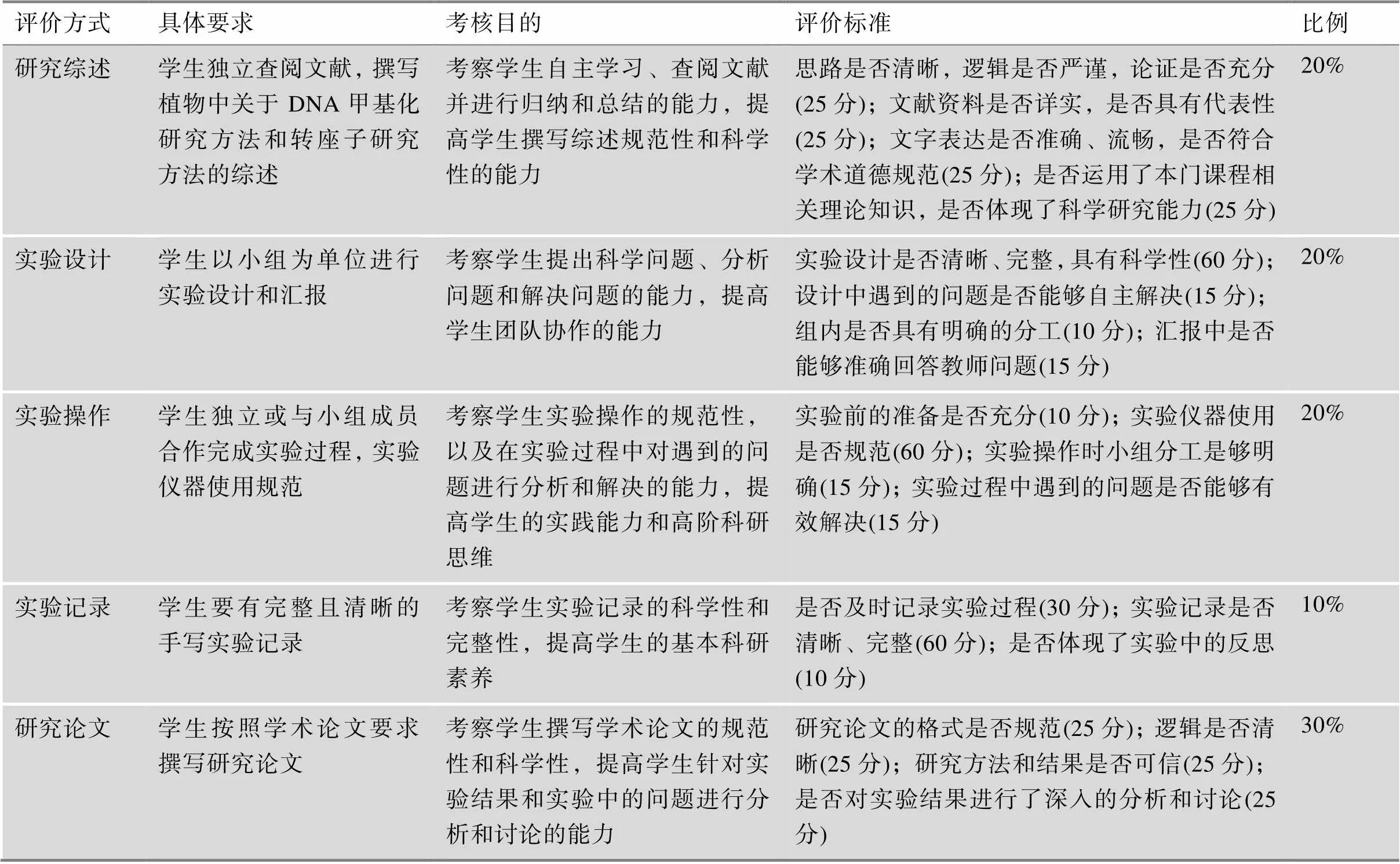
表2 课程评价方式
4 课程实践中的教学反馈和反思
为更好地了解科研转化实验的教学效果,自2020年春季起,通过问卷方式对修完本课程的学生进行调查,问卷通过问卷星APP进行发布,包括四轮教学实践(2020春、2021春、2022春和2022秋)。问卷共计19道题,其中1~15为主观题,重点关注学生对课程教学内容、教学模式的设置、考核方式的合理性、自身科研思维和科研能力提高等的满意度,每道题设置5个选项,分别为“非常不同意”、“不同意”、“一般”、“同意”和“非常同意”,以选择后两项即“同意”和“非常同意”学生比例的和作为学生满意度指标;16和17题为分类选择题,关注学生在实验课后的自我反思和反馈;18和19题为开放性问题,关注学生对课程的建议,为课程后续改进提供依据。
4.1 学生对课程的满意度
通过主观题的设置调查学生对该课程的满意度,从表3中可以看出本实验内容的设置突出了研究型教学内容(问题1),课程的教学内容和模式(问题2、3)充分调动了学生自主学习、主动思考和解决问题的能力(问题4、7),在实验中学生能够明确实验中需要注意的关键点(问题5),整个实验课程的设置以学生为主导(问题6),学生通过小组协作(问题4、5、14)完成实验,考核方式的多元化设置(问题8、9)有助于学生更加注重实验过程中的严谨性,学生高度认可这种学习方式(问题10),认为能够有效培养他们的科研思维和创新能力(问题11),并且学生认同高阶挑战性的课程能够激发他们的学习兴趣(问题12),学到更多的知识(问题13)。以上结论来自于前14个问题的满意度,表3中除个别学期个别问题的满意度在85%左右外,其余满意度都在90%以上,甚至有的达到100%,这说明总体上学生高度认可这种教学模式。值得注意的是,学生对问题15的满意度明显偏低,这说明高阶挑战性的研究型实验内容的设置增加了学生的课业负担,这也从侧面反应了综合性创新实验的一个特点,学生需要花费更多的精力和时间去完成这门课,但同时也让学生领悟了科研中所需要的努力、付出和坚持。
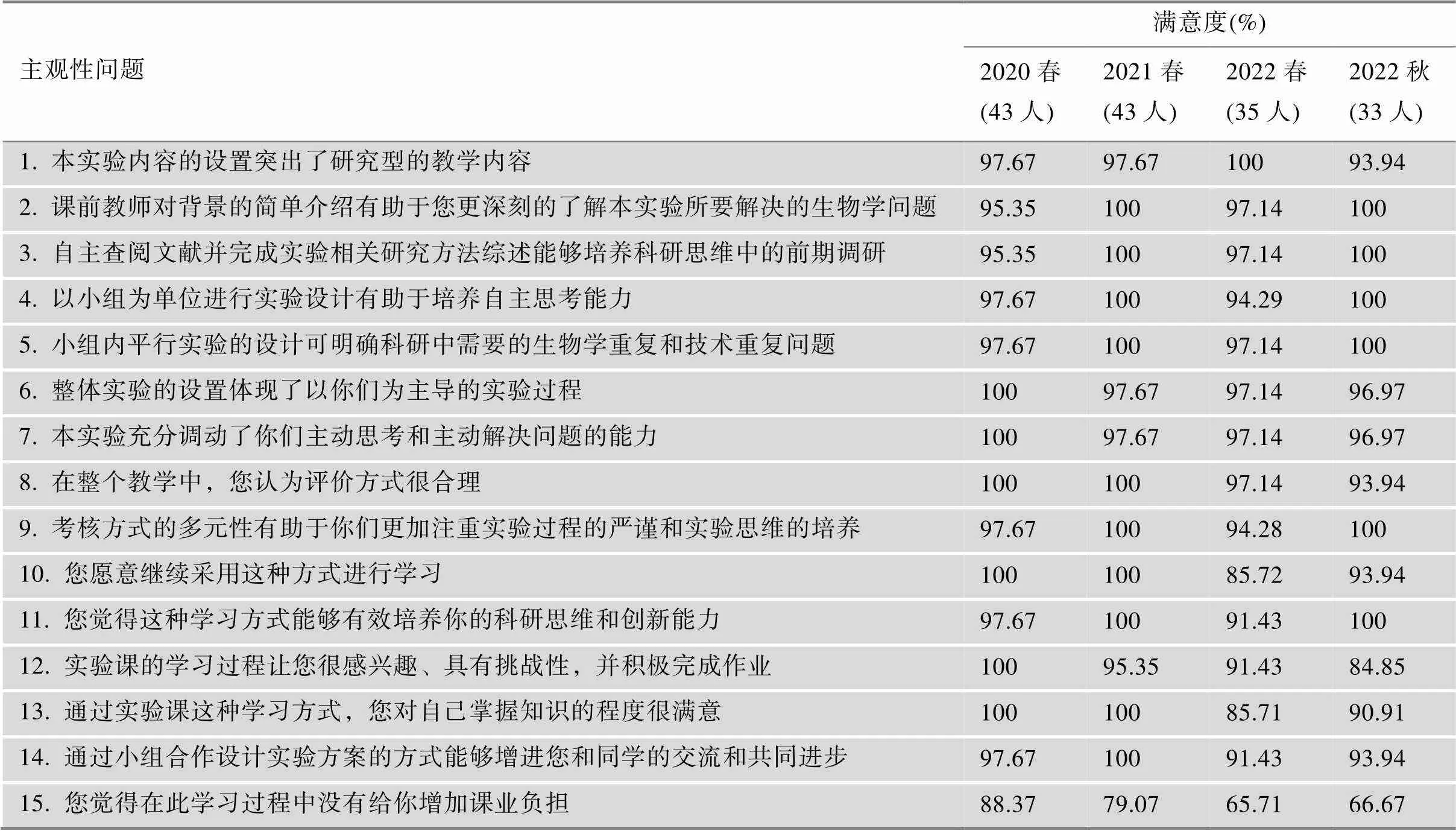
表3 学生满意度调查
4.2 学生的自我反思
课程完成后,学生通过自我反思(图4A,问题16:在本实验课中,您认为影响自己学习效果的因素是什么?),认为自身对实验中生物学问题理解的不够深入对学习效果的影响最大,其次是自主阅读文献能力不足,而对教师的依赖性过大及实验操作能力对学习效果的影响则相对较小,这也正是综合性探究实验与以往基础验证性实验最大的区别,学生作为课程的主导,要具有主动探究和主动思考的能力,通过本实验课的实践让学生认识到目前自身存在的不足,有助于其在今后学习中的改进和提高。同时,教师根据学生反馈,在2022年春季加强了课程相关理论内容的讲解,在一定程度上提高了学生对生物学问题的理解能力。但也注意到此项依然是学生存在的主要问题,在今后的授课中教师将在理论课中增加答疑讨论环节,并在表观遗传学理论课中对涉及实验课内容的部分以科研案例的形式进行详细讲解,来帮助学生更好地理解实验中的科学问题。
通过本实验课的训练(图4B,问题17:课程结束后,您觉得哪一项能力提高的最多?),学生认为其科研能力、问题解决能力和合作能力提高的最多。从图中可以看出在所调查的4个学期中此3项始终位列前三,学生的批判思维和创新能力也有所提高,但其选择比例较低的原因可能与本题设置有关,此题设置为单选题,学生只能从中选择一项,而实际上科研能力和问题解决能力的提高就包含批判性思维和创新能力的提高,因此通过学生的反馈,可以看出本课程达到了提高学生科研能力的目的。
4.3 学生对课程的建议
开放性问题18和19由学生自由回答,重点关注学生对课程的建议。对于问题18:本实验课的教学模式你喜欢哪部分?学生普遍反馈喜欢在教师提供一定的引导和启发下,让他们以小组合作方式进行自主探索和自主设计的教学模式。对于问题19:您希望以后的课程在哪些方面得到改善?在2020年和2021年春季的调查中,学生反馈教师理论课讲解时间偏少,建议增加理论课学时,因此在2022年春季和秋季的实验课中,理论课由之前的2学时调整为4学时。此外在2022年秋季的调查中,学生反馈希望教师增加对实验数据处理部分的指导,并增加小组间的讨论环节。对于此反馈,教师拟在下一轮的教学实践中,增加实验后的成果汇报部分,师生共同对实验结果进行分析和讨论,培养学生的批判性思维。
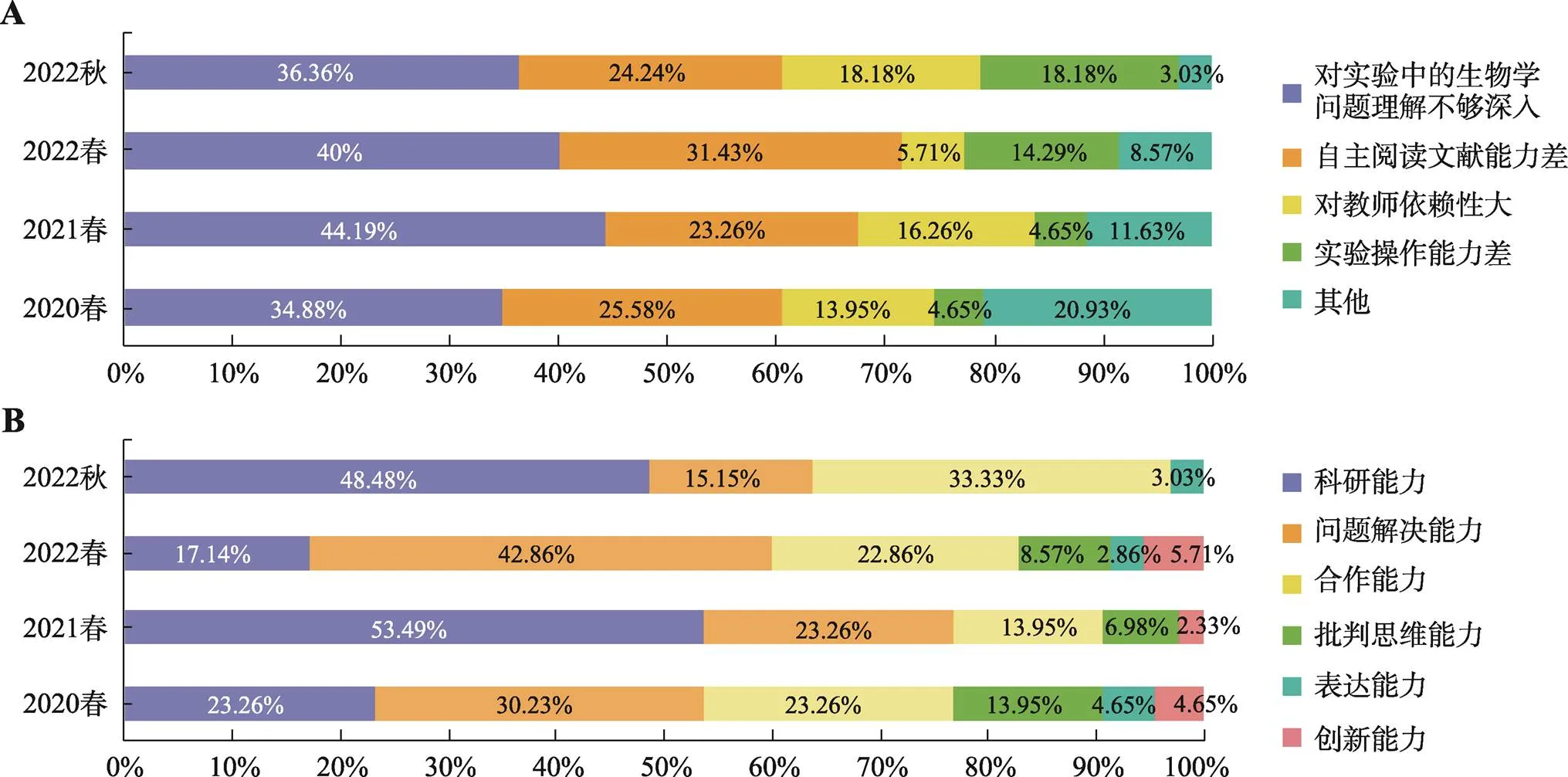
图4 学生的自我反思
A:问题16:在本实验课中,您认为影响自己学习效果的因素是什么?B:问题17:课程结束后,您觉得哪一项能力提高的最多?
4.4 教师的实践反思
通过连续4年的调查反馈,教师及时掌握了学生在学习中遇到的问题和学习需求,及时调整授课模式,灵活调整课堂内容,提高了实验课的教学质量。除以上学生反馈的建议外,教师在多轮授课中也发现本课程存在可以进一步改进的部分,即如何建立转座子活性与其自身或临近区域DNA甲基化关系,要求学生自主选择转座子进行转座活性与DNA甲基化相关性的研究。本部分的开放性和发散性极高,学生可以在水稻全基因组范围选择转座子,这就存在着选择的转座子的差异,转座子是否发生转座激活的差异,转座子自身甲基化状态的差异、转座子原位点是否存在DNA甲基化、转座子激活位点的临近序列的甲基化状态的差异等,每组学生的选择不同,结果就不同。若学生选择用于分析的片段本身不存在DNA甲基化,则无法建立甲基化状态和转座子活性的关系。为了提高本部分的实验成功率,教师要求学生在具体实验方案确定时对选择的转座子进行详细介绍,并且要对进行DNA甲基化分析区段的甲基化情况状态进行确定,可利用实验室发表文献中的甲基化组数据进行查询[1,2],这在一定程度上提高了本部分实验的成功率。
除此之外,本课程也有一些新发现,如在检测转座子激活的实验中,学生在–/–中发现了新的跳出位点,经过材料基因型的确认以及3次重复,证实该结果是可信的。此时学生提出问题:为什么相同基因型的愈伤组织中的活性不同?其他位点的活性是否也发生了改变?其他转座子的转座活性是否也有不同?造成这些差异的原因是什么?针对学生的疑问,教师引导学生进一步思考:“愈伤组织在连续多代的培养中,其基因组、DNA甲基化组、转录组、蛋白质组、代谢组等会发生怎样的变化?这些变化与其再生能力是否相关?”学生可以在课程结束后自主查阅文献,甚至可以进行相应的实验设计来进行进一步的探究。由此本课程通过一个综合性实验的探索引发了学生更深、更广的思考,不断开拓学生的科研思维,培养学生创新能力。
5 结语
前沿科研成果向一线教学转化时,要考虑育人目标以及学校可实现的软、硬件设施,实验周期和实验成本等。本文通过具体的科研转化,将表观遗传学研究领域的热点研究问题和实验技术转化为综合型创新实验课程,在完善专业教学体系基础上,实现了课程的高阶性,具有一定的挑战度。该课程将表观遗传修饰上游关键酶基因突变与转座子活性和特定序列的DNA甲基化状态关联,学生在基因组范围内自主选择感兴趣的转座子进行探究,能够更好帮助学生理解表观遗传修饰在维持基因组稳定性中的重要作用。本实验与华南农业大学设计的表观遗传实验显著不同,后者重点关注一个基因自身DNA甲基化修饰变异引起的基因表达改变和表型变异[29]。本课程在具体实施中要充分考虑本科生的知识水平和科研能力,进行有的放矢的引导,让学生作为科研的主角,课程的重点不在于学生是否能够得到预期实验结果,更注重的是让学生学会进行科学研究,提高学生独立探索、独立思考和独立解决问题的科研能力,这才是通过实验教学培养拔尖人才的重要举措。
[1] Hu LJ, Li N, Xu CM, Zhong SL, Lin XY, Yang JJ, Zhou TQ, Yuliang AZ, Wu Y, Chen YR, Cao XF, Zemach A, Rustgi S, von Wettstein D, Liu B. Mutation of a major CG methylase in rice causes genome-wide hypomethylation, dysregulated genome expression, and seedling lethality., 2014, 111(29): 10642–10647.
[2] Hu LJ, Li N, Zhang ZB, Meng XC, Dong QL, Xu CM, Gong L, Liu B. CG hypomethylation leads to complex changes in DNA methylation and transpositional burst of diverse transposable elements in callus cultures of rice., 2020, 101(1): 188–203.
[3] Berger SL, Kouzarides T, Shiekhattar R, Shilatifard A. An operational definition of epigenetics., 2009, 23(7): 781–783.
[4] Allis CD, Jenuwein T. The molecular hallmarks of epigenetic control., 2016, 17(8): 487–500.
[5] Li B, Carey M, Workman JL. The role of chromatin during transcription., 2007, 128(4): 707–719.
[6] Feinberg AP. The key role of epigenetics in human disease prevention and mitigation., 2018, 378(14): 1323–1334
[7] Ashe A, Colot V, Oldroyd BP. How does epigenetics influence the course of evolution?, 2021, 376(1826): 20200111.
[8] Lieberman-Lazarovich M, Kaiserli E, Bucher E, Mladenov V. Natural and induced epigenetic variation for crop improvement., 2022, 70: 102297.
[9] Zhang L, Lu QJ, Chang C. Epigenetics in health and disease., 2020, 1253: 3–55.
[10] Phillips T. The role of methylation in gene expression., 2008, 1(1): 116.
[11] Moore LD, Le T, Fan GP. DNA methylation and its basic function., 2013, 38(1): 23–38.
[12] Gallusci P, Dai ZW, Génard M, Gauffretau A, Leblanc- Fournier N, Richard-Molard C, Vile D, Brunel-Muguet S. Epigenetics for plant improvement: current knowledge and modeling avenues., 2017, 22(7): 610–623.
[13] Law JA, Jacobsen SE. Establishing, maintaining and modifying DNA methylation patterns in plants and animals., 2010, 11(3): 204–220.
[14] Cao XF, Jacobsen SE. Locus-specific control of asymmetric and CpNpG methylation by the DRM and CMT3 methyltransferase genes., 2002, 99(Suppl 4): 16491–16498.
[15] Stroud H, Greenberg MV, Feng SH, Bernatavichute YV, Jacobsen SE.Comprehensive analysis of silencing mutants reveals complex regulation of themethylome., 2013, 152(1–2): 352–364.
[16] Zemach A, Yvonne Kim M, Hsieh PH, Coleman-Derr D, Eshed-Williams L, Thao K, Harmer SL, Zilberman D. Thenucleosome remodeler DDM1 allows DNA methyltransferases to access H1-containing heterochromatin., 2013, 153(1): 193–205.
[17] Cokus SJ, Feng SH, Zhang XY, Chen ZG, Merriman B, Haudenschiil CD, Pradhan S, Nelson SF, Pellegrini M, Jacobsen SE. Shotgun bisulphite sequencing of thegenome reveals DNA methylation patterning., 2008, 452(7184): 215–219.
[18] Lister R, O’Malley RC, Tonti-Filippini J, Gregory BD, Berry CC, Harvey Millar A, Ecker JR. Highly integrated single-base resolution maps of the epigenome in., 2008, 133(3): 523–536.
[19] Mathieu O, Reinders J, Caikovski M, Smathajitt C, Paszkowski J. Transgenerational stability of theepigenome is coordinated by CG methylation., 2007, 130(5): 851–862.
[20] Pavlopoulou A, Kossida S. Plant cytosine-5 DNA methyltransferases: structure, function, and molecular evolution., 2007, 90(4): 530–541.
[21] Yamauchi T, Johzuka-Hisatomi Y, Fukada-Tanaka S, Terada R, Nakamura I, Iida S. Homologous recombination-mediated knock-in targeting of the MET1a gene for a maintenance DNA methyltransferase reproducibly reveals dosage-dependent spatiotemporal gene expression in rice., 2009, 60(2): 386–396.
[22] Yamauchi T, Johzuka-Hisatomi Y, Terada R, Nakamura I, Iida S. The MET1b gene encoding a maintenance DNA methyltransferase is indispensable for normal development in rice., 2014, 85(3): 219–232.
[23] Saze H, Mittelsten Scheid O, Paszkowski J. Maintenance of CpG methylation is essential for epigenetic inheritance during plant gametogenesis., 2003, 34(1): 65–69.
[24] Kankel MW, Ramsey DE, Stokes TL, Flowers SK, Haag JR, Jeddeloh JA, Riddle NC, Verbsky ML, Richards EJ.MET1 cytosine methyltransferase mutants., 2003, 163(3): 1109–1122.
[25] Kidwell KK, Osborn TC. Simple plant DNA isolation procedures. In: Beckmann JS, Osborn TC, eds. Plant Genomes: Methods for Genetic and Physical Mapping. Springer, Dordrecht, 1992.
[26] Jiang N, Bao ZR, Zhang XY, Hirochika H, Eddy SR, McCouch SR, Wessler SR. An active DNA transposon family in rice., 2003, 421: 163–167.
[27] Kikuchi K, Terauchi K, Wada M, Hirano HY. The plant MITEis mobilized in anther culture., 2003, 421: 167–170.
[28] Shan XH, Ou XF, Liu ZL, Dong YZ, Lin XY, Li XW, Liu B. Transpositional activation ofin an asymmetric nuclear somatic cell hybrid of rice andwas accompanied by massive element loss., 2009, 19: 1325–1333.
[29] Zhang XQ, Li N, Xie XM. Design and exploration of epigenetic comprehensive experiments., 2021, 43(12): 1179–1187.张向前, 李楠, 解新明. 表观遗传学综合性实验设计与探讨. 遗传, 2021, 43(12): 1179–1187.
Epigenetics comprehensive experimental course based on the integration of science and education to cultivate students' ability of cutting-edge innovation
Xiufang Ou, Ying Wu, Ning Li, Lili Jiang, Bao Liu, Lei Gong
The integration of science and education is an effective way for universities to cultivate students in cutting-edge innovative interests. Epigenetics is the expansion of classical genetics, the corresponding experimental courses of which have not been integrated into the current teaching system. In this paper, by taking advantage of our laboratory's research on the DNA methylation maintenance gene,in rice, we have integrated our innovative findings in the education curriculum, and built a comprehensive teaching system on experimentation research, which greatly stimulates the curiosity of the students. Taking themutants and its isogenic wild-type rice plants as experimental materials, this course has successfully demonstrated a causal link between genetic mutation and epigenetic variation, a topic widely interested by the students in learning genetics and epigenetics. Through the practice of this course, students have a deeper understanding of the important role of epigenetic modifications, their scientific research capabilities have been greatly improved, thereby strongly supporting the cultivation of top innovative talents among the students.
integration of science and education; epigenetics comprehensive experiment; experimental education
2023-07-03;
2023-11-15;
2023-11-16
吉林省高等教育教改研究课题立项(编号:JLO4169120190727102937,JLJY202393805644,JG2020008)资助[Supported by the Higher Education Teaching Reform Research Topic of Jilin Province (Nos. JLO4169120190727102937, JLJY202393805644, JG2020008)]
欧秀芳,博士,副教授,研究方向:表观遗传学。E-mail: ouxf074@nenu.edu.cn
宫磊,博士,教授,研究方向:进化生物学。E-mail: gongl100@nenu.edu.cn
10.16288/j.yczz.23-179
(责任编委: 陈德富)