大曲高温放线菌Thermoactinomyces daqus CICC 10681的DNA(G+C)含量分析
张 欣,姚 粟,刘 洋,刘 勇,张 露,程 池*
(中国食品发酵工业研究院 中国工业微生物菌种保藏管理中心,北京 100015)
(G+C)含量是指生物体脱氧核糖核酸(deoxyribonucleic acid,DNA)中鸟嘌呤(guanine,G)和胞嘧啶(cytosine,C)占整个碱基总量[G+C+腺嘌呤(adenine,A)+胸腺嘧啶(thymine,T)]的摩尔分数,即(G+C)摩尔分数为(G+C)/(G+C+A+T)。(G+C)摩尔分数是微生物物种的重要遗传指征,尤其是细菌,因其(G+C)摩尔分数范围较宽,可达20%~80%[1-3],且每一种细菌(G+C)含量变化范围很小,比较稳定,不受菌龄、生长环境等因素影响。因此,(G+C)含量分析已成为微生物遗传学分类的一种重要方法,在鉴别细菌种间和种属间关系以及建立一个新的分类单位时具有重要意义[4-5]。
(G+C)含量测定方法主要有热变性温度法和高效液相色谱法[6-8]。热变性温度法是1961年MARMUR J等[8]提出的用于测定(G+C)含量的一种物理方法,主要基于双链DNA发生热变性过程中所呈现的增色效应,通过检测波长260 nm处的吸光度值变化,获得双链DNA的溶解曲线和解链温度(Tm),再根据Tm与(G+C)含量的经验公式换算得到(G+C)摩尔分数[9]。Tm间接反映了菌株G-C碱基对的含量,因为3个氢键连接的G-C碱基对断裂需要的温度高于两个氢键连接的A-T碱基对,因此DNA中G-C碱基对含量越多,解链温度Tm也就越高[6]。热变性温度法得到的DNA(G+C)摩尔分数由于是物理参数解链温度通过经验公式换算而成,因此间接地反映DNA 的(G+C)含量。
1989年MESBAH M等[7]首次提出利用液相色谱精确测定DNA的(G+C)含量,DNA经过水解(酸解或酶解)处理,再借助色谱分离技术将水解产物(碱基或脱氧核苷)从色谱柱上逐一分离开,并对目标碱基成分进行定量检测。近年来随着色谱技术的发展,定量分析的灵敏度和准确度大大提高,高效液相色谱法直接定量测定目标碱基,得到的(G+C)含量精确度更高。
此外,通过对微生物进行全基因组测序,无疑也能从中获得DNA的(G+C)摩尔分数,但全基因组测序费用昂贵,费时费力,主要用于重要的功能菌株的遗传学分析,不适合用于(G+C)摩尔分数的常规测定。
本研究使用热变性温度法和液相色谱法对大曲高温放线菌(Thermoactinomyces daqus)CICC 10681的(G+C)摩尔分数进行了分析[10],并首次通过该菌株的全基因组测序结果,对热变性温度法和液相色谱法的测定结果进行了验证。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
参考菌株Escherichia coliK12(CICC 10424)、Escherichia coliDH5α(CICC 10399);样品菌株大曲高温放线菌(Thermoactinomyces daqus)CICC 10681:中国工业微生物菌种保藏管理中心。
1.1.2 试剂
标准脱氧核苷:鸟嘌呤脱氧核苷(deoxyriboguanosine monophosphat,dGMP)、胸腺嘧啶脱氧核苷(deoxythymidine,dTMP):生工生物工程有限公司。三乙胺、甲醇(色谱级):百灵威科技有限公司;S1核酸酶和小牛小肠碱性磷酸酶(calf intestinal alkaline phosphatase,CIAP):宝生物(大连)有限公司;其余试剂均使用国产分析纯试剂。
0.1×柠檬酸钠(saline sodium citrate,SSC)缓冲液的配制:175.3 g NaCl和88.2 g柠檬酸钠溶于1 L ddH2O,再稀释200倍,调节pH 7.0。
1.2 仪器与设备
DU 800核酸蛋白分析仪:美国Beckman公司;Ultimate 3000 高效液相色谱仪:赛默飞世尔科技有限公司;SK8200LHC超声波清洗器:上海科导超声仪器有限公司;PTFE HPLC用溶剂过滤器:北京迈瑞达科技有限公司。
1.3 方法
1.3.1 细菌基因组DNA提取
主要按照MARMUR J等[11]的方法提取细菌基因组DNA,并做了少量改进,具体方案如下:
(1)称取离心收集到的湿菌体1.0 g,重悬于6 mL氯化钠-Tris-EDTA缓冲液(sodium chloride-tris-EDTA,STE)缓冲液(10 mmol/L Tris-HCl,10 mmol/L NaCl,1 mmol/L EDTA),振荡混匀。(2)加入60 μL(100 mg/mL)溶菌酶,10 μL核糖核酸酶(ribonuclease,RNase)A(12.5 mg/mL)37 ℃水浴2 h。(3)加入660 μL 10%十二烷基磺酸钠(sodium dodecyl sulfate,SDS)、80 μL蛋白酶K,50 ℃温浴2 h。(4)加入1.7 mL5 mol/L NaCl,使终浓度为1 mol/L,充分混匀。(5)用等体积的酚/氯仿/异戊醇(25∶24∶1,V/V)混合液反复抽提至无蛋白分层。(6)取上清,加0.6倍体积的预冷异丙醇,得到沉淀基因组DNA,用体积分数为70%的乙醇进行脱盐处理后自然风干。(7)用重蒸水或0.1×SSC充分溶解基因组DNA,并用DU800核酸蛋白分析仪测定检测DNA纯度。
1.3.2 热变性温度法测定(G+C)含量
提取的基因组DNA溶解于0.1×SSC中,调节其浓度为5~15 μg/mL。以大肠杆菌(Escherichia coli)K12为参考菌株,参照LI J等[6]的方法,采用DU 800核酸蛋白分析仪,设置升温范围50~95 ℃,升温速度1.0 ℃/min,实时检测升温过程中DNA样品在波长260 nm处的吸光度值,绘制解链曲线。通过DU 800软件分析得到解链温度Tm值,通过公式(G+C)含量=(Tm样品-Tm参考)×2.08+51.2[2]计算出菌株基因组DNA的(G+C)摩尔分数。
1.3.3 高效液相色谱法测定菌株的碱基组成
以大肠杆菌(Escherichia coli)DH5α为参考菌株,参照MESBAH M等[7,12]的方法并加以优化,主要步骤如下:(1)将提取的基因组DNA溶解于重蒸水中,调节其浓度为100~200 μg/mL。(2)取70 μL DNA溶液于1.5 mL离心管中,100 ℃煮沸5 min,立即冰浴。(3)依次添加5 μL乙酸钠(0.3 mol/L,pH5.1)、5 μL S1核酸酶(1 U/5 μL),37 ℃温浴2 h。(4)添加10 μL小牛小肠碱性磷酸酶(1 U/10 μL),水解过夜。(5)酶解液于13 000 r/min离心10 min,上清立即转入色谱进样瓶,进行高效液相色谱仪分析。优化的色谱条件为:采用C18(4.6 mm×250 mm,5 μm)色谱柱,88%三乙胺和12%甲醇(pH 5.1)作为流动相,检测波长260 nm,流速1.0 mL/min,进样量15 μL,色谱分离时间60 min。
用软件Chromeleon 7.10 SR1对色谱图进行分析,获得DNA酶解液中鸟嘌呤脱氧核苷(dGMP)和胸腺嘧啶脱氧核苷(dTMP)对应的峰面积。(G+C)含量的计算如下:
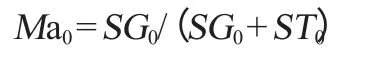
式中:Ma0为参考菌株的(G+C)表观摩尔分数,%;SG0、ST0为参考菌株DNA水解产物经HPLC分离所获得的dGMP、dTMP的峰面积。
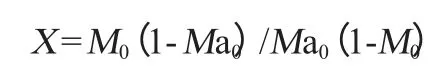
式中:X为校正系数;M0为参考菌株的实际摩尔分数;Ma0为参考菌株的(G+C)表观摩尔分数,%。
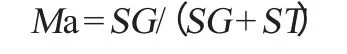
式中:Ma为样品菌株的(G+C)表观摩尔分数;SG、ST为样品菌株DNA水解产物经HPLC分离所获得的dGMP、dTMP的峰面积。
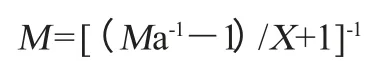
式中:M为样品菌株的(G+C)摩尔分数,%;X为校正系数;Ma为样品菌株的(G+C)表观摩尔分数,%。
1.3.4 全基因组测定
大曲高温放线菌株CICC10681的全基因组序列测定由北京诺赛基因组研究中心完成。
2 结果与分析
2.1 DNA 纯度检测
充分溶解的菌株基因组DNA用1%的琼脂糖凝胶电泳检测,结果见图1。由图1可知,DNA条带清晰,无拖尾。DU80核酸蛋白分析仪检测结果显示所有菌株基因组DNA OD260nm/OD280nm>1.8,OD260nm/OD230nm>2.0,表明提取的基因组DNA纯度较高,无蛋白及小分子盐等杂质污染。

图1 菌株基因组DNA的琼脂糖凝胶电泳图谱Fig.1 Agarose gel electrophoresis of strain genomic DNA
2.2 热变性温度法测定CICC 10681基因组DNA的(G+C)含量
菌株CICC 10681和参考菌株Escherichia coliK12的热变性曲线如图2所示,加热温度范围50~95 ℃,升温速度1 ℃/min。

图2 标准菌株(A)及样品菌株(B)在0.1×SSC溶液中的热变性曲线Fig.2 Thermal denaturation curve of standard strains(A) and sample strains(B) in 0.1×SSC solution
DU800软件分析得到CICC 10681和参考菌株Escherichia coliK12的基因组DNA的Tm值分别为74.4℃和76.4 ℃。根据公式:(G+C)摩尔分数=(Tm样品-Tm参考)×2.08+51.2,计算得到大曲高温放线菌(Thermoactinomyces daqus)CICC 10681的(G+C)摩尔分数=47.0%。
2.3 HPLC 法测定CICC 10681基因组DNA的(G+C)含量
HPLC得到的菌株脱氧核苷色谱分离结果见图3,参照菌株E.coliDH5α和CICC 10681 基因组DNA酶解液的色谱图中鸟嘌呤脱氧核苷(dGMP)、胸腺嘧啶脱氧核苷(dTMP)分离效果良好,峰形正常,无干扰峰。色谱工作站分析得到大肠杆菌(E.coli)DH5α和大曲高温放线菌(Thermoactinomyces daqus)CICC 10681基因组DNA的dGMP和dTMP对应的峰面积见表1。
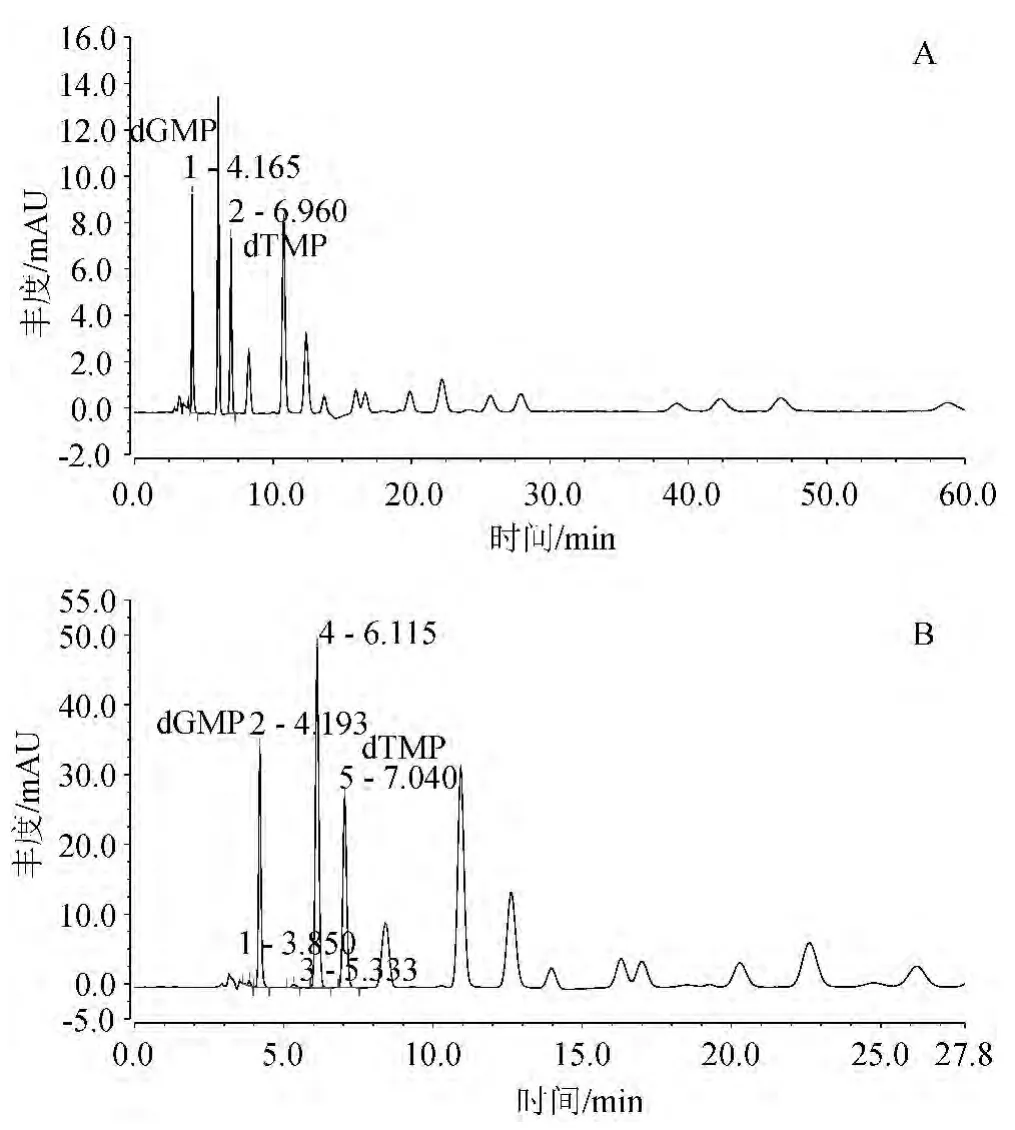
图3 标准菌株(A)及样品菌株(B)DNA的脱氧核苷色谱图Fig.3 Deoxyribonucleoside chromatogram of DNA instandard strain(A) and sample strain(B)

表1 HPLC分析得出的样品脱氧核苷成分的保留时间及峰面积Table 1 Retention time and peak area of sample deoxynucleoside by HPLC analysis
根据公式,参照菌株大肠杆菌(E.coli)DH5α的表观摩尔分数Ma0=SG0/(SG0+ST0)=46.021 5%;校正系数X=M(1-Ma0)/Ma(1-M0)=1.270 639(大肠杆菌(E.coli)DH5α的实际摩尔分数,M0,=52%);菌株大曲高温放线菌(Thermoactinomyces daqus)CICC 10681基因组DNA的(G+C)摩尔分数为49.3%。
2.4 全基因组测序分析菌株CICC 10681的(G+C)含量
采用双端测序法,用软件CLC Genomics Workbench 5.0.1 进行从头测序(De novo sequencing),共得到93个片段,拼接后总碱基数为3.46×106个,(G+C)摩尔分数为49.0%。
菌株CICC 10681是本课题组从扳倒井高温大曲曲块中分离到的一株高温放线菌,经分类学研究表明其为高温放线菌属内一新种,定名为大曲高温放线菌(Thermoactinomyces daqus)[10]。目前,高温放线菌属正式公布的有2个种,普通高温放线菌(Thermoactinomyces vulgaris)和中间型高温放线菌(Thermoactinomyces intermedius)。据权威文献报道,这两个种模式株的(G+C)摩尔分数均为48%(HPLC法)[13],本研究测得的大曲高温放线菌CICC 10681的(G+C)含量47.0%(Tm法)、49.3%(HPLC法),与高温放线菌属的(G+C)含量接近,符合该属耐高温低(G+C)含量的特征。这与其他放线菌属高(G+C)含量(65%~75%)的特征差异明显(超过20%),正基于对(G+C)含量及重组DNA(recombinant DNA,rDNA)序列等遗传学分析,高温放线菌属的分类学地位发生了明显的改变,从最早的放线菌科转移到新的分类单元高温放线菌科中[14-15]。
从2种方法的测定结果看,Tm法和HPLC法测定的CICC 10681的(G+C)摩尔含量分别为47.0%(Tm法)和49.3%(HPLC法),与该菌株的全基因组测序结果(49.0%)相比,Tm法的结果存在一定的偏差。主要原因可能有:(1)Tm法得到的(G+C)含量是通过Tm值与(G+C)含量的经验公式得到,这种换算关系因DNA缓冲体系的浓度、pH的不同,差异显著。(2)DNA纯度不高或发生轻微降解,尤其是核糖核酸(ribose nucleic acid,RNA)的污染直接对结果产生偏差。另外,Tm法忽略了4种碱基之外的其他稀有碱基,且在Tm值较低或较高时,仪器精密度对结果的影响比较明显。因此,Tm法对试剂的准确配制、DNA的纯度和仪器的要求比较高。而HPLC法直接定量检测DNA的碱基组分,排除了稀有碱基、修饰碱基和RNA的干扰,色谱灵敏度高于分光光度法,测得的结果与全基因组测序的结果非常接近。而且HPLC法能实现大量样品的连续化处理,测定效率高。因此,HPLC法更适合用于微生物DNA(G+C)含量的准确描述和大量样品的测定。
3 结论
对大曲高温放线菌(Thermoactinomyces daqus)CICC 10681的分析表明,Tm法和HPLC法测定的(G+C)摩尔分数分别为47.0%(Tm法)和49.3%(HPLC法),与全基因组测序的结果(49.0%)基本一致,符合高温放线菌属的低(G+C)含量特征。HPLC法灵敏度高,结果准确,适用于微生物分类学中对微生物(G+C)含量的准确描述。
[1]JOHNSON J L.Use of nucleic acid homologies in the taxonomy of anaerobic bacteria[J].Int J Syst Bacteriol,1973,23(4):308-315.
[2]GOODFELLOW M,O'DONNELL A G.Handbook of new bacterial systematics[M].London:Academic Press Ltd.,1993.
[3]VANDAMME P,POT B,GILLIS M,et al.Polyphasic taxonomy,a consensus approach to bacterial systematics[J].Microbiol Mol Biol Rev,1996,60(2):407-438.
[4]LEE K Y,WAHL R,BARBU E.Contenuen bases puriqueet pyrimidiques des acides deoxyribonucleiques des bacteries[J].Ann Inst Pasteur,1956,91(2):212-224.
[5]林万明.细菌分子遗传学分类鉴定法[M].上海:上海科学技术出版社,1990.
[6]LI J,QIN S,YOU Z Q,et al.Melghirimyces profundicolussp.nov.,isolated from a deep-sea sediment[J].Int J Syst Evol Microbiol,2013,63(11),4552-4556.
[7]MESBAH M,PREMACHANDRAN U,WHITMAN W B.Precise measurement of the G+C content of deoxyribonucleic acid by high-performance liquid chromatography[J].Int J Syst Bacteriol,1989,39(2):159-167.
[8]MARMUR J,DOTY P.Determination of the base composition of deoxyribonucleic acid from its thermal denaturation temperature[J].J Mol Biol,1962,5(1):109-118.
[9]MANDEL M,IGAMBI L,BERGENDAHL J,et al.Correlation of melting temperature and cesium chloride buoyant density of bacterial eoxyribonucleic acid[J].J Bacteriol,1970,101(2):333-338.
[10]YAO S,LIU Y,ZHANG M J,et al.Thermoactinomyces daqussp.nov.,a thermophilic bacterium isolated from high temperature Daqu[J].Int J Syst Evol Microbiol,2014,64(1):206-210.
[11]MARMUR J,DOTY P.Thermal renaturation of deoxyribonucleic acids[J].J Mol Biol,1961,3(5):585-594.
[12]LEE Y J,WAGNER I D,BRICE M E,et al.Thermosediminibacter oceanigen.nov.,sp.nov.andThermosediminibacter litoriperuensissp.nov.,new anaerobic thermophilic bacteria isolated from Peru Margin[J].Extremophiles,2005,9(5):375-383.
[13]YOON J H,KIM I G,SHIN Y K,et al.Proposal of the genusThermoactinomyces sensu strictoand three new genera,Laceyella,ThermoflavimicrobiumandSeinonella,on the basis of phenotypic,phylogenetic and chemotaxonomic analyses[J].Int J Syst Evol Microbiol,2005,55(1):395-400.
[14]YOON J H,PARK Y H.Phylogenetic analysis of the genusThermoactinomycesbased on 16S rDNA sequences[J].Int J Syst Evol Microbiol,2000,50(3):1081-1086.
[15]YOSHIHIDE M,ATSUKO K,SATORU M.Mechercharimyces mesophilusgen.nov.,sp.nov.andMechercharimyces asporophorigenenssp.nov.,antitumour substance-producing marine bacteria,and description ofThermoactinomycetaceaefam.nov.[J].Int J Syst Evol Microbiol,2006,56(12):2837-2842.

