干酪乳杆菌刺激小鼠肠道的转录组学分析
程荣叶, 李树东, 秦 达, 杨启尧, 李 颖, 余丽芸*, 侯喜林*
(1.黑龙江八一农垦大学 生命科学技术学院,黑龙江 大庆 163319;2.黑龙江八一农垦大学 动物科学技术学院,黑龙江 大庆 163319)
干酪乳杆菌(Lactobacilluscasei)是一种哺乳动物胃肠道宿主菌,在过去几十年中,因其益生特性在临床和动物模型中被广泛研究[1]。但是其干预肠道菌群,加强免疫反应的详细机制还不清楚,为了探究这个问题,拟通过干酪乳杆菌口服实验动物鼠,进行肠道的转录组学测序,分析发生显著变化的表达基因,挖掘这些基因在刺激免疫细胞分化成熟等方面的可能通路,为揭示这个科学问题提供参考。近20年来,益生菌的研究取得了突破性进展,Toh等[2]研究报道了干酪乳杆菌作为对人类和家畜的一种益生菌,能够稳定肠道微生物群落,减少病原微生物入侵,减轻或预防由细菌和病毒引起的腹泻,从而使肠道运动障碍达到一种正常化的状态。口服免疫后,干酪乳杆菌携带抗原物质附着在肠道和泌尿生殖系统中,刺激机体产生免疫反应,进一步阻碍病原微生物的入侵和黏附,减少了炎症反应,并且干酪乳杆菌还可激活机体相关免疫细胞的活性,包括巨噬细胞、NK细胞和T淋巴细胞[3-4]。除此之外,干酪乳杆菌进入机体后还可以产生一些富含抑菌活性的多肽或前体多肽,抑制有害菌感染机体,促进肠道内益生菌的增殖,进而发挥益生作用[5]。重要的是重组乳酸菌增强了黏膜表面和血清中的lgA、IgM和IgG的水平,诱导产生TNF-α、IFN-γ等细胞因子,继而加强体液免疫和细胞免疫[6-8]。特别是组学手段为深入研究提供了目标和方向,加快了益生菌机制的研究进程。Wang等[9]采用宏基因组和宏转录组测序揭示了干酪乳杆菌的体内表达模式,发现摄入的益生菌必须改变其转录模式才能在人类肠道中生存和适应,时间依赖性的激活模式研究表明益生菌和人类肠道微生物之间存在高度动态的串扰。转录组学确定了驱动嗜酸乳杆菌(Lactobacillusacidophilus)在肠道适应性和活性的可能基因,揭示了这种益生菌促进抗炎反应、维持肠上皮稳态和调节宿主昼夜代谢轴的作用[10]。同样的高通量转录组分析揭示了芽胞杆菌(Bacillus)对鸡的免疫、肠道屏障系统和代谢具有改善作用[11]。但是干酪乳杆菌在刺激肠道组织后产生的转录因子功能还没有完全被揭示,有待进一步的验证和研究。转录组学研究已经取得了一些成果,揭示了一些转录因子、调控蛋白的作用,例如脂氧合酶(Aloxe)3基因编码的一种新型代谢调控蛋白-表皮型Aloxe3的发现[12];白细胞分化抗原6 (Cluster of differentiation 6,CD6)鉴定为富含半胱氨酸的清道夫受体糖蛋白超家族中的一员[13],在胸腺发育、T细胞活化和机体的免疫应答中均起着至关重要的作用[14-16]。在药物代谢中,肝脏含有发挥重要作用的多种药物代谢酶,例如羧酸酯酶(Carboxylesterase,CES )组成一个多基因家族,其基因产物多数分布在机体的各个组织和细胞的内质网中,这些酶以十分高效的催化去水解内源性和外源性物质[17]。ZBTB16基因属于抑癌基因家族成员之一,最早发现于早幼粒细胞白血病的研究中,故称之为早幼粒细胞白血病锌指蛋白(ZBTB16或PLZF或ZNF145)[18],该基因可在人的肺组织中表达[19-21]。糖蛋白非转移性黑色素瘤蛋白B(GPNMB)是一种跨膜糖蛋白,因其在黑色素瘤细胞系中高表达且转移性低而得名,由于GPNMB在成骨细胞分化和增加骨矿物质沉积中的作用,也被称为骨激活素[22-24]。OX40(CD134)及其配体OX40L(CD252)是TNF家族的成员,在CD4+、CD8+T细胞及其他几种淋巴和非淋巴细胞上均有表达[25]。UBASH3A是蛋白络氨酸磷酸激酶家族中的一员,包含三个结构域:N-末端UBA(泛素相关)、SH3(Src同源性3)和C末端组氨酸磷酸酶PGM结构域[26-27]。LZTS1亮氨酸拉链肿瘤抑癌基因1位于染色体8p22上[28-29]。研究发现Nanos1基因作为转录后阻遏子参与调节翻译过程[30]。孕激素和脂肪Q受体7(PAQR7)是膜孕激素受体中不可或缺的一员[31]。血清和糖皮质激素诱导的蛋白激酶1(SGK1)被鉴定为一种促肿瘤基因[34-35],是蛋白激酶“AGC”亚家族的成员,与AKT激酶家族具有结构和功能的相似性,也称为Akt[32-33]。TRIM46基因属于三重基序(Tripartite Motif,TRIM)蛋白家族成员,也称为GENEY,位于人染色体1q21上[36-37]。 这些基础研究提示益生菌调节肠道微生物群落的同时,有可能引起肠道组织的某些转录因子发生变化,进而调节免疫细胞的活性。因此,本研究以干酪乳杆菌为实验材料灌胃小鼠,取不同时间节点的肠道样本进行高通量转录组学测序,利用NGS分析干酪乳杆菌刺激小鼠肠道的转录组数据,对不同时间节点的转录组数据通过GO功能富集和KEGG通路富集,对富集的相关功能的差异基因进行生物信息学分析,进一步利用实时荧光定量技术(qRT-PCR)验证相关差异基因的表达。
1 材料与方法
1.1 材料
1.1.1 供试菌株 试验用菌株干酪乳杆菌CICC6105(LactobacilluscaseiCICC6105)购自中国工业微生物菌种保藏中心,-80 ℃冰箱保存。
1.1.2 实验动物 20只雌性 Balblc小鼠(SPF级)购自北京维通利华动物技术有限公司,为5~6周龄,实验开始前在无菌环境中适应性饲养一周,自由饮食。
1.1.3 主要试剂及仪器设备 蛋白胨、牛肉膏、酵母膏、吐温-80、琼脂购自Sigma公司;葡萄糖、无水乙酸钠、柠檬酸三胺、磷酸氢二钾、硫酸镁、硫酸锰、碳酸钙购自国药集团; SYBR Green I核酸染料和HiScript III Reverse Transcriptase逆转录试剂盒购自诺唯赞公司; Recombinant RNasin Ribonuclease Inhibitor购自Promega公司。实时荧光定量PCR仪(LightCycler®96, 瑞士罗氏生物公司);微量紫外分光光度计(Evolution 220, 美国 ThermoFisher 公司);电热恒温培养箱(DRP-9162, 上海森信实验仪器有限公司);空气浴震荡器(HZQ-C, 哈尔滨市东联电子技术有限公司);旋涡震荡器(QL-861, 海门市其林贝尔仪器制造有限公司);台式低温冷冻高速离心机(A-14C-1EU, 德国 Sigma 公司)。
1.2 方法
1.2.1 试验动物模型分组 选取20只体重相近状态良好的5~6 周龄SPF级Balb/c小鼠,在相同饲养环境条件下随机将其分为两组,分别为对照组(NTC)、干酪乳杆菌组(TOA)。NTC组:用生理盐水饲喂小鼠30 d,分别在第10天、20天、30天取3只小鼠的小肠组织;TOA组:用浓度为5×1011cfu/mL的干酪乳杆菌生理盐水悬液200 μL饲喂小鼠30 d,分别在第10天、20天、30天取3只小鼠的小肠组织。
1.2.2 干酪乳杆菌悬液的制备 将冻存的干酪乳杆菌菌株从-80 ℃冰箱取出,取出后将冻存菌平板划线接种于MRS固体培养基中,37 ℃培养12 h后,挑取单菌落在5 mL MRS液体培养基中37 ℃静置培养过夜,再以2%(体积分数)的比例接种于100 mL进行扩大培养,5 000 r/min,4 ℃离心10 min收集菌体,菌体用生理盐水洗涤,重复3次。然后进行平板细菌计数,再用生理盐水将菌体稀释为5×1011cfu/mL,对小鼠进行每天一次的灌胃。
1.2.3 小鼠肠道的提取 对各组小鼠进行12 h禁食,禁食后,利用颈椎脱臼法处死小鼠,随后将处死的小鼠放置浓度为75%的酒精中消毒,在无菌的超净工作台解剖小鼠,剪取胃幽门至回盲部的小肠,放入含有生理盐水的平皿中,使用含有生理盐水的注射器对小肠中的内容物进行洗涤,重复3次,洗涤后将小肠放入冻存管中。将冻存管放置液氮中速冻30 min,随后将小肠保存至-80 ℃冰箱中,待整体试验取样完成后,将小肠用干冰运输送至上海派森诺生物技术有限公司。
1.2.4 差异基因分析 小肠样本经测序公司测序后返回,测序 Raw counts 数据采用 DESeq2 分析,得到显著差异的基因,将筛选条件设为Pvalue≤0.05,且差异倍数|Fold change|>1 。随后将表达差异分析结果进行可视化,通过热图可视化体现样本间差异表达情况。
1.2.5 差异基因的GO和KEGG富集分析 为了研究干酪乳杆菌刺激小鼠肠道后产生的差异基因功能,利用派森诺平台(https://www.genescloud.cn/login)对筛选出的差异基因做基因本体分析(GO)和KEGG通路分析,差异基因显著富集结果利用柱状图对其进行可视化。
1.2.6 基本特性分析 使用在线网站ExPASy(https://web.expasy.org/protparam/)对14个基因分子量、等电点、不稳定系数和脂溶系数进行分析;利用Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)在线网站对基因编码蛋白进行亚细胞定位分析;使用NCBI的Conserved-Domains数据库对14个差异表达基因进行蛋白的功能结构域预测分析。
1.2.7 染色体定位分析 从NCBI数据库中获得的小鼠基因组gff文件,利用Tbtools软件对14个差异表达基因进行染色体定位分析。
1.2.8 基因结构与保守基序分析 使用在线网站MEME (http://meme-suite.org/tools/meme)对14个基因的保守基序进行预测;使用Gene Structure Dispaly Server(http://gsds.gao-lab.org/)在线分析基因结构,得出基因中CDS (Sequence coding for amino acids in protein)分布情况。
1.2.9 qRT-PCR表达分析 利用NCBI primer designer,以14个差异表达基因的核苷酸序列为模板设计荧光定量引物,扩增长度控制在80 ~ 150 bp以内,SDHA作为内参基因,引物合成由生工生物工程(上海)股份有限公司完成(表1)。qRT-PCR反应时以反转录的第一链cDNA稀释10倍作为模板。反映体系参照诺唯赞公司的HiScript III Reverse Transcriptase说明书,利用 Lightcycler 96 Real-time PCR System进行 qPCR。所有实验数据按照平均数±标准方差计算;数据分析均使用 Graphpad prism 9.0 软件进行;组间显著性差异使用 Student′st-test 法进行分析;P< 0.05 代表组间显著(*P< 0.05, **P< 0.01 和 ***P< 0.001, ****P< 0.000 1)。
2 结果与分析
2.1 表达差异基因分析
为了分析处理组与对照组之间差异基因的表达情况,对D10NTC vs.D10TOA 组、D20NTC vs. D20TOA组、D30NTC vs. D30TOA 组的差异基因利用 Deseq2 包对原始 counts 数据进行分析,阈值设置为P≤ 0.05 和|log2(Fold change)|>1,结果发现D10NTC vs. D10TOA 组、D20NTC vs. D20TOA组、D30NTC vs.D30TOA 组中分别有176、32、261个基因发生变化。其中在 D10NTC vs.D10TOA 组有 59 个基因上调与 117 个基因下调;D20NTC vs. D20TOA 组有 23 个基因上调与 9 个基因下调;D30NTC vs. D30TOA 组有 181 个基因上调与 80 个基因下调;利用热图对上述差异基因进行可视化(图1)。
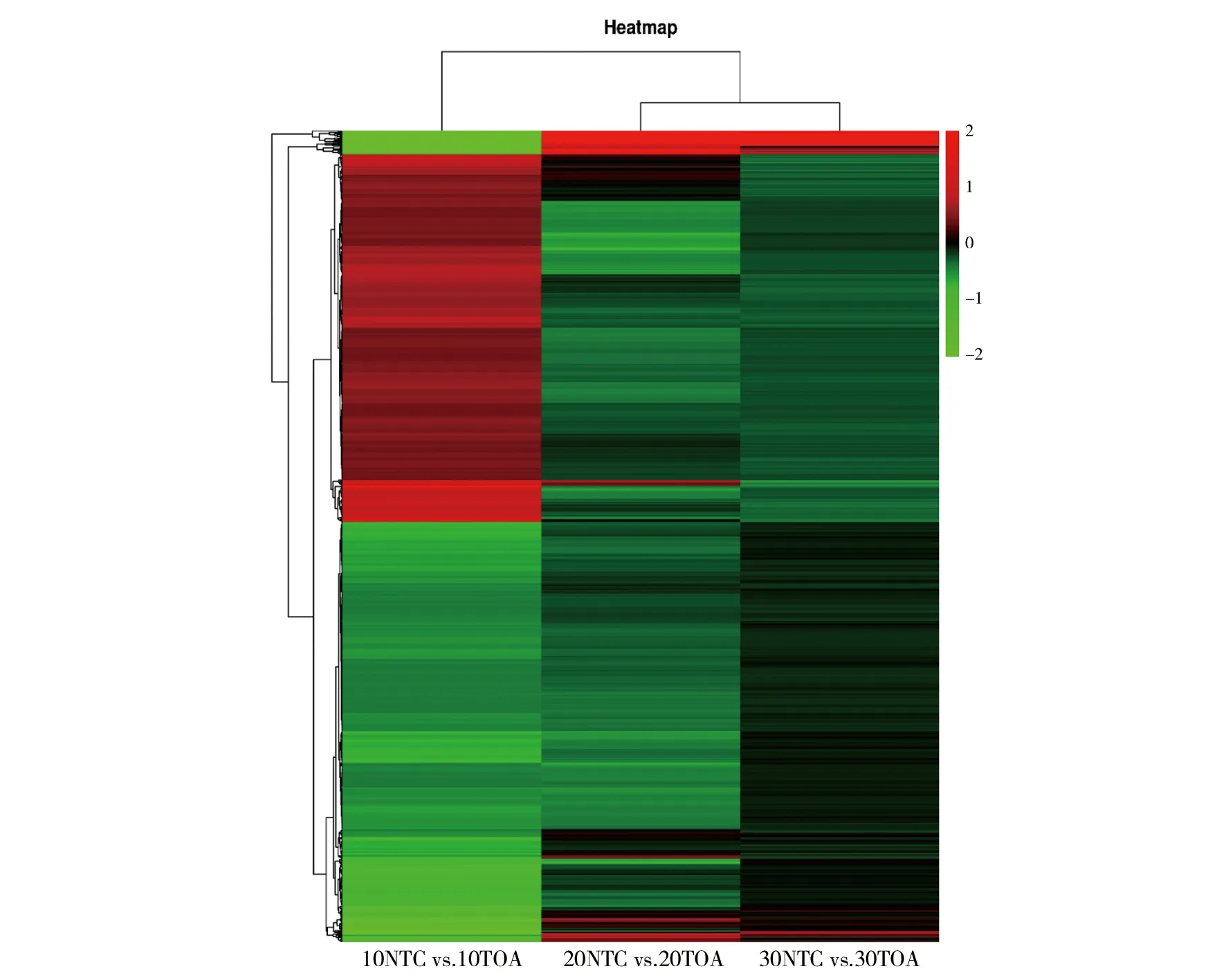
图1 各组之间差异基因表达热图Fig.1 The heat map of differential gene expression among groups
2.2 GO功能富集分析结果
对差异基因的生物进程进行GO功能富集分析,干酪乳杆菌组(TOA)和生理盐水组(NTC)的GO富集分析结果(图2A、图2B、图2C):D10NTC vs. D10TOA 组表达的差异基因主要富集在免疫系统负调控、细胞分化、细胞黏附的调节、T细胞激活等进程;D20NTC vs. D20TOA 组表达的差异基因主要富集在淋巴细胞活化、调节细胞分化进程和T细胞激活等进程;D30NTC vs. D30TOA组表达的差异基因主要富集在免疫反应调节过程、免疫系统调节正调控、T细胞激活等进程。以上分析结果发现与细胞免疫相关的功能基因有Gpnmb、Hist1h4c、Zbtb16、Hist2h4、Sgk1、Aloxe3、Cspg5、Lzts1、Paqr7、Trim46、Ces1d、Nanos1、Cd6、OX40,故后续实验将选取这14个基因进行验证。


图2 干酪乳杆菌组与空白组GO功能富集柱状图Fig.2 Histogram of GO function enrichment in Lactobacillus casei group and blank groupA: D10NTC vs.D10TOA组柱状图; B: D20NTC vs. D20TOA组柱状图; C: D30NTC vs. D30TOA组柱状图;D:14个差异表达基因热图 A: D10NTC vs. D10TOA; B: D20NTC vs. D20TOA; C: D30NTC vs. D30TOA;D: Heat map of 14 differentially expressed genes
2.3 KEGG富集分析结果
对差异基因进行KEGG通路富集分析,干酪乳杆菌组(TOA)和生理盐水组(NTC)的KEGG分析结果(图3):D10NTC vs. D10TOA 组的通路主要富集在PPAR信号通路、半乳糖代谢、脂肪的消化和吸收类固醇激素的生物合成等(图3A);D20NTC vs. D20TOA 组主要富集在皮质醇的合成和分泌、脂肪细胞因子信号通路、胆固醇代谢、烟酸和烟酰胺代谢等(图3B);D30NTC vs. D30TOA 组主要富集在细胞黏附分子(CAM)、B细胞受体信号通路、趋化因子信号通路、C型凝集素受体信号通路等(图3C)。

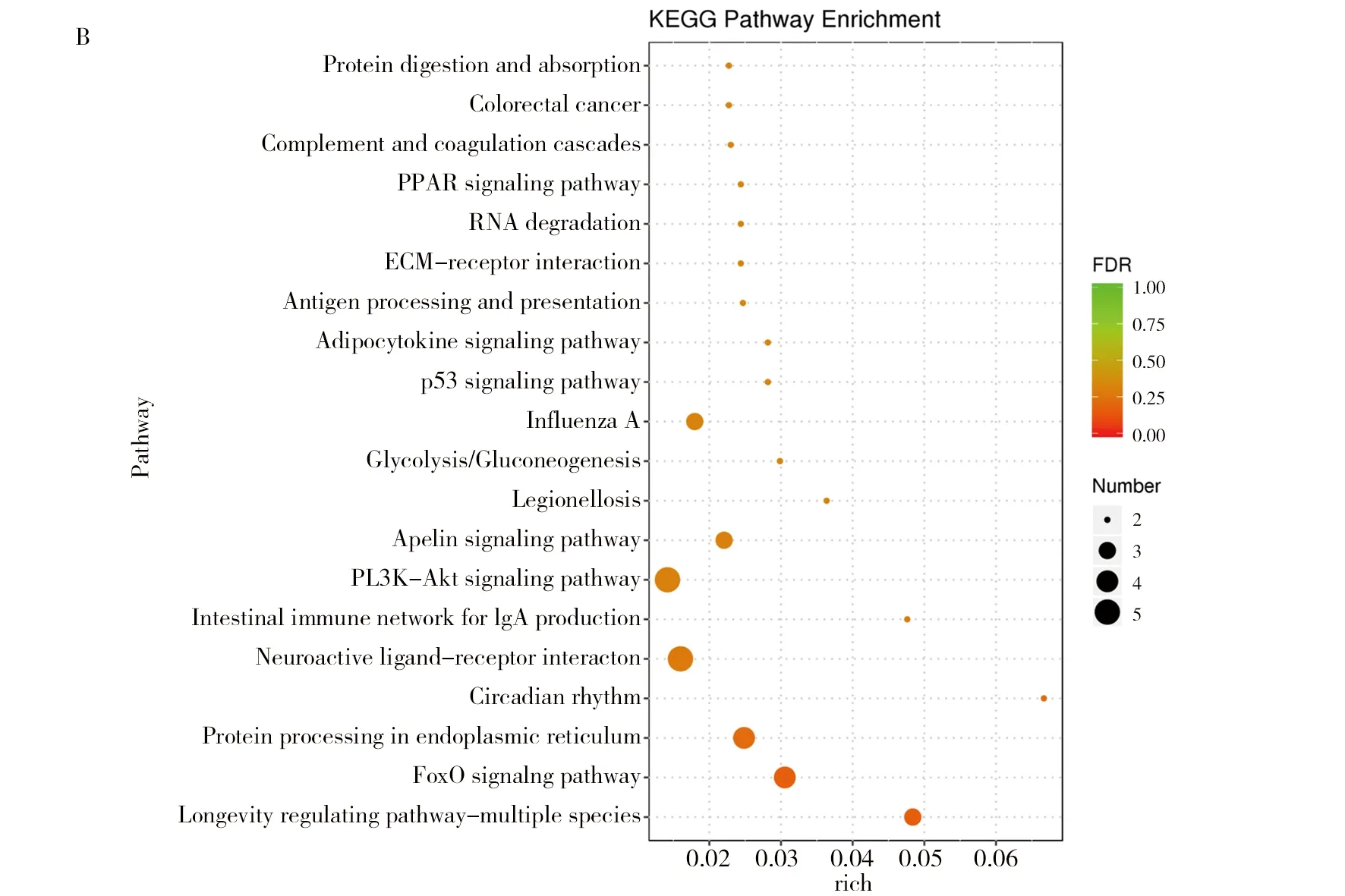

图3 干酪乳杆菌组与空白组KEGG通路富集气泡图Fig.3 The enrichment Bubble Diagram of KEGG pathway in L. casei group and blank groupsA:D10NTC vs.D10TOA;B:D20NTC vs.D20TOA;C:D30NTC vs.D30TOA
2.4 基本特性分析结果
供试的14个差异表达基因的蛋白质长度为103~759 aa,分子量在11.37~83.45 kDa之间,其中ENSMUSG00000042766(Trim46)编码的蛋白长度最大为759 aa,ENSMUSG00000060678(Hist1h4c)和ENSMUSG00000091405(Hist2h4)蛋白最小为103 aa;等电点pI在4.42~11.36之间,其中ENSMUSG00000060678(Hist1h4c)和ENSMUSG00000091405(Hist2h4)的pI最大,ENSMUSG00000032482(Cspg5)的pI最小;亚细胞定位预测表明,它们分别定位在细胞质、溶酶体、高尔基体、内质网、细胞外和细胞核内,均为不稳定的脂溶性蛋白,且均含有功能结构域(表2)。
2.5 染色体分析结果
14个差异表达的基因分布于小鼠染色体的3、4、6、8、9、10、11、13、和19号染色体上,其中Trim46和Hist2h4基因位于3号染色体,Paqr7和OX40基因位于4号染色体,Gpnmb基因位于6号染色体上,Lzts1和Ces1d基因位于8号染色体上,Zbtb16和Cspg5基因位于9号染色体上,Sgk1基因位于10号染色体上,Aloxe3基因位于11号染色体上,Hist1h4c基因位于13号染色体上,Cd6和Nanos1基因位于19号染色体上,从分布结果可以看出基因在染色体分布较为分散,没有出现聚集并形成基因簇的现象(图4)。

图4 染色体定位Fig.4 Chromosomal localization
2.6 基因结构与保守基序分析结果
基因上的保守基序Motif分布稀疏,其中Hist2h4和Hist1h4c的保守基序分布及种类相似度高,保守基序模型较为保守。其余基因保守基序相似性不高,表明它们之间不具有类似的功能(图5 A)。Hist1h4c、Hist2h4不含CDS序列,Gpnmb、Zbtb16、Aloxe3和Ces1d含有一个CDS序列,Sgk1、Cspg5、Paqr7、Trim46、Nanos1、Cd6和OX40含有2个CDS序列,Lzts1基因均含有3个CDS序列 (图5 B)。

图5 基因结构、保守基序可视化分析Fig.5 Visual analysis of gene structure and conserved motifsA:基因结构; B:保守基序A:Gene structure; B:Conserved motif
2.7 qRT-PCR分析结果
为验证 RNA-seq 分析结果,利用实时荧光定量技术(qRT-PCR)对筛选出来的14个基因进行验证,分析在不同时间差异基因的表达水平,结果发现,其中Hist1h4c、Hist2h4、Cspg5、Cd6、Gpnmb、Trim46和Nanos1基因的表达模式一致,在不同时间处理下的整体表达水平都比较低,组间表达不显著。Sgk1、Ces1d、Lzts1、Paqr7、Aloxe3、Zbtb16和OX40整体表达水平较高,表达峰值明显与转录组数据相符。其中Sgk1基因在第20天和第30天有显著差异,Ces1d、Lzts1、Paqr7、Aloxe3、Zbtb16基因在第10天和第20天有显著差异,OX40基因在第10天、第20天和第30天均有显著差异(图6)。

3 讨 论
Jung等[38]发现干酪乳杆菌能预防病原体感染、对机体有保护和预防癌症等作用。Devi等[6]发现干酪乳杆菌进入机体后,刺激黏膜激活机体免疫系统,影响特异性免疫应答能力,促进T、B细胞的增殖和成熟。肠道属于最早接触环境因素(食物、毒素和病原体等)的主要器官之一,具备近端到远端的黏膜结构梯度和营养处理能力[39]。干酪乳杆菌对肠道的研究具有重要的意义,但是目前干酪乳杆菌刺激小鼠肠道,其对免疫细胞的影响还不清楚,因此,我们的研究旨在揭示干酪乳杆菌干预肠道后什么基因变化,能够激活T细胞,致使T细胞增殖;T细胞激活对黏膜免疫有什么样的作用效果?本研究通过RNA-seq测序探究了干酪乳杆菌刺激小鼠肠道后基因表达谱的变化,发现7个基因功能主要与免疫系统过程、固有免疫反应、T细胞激活等相关,为进一步深入研究奠定了基础。
在建立干酪乳杆菌刺激小鼠肠道模型中,发现干酪乳杆菌饲喂10 d时,饲喂组对比对照组的上调基因数量比下调基因少,饲喂到20 d、30 d时,上调基因数量相比下调基因的数量增多。这种现象的原因可能是饲喂干酪乳杆菌10 d后,小鼠肠道内菌群的变化还没有趋于稳定,有些作用还没有显现。但是继续饲喂干酪乳酸菌之后,随着饲喂时间的增加,小鼠可能产生耐受反应,小鼠肠道内益生菌群趋于稳定,有些基因开始上调。干酪乳酸菌在小鼠肠道中为优势菌属,刺激小鼠肠道细胞上调相关基因,类似的研究报道表明,当饲喂益生菌之后,鸡肠道上调基因数目相比下调基因数目增多[40],但具体机制需要进一步深入研究。对差异基因进行生物进程富集分析,显示差异基因多数富集在T 细胞活化、免疫调节负调控、细胞分化和先天免疫应答的调节上。KEGG通路富集显示和PPAR信号通路、半乳糖代谢、B细胞受体信号通路、趋化因子信号通路、C型凝集素受体信号通路相关。以上结果进一步说明了干酪乳杆菌刺激小鼠肠道可能通过促进T细胞激活、细胞分化等以增强免疫系统能力。Lian等[41]研究结果表明抑制CES1(Ces1d的人类同源基因)可能是预防和治疗非酒精性脂肪肝(NAFLD)的一个新的药理学靶点。Kawaue等[42]研究证明Lzts1可作为细胞动力学的主要调节器发挥作用,在进化过程中增加了大脑结构的复杂性。Tan等[43]证明PAQR7可能介导孕酮诱导BPBC EMT逆转,由孕酮激活的PAQR7与小窝蛋白-1相互作用(Cav-1)或调节表皮生长因子的活性受体(EGFR),使PI3K/Akt信号通路失活,最终抑制BPBC中的EMT。Higgins等[44]研究发现Aloxe3作为一种潜在的新的肝细胞禁食反应效应器,利用PPARy介导和多效性效应来增强肝脏和整个宿主的代谢,作为改善代谢性疾病的一个有希望的靶点。对于显著表达的PLZF基因(早幼粒细胞白血病锌指蛋白),有研究发现其对Naïve T细胞的正常发育起着至关重要的作用[45-47]。 Zhang等研究发现,OX40调节T细胞的增殖,主要在后期对维持细胞存活发挥着至关重要的作用[48-49]。以上基因在乳酸菌刺激肠道后表达也显著上升,和肠道黏膜免疫有什么相关性还有待于进一步验证。
本研究用干酪乳杆菌刺激小鼠肠道,转录组测序的生物信息学分析发现了与T细胞激活相关的功能基因,同时也发现细胞黏附分子(CAM)、B细胞受体信号通路发生了显著变化,初步表明干酪乳杆菌对机体的免疫过程发挥着重要的作用,为后续深入详细研究干酪乳杆菌对免疫系统的作用机制提供参考。

