基于多维组学数据的玉米农艺和品质性状预测研究
杨静蕾 吴冰杰 王安洲 肖英杰,2,*
1 作物遗传改良全国重点实验室 / 华中农业大学, 湖北武汉 430070; 2 湖北洪山实验室, 湖北武汉 430070
杂种优势利用和基于表型选择的传统育种为粮食产量的提升作出了巨大贡献。近年来, 气候变化和人口增加给全球粮食安全带来了严峻挑战, 预计到2050 年, 世界人口将增至95 亿, 届时需要多生产70%的粮食才能满足人口需求[1-2], 因此亟需新的育种技术和方法来提高作物产量[3-4]。高通量基因分型平台和表型平台、基因组选择、机器学习等新技术,为育种效率的提升和作物新品种的培育提供了新的契机[5]。
Meuwissen 在 2001 年首次提出基因组选择(genomic selection, GS)或基因组预测的概念[6], 其核心是利用参考群体的全基因组分子标记基因型数据与表型数据建立统计模型, 在表型未知而基因型已知的候选群体中, 利用该模型估计每个材料的基因组育种值, 进而在早期实施选择, 大幅度提高了遗传增益。GS 在理论上类似于MAS (marker assisted selection), 但MAS 仅利用少量显著性标记对主效基因进行选择。动植物重要性状一般为数量性状, 由大量微效基因控制, MAS 无法捕捉到微效基因的贡献, 因此对数量性状改良的作用有限。而基于覆盖全基因组的高密度标记, 直接估计全基因组中的所有标记效应的基因组选择, 可以捕获具有微小影响的遗传位点[7], 能够更好地解释表型变异, 提高复杂农艺性状地预测精度和选择效率。
随着下一代测序技术和高密度单核苷酸多态性(single nucleotide polymorphism, SNP)基因分型技术的快速发展, 目前, 基因组预测已经成为革命性的育种手段。基因组选择的首次利用是在奶牛中, 极大缩短了奶牛选育的世代间隔, 后来又成功用于猪、羊等主要动物的育种[8-9]。据报道[10], 动物中基因组预测的精度可达0.8, 这为基于预测结果对动物幼崽进行早期筛选, 加快种畜选育提供了可能。在玉米、水稻、小麦等作物中[11-14], 也陆续开展了该类研究, 比如, Wang 等[13]在水稻杂交育种中开发的MV-ADV 模型有助于对低遗传力性状(如产量)进行基因组预测; Ma 等[14]曾使用不同遗传结构的群体构建训练集对大豆百粒重进行全基因组预测, 预测精度最高可达0.75; Charmet 等[15]评估了基因组选择在小麦3 个育种群体的3 个性状的表现, 具有较高遗传力的抽穗期, 其预测精度可高达0.7。
在玉米中, 有研究发现, 基因组选择优于分子标记辅助选择和基于系谱的传统表型选择, 可显著提高低遗传力性状的预测精度和选择效率[16-17]。Cao等[18]利用玉米的1 个关联群体和3 个双单倍体群体,对其焦油斑点病抗性性状进行GS 研究, 发现预测精度受训练群体大小和标记密度影响, 但总体而言预测精度较高, 说明GS 对玉米抗病性筛选具有较强的应用潜力。基于具有热带血缘的22 个玉米双亲分离群体的基因型和干旱和正常情况的表型数据,Zhang 等[19]对产量、株高和开花期3 个性状进行基因组预测, 发现使用200 个SNPs 标记及50%群体作训练集时, 3 个性状的预测精度分别是0.28、0.32 和0.29, 且随着训练群体大小和标记密度的增加, 预测精度也会随之提升。目前, GS 已在玉米的多种遗传和育种群体中得到广泛应用[20-22]。
统计模型是基因组预测的核心, 影响基因组预测精度。常用的 GS 模型有 LSE (least-squares estimation)、BLUP (best linear unbiased prediction)、Bayes (bayesian analysis)、LASSO (least absolute shrinkage prediction operator)等[23]。其中 rrBLUP(ridge regression best linear unbiased prediction)的利用最为广泛, 其是利用训练群体估计标记效应, 在预测群体中将标记效应累加, 进而预测未知表型个体的基因组育种值。据报道, 基于该方法对玉米的开花时间进行GS, 预测精度可达0.64[24]。Bhering等运用模拟数据集评估rrBLUP、GBLUP (genomic best linear unbiased prediction)、Bayesian LASSO 三种统计方法的基因组预测性能, 发现该方法在分析效率和预测精度上表现均最好[25]。Yan 等[26]基于rrBLUP、Bayes 和RF (random forest) 3 种统计方法,对玉米籽粒中镉元素含量进行预测, 预测精度分别达到0.89、0.83 和0.75, 其中rrBLUP 方法的预测精度最高。LASSO 由Tibshirani 于1996 年首次提出[27],该方法通过最小化残差平方和的约束, 实现对高维数据的变量选择, 从而在基因表达分析中被广泛应用, 具有较高的预测准确性、良好的解释能力和稳健性。在利用卫星遥感数据预测玉米年产量变化的研究中, LASSO 的预测精度达0.78[28]。在陆地棉纤维品质的预测研究中, Islam 等[29]发现 Bayesian LASSO 比 GBLUP、rrBLUP、Bayes、PKHS(reproducing kernel space) 4 种统计模型的预测精度要更高。Tsai 等[30]使用春大麦和冬小麦2 套数据验证rrBLUP 和Bayesian LASSO 两种模型的预测性能,发现在春大麦中, Bayesian LASSO 对白粉病和产量的预测精度高于rrBLUP, 而在冬小麦中, 2 个模型对产量的预测精度相似。以上研究表明, 对于不同性状、不同物种和群体, 基因组预测方法表现可能存在差异, 但是rrBLUP 与LASSO 模型在众多统计模型中表现出更好的性能, 于是本研究主要基于以上2 种统计模型进行GS 研究。但前人研究往往基于少量性状和同种类型数据, 对农艺和品质性状的预测能力系统评估还较缺乏。
近年来, 随着多维组学技术的迅速发展, 植物在转录、翻译和代谢水平上的变化都可以进行定量检测。Azodi 等[24]基于转录组数据和基因组数据来构建开花时间的预测模型, 通过评估模型特征的重要性, 发现模型中最重要的2个特征都为转录组特征, 验证了转录组数据相比基因组数据对预测模型的重要性, 这也表明转录组提升基因组预测方面的巨大潜力。此外, Zhang 等[31]对由385 个自交系组成的玉米关联群体进行全基因组代谢物分析, 共检测到1035 种显著变化的代谢物, 发现其中15 种代谢物性状可解释超过60%的玉米苗期干旱处理后存活率表型变异。Qin 等[32]曾通过代谢物分析发现硼元素通过增加抗氧化酶的活性和改变代谢产物, 减轻了镉毒害对小麦的影响。Hu 等[33]发现使用转录组、代谢组数据对水稻产量的预测精度可达 0.4869 和0.4593, 远高于基因组预测的精度, 这说明对于受微效多基因控制的产量性状, 转录组和代谢组数据可能会提供比基因组更丰富的信息进行预测。
目前, 在玉米中, 利用多组学数据对玉米重要性状的预测研究报道较少, 在不同模型和不同性状上的系统评估也较为缺乏。为系统研究组学数据对玉米性状预测的效果, 本研究以具有广泛多样性的368 个玉米自交系的基因组、转录组和代谢组数据,基于rrBLUP 与LASSO 模型, 对55 个农艺和品质性状进行预测分析, 系统评估了各组学数据和统计模型, 对农艺性状和品质性状预测能力的差异, 为后续玉米重要性状的基因组育种提供了理论依据。
1 材料与方法
1.1 试验材料
1.1.1 群体及表型数据 本研究使用来自368 个不同玉米自交系组成关联群体作为试验材料, 分别在海南(三亚, 18°25′N, 109°51′E, 2010 年)、云南(昆明, 24°25′N, 102°30′E, 2011 年)和重庆(29°25′N,106°50′E, 2011 年) 3 个地点进行田间试验。根据系谱信息, 将所有自交系分为2 组, 即温带(temperate,TEM)和热带/亚热带(tropic/sub-tropic, TST)。所有自交系根据不完全随机区组试验设计, 单行种植在试验地中。对收获的玉米植株进行表型鉴定, 获取包括株高、穗行数在内的20 个农艺性状和包括籽粒生育酚、脂肪酸含量在内的35 个品质性状。本研究使用的表型数据均来自于已发表文章[32-34]。对表型数据进行预处理, 删除各表型的异常值并计算55 个性状的描述性统计。
1.1.2 基因组和转录组数据 本研究所用群体已使用Illumina Maize SNP50 芯片和转录组进行测序,并鉴定到103 万个覆盖全基因组的高质量SNPs 和28,769 个基因的表达数据[34-36]。本研究从中随机挑选5 万个SNPs 作为基因组数据; 将基因表达数据进行标准化处理后作为转录组数据。基因组和转录组数据将用于后续的多组学预测研究。
1.1.3 代谢组数据 本研究使用群体的成熟籽粒已经进行了靶向和非靶向代谢组分析。其中靶向代谢组是验证测试样品中是否存在目标代谢物的检测方式, 本研究使用的靶向代谢组是指以关联群体在云南和重庆2 个环境的成熟玉米籽粒为测试样品,检测其中部分氨基酸含量所形成的数据集: 包括17种重要的氨基酸、47 个氨基酸的衍生性状以及它们各种氨基酸的总和, 共计130 种靶向代谢物[37]。非靶向代谢组是通过对生物体内代谢物进行全面分析,找出差异代谢物的研究方式。本研究使用的非靶向代谢组是关联群体在海南、云南、重庆3 个环境下种植, 对成熟籽粒进行基于气相色谱或液相色谱结合质谱的代谢物分析获取的数据, 在3 个环境下共鉴定到2031 种代谢物[38]。以上代谢物数据将用于本研究的多组学预测研究。研究中进行了靶向代谢物与非靶向代谢物的整合, 通过逐步整合得到5 个数据集, 包括海南非靶向代谢物的数据集E1; 海南和云南非靶向代谢物的数据集E1+E2; 海南、云南和重庆非靶向代谢物的数据集E1+E2+E3; 全部非靶向代谢物和重庆靶向代谢物的数据集 E1+E2+E3+CQ; 所有非靶向代谢物和靶向代谢物的数据集E1+E2+E3+CQ+YN。
1.2 基因组预测的统计模型
1.2.1 岭回归最佳线性无偏估计(rrBLUP) 该方法是基因组预测最常用的模型之一, 它通过在训练群体中估计标记效应, 结合预测群体的基因型信息将标记效应累计, 最终获得预测群体的个体估计育种值。该方法在构建标记效应矩阵时, 赋予部分标记较大权重, 从而放大有效标记的贡献, 弱化无效标记的作用, 以使预测结果更符合实际[39]。本研究使用基于R 语言的“rrBLUP”软件包实现模型构建,此软件包含3 个函数: A.mat 函数主要用于构建加性效应矩阵; mixed.solve 函数主要用于混合模型的参数求解, 可以用于预测标记效应或育种值; kin.blup函数使用加性效应关系矩阵预测基因型值, 进而预测表型。本研究首先使用A.mat 函数计算加性效应矩阵, 再通过kin.blup 函数进行表型预测, 计算预测结果与真实值的皮尔逊相关系数(Pearson correlation efficiency,r), 即预测精度。
1.2.2 最小绝对收缩选择算子(LASSO) 该模型通过构造一个惩罚函数得到一个较为精炼的模型,使它收缩一些系数, 同时设定一些系数为零, 以实现对变量的选择和对模型复杂程度的降低。因其对数据的要求极低, 无论变量是连续还是离散的, 都能用LASSO 进行处理。本研究利用R语言中“lattic”软件包进行LASSO 预测, 首先使用createFolds 函数将数据随机分为5 份, 再基于训练集使用train 函数中的LASSO 参数进行模型构建, 然后基于测试集的组学数据使用predict 函数进行表型预测。模型的预测精度, 为预测结果与真实值的皮尔逊相关系数。
1.2.3 基因组预测的交叉验证 本研究使用5 折交叉验证计算模型的预测值与真实值的相关性, 并将5 次重复交叉验证结果的均值来评估模型的性能。该方法将数据集随机切分为5 个互不相交且大小相同的子集, 使用其中4 个子集训练模型, 剩下的1 个子集当作测试集测试模型。将上述步骤重复5 次, 每次挑选不同的子集作为测试集, 训练得到5个模型, 共得到5 个预测值与真实值的皮尔逊相关系数, 对这5 次的皮尔逊相关系数取平均值得到一个交叉验证的结果。该方法会使所有数据参与到训练和预测中, 在最大化利用数据训练模型的同时避免过拟合的现象。
2 结果与分析
2.1 基于基因组数据分析对农艺性状和品质性状的预测差异
基于基因组数据和rrBLUP 模型, 研究发现, 品质性状的平均预测精度(r=0.628)明显优于预测农艺性状(r=0.504) (图1-A)。在农艺性状中, 仅有5 个性状的预测精度高于0.6, 分别是粒长(kernel length)、散粉期(pollen shed)、雄穗分枝数(tassel branch number)、穗位叶长(ear leaf length)、抽雄期(heading date), 其中粒长的预测效果最好, 达到0.689 (图1-B)。在品质性状中, 60%以上的性状预测精度大于0.6,其中籽粒含油量(OIL)的预测效果最好, 达到0.887,预测效果最佳的前5 个性状分别是含油量(OIL)、油酸(C18_1)、亚油酸(C18_2)、花生烯酸(C20_1)、花生酸(C20_0), 预测精度均高于0.7 (图1-C)。
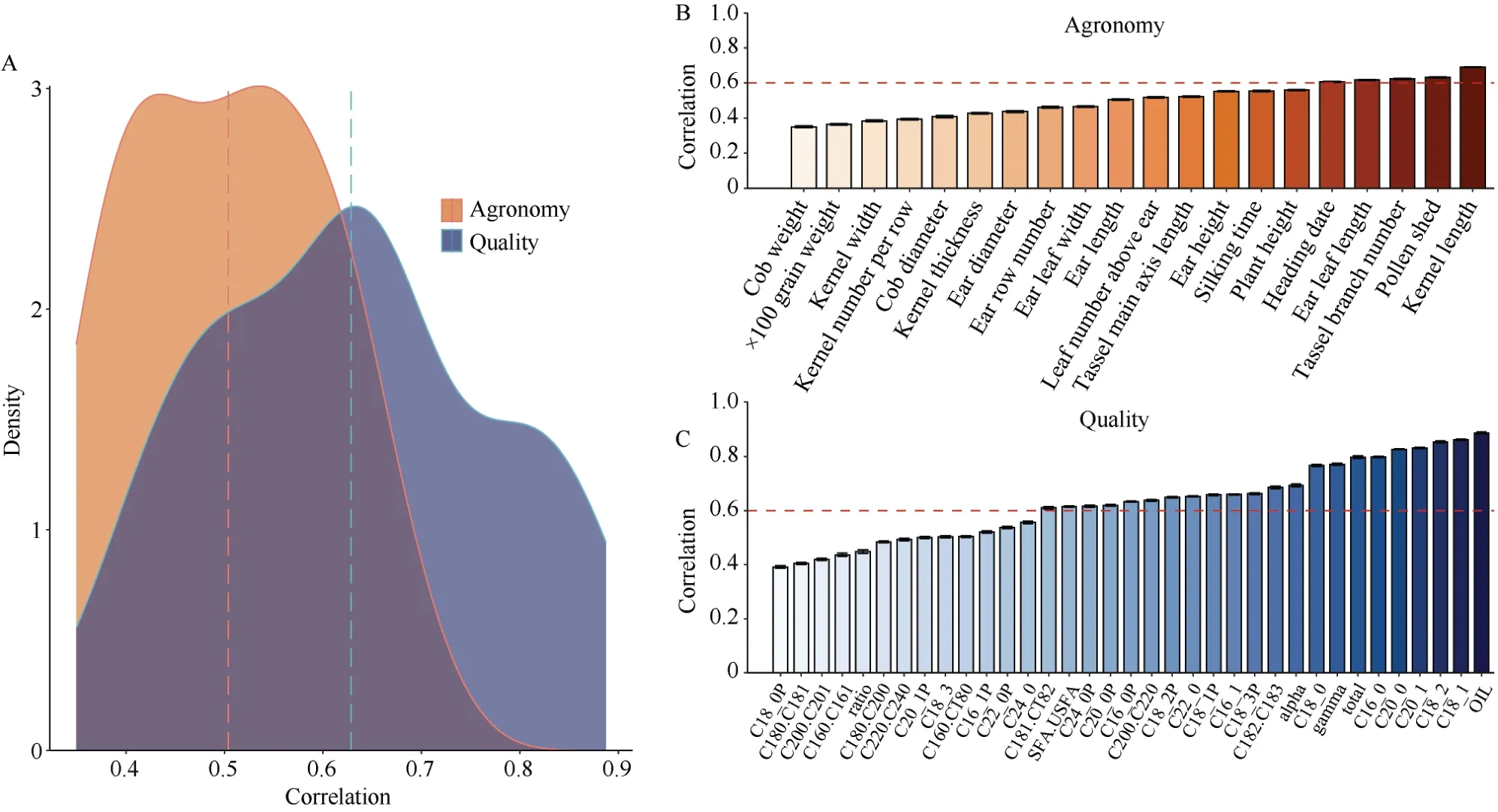
图1 基于基因组数据分析对农艺性状和品质性状的预测差异Fig.1 Prediction differences in agronomic traits and quality traits based on genomic data analysis
2.2 不同组学数据对性状预测的差异
对比分析基于基因组、转录组、代谢组预测不同类型性状的结果, 研究发现, 对于农艺性状,基因组数据的预测精度都显著高于转录组的预测精度(r=0.504 vsr=0.459,P=4E-3)。此外, 转录组数据的预测精度显著高于代谢组的预测精度(r=0.459 vsr=0.333,P=5.39E-6)。品质性状也遵循该趋势(图2-A)。
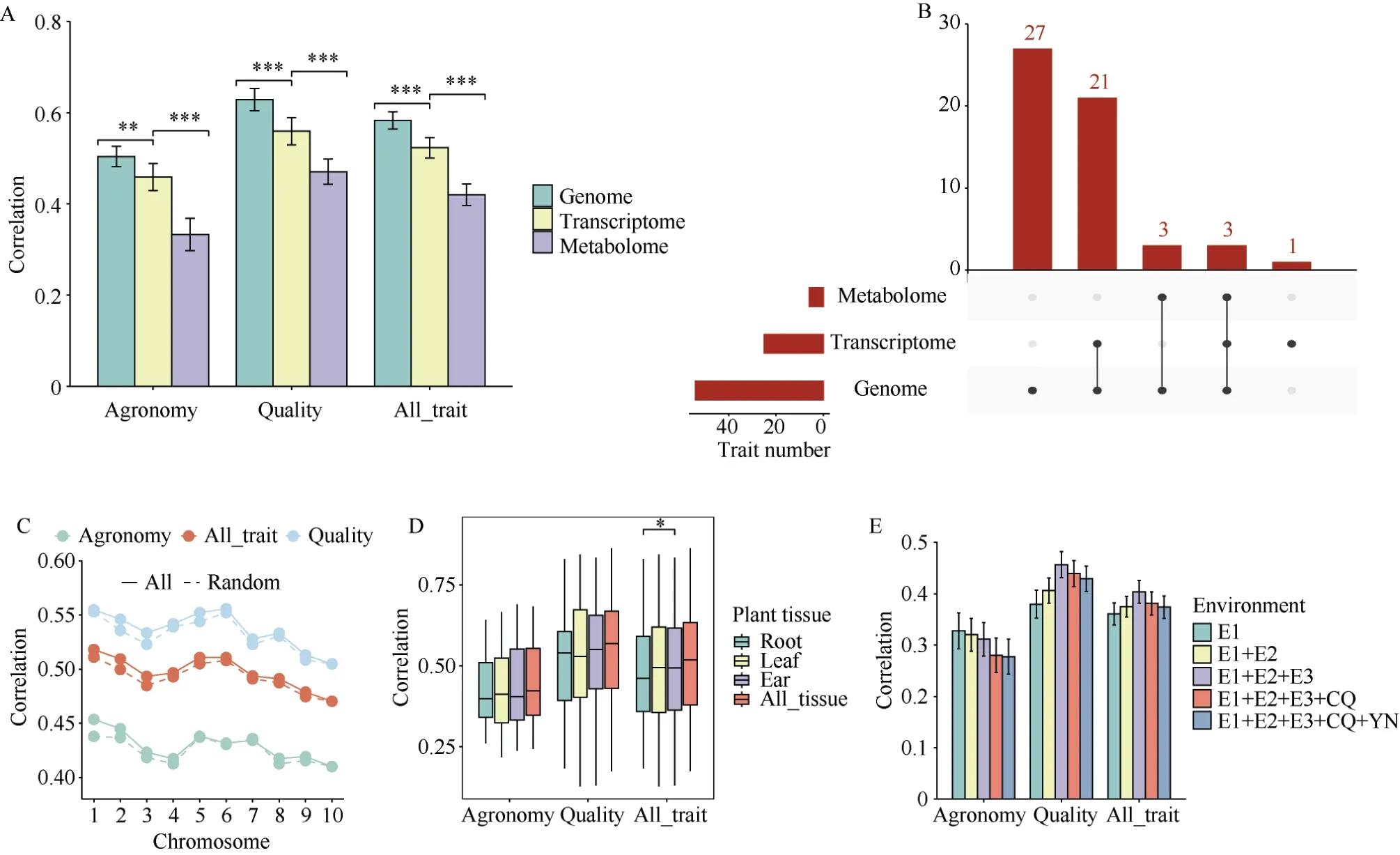
图2 不同组学数据对性状预测的差异Fig.2 Differences in trait prediction between omics data
此外, 相比于其他组学数据, 基因组数据对27个性状的预测精度最高。在这27 个性状中, 花生烯酸(C20_1)利用基因组数据的预测精度高达 0.832,明显高于该性状基于转录组数据和代谢组数据的预测结果(0.776 和0.638)。其中, 软脂酸与棕榈油酸的比例(C160.C161)呈现出基因组数据和转录组数据的预测差值最大(rdiff.=0.251), 雄穗主轴长(Tassel main axis length)呈现出基因组数据和代谢组数据的预测差值最大(rdiff.=0.419), 同时该性状也表现出转录组数据和代谢组数据的最大预测差值(rdiff.=0.275)。我们发现, 有21 个性状使用基因组数据和转录组数据的预测精度没有明显差异(rdiff.<0.05),但均优于代谢组数据的预测精度。其中, 籽粒含油量(OIL)表现出基因组数据和转录组数据的最高的预测精度(r=0.887 和r=0.862), 明显高于代谢组对该性状的预测精度(r=0.734)。油酸含量(C18_1P)、亚油酸含量(C18_2P)、油酸与亚油酸的比例(C181.C182)的基因组和代谢组数据预测精度没有明显差异, 但均显著高于转录组数据的预测精度, 同时,我们发现代谢组预测精度在各组学中达到最高。吐丝期(silking stage)使用转录组数据的预测精度达到0.606, 明显优于基因组和代谢组数据的预测精度(0.555 和0.526)。此外, 粒长(kernel length)被基因组、转录组和代谢组数据预测精度都非常高(r>0.68)(图2-B)。
通过分析不同染色体标记的预测差异, 我们发现, 农艺性状、品质性状都呈现出了相同的预测趋势。1 号染色体(n=7976)、5 号染色体(n=6093)和6号染色体(n=4098)的标记预测精度高, 9 号染色体(n=3703)和10 号染色体(n=3480)的标记预测精度低,这可能说明9 号、10 号染色体预测精度低是因为染色体上标记数目少(图2-C)。为了进一步验证这个猜想, 本研究以10 条染色体中最少的标记数目为参考,对染色体的标记进行随机挑选, 并分别进行表型预测, 将结果进行比较, 发现仍为1 号染色体、5 号染色体、6 号染色体上的标记预测效果最佳, 9 号染色体和10 号染色体上的标记预测效果较差。将随机预测结果与染色体上所有标记预测结果相比, 发现所有标记预测结果略微优于挑选标记预测结果, 同时仍呈现出1 号染色体、5 号染色体、6 号染色体上的挑选标记预测效果最佳, 9 号染色体和10 号染色体上的挑选标记预测效果最差, 这说明1 号染色体、5号染色体和6 号染色体上可能存在对表型变异贡献最大的标记。因此, 研究初步推测在训练基于基因组数据的基因组预测模型时, 加大1 号染色体、5 号染色体、6 号染色体上的标记数目, 可以提升全基因组数据的预测效果。
基于基因高表达的玉米组织, 本研究将转录组数据分为根高表达基因集Root (n=6322)、叶片高表达基因集 Leaf (n=3101)和穗高表达基因集 Ear(n=2577)三大类, 以及所有基因集(n=18,548)。结果发现, 使用3 种组织数据预测2 种性状的结果表现趋势略微不同, 对于农艺性状而言, 穗高表达基因集的预测结果最好(r=0.435), 叶片高表达基因集的预测结果其次(r=0.434), 根高表达基因集的预测效果最差(r=0.428); 对于品质性状而言, 叶片高表达基因集的预测结果最好(r=0.534), 穗高表达基因集的预测结果其次(r=0.523), 根高表达基因集的预测效果最差(r=0.518) (图2-D)。研究进一步对比基于不同组织预测2 种类型性状的结果, 发现3 种组织数据集中均呈现出品质性状的预测结果优于农艺性状。
通过逐步整合不同环境的代谢物数据, 得到5个数据集, 分别是 E1、E1+E2、E1+E2+E3、E1+E2+E3+CQ、E1+E2+E3+CQ+YN, 分别用以上数据集分别预测农艺性状与品质性状, 发现呈现不同的预测趋势。对于农艺性状, 数据集增大伴随着预测精度的下降, 对于品质性状, 数据集增大伴随着预测精度的上升(图2-E)。为了解释更多环境的代谢物会降低农艺性状预测精度的现象, 我们分析了5 个环境的代谢物与农艺和品质性状的相关性。统计结果发现, 与农艺性状正相关和负相关的代谢物数目比较接近, 因此整合不同代谢数据可能会存在贡献抵消的情况, 而导致预测精度下降; 对品质性状而言, 不同环境代谢物负相关数目远小于正相关, 因此整合数据预测有更大潜力提升预测精度。
2.3 不同模型对性状预测的影响
总体来说, rrBLUP 相比于LASSO 模型对不同性状的预测精度更高, 但对于某些品质性状, LASSO模型的预测效果更佳。具体来说, 基于基因组数据,发现对于55 种性状rrBLUP 均为最佳模型。基于转录组数据, rrBLUP 对于20 个农艺性状是最优预测模型, 对品质性状中的大多数性状为最优模型, 而软脂酸与棕榈油酸的比例(C160.C161)和生育酚比值(ratio)则是LASSO 为最佳预测模型。基于代谢组数据, LASSO 对于4 个农艺性状和8 个品质性状是最优模型。在农艺性状中LASSO 预测效果最佳的性状是株高(Plant height) (r=0.331), 品质性状中预测效果最佳的性状是亚麻酸含量(C18_3P) (r=0.622)(图3-A)。基于上述结果, 我们初步推测rrBLUP 适合用于预测以农艺性状为代表的数量性状, LASSO更适合用于预测以品质性状为代表的质量性状。这可能是由于LASSO 模型本质是进行变量选择, 该方法很难捕获到微效基因的效应, 而对于由主效基因控制的质量性状, 该方法可以放大主效基因的作用,以实现更好的预测效果。

图3 不同模型和数据组合对性状预测的整合评估Fig.3 Integrated evaluation of models and omic-data combinations on trait prediction
为探究不同模型之间精度变化规律, 本研究设定预测精度为0~0.3 为低精度、0.3~0.6 为中精度、0.6~1.0 为高精度(图3-B)。利用基因组数据, 发现使用rrBLUP 模型时所有性状的预测精度均高于0.3,其中27 个性状预测精度较高。使用LASSO 模型时,41 个性状预测精度中等或较高。其中, 4 个性状被rrBLUP 和LASSO 预测均较高, 均为品质性状, 分别是油酸(C18_1)、亚油酸(C18_2)、油分浓度(OIL)、生育酚总量(total)。利用转录组数据, 发现使用rrBLUP 模型时 18 个性状预测精度较高。使用LASSO 模型, 有39 个性状预测精度中等或较高。其中1 个性状-gamma 生育酚(gamma)被rrBLUP 和LASSO 预测精度均较高。利用代谢组数据, 发现使用rrBLUP 模型时40 个性状预测精度中等或较高,其中, 10 个性状预测精度较高。使用LASSO 模型时,37 个性状预测精度中等或较高, 其中, 6 个性状预测精度较高(图3-B)。这些结果表明, 针对不同类型性状, 不同组学数据在rrBLUP 和LASSO 模型下表现出不同的精度变化模式。
通过整合分析, 因此我们鉴定出不同性状的最佳预测的数据和模型组合(图3-C)。结果发现, 45 个性状的最优预测组合为基因组数据和rrBLUP 模型(Geno_rrBLUP), 其中22 个性状预测精度大于0.6,其中预测精度最高的性状为籽粒含油量(OIL) (r=0.887)。3 个性状的最优预测组合为转录组数据和rrBLUP 模型(Tran_rrBLUP), 预测精度均大于0.6,其中预测精度最高的性状为散粉期(Pollenshed)(r=0.651)。生育酚比值(ratio)最优预测组合为转录组数据和 LASSO 模型(Tran_LASSO), 预测精度为0.459; 3 个性状的最优预测组合为代谢组数据和rrBLUP 模型(Meta_rrBLUP), 预测精度均大于0.6,精度最高的性状为油酸含量(C18_1P) (r=0.667); 3 个性状的最优预测组合为代谢组数据和LASSO 模型(Meta_LASSO), 其中预测精度最高的性状是软脂酸与硬脂酸的比例(C160.C180) (r=0.522)。此外, 结果显示, 仅在农艺性状中出现转录组数据为最优预测组合的数据来源, 仅在品质性状中出现代谢组数据为最优预测组合的数据来源。这说明, 转录组数据对农艺性状预测更有价值, 代谢组数据对品质性状预测作用更大。
2.4 材料异质性对不同组学预测的影响
本研究的368 个自交系根据系谱来源可分为温带(temperate, TEM)材料和热带(tropic/sub-tropic,TST)材料。基于基因组、转录组和代谢组数据, 利用rrBLUP 模型对温热带材料进行预测分析。研究发现,训练集和测试集的材料类型相同时(即温带预测温带, TEM-TEM; 或热带预测热带, TST-TST; 精度0.211~0.582), 3 种组学数据预测效果均优于训练集和测试集类型不同的情况(即温带预测热点, TEMTST; 或热带预测温带, TST-TEM; 精度0.120~0.197)(图4-A)。同时, 研究发现, 使用TEM 预测TEM 的效果明显优于使用TST 预测TST 的效果, 而使用TST 预测TEM 的效果略优于使用TEM 预测TST 的效果(图4-B), 这可能是由于热带材料的变异程度比温带材料的变异程度丰富, 从而使得用TST 训练集很难覆盖TST 测试集的遗传变异, 但是包含了TEM中的大部分变异。除此之外, 研究发现使用归属于TST 的代谢组数据训练模型时, 预测TEM 与预测TST 的精度相差无几, 仅有0.015, 这个结果说明在使用亲缘关系较远的材料进行预测时, 可以加入代谢组学数据进行模型的构建, 可能会提升预测精度。
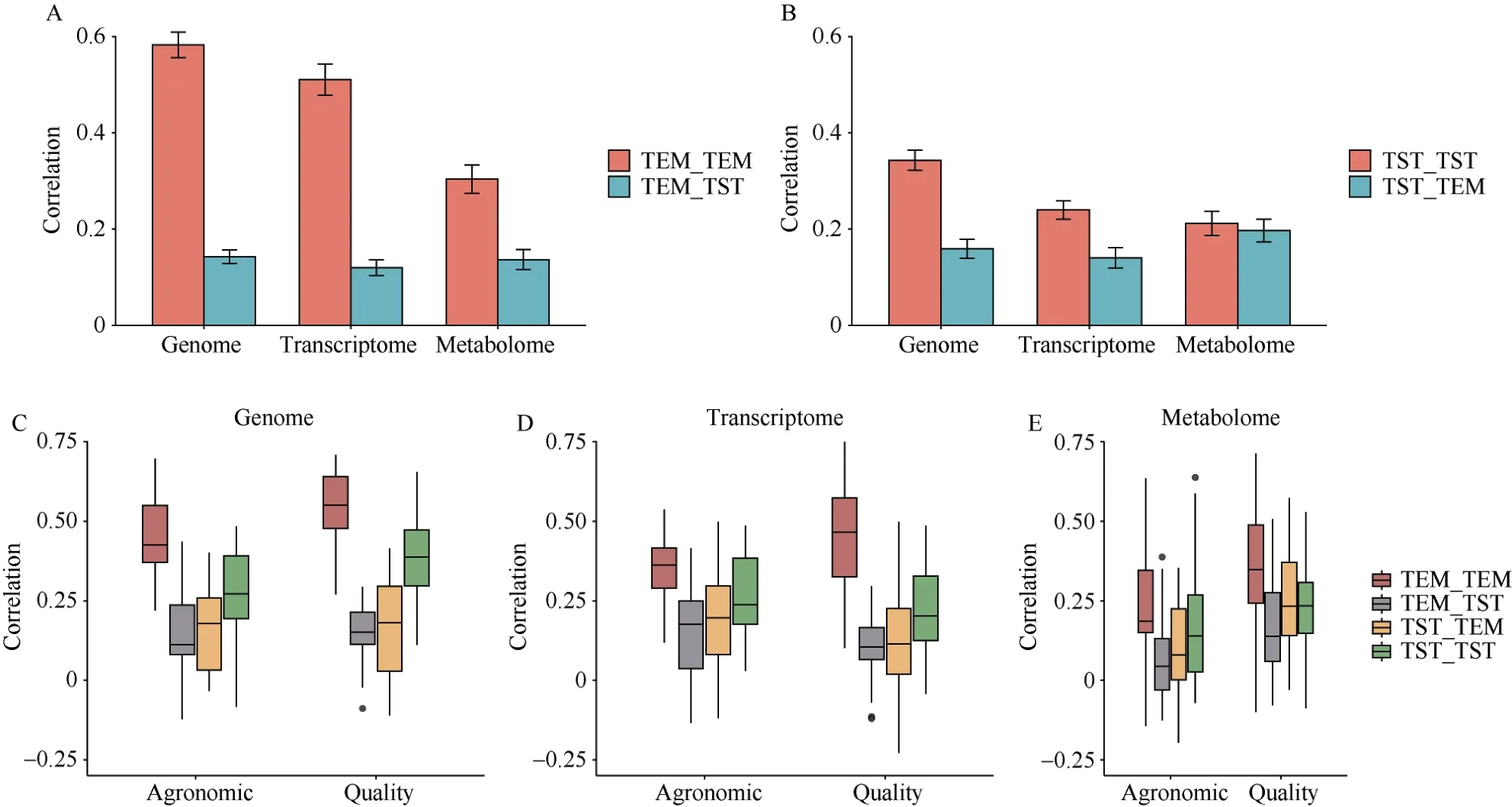
图4 材料系谱差异对不同组学预测的影响Fig.4 Influence of material genealogy on prediction by omics data
对农艺和品质性状, 材料异质性对不同组学预测进行分析。对比发现, 基于基因组数据(图4-C)和代谢组数据(图4-E)的预测结果, 在4 种不同训练集和测试集的组合中, 均表现出品质性状的预测效果优于农艺性状的预测结果; 基于转录组数据的预测结果(图4-D), 发现除了TEM-TEM 的组合之外, 其余3 种组合均表现出农艺性状的预测效果优于品质性状的预测结果。此外, 研究发现基于代谢组数据的预测结果也表现出 TST-TST 的预测结果与TST-TEM 的结果相当, 而对于品质性状, TST-TEM的预测均值(r=0.255)略高于TST-TST (r=0.238)。
3 讨论
本研究基于基因组、转录组和代谢组数据, 使用rrBLUP 和LASSO 两种统计模型, 对玉米的农艺性状和品质性状进行了基因组预测分析。结果发现,基于基因组数据, 品质性状的预测能力整体高于农艺性状。对比不同组学数据, 2 种类型性状均呈现基因组数据预测精度高于转录组数据预测精度, 转录组数据预测精度高于代谢组数据预测精度的预测趋势。对比不同模型, rrBLUP 在大多数农艺性状中表现出了良好的预测效果, 而LASSO 在一些品质性状中预测效果更好。整合分析发现, 转录组数据对农艺性状预测更有价值, 代谢组数据对品质性状预测作用更大。此外, 使用亲缘关系较远的材料进行预测时, 加入代谢组学数据进行模型构建, 可提升预测精度。
由于数据和模型的局限性, 本研究还存在值得改进和深入分析的地方。对于不同组学数据, 由于转录组和代谢组数据维度要远小于基因组数据, 同时, 本研究使用的转录组数据仅来自玉米籽粒, 可能忽略了某些组织特异性基因的表达差异。此外,Zheng 等[40]曾证明禾本科的组蛋白修饰在植物生长和胁迫适应中发挥着重要作用, 这表明表观遗传修饰在植物生长中的重要性, 而在本研究中未涉及到表观组学的预测分析。因此, 未来可通过不断补充组学数据类型和扩充组学数据维度, 进一步提高预测精度。对于不同模型, 本研究仅选择了2 种稳健的统计模型rrBLUP 与LASSO 进行分析和比较, 发现rrBLUP 更适合用于预测以农艺性状为代表的数量性状, LASSO 更适合用于预测以品质性状为代表的质量性状。同时, Campos 等[41]曾验证相同的数据集预测相同的目标性状可能会出现多个同样好的预测模型, 很难直接确定GS 的最优统计方法。因此,即使rrBLUP 和LASSO 在GS 中表现出更优的性能,未来仍可以尝试更多机器学习或神经网络算法, 以更好地学习多组学数据内部的复杂非线性互作关系[24]。此外, 本研究仅是基于单个组学的预测效果分析, 而已有报道利用遗传标记作为变量并结合转录组和代谢组数据, 进行 3 层遗传特征学习的MLLASSO (multilayered least absolute shrinkage and selection operator)模型, 产量的预测精度(R2)可由0.1588 提升至0.2451[33], 表明基于中心法则, 借助机器学习算法优势, 建立时序性的多模态多组学预测模型[33], 是合理利用多组学数据精确预测玉米复杂性状的潜在解决方案。
4 结论
本研究基于rrBLUP 和LASSO 两种统计模型,分别利用基因组、转录组、代谢组数据进行基因组预测分析, 系统测试了多个玉米性状在不同组学数据下预测能力的差异, 为多维组学数据进行基因组预测分析提供了数据基础和参考依据。

